基于高通量测序分析虾夷扇贝柱菌群结构及腐败优势菌
2018-10-31江艳华王联珠许东勤李风铃翟毓秀
江艳华,王联珠,许东勤,2,李风铃,翟毓秀,张 媛,姚 琳,*
(1.中国水产科学研究院黄海水产研究所,农业部水产品质量安全检测与评价重点实验室,山东 青岛 266071;2.上海海洋大学食品学院,上海 201306;3.獐子岛集团股份有限公司,辽宁 大连 116011)
虾夷扇贝(Patinopecten yessoensis)隶属于软体动物门、瓣鳃纲、异柱目、扇贝科、扇贝属,是一种冷水性贝类,经过近20 a的养殖推广,目前已在我国渤海及黄海北部形成规模化和产业化养殖,创造了数十亿元的产值,是我国北方最重要的经济海水养殖贝类之一[1]。虾夷扇贝个体较大、味道鲜美、营养丰富,深受消费者的青睐[2]。虾夷扇贝产品形式多样,大多以贝柱为主,包括鲜冻贝柱、干贝、即食贝柱等。由于虾夷扇贝柱组织细嫩、含水量高,在储运过程中极易发生腐败变质,即使在较低温环境下,有些微生物仍然可以繁殖,导致贝柱的变质[3]。然而,微生物对虾夷扇贝柱品质变化的影响及作用机制目前尚不清楚。因此,有必要分析虾夷扇贝柱中微生物的菌群结构及其在贮藏过程中腐败后的菌群结构,了解相关微生物种类,为进一步研究微生物在虾夷扇贝柱品质劣化中的作用提供基础资料。
传统的微生物菌群分析采用培养分离法,然而大多数环境微生物对培养条件要求苛刻,能够培养分离出的微生物仅占样品的1%~10%,无法解析微生物的组成及丰度情况[4]。随着宏基因组学概念的提出和高通量测序技术的发展,使样品中不可培养的微生物、低丰度的微生物均能被检测出,更完整地反映样品中微生物的群落特征,使菌群的分析更准确和快速,目前已经得到广泛的应用[5-8]。本研究采用宏基因组结合Illumina MiSeq高通量测序技术分析虾夷扇贝柱的菌群和腐败菌群,同时与平板分离结合高通量测序技术获得可培养菌群和腐败菌群的结构特征进行比对,为进一步探讨微生物与虾夷扇贝柱品质的相关性研究提供参考基础。
1 材料与方法
1.1 材料与试剂
虾夷扇贝由獐子岛集团股份有限公司提供,为2016年5月采捕,规格为2 a龄,健康状态良好。扇贝于低温环境下运抵实验室后立即处理,处理前仍保持鲜活状态。
2216E琼脂培养基 北京陆桥技术股份有限公司;土壤基因组DNA提取试剂盒、细菌基因组DNA提取试剂盒 美国Omega公司;10×PCR buffer、dNTPs、Taq酶日本Takara公司;Qubit 2.0 DNA检测试剂盒 美国Life公司;Agencourt AMPure XP磁珠核酸纯化试剂盒德国Beckman公司。
1.2 仪器与设备
T1型聚合酶链式反应(polymerase chain reaction,PCR)扩增仪 德国Biometra公司;PowerPac型电泳仪美国Bio-Rad公司;Infinity3000型凝胶成像系统 法国Vilber公司;Q32866型Qubit®2.0荧光计 美国Invitrogen公司;MiSeq高通量测序仪 美国Illumina公司;3K15型台式离心机 德国Sigma公司。
1.3 方法
1.3.1 样品处理
用洁净水将虾夷扇贝外壳冲洗干净,采用无菌手术刀小心将贝柱与其他组织分离开,用无菌水冲洗扇贝柱3 遍。将扇贝柱分别放入无菌样品袋中,置于4、15、25 ℃放置至样品腐败(其中4 ℃放置12 d、15 ℃放置4 d、25 ℃放置2 d,以感官和挥发性盐基氮作为判定依据)。新鲜扇贝柱和不同温度下腐败的扇贝柱分别匀浆后用于基因组DNA的提取。
1.3.2 宏基因组DNA的提取
用土壤基因组DNA提取试剂盒提取新鲜扇贝柱及不同温度下腐败扇贝柱的宏基因组DNA,新鲜样品的宏基因组DNA记为BZ,4、15、25 ℃腐败样品的宏基因组DNA分别记为BZ4、BZ15、BZ25。
1.3.3 可培养细菌基因组DNA的提取
无菌操作称取新鲜样品及25 ℃腐败样品各25 g,分别加入225 mL无菌生理盐水,匀浆后制成1∶10匀液,进行10 倍梯度稀释,取适宜稀释度匀液100 μL涂布2216E琼脂平板,30 ℃培养48 h。用无菌的TE缓冲液收集平板上所有菌落,8 000 r/min离心20 min,沉淀用细菌基因组DNA提取试剂盒提取基因组DNA,即为样品中可培养细菌菌群的基因组DNA[9]。新鲜样品的可培养细菌菌群的基因组DNA记为BZC,腐败样品的可培养细菌菌群的基因组DNA记为BZC25。
1.3.4 PCR扩增及高通量测序
将提取的基因组DNA作为模板,扩增16S rDNA序列的V3-V4可变区,引物序列上加有接头序列和测序引物序列,以适应MiSeq测序平台使用,同时正向引物上接有不同碱基的标签(barcode)序列以区分不同样品,引物如下:
341F:5’-CCCTACACGACGCTCTTCCGATCTG(barcode)CCTACGGGNGGCWGCAG-3’,805R:5’-GACTGGAGTTCCTTGGCACCCGAGAATTCCAGAC TACHVGGGTATCTAATCC-3’[10]。
通过两轮PCR扩增并完成接头序列的连接。第1轮PCR体系为50 μL反应液,含10×PCR buffer 5 μL,2.5 mmol/L dNTPs 2.0 μL,50 μmol/L引物各0.5 μL,5 U/μL Taq酶0.5 μL,DNA模板10 ng。PCR条件为:94 ℃预变性3 min;94 ℃变性30 s,45 ℃退火 20 s,65 ℃延伸30 s进行5 个循环;之后94 ℃变性20 s,55 ℃退火 20 s,72 ℃延伸30 s进行20 个循环,最后72 ℃延伸5 min。第2轮PCR体系同第1轮,模板为20 ng,PCR条件为:95 ℃预变性30 s,然后95 ℃变性15 s,55 ℃退火15 s,72 ℃延伸 30 s进行5 个循环,最后72 ℃延伸5 min。PCR结束后,利用琼脂糖电泳进行鉴定,采用磁珠核酸纯化试剂盒对PCR产物进行回收。利用Qubit2.0 DNA检测试剂盒对DNA进行定量,依托生工生物工程(上海)股份有限公司的Illumina MiSeq测序平台进行高通量测序。
1.4 数据分析
对MiSeq测序得到的数据,首先取出引物接头序列,再根据PE reads之间的overlap关系,将成对的reads拼接成一条序列,随后根据barcode区分样品得到各样本数据,对各样本序列进行质量控制,去除序列末端质量值20以下的序列,切除含N部分序列,并去除数据中的短序列,再对低复杂度序列进行过滤[11]。将多条序列按其序列间的距离进行聚类,对相似性在0.97以上的序列进行归并,生成操作分类单元(operational taxonomic units,OTUs)。根据聚类分析结果,计算ACE、Chao1、Shannon、Simpson指数进行alpha多样性分析,其中ACE、Chao1指数是对菌群丰度进行评估,Shannon、Simpson指数是对菌群多样性进行评估[12]。采用RDP classifier软件对序列进行物种分类,根据分类学分析比对结果,在门、属等水平上对样品的菌群结构进行种类和丰度分析[13]。
2 结果与分析
2.1 基因组DNA提取及PCR扩增
提取的宏基因组DNA和可培养细菌基因组DNA的条带清晰,有明显主带,其中宏基因组DNA有弥散拖尾现象。以提取的基因组DNA作为模板,用16S rDNA V3-V4区的通用引物均扩增出目的片段(图1),条带清晰,满足后续测序实验的要求。
图1 PCR扩增产物电泳图Fig. 1 Electrophoretogram of PCR amplified products
2.2 菌群测序结果质量分析

表1 虾夷扇贝柱新鲜及腐败样品菌群的alpha多样性比较Table 1 Alpha diversity of bacterial fl ora in fresh and spoiled adductors of P. yessoensis
样品的基因组DNA经PCR扩增16S rDNA V3-V4区后进行高通量测序,经统计分析获得序列的alpha多样性,结果见表1。所有样本的测序覆盖率均在93%以上,表明样品中序列未被测到的概率较低。所测试的样本中,新鲜样品的宏基因组(BZ)的测序量和OTUs数量最多,ACE和Chao1指数最低,表明新鲜样品中菌群相对丰度最低,而Shannon指数最高、Simpson指数最低,表明菌群的多样性最高。
2.3 细菌菌群结构分析
2.3.1 基于门水平的细菌菌群结构分析

表2 虾夷扇贝柱新鲜及腐败样品菌群在门水平上的分布Table 2 Relative abundance of bacterial phyla in fresh and spoiled adductors of P. yessoensis%
宏基因组DNA样本序列经RDP classifier软件进行分类分析,在门水平上,如表2所示,新鲜样品中的总细菌菌群主要包含变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、浮霉菌门(Planctomycetes)和拟杆菌门(Bacteroidetes),相对丰度分别为47.92%、14.99%、12.23%和7.58%,还包括少量的未分类菌(unclassified)、酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)、Ignavibacteriae、柔膜菌门(Tenericutes)等;样品在4、15 ℃腐败后的菌群以变形菌门(Proteobacteria)为主,相对丰度分别为98.38%和99.78%,在25 ℃腐败后的菌群则以变形菌门、梭杆菌门(Fusobacteria)和厚壁菌门为主,相对丰度分别为69.97%、18.94%和11.06%。
对可培养细菌DNA样本进行测序后发现,新鲜样品中的可培养细菌菌群99.98%归属于变形菌门,在25 ℃腐败后的可培养菌群以变形菌门(82.57%)和厚壁菌门(17.40%)为主。
2.3.2 基于属水平的细菌菌群结构分析
在属水平上的统计分析结果见表3。基于宏基因组DNA测序的结果显示,新鲜样品中的细菌菌群包含的物种多达100余种,其中相对丰度大于1%的有未分类菌(unclassified)、Candidatus kuenenia、弧菌属(Vibrio)、别弧菌属(Aliivibrio)、芽孢杆菌属(Bacillus)、科尔韦尔氏属(Colwellia)、肠球菌属(Enterococcus)、南极菌属(Moritella)、希瓦氏菌属(Shewanella)、黏着杆菌属(Tenacibaculum)、陶厄氏菌属(Thauera)、发光杆菌属(Photobacterium)、弓杆菌属(A r c o b a c t e r)、假交替单胞菌属(Pseudoalteromonas)、Ignavibacterium、极地杆菌属(Polaribacter)、Gp4,占总量的75.35%。当样品在4、15 ℃和25 ℃放置腐败后,与新鲜样品比较,菌群多样性降低、菌群结构发生了变化,对于一些在新鲜样品中占比较高的菌,如C. kuenenia在腐败后未有检出;而在新鲜样品中含量较低的发光杆菌属、别弧菌属、假交替单胞菌属、乳酸菌属(Lactobacillus)、梭杆菌属(Fusobacterium)等则显著增多。4 ℃的腐败优势菌为发光杆菌属(46.91%)、别弧菌属(36.82%)和假交替单胞菌属(11.01%),15 ℃的腐败优势菌为发光杆菌属(46.97%)和别弧菌属(43.33%),25 ℃的腐败优势菌为发光杆菌属(41.94%)、别弧菌属(23.10%)、梭杆菌属(18.61%)和乳酸菌属(10.60%)。
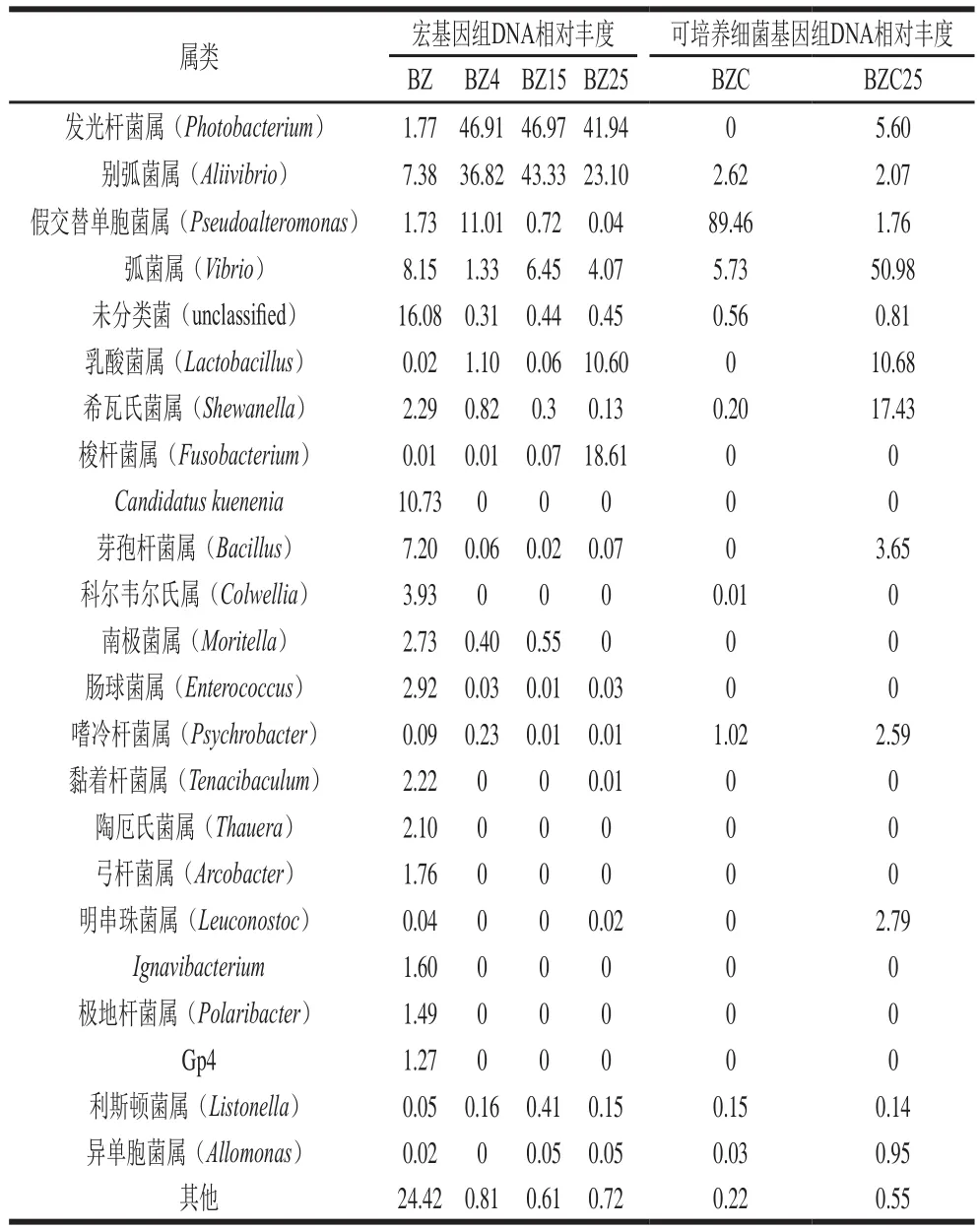
表3 虾夷扇贝柱新鲜及腐败样品菌群在属水平上的分布Table 3 Relative abundance of bacterial genus in fresh and spoiled adductors of P. yessoensis%
对可培养细菌DNA样本进行测序后发现,新鲜样品的可培养细菌菌群多样性降低,89.46%为假交替单胞菌属,此外含有少量弧菌属、别弧菌属、嗜冷杆菌属(Psychrobacter)等。在25 ℃放置腐败后,可培养的腐败优势菌为弧菌属(50.98%)、希瓦氏菌属(17.43%)、乳酸菌属(10.68%)。结果发现,在总细菌菌群中占优势的发光杆菌属、别弧菌属和梭杆菌属在可培养细菌菌群中比例大大下降,说明这几类菌不适宜在分离培养基上生长,而弧菌属、希瓦氏菌属、乳酸菌属则易培养分离,成为可培养的腐败优势菌。
3 讨 论
由于环境中微生物物种多样性极其丰富,传统培养分离法能够分离出的微生物仅占样品中总菌群的很小一部分。宏基因组学概念的提出,使环境中不可培养的微生物、低丰度的微生物等均能被检测出,更完整地反映样品中微生物的群落特征[6]。高通量测序技术能够一次对几十万到几百万条DNA分子进行序列测定,目前的Illumina MiSeq二代测序技术具有高准确性、高通量、高灵敏度和低运行成本等特点,结合宏基因组学技术,使环境中微生物菌群的分析变得更加准确和快速,在生态、医药、农业、食品等各个领域得到广泛应用[5,14-15]。
本实验通过宏基因组结合Illumina MiSeq高通量测序技术分析了新鲜虾夷扇贝柱的总菌群结构及在4、15 ℃和25 ℃腐败后的总菌群结构,其中4 ℃代表冷藏保存温度,15 ℃代表工厂加工环境温度,25 ℃代表室温。结果发现,新鲜虾夷扇贝柱中的细菌菌群多样性显著高于腐败样品的细菌菌群。当样品放至腐败后,菌群结构发生了变化,在4、15 ℃和25 ℃三个温度条件下的腐败优势菌均含有发光杆菌属和别弧菌属,此外,在4 ℃条件下,假交替单胞菌属相对丰度也较高,在25 ℃条件下,优势菌群还包括梭杆菌属和乳酸菌属。为与宏基因组样本的结果进行比对,本实验还采用平板分离结合高通量测序分析了新鲜样品中可培养菌群及25 ℃条件下腐败的可培养菌群结构。新鲜虾夷扇贝柱可培养菌群的多样性显著低于宏基因组测序获得的总细菌菌群,菌群结构也显著不同,例如可培养细菌中占89.46%的假交替单胞菌属仅占总细菌菌群的1.73%。25 ℃条件下的可培养腐败菌群与总腐败菌群多样性没有显著差异,优势菌群则不同,以弧菌属、希瓦氏菌属和乳酸菌属为主。虽然不同细菌由于生长速率不同,在琼脂平板上的菌落大小会稍有差异,提取平板上收集的菌落的基因组DNA不能准确反映样品中各种细菌的比例,但该方法省时省力,可以确定样品中可培养细菌的种类,并粗略估算它们的相对丰度,结果可作为参考。
水产品的腐败变质主要是由微生物产生的酶对蛋白质等的分解作用引起,在酶的作用下,蛋白质分解为氨基酸,然后进一步分解为胺类物质,最后生成硫化氢、氧化三甲胺等[16]。研究表明,在水产品腐败过程中只有少数部分特定种类细菌占主导地位,造成产品的腐败劣变,这类细菌被称为特定腐败菌[17]。发光杆菌属属于变形菌门、弧菌科,窦妍等[18]的研究发现发光杆菌属广泛存在于健康和患病的虾夷扇贝柱中,而在本实验中,发光杆菌则主要存在于腐败的扇贝柱中,新鲜扇贝柱中的丰度则很低,可能与不同养殖区域的样品有关。有研究报道,发光杆菌能够分解蛋白产生氧化三甲胺,是水产品的特定腐败菌[19],本实验不同温度下腐败样品的总菌群中,发光杆菌丰度均大于40%,表明该菌可能是虾夷扇贝柱的特定腐败菌。然而,测定的可培养细菌菌群中,发光杆菌丰度极低甚至在新鲜样品中未检测出,表明这类菌不易培养。假交替单胞菌属是1995年从交替单胞菌属(Alteromonas)中分出来的一个新属[20],该菌仅分布于海洋环境中,能产生蛋白酶等,已被证实是鱼贝类低温贮藏过程中的特定腐败菌,分解水产品后能够产生强烈的氨臭味[17,21]。但是目前关于该菌的研究,尤其在引起水产品腐败变质方面的报道较少。本实验发现,假交替单胞菌属在低温腐败样品的总菌群中相对丰度较高,表明该菌较适合低温的环境,在新鲜样品的可培养菌群中相对丰度高达近90%,而在腐败样品中可培养的比例则极低,表明这类菌大部分不可培养。乳酸菌属是一类可以发酵碳水化合物,并产生大量乳酸的细菌,广泛存在于人和动物肠道以及食品中,多数作为益生菌使用,但实际上乳酸菌能够分解蛋白质以及糖类,可引起肉类的腐败变质[22],但目前鲜见乳酸菌引起水产品腐败的相关研究报道。本实验中,乳酸菌在25 ℃腐败后的总菌群和可培养菌群中的丰度结果一致,表明该菌可能是该温度下的特定腐败菌,需要进一步实验证实。别弧菌属属于变形菌门、弧菌科,据报道该菌可能引起水产动物的疾病[18,23],梭杆菌属属于梭杆菌门、梭杆菌科,常寄生于人或动物的口腔、上消化道、肠道及泌尿生殖道和土壤中,目前鲜见别弧菌和梭杆菌引起水产品腐败的相关报道,然而,扇贝柱在不同温度下腐败后,别弧菌在总菌群中的相对丰度均很高,在25 ℃梭杆菌的相对丰度也较高,而别弧菌在可培养菌群中的相对丰度则极低,梭杆菌则不可培养,它们是否是扇贝柱的特定腐败菌,还需要进一步证实。已有的报道表明,弧菌属的细菌在海洋环境中广泛存在,种类多,是海洋环境和生物体内最常见的一类条件致病菌,此外,弧菌还具有较强的蛋白质分解能力,是水产品的特定腐败菌[17,24]。希瓦氏菌属属于变形菌门、弧菌科,能够分解蛋白产生氧化三甲胺和硫化氢,造成鱼、虾、贝等水产品腐败和不良气味产生,是目前研究得最多的水产品特定腐败菌之一[25-28]。通过实验发现,弧菌属、希瓦氏菌属和乳酸菌属的细菌在虾夷扇贝柱的可培养菌群中的相对丰度较高,尤其是弧菌的比例高达50%,表明这几类细菌易培养分离。
虾夷扇贝生活在海洋环境中,能够附着或通过滤食富集海洋环境的微生物,这些微生物在加工或储运过程中影响着产品的质量安全[29-30]。本实验通过两种方法测定了虾夷扇贝柱在新鲜及腐败状态下的总菌群结构和可培养菌群结构,两种方法获得的结果不同。通过平板分离获得的可培养菌为活菌,可以进一步研究菌株的致腐能力及机制,目前在防腐保鲜的研究中仍占有重要地位。基于宏基因组获得的菌群结构多样性和丰度更完整地反映样品中实际菌群的特征,但部分优势菌不易培养分离,获得的优势菌群是否为特定腐败菌还需要进一步验证,如果经验证腐败优势菌与品质劣化呈正相关性,该方法将在水产品的品质变化及保鲜技术研发中得到更广泛应用。下一步将通过开展基于宏基因组的高通量测序技术分析虾夷扇贝柱在腐败过程中菌群变化与品质下降的相关性、腐败优势菌对扇贝柱的降解作用等相关研究,从而揭示微生物在虾夷扇贝柱品质劣化中的作用机制,并为虾夷扇贝柱保鲜工艺的研究提供基础。
