人工养殖下达氏鳇幼鱼肠道菌群组成分析
2018-09-18吴金平岳华梅李创举乔新美陈细华
阮 瑞,吴金平,李 营,岳华梅,李创举,乔新美,熊 伟,陈细华
(中国水产科学研究院长江水产研究所,武汉 430223)
细菌、古细菌、病毒、真菌以及原生动物等肠道微生物在宿主生长发育过程中起重要作用,不仅可以促进宿主的免疫系统发育,还能辅助宿主营养吸收[1,2]。对脊椎动物肠道微生物群落结构及功能的认识主要来源于对哺乳动物的研究。近年来,随着生物技术的发展和对肠道微生物重要性的认识,有关鱼类肠道微生物的组成、功能、影响因子以及调控方法等研究也越来越多[2-4]。
细菌是肠道微生物的最主要组成部分。鱼类肠道菌群主要由变形菌门(Proteobacteria)、梭杆菌门(Fusobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)和疣微菌门(Verrucomicrobia)等组成[5-7]。鱼类肠道微生物不仅可以分泌多种消化酶(如:蛋白酶、纤维素酶、磷酸酶等)促进鱼类消化吸收营养物质[8],而且还可以合成多种维生素,如维生素B12[3]。另外,稳定的肠道微生物结构可以有助于保护鱼类免受病原微生物通过肠道侵害[9]。
鲟是目前地球上最古老的辐鳍鱼类,被誉为活化石。鲟肉质鲜美,其鱼卵制作的鱼子酱营养价值极高。随着鲟养殖业迅速发展,关于其对氨基酸、脂肪酸、糖类、维生素、矿物元素的需要量进行了初步研究,表明不同种类的鲟对蛋白质和脂肪需要量以及不同蛋白源和脂肪源的利用率存在差异[10]。但仍缺乏关于如何提高饲料氮磷利用率以及功能性饲料配制的研究[11]。掌握鲟肠道微生物的确切组成及相应功能,将能够为改善其肠道微生物类群以促进其消化吸收和健康生长、减少氮磷排放等功能性饲料配制提供参考。因此,本研究拟利用16S rRNA扩增子进行高通量测序技术对达氏鳇(Husodauricus)幼鱼肠道内容物菌群结构进行初步分析,为全面深入了解鲟不同种类、不同生长阶段的肠道微生物群落结构提供帮助,也为今后探讨鲟肠道微生物在提高免疫力、生产性能、饲料利用率以及蛋白源替代等研究提供基础。
1 材料和方法
1.1 样品采集与DNA提取
养殖试验在中国水产科学研究院长江水产研究所太湖试验场(湖北荆州)进行。使用地下水在玻璃纤维平底圆池(直径105 cm)中进行饲养,每个养殖池中养殖30尾,共有三个养殖池。利用实验室配制的饲料对达氏鳇幼鱼进行每天(8:00和15:00)两次投喂。养殖期间水温为18~20 ℃。饲养饲料配方如表1所示。4周后每个养殖池采集2尾共采集6尾达氏鳇幼鱼后肠内容物,迅速将样品放入液氮中冻存1 h后,转入-80 ℃冰箱长期保存。达氏鳇幼鱼个体信息如表2所示。样品DNA提取按照QIAamp®PowerFecal®DNA Kits(QIAGEN,Germany)说明书进行操作。
1.2 PCR扩增与高通量测序
将提取的DNA样品送往北京诺禾致源科技股份有限公司进行建库和16S rRNA扩增子高通量测序。流程为:利用带有序列标签的16S V3-V4变异区引物(341F:5′-CCTAYGGGRBGCASCAG-3′,806R:5′-GGACTACNNGGGTATCTAAT-3′)[12,13]对提取样品DNA 进行扩增;并对扩增产物进行目的条带割胶回收以及构建测序文库;最后通过Thermofisher公司的Ion S5TM XL测序系统进行单末端高通量测序。
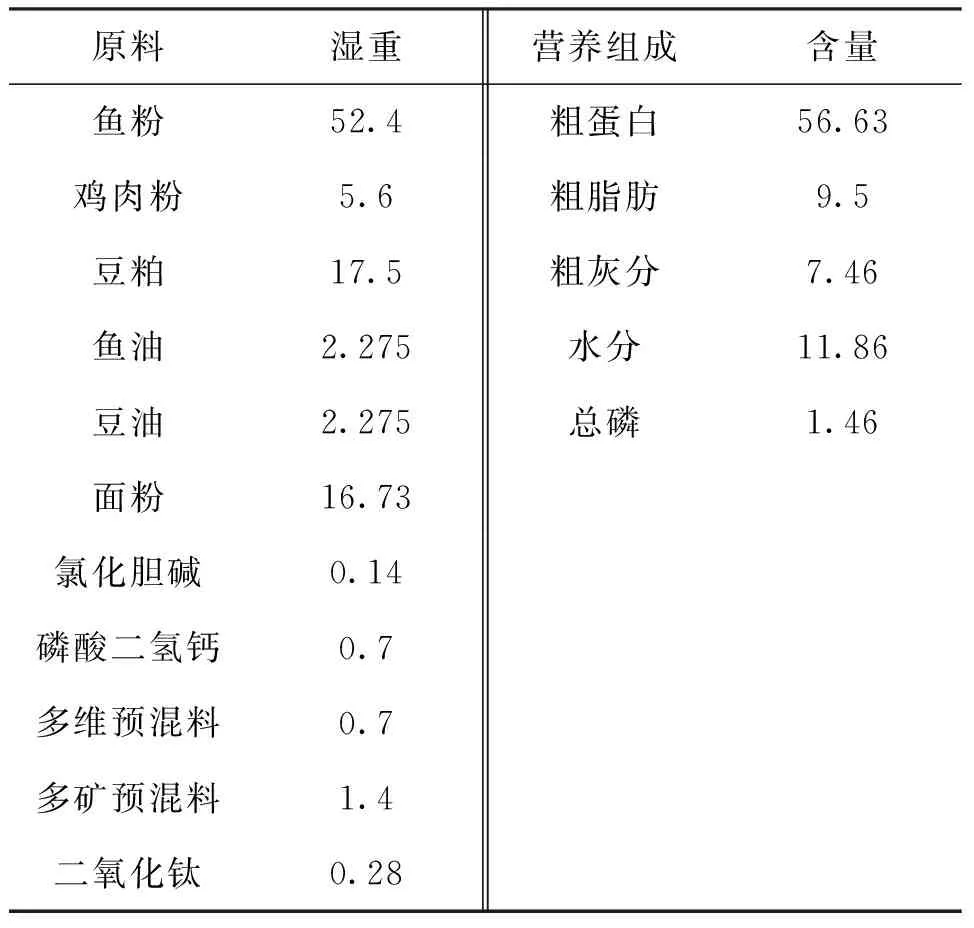
表1 饲料配方及营养成分Tab.1 Formulation and nutrient contents of experimental diets %
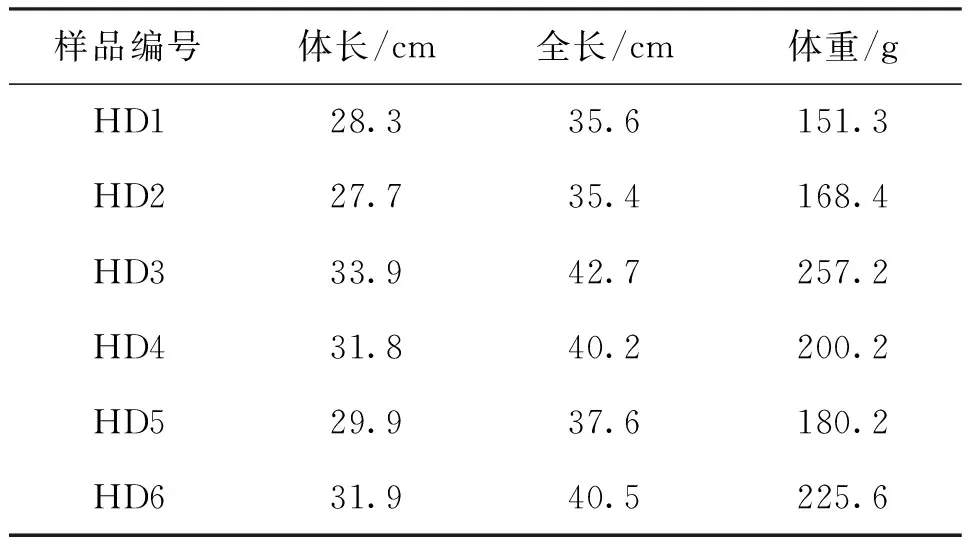
表2 样品信息Tab.2 Information of six H.dauricus
1.3 数据分析
首先使用Cutadapt(V1.9.1)[14]对测序原始数据(Raw Reads)进行低质量序列剪切和根据序列标签将得到的序列拆分到每个样品,并将序列标签和引物序列去除。然后将上述得到的序列通过UCHIME Algorithm去除嵌合体序列,得到有效序列(Clean Reads)[15,16]。利用Uparse(V7.0.1001)软件[17]对Clean Reads按相似性为97%进行操作分类单元(Operational Taxonomic Units,OTUs)聚类,同时将含有小于10条Clean Reads的OTUs去除,并选取出现频次最高的Clean Read作为该OTU的代表序列。随后用Mothur方法与SILVA(http://www.arb-silva.de/)的SSUrRNA数据库进行物种注释分析(设定阈值为0.8~1)[18,19]。最后以样品中数据量最少的为标准进行均一化处理,利用Qiime(V1.9.1)软件进行后续数据分析。
2 结果
2.1 16S rRNA高通量测序
6尾人工养殖达氏鳇幼鱼后肠内容物经DNA提取、16S rRNA的V3-V4引物扩增及高通量测序,共获得558 610条Raw Reads和541 575条Clean Reads。每个样品的Raw Reads、Clean Reads和平均长度如表3所示。去除不能够被注释以及含有小于10条Clean Reads的OTUs,最后每个样品以84 436条Clean Reads参与后续分析。6个样本总共被划分成73个OTUs,而每个样品具体含有的OTUs见表3,共有29个核心OTUs。通过Qiime软件计算每个样品的测序深度指数(Good’s coverage)都大于99.99%(表3),表明了每个样本到达了足够的测序覆盖度且本次检测到的OTUs也足够代表该批次抽样群体。同时样本稀释曲线(Rarefaction curve)趋向平坦表明测序数据量是足够的(图1)。高通量测序原始数据已上传至NCBI的Sequence Read Archive(SRA)数据库,收录号为:PRJNA451304。
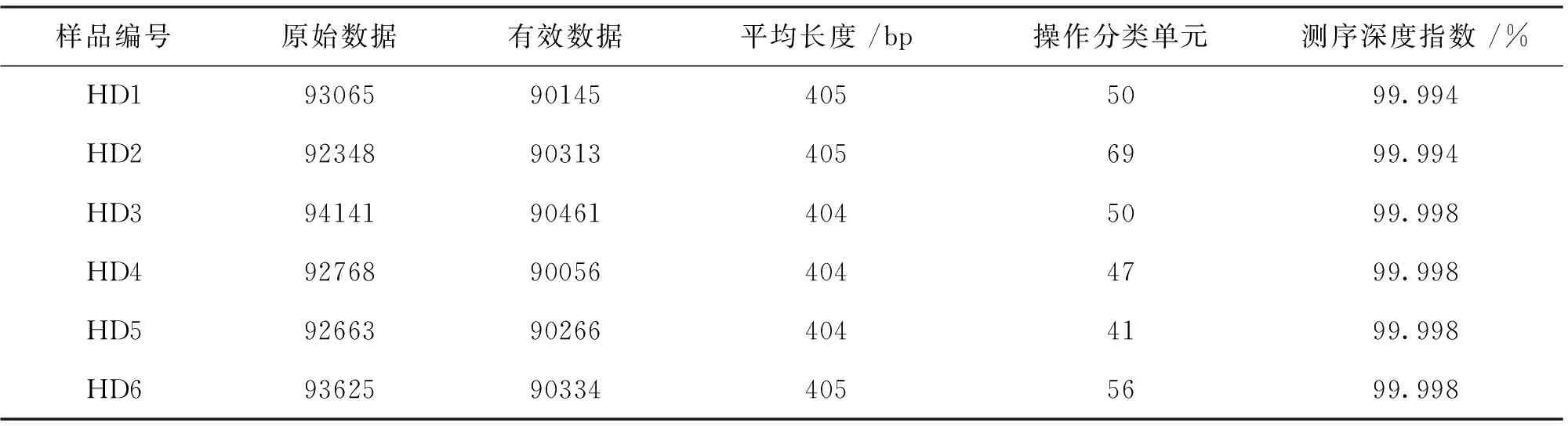
表3 高通量测序数据基本信息表Tab.3 Summary of high-throughput sequencing from six intestinal contents of H.dauricus
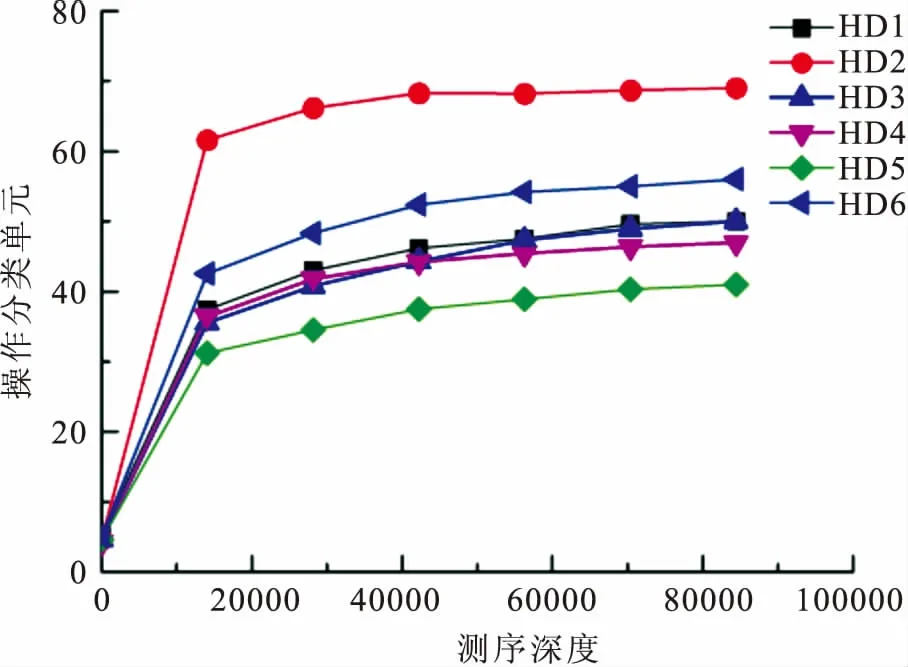
图1 稀释曲线Fig.1 Rarefaction curve of OTUs
2.2 达氏鳇幼鱼肠道菌群组成分析
通过对6尾达氏鳇幼鱼肠道内容物菌群进行物种注释发现73个OTUs主要划分为7个门,分别为厚壁菌门(Firmicutes)占98.253%、梭杆菌门(Fusobacteria)占1.225%、变形杆菌门(Proteobacteria)占0.293%、柔膜菌门(Tenericutes)占0.166%、拟杆菌门(Bacteroidetes)占0.045%、蓝藻门(Cyanobacteria)占0.011%、放线菌门(Actinobacteria)占0.007%。在门分类水平下,不同样品中的菌群相对丰度的结果也表明厚壁菌门在达氏鳇人工养殖幼鱼的肠道内容物中占有绝对优势(图2A)。
在目分类水平下,6个样品整体上主要分为7个目,分别为梭菌目(Clostridiales)占98.216%、梭杆菌目(Fusobacteriales)占1.225%、肠杆菌目(Enterobacteriales)占0.293%、支原体目(Mycoplasmatales)占0.166%、拟杆菌目(Bacteroidales)占0.045%、芽孢杆菌目(Bacillales)占0.026%和乳杆菌目(Lactobacillales)占0.009%。梭菌目、芽孢杆菌目和乳杆菌目都属于厚壁菌门,但是梭菌目在目分类水平下是绝对优势菌群,其具体在每个样品中所占比例如图2B所示。梭菌科(Clostridiaceae)和狭义梭菌属(Clostridium_sensu_stricto_1)是在科和属分类水平下的优势菌群,分别占各自分类中的98.137%和92.892%。梭菌科在每个样品中所占的比例都大于92.5%(图2C),狭义梭菌属在每个样品中所占的比例都大于82%(图2D)。除了优势菌群狭义梭菌属外,含量相对较多的还有3.99%的分节丝状菌(Candidatus_Arthromitus)、1.22%的鲸杆菌属(Cetobacterium)等(图2D)。通过对梭菌属进一步进行注释,发现绝大部分被注释到Clostridium colicanis。

图2 物种相对丰度柱状图Fig.2 Relative abundance at phylum,order,family and genus level for the intestinal microbiota from six aquaculture H.dauricus A是在门分类水平上的相对丰度柱状图,B是在目分类水平上的相对丰度柱状图,C是在科分类水平上的相对丰度柱状图, D是在属分类水平上的相对丰度柱状图。横坐标代表样品,纵坐标表示相对丰度。
3 讨论
本研究对人工养殖的达氏鳇幼鱼肠道内容物菌群组成与多样性进行分析,结果显示每个样品按84436条Clean Reads进行数据抽平处理后,可以划分为73 OTUs,属于7个门,其中厚壁菌门是绝对优势菌群,其余6个门菌群只占1.75%。已有研究表明大部分鱼类的肠道微生物按照相对丰度由大到小主要由变形杆菌门、梭杆菌门、厚壁菌门,以及含量比例较少的拟杆菌门和放线菌门等组成[3,20],例如:斑马鱼(Daniorerio)[21]、草鱼(Ctenopharyngodonidellus)、鲤(Cyprinuscarpio)[22,23]、褐点石斑鱼(Epinephelusfuscoguttatus)、游鳍叶鲹(Atulemate)[20]。但是,大西洋鲑(SalmosalarL.)的肠道微生物主要由厚壁菌门(50%)、变形杆菌门(23.1%)和柔膜菌门(6.9%)组成[24]。在本研究中,人工养殖的达氏鳇幼鱼的肠道内容物菌群则以厚壁菌门为最主要的菌群(占到98.253%),暗示了不同种类的鱼类肠道微生物在门分类水平上的组成存在差异。
Liu等[23]对团头鲂(Megalobramaamblycephala)、草鱼、鳜(Sinipercachuatsi)、翘嘴鲌(Culteralburnus)、鲤、鲫(Carassiusauratus)、鲢(Hypophthalmichthysmolitrix)和鳙(Hypophthalmichthysnobilis)肠道菌群进行研究发现,食性可能影响鱼类肠道菌群的组成,在属的水平上,草食性鱼肠道中主要以具有降解纤维素功能的梭菌属、柠檬酸杆菌属(Citrobacter)和纤毛菌属(Leptotrichia)为主,而在肉食性鱼肠道中主要菌群是鲸杆菌属和产蛋白酶菌(Halomonas)。本研究发现达氏鳇幼鱼肠道内容物菌群中优势菌群梭菌属与草食性鱼偏相似,同时也发现其菌群分布有少量的鲸杆菌属。这可能是与达氏鳇食性相关,因为鲟鱼为亚冷水性、杂食偏肉食性鱼类。
鲟鱼肠道、胃和肝脏组织含有淀粉酶,能够利用淀粉、葡萄糖、半乳糖和麦芽糖等糖类[25-27]。Geraylou等[28]通过饲料添加2%阿拉伯低聚木糖饲喂西伯利亚鲟(Acipenser baerii)12周后,发现能够提高西伯利亚鲟短链脂肪酸的产生和提高其免疫力,并且能够增加肠道中乳酸菌和梭菌属丰度。本研究发现梭菌属在达氏鳇幼鱼肠道菌群是绝对优势菌群,达到92.892%。而试验中饲喂的人工配合饲料中含有16.73%的面粉,可能是造成达氏鳇幼鱼肠道内容物含有高丰度的梭菌属的原因。Greetham等[29]对C.colicanis的功能鉴定发现其可以利用多种糖分产生乳酸盐、醋酸盐、甲酸盐。而短链脂肪酸对宿主肠道健康十分重要[30]。另外,鲸杆菌属能够产生大量的维生素B12,并且能够抑制潜在病原菌生长[31]。而本实验中达氏鳇幼鱼肠道菌群主要包括梭菌属,还有少量的鲸杆菌属,关于其在达氏鳇肠道中的具体功能还需要进一步验证,但为今后深入了解鲟鱼肠道微生物组成及功能、开展鲟鱼饲料添加剂等研究提供了基础。
4 结论
本研究利用16S rRNA扩增子高通量测序方法对6尾在已知饲料配方下饲养4周的达氏鳇幼鱼肠道内容物进行菌群结构和多样性分析,结果表明该饲养条件下的达氏鳇幼鱼肠道微生物的绝对优势菌群属于厚壁菌门、梭菌目、梭菌科、梭菌属,此外还包含有分节丝状菌、鲸杆菌属等少量菌群。
