基于生物信息学的条斑紫菜microRNAs及其靶标预测
2018-08-17尹旭岗吕俊平南芳茹谢树莲
尹旭岗,冯 佳,吕俊平,刘 琪,南芳茹,谢树莲,高 帆
(山西大学生命科学学院,山西 太原 030006)
条斑紫菜 (Porphyra yezoensis) 属红藻纲(Florideophyceae)红毛菜科(Bangiaceae)紫菜属,富含蛋白质、多糖以及维生素,可供食用或药用,多分布于日本、韩国及中国的北方,是我国重要的经济海藻之一[1]。随着环境条件的日益恶化,作为大型海洋经济红藻之一的条斑紫菜,面临着种质退化、品质降低和资源减少的危机。为缓解以上危机,条斑紫菜新品种的选育日益紧迫,而基因后转录水平的遗传改良是实现高效分子育种的必要途径。作为抑制基因后转录水平表达的非编码RNA,microRNA(miRNA)在植物的生长发育、新陈代谢及胁迫响应等方面起重要的调控作用。传统实验方法鉴定miRNAs较繁琐,对于非编码RNA研究较薄弱的条斑紫菜,利用生物信息学技术预测miRNAs及其靶标可极大地提高鉴定效率。
miRNA是一类具有基因后转录水平调控功能的内源性非编码小RNA。LEE等[2]在秀丽隐杆线虫(Caenorhabditis elegans)中首次发现了这类特殊的RNA分子。在相关酶及蛋白因子的作用下,原始miRNA经过2次剪切,最终形成长度约21 nt左右的成熟体,并通过切割抑制与翻译抑制2种方式实现对基因的转录后调控[3]。研究证实,miRNAs在植物生长发育、新陈代谢、胁迫响应、信号转导及其他生命活动中均发挥着重要的调控作用[4]。自2002年首次在模式植物拟南芥中发现miRNA以来,植物miRNA研究已有了快速的发展[5]。截至2018年1月,研究者们已在34种植物中发现miRNA成熟体序列约8 500条(miRBase 21.0[6]。作为一种重要的模式经济海藻,条斑紫菜miRNAs研究起步较晚。2010年,LIANG等[7]利用高通量测序联合生物信息的方法鉴定到231条条斑紫菜miRNAs,其中,7条为物种特异miRNAs;2012年,HE等[8]通过对条斑紫菜孢子体和配子体小RNA的高通测序,鉴定到14条新的miRNAs;之后再无该海藻miRNAs的相关报道。
目前,获得miRNAs的主要方法有直接克隆[9]、正向遗传[2]、深度测序[10]和生物信息[11]。与动物相比,植物间的miRNA序列相对保守,在考虑试验成本及鉴定效率的前提下,利用生物信息学技术预测植物的miRNAs及其靶标基因是可行的[12]。
本研究以条斑紫菜为研究对象,已公布植物miRNAs为探针,以条斑紫菜ESTs为模板,利用生物信息技术预测条斑紫菜miRNAs及其前体二级结构,对miRNA靶标基因及其功能进行预测,分析了条斑紫菜miRNA序列的保守性与多态性。研究结果进一步扩充了紫菜miRNA及其靶基因信息数据,为条斑紫菜功能的开发与应用、后转录水平改良条斑紫菜品质奠定了理论基础。
1 材料和方法
1.1 序列获取
miRBase 21.0数据库(http://www.mirbase.org/)[6]下载所有植物miRNAs及其前体序列(pre-miRNAs),去除冗余后形成miRNA搜索序列。Genbank(https://www.ncbi.nlm.nih.gov/) 和 Rfam(http://rfam.sanger.ac.uk)数据库[13]分别下载已公布条斑紫菜的SRA和RNA数据,作为miRNA模板序列。从文献下载已公布条斑紫菜的miRNAs相关信息数据。Genbank数据库下载紫菜mRNAs和ESTs用于条斑紫菜miRNA靶基因预测。
1.2 应用软件
序列比对软件应用BLAST 2.2.24(https://blast.ncbi.nlm.nih.gov/Blast.cgi)[14]。用 MFOLD(http:∥mfold.rna.albany.edu/)结合 Mireap(http://sourceforge.net/projects/mireap/)在线预测pre-miRNA二级结构[15-16]。用 psRNATarget(http://plantgrn.noble.org/psRNATarget/)预测miRNA对其靶标的抑制方式[17]。Target-Finder(http://targetfinder.org/)预测 miRNA潜在的靶基因。miRNA保守性用Clustal W(http://www.clustal.org/clustal2/)[18]结合 WebLoGO(http://weblogo.berkeley.edu/logo.cgi)在线工具进行分析[19],miRNA 前体的系统进化树用Clustal W结合MEGA 5.0(https://www.megasoftware.net/)进行构建[20]。
1.3 条斑紫菜miRNA成熟体预测
将miRNA搜索序列(22 431条pye-ESTs)与miRNA模板序列(已公布植物miRNAs)进行序列联配及BLAST比对,根据以下原则对条斑紫菜miRNAs序列进行筛选[21]:(1)错配碱基数不超过3 个;(2)百分比不超过(L-4+m)/L,其中,L是长度(碱基数),m是不一致的碱基数;(3)去除tRNA和rRNA及蛋白编码序列;(4)(A+U)占比30%~70%。
1.4 条斑紫菜miRNA前体预测
为进一步确认预测的条斑紫菜miRNA成熟体序列是否为条斑紫菜miRNAs,还须对其前体序列及二级结构进行预测。据统计,植物pre-miRNA序列长度约(144.57±56.91)nt[22]。以成熟体序列为核心,上下游序列各延伸120 nt作为pre-miRNA预测的源序列。根据以下原则预测pre-miRNA二级结构[23]:(1)5′和 3′端不超过 2 nt的悬挂;(2)loop 或泡状凸起大小不超过4 nt;(3)miRNA和 miRNA*必须位于“发夹”结构的一条臂上;(4)最小折叠自由能(MFE)不超过 -75.3 kJ/mol;(5)错配碱基数不超过4;(6)miRNA成熟体与其互补链间无loop或gap现象。符合以上原则即可确定为条斑紫菜miRNAs。
1.5 条斑紫菜miRNA靶基因预测
将条斑紫菜miRNAs与紫菜ESTs及mRNAs进行BLASTn比对,根据以下原则预测miRNA靶标序列[24]:(1)错配碱基数不超过 4 个;(2)2~12 位点间错配碱基数不超过1个,其中,10~11位点间无碱基错配;(3)在位点13~21之间错配碱基数不超过4个,且不能有 2个连续的错配碱基;(4)pre-miRNA具有最低的MFE和最高MFEI。获得miRNA靶标序列后,根据miRNA靶标的9~11 nt位点间是否完美互配,预测miRNA是否为切割抑制方式。BLASTp结合Pfam数据(http://pfam.sanger.ac.uk/)[25],预测并分析miRNA靶基因及其功能。
1.6 条斑紫菜miRNAs的保守性与miR164s系统进化分析
利用序列联配分析条斑紫菜已公布miRNAs的保守性,搜索保守性较高的区域,绘制条斑紫菜miRNA保守序列图谱。根据条斑紫菜已公布的pre-miRNAs,以贝叶斯算法[26]构建包含条斑紫菜在内的所有植物miR164s系统进化树,分析各miR164s间的系统进化关系,推测潜在的miR164基因家族。
2 结果与分析
2.1 条斑紫菜miRNAs及其前体序列特征分析
根据植物miRNA成熟体序列的保守性,本研究从15条候选序列中共预测到5条新的条斑紫菜miRNAs(表1),筛选率为33.3%。由表1可知,预测miRNAs的长度为21,22 nt,符合植物miRNA长度标准[27]。预测的条斑紫菜miRNAs前体序列的(A+U)含量百分比在19.1%~57.1%,基本符合植物miRNA的AU碱基含量占比[21]。前体序列二级结构均呈典型的茎环状,成熟体序列均位于一条臂上,其中,3条位于5′端,2条位于3′端。前体序列长度在 136~293 nt,平均 209 nt,大于动物(70~80 nt)的长度[22]。有研究表明,为维持miRNA前体构型的稳定性,其二级结构的MFE值应小于-75.3 kJ/mol,MFEI值应不小于0.85[22]。本研究获得的条斑紫菜pre-miRNAs的 MFE 值在 -76.6~-128.6 kJ/mol,MFEI值在1.46~3.87,均符合pre-miRNA二级结构的能量阈值要求(图1)。综合以上特征,本研究预测的条斑紫菜miRNAs信息基本可信。

表1 预测的条斑紫菜miRNAs信息
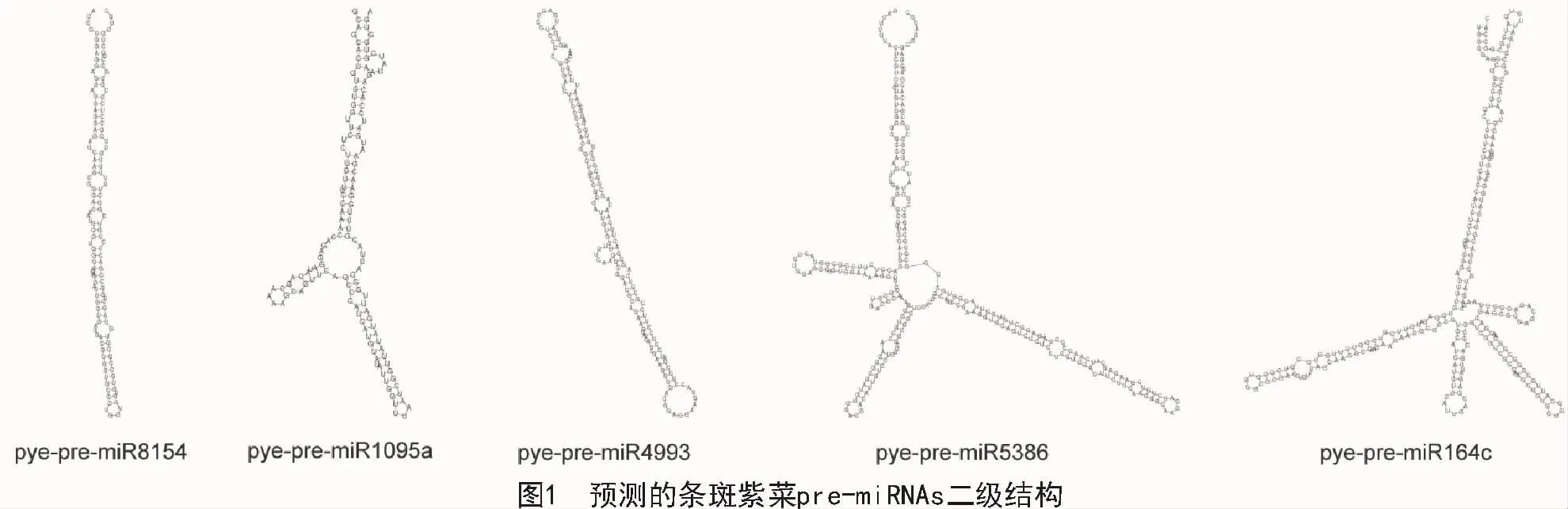
2.2 条斑紫菜miRNAs的保守性
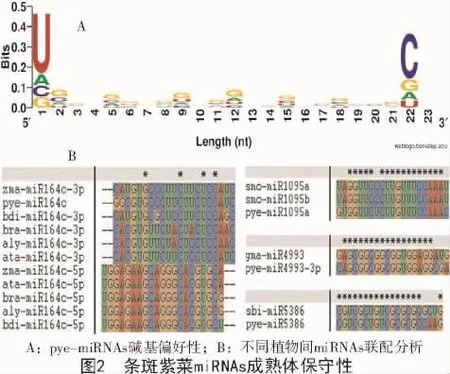
根据本研究获得的条斑紫菜miRNA数据结合LIANG等[7]和HE等[8]的数据,对条斑紫菜miRNAs的各位置碱基的偏好性进行分析。由图2-A可知,条斑紫菜miRNAs碱基体现了首尾偏好特征,首位碱基偏好U(符合植物miRNA首位碱基偏好)[10],末位碱基偏好C,其余位置无明显的碱基偏好性。将研究所得5条pye-miRNAs与已知植物miRNAs进行序列联配,结果显示,条斑紫菜miR164c与其他10种植物间保守性较高,同源一致度达96.6%;此外,pye-miR8154 与 cpa-miR8154,pye-miR1095a与smo-miR1095a/b,pye-miR4993与 gma-miR4993-3p(图2-B)的同源一致度均超过98%。研究结果证实了条斑紫菜与其他不同植物物种间miRNA成熟体序列的保守性特征。
2.3 条斑紫菜miRNA靶标及其靶基因功能的预测
因植物中多数miRNAs几乎与其靶标位点严格地互补配对[28],依此原则本研究共预测到pyemiRNAs的靶标位点64个,其中,pye-miR4993-3p预测到的靶标最多(58个),而pye-miR164c中未预测到任何靶标位点(表2)。序列联配分析结果表明,pye-miRNAs与其靶标的错配率仅为2.3%。psRNATarget预测pye-miRNAs对其靶基因的主要抑制类型为切割抑制。在细胞的特定部位,往往有某种蛋白质的高丰度表达或者特异性表达[29]。根据miRNA靶基因注释信息及蛋白质序列比对结果,条斑紫菜中共预测到可靠的miRNA靶基因5条,功能涉及代谢和生长发育(表2),其余靶基因产物均为未知的假定蛋白,靶基因预测结果尚不能确定。

表2 预测的条斑紫菜miRNAs靶标信息
2.4 条斑紫菜miR164的系统进化
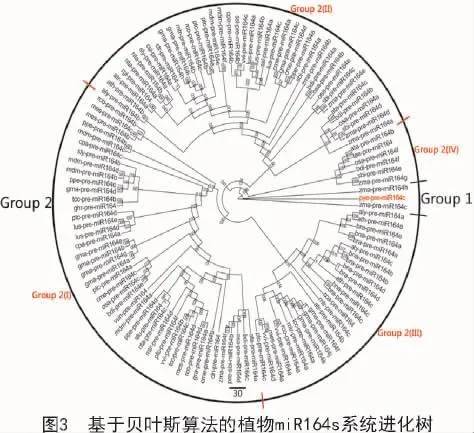
利用已预测条斑紫菜及已公布植物miR164前体序列,对条斑紫菜miR164在植物间的系统进化关系进行分析。由图3可知,所有pre-miR164s被明显地分成2个大组。第一大组仅含有包括pye-premiR164c,zma-pre-miR164c 和 zma-pre-miR164h这3个pre-miR164基因家族子序列,表明三者间有较近的进化距离及较低的碱基替代率。第二大组含有的pre-miR164基因家族成员数最多(116个家族成员),其又可分为4个亚组。第I亚组含44个pre-miR164s,分别属于包括水稻、高粱、玉米、山羊草在内的24个植物物种。第II亚组含41个pre-miR164s,分别属于包括互叶梅、地黄、烟草、番茄在内的23个植物物种。第III亚组含26个pre-miR164s,分别属于包括蒺藜苜蓿、芜菁、油菜、拟南芥在内的13个植物物种。第IV亚组含6个pre-miR164s,分别属于二穗短柄草、小麦、水稻、山羊草、玉米和高粱6个植物物种。由前体序列的进化树中可以看出,进化树整体较为分散,尤其是第二大组除可分为4个亚组外,各亚组又形成了各自的分支。从miR169s宿主物种分布来看,miR164s的进化与物种进化并不同步。有些亲缘关系较远的物种可以聚到同一分支上,如pye-pre-miR164c和zma-pre-miR164h/g;而同一物种的miR169家族成员之间却相距较远,如玉米的6个miR164家族成员分布在除第二大组第III亚组外的其他各组中。
3 讨论
目前,利用生物信息学技术预测紫菜miRNAs及靶标是一种成本较低且便捷的研究手段,但该方法也存在序列筛选率低(本研究仅预测到5条新的pye-miRNAs)及必要的试验验证等缺陷。尽管pye-miRNAs分析结果表明其序列信息基本可信,但进一步的实时定量RT-PCR[30],RNA干扰[31],基因过表达[32]或Northern blot[33]等仍有必要。为进一步挖掘调控条斑紫菜特定功能的miRNA分子,不同控制条件下的小RNA文库测序需继续进行。尽管基于第2代高通量测序技术的条斑紫菜miRNAs的鉴定已有报道[7-8],但随着第3代高通量测序技术的逐步完善,更丰富、更准确的pye-miRNAs信息将被挖掘。
由于miR164s是植物中古老的、家族成员较多的保守miRNA基因家族[34],考察条斑紫菜miR164在不同植物间的系统进化地位很有必要。然而,研究结果显示了miR164家族成员在不同植物间的多态性与随机性特征,且与物种进化不同步。这可能与不同植物基因组的复制事件以及miR164前体序列的产生(可产生于前体序列的5′或3′端)与自身的变异有关,表明了植物miRNA前体的复杂性,推测该家族的基因可能以不同的方式和速度进化。
研究已证实,miR164s对拟南芥[35]、番茄[36]及水稻[37]的分生组织、辅助分生组织及叶缘锯齿状的形成起重要的调控作用。miR164的靶基因为CUC(Cup Shaped cotyledon)基因家族,控制植物器官分生与边界的形成[38]。本研究未在条斑紫菜中鉴定到miR164靶基因,运用降解组测序及RACE的方法继续对其靶基因进行鉴定,将有利于条斑紫菜叶状体形态形成的深入研究,为紫菜后转录水平的分子选育及其功能的开发利用奠定理论基础。
综上,本研究将为条斑紫菜这一重要紫菜品种的资源保护与利用、后转录水平的品质改良提供重要的参考。
