食品源单增李斯特菌喹诺酮类抗生素耐药机制研究
2018-08-09程健恒陈谋通陈月桃张菊梅吴清平
程健恒, 陈谋通, 陈月桃,2, 张菊梅, 吴清平,*
(1.广东省微生物研究所/省部共建华南应用微生物国家重点实验室/广东省菌种保藏与应用重点实验室/广东省微生物应用新技术公共实验室,广东广州 510070;2.华南农业大学食品学院,广东广州 510642)
单核细胞增生李斯特菌(Listeria monoocytogens,LM),是一种革兰氏阳性兼性厌氧食源性致病菌,被世界卫生组织列为四大食源性致病菌之一。LM可感染人畜发病,可导致脑膜炎、败血症、流产和肠胃炎等严重李斯特菌病,致死率为20% ~30%,易感人群为老人、孕妇、新生儿及免疫力低下的人群。
喹诺酮类药物是指含有4-喹诺酮类母核的合成抗菌药物,是现今临床治疗细菌感染性疾病的重要药物。尽管喹诺酮类药物未列为临床治疗李斯特菌病的一线抗生素,但是由于此类药物在临床上使用的频率越来越高,间接上导致了耐受喹诺酮类LM的出现[1]。第三代喹诺酮类药物具有良好的组织渗透性、抗菌活性强、不良反应少,被广泛应用于人医、兽医和渔业等方面,常用品种有左氧氟沙星、环丙沙星、氧氟沙星、恩诺沙星等。国内外研究表明,LM对喹诺酮类抗生素的耐药情况较为严重[2-5]。
前期研究表明,细菌对喹诺酮类耐药的机制主要有3种:1)由于编码促旋酶(gyrA和gyrB编码)或扑拓异构酶Ⅳ(parC和parE)基因的喹诺酮耐药决定区(quinolone resistance-determining region,QRDR)发生突变,导致氨基酸置换,使得抗生素不能与靶酶结合而耐药[6]。在革兰氏阴性菌中,促旋酶比扑拓异构酶更敏感,因此革兰氏阴性菌耐喹诺酮类菌株一般在gyrA和gyrB的位点上发生突变(主要在gyrA上);但在革兰氏阳性菌中,扑拓异构酶Ⅳ通常是喹诺酮类抗生素的首要目标,因此,革兰氏阳性菌的喹诺酮类耐药突变首先在parC上发生[7]。2)喹诺酮类耐药机制是通过改变膜的通透性或通过外排泵作用降低细胞内抗生素浓度,从而造成喹诺酮类抗生素耐药[7-9]。目前研究发现,LM有两个外排泵基因与耐药性相关,其中一个是mdrL,能泵出大环内酯类、头孢噻肟类等抗生素。通常情况下,喹诺酮类抗生素也能被MDR系统泵出,但由于底物特异性较低,当MDR系统过度表达时,会赋予细菌低水平的喹诺酮类耐药[7]。另一个外排泵基因lde,它对LM产生喹诺酮耐药发挥着重要作用[1]。3)由质粒介导的(plasmid-mediated quinolone resistance,PMQR)喹诺酮类耐药。PMQR基因通常位于移动质粒上,并可通过水平基因转移过程中在细菌细胞间传播。目前发现的质粒介导喹诺酮类耐药基因有3种,分别是qnr,aac(6')-lb-cr以及质粒介导的外排泵基因qepA和oqxAB[10]。
本研究对全国代表性地区分离的945株食源性LM进行喹诺酮类抗生素敏感性分析,研究32株耐受喹诺酮类抗生素的分离菌株携带耐药基因、外排泵和耐药质粒的情况,并在此基础上进行遗传多样性分析,旨在探讨LM耐受喹诺酮类抗生素的潜在分子机制,为预防和控制LM提供数据参考。
1 材料与方法
1.1 实验材料
1.1.1 菌株及来源
自2012年7月至2016年7月,本实验室从全国43个代表性城市或地区共分离筛选出食源性单增李斯特菌945株,用终浓度为15%的甘油管保藏于-40℃冰箱中备用。
1.1.2 培养基与试剂
李斯特菌显色培养基、脑心浸出液培养基等培养基、一次性接种环,广东环凯微生物科技有限公司;盐酸环丙沙星,中国食品药品检定研究院技术服务部标准品;利血平,上海阿拉丁生化科技股份有限公司;PrimeSTARⓇHSDNA Polymerase,宝生物工程(大连)有限公司;2×DreamTaq Green PCR Master Mix,Thermo Fisher Scientific公司;Trans2K DNA marker,北京全式金生物技术有限公司;GoldViewⓇ核酸染料,北京索莱宝(Solarbio)公司;细菌基因组DNA提取试剂盒,广州美基生物科技有限公司;电泳级琼脂糖,西班牙Biowest公司;抗生素药敏纸片,英国Oxoid公司;96孔板,美国康宁公司;PCR引物均由英潍捷基(上海)贸易有限公司合成。
1.1.3 仪器与设备
DNP-9162型电热恒温培养箱、HK-C2S型单人双面垂直洁净工作台,广东环凯微生物科技有限公司;ZEALWAY GI-54TW型高压灭菌锅,美国致微仪器股份有限公司;EPOCH2型微孔板分光光度计,美国伯腾仪器有限公司;BiometraTone 96G型PCR仪,德国耶拿分析仪器股份公司;Tanon2500型凝胶成像系统,上海天能科技有限公司;LightCycler 96型荧光定量PCR仪,美国罗氏(Roche)公司;游标卡尺,广西桂林广陆数显仪器厂。
1.2 实验方法
1.2.1 抗生素敏感性分析
由于目前美国临床实验室标准化委员会(Clinical and Laboratory Standards Institute,CLSI)指南中尚未制定李斯特菌抗生素敏感测试的参考标准,因此采用CLSI 2017年版(M100-S27)的葡萄球菌属临界值判断标准作为参考(表1),采用KB法对本实验室保存的945株食源性单核细胞增生李斯特菌进行喹诺酮类抗生素敏感性分析。金黄色葡萄球菌ATCC 25923和大肠杆菌 ATCC 25922作为药敏纸片质量控制菌株。采用游标卡尺测定抑菌圈大小,精确至0.01 mm。筛选出的喹诺酮类耐药的单增李斯特菌用于后续实验。
1.2.2 最小抑菌浓度实验
利用去离子水配制质量浓度为256μg/mL的环丙沙星母液,并用针筒过滤器(孔径0.22μm)过滤后放置于-20℃冰箱备用。实验用菌株37℃过夜培养后,利用BHI无菌液体培养基稀释制备浓度为105CFU/mL的初始菌液。首先在96孔板加入100 μL无菌BHI培养基,吸取100μL环丙沙星母液在96孔板上,使用二倍稀释法进行稀释。每孔接种100μL的初始菌液,环丙沙星终质量浓度分别为64、32、16、8、4、2、1、0 μg/mL,Parafilm 封口膜封板后于 37 ℃培养18 h,使用EPOCH2微孔板分光光度计读取每个孔的OD595吸光值。根据OD595吸光值判定,培养基中未见菌株生长的环丙沙星最低浓度值即为最小抑菌浓度(minimal inhibit concentration,MIC)值。

表1 喹诺酮类抗生素药敏判断标准Tab.1 Criterion of quinolone resistance in Listeria monocytogenes
1.2.3 PCR扩增QRDR和PMQR基因
按照细菌基因组提取试剂盒的操作步骤提取LM的基因组作为扩增模版,其中QRDR相关基因gyrA、gyrB,parC、parE、fepR 的扩增引物和反应条件如表2。PCR体系为50μL,包含5×Prime STAR buffer(Mg2+Plus)10μL,dNTP mixture(4 mmol/L)4μL,正向引物与反向引物各0.3μmol/L,2μL基因组以及1.25 UTaq DNA聚合酶。扩增条件为:94℃预变性5 min,94℃变性30 s,退火1 min,72℃延伸1 min,35个循环后,72℃延伸10 min。扩增产物经测序后,使用DNAMAN软件对其序列进行拼接。以单增李斯特菌标准菌株EGDe基因组序列作为参考序列,利用Mega 7.0软件对QRDR基因的测序序列进行BLAST比对分析,以确定其QRDR基因突变情况。
PMQR 相关基因 qnrA、qnrB、qnrD、qnrS的扩增引物与反应条件如表3。扩增体系为20μL,包含10μL 2×Dream Taq Green PCR Master Mix,正向引物与反向引物各0.3μmol/L以及2μL基因组DNA。扩增条件为94℃预变性5 min,94℃变性30 s,退火1 min,72℃延伸1 min,30个循环后,72℃延伸10 min。产物利用1.5%琼脂糖凝胶(含0.005%goldview staining)电泳进行分离(100 V,40 min),利用Tanon 2500UV照相系统拍照并保存为TIFF格式。

表2 LM的QRDR相关基因引物Tab.2 Primers of QRDR genes in Listeria monocytogenes

表3 LM的PMQR相关基因引物Tab.3 Primers of PMQR genes in Listeria monocytogenes
1.2.4 外排泵lde检测及抑制实验
对32株喹诺酮类抗生素耐药的单增李斯特菌的基因组进行PCR扩增,检测外排泵基因lde,引物由本研究设计(ldeF:5′-ATAGGCTGGGTAAGTGCTGCTGC-3′,ldeR: 5′-ATTTGCGTCGTCTAAACTCTCAGGC-3′)。 扩增体系为 20μL,包含 10μL 2×Dream Taq Green PCR Master Mix,正向引物与反向引物各0.3μmol/L以及2μL基因组DNA。扩增条件为94℃预变性5 min,94℃变性30 s,60℃退火1 min,72℃延伸1 min,30个循环后,72℃延伸10 min。产物利用1.5%琼脂糖凝胶(含0.005%goldview staining)电泳进行分离(100 V,40 min),利用Tanon 2500UV照相系统拍照并保存为TIFF格式。
利用三氯甲烷溶解利血平后再加入无水乙醇制备终质量浓度20 mg/mL的利血平母液,利用去离子水配制质量浓度为256μg/mL的环丙沙星母液。两种溶液用针筒过滤器(孔径0.22μm)过滤后放置于-20℃冰箱备用。实验用菌株37℃过夜培养后,利用BHI无菌液体培养基稀释制备浓度为105CFU/mL的初始菌液。首先在96孔板上加入100 μL无菌BHI培养基,吸取100μL环丙沙星母液在96孔板上,使用二倍稀释法进行稀释。每孔接种100μL的初始菌液,并加入终质量浓度为20μg/mL的利血平,Parafilm封口膜封板后37℃培养18~20 h,使用EPOCH2微孔板分光光度计读取每个孔的OD595吸光值。根据OD595吸光值进行判断,培养基中未见菌体生长的环丙沙星最低浓度值即为MIC值。加入利血平后MIC值下降4倍及以上的菌株视为外排泵阳性菌株。
1.2.5 遗传多样性分析实验
利用多重PCR进行耐受喹诺酮类抗生素的LM分离菌株血清组分析[16]。该多重PCR可将13种血清型的单增李斯特菌分为血清组Ⅰ.1(血清型1/2a和3a),Ⅰ.2(血清型1/2c和3c),Ⅱ.1(血清型4b,4d和4e),Ⅱ.2(血清型1/2b,3b和LM)和Ⅲ(血清型4a和4c)。反应体系为50μL,包含25.0 μL 2×DreamTaq Green PCR Master Mix,prs正反向引物各 0.15 μmol/L,Lmo0737、Lmo1118、ORF2819、ORF2110正反向引物各0.3μmol/L以及2μL基因组DNA,用灭菌ddH2O补齐至50μL。扩增条件为94℃预变性3 min;94℃变性30 s,53℃退火70 s,72℃延伸70 s,35个循环后,72℃延伸10 min。产物利用1.5%琼脂糖凝胶(含0.005%gold-view staining)电泳进行分离(100 V,40 min),利用 Tanon 2500UV照相系统拍照并保存为TIFF格式。
通过扩增LM的7个管家基因(abcZ,bglA,cat,dapE,dat,ldh和lhkA)进行MLST分析,管家基因的引物序列[17],ldh引物由本研究设计(ldhF:5′GACAGAACAATTGGGGATGCAATG;ldhR:AACGCCGTAGAATGTAGCGCCT)。PCR体系为50μL,包含5×PrimeSTAR buffer(Mg2+Plus)10μL,dNTPmixture(10 mmol/L)4μL,正向引物与反向引物各0.3 μmol/L,2μL基因组DNA以及1.25 U DNA聚合酶。PCR扩增条件:94℃预变性4 min,94℃变性30 s,52℃退火30 s(bglA为45℃以及ldh为60℃),72℃延伸2 min,35个循环之后,72℃延伸10 min。扩增产物进行双向测序,利用法国巴斯德研究所Listeria MLST数据库(http:∥bigsdb.pasteur.fr/listeria/listeria.html)进行MLST分析。基于MLST的7个管家基因(abcZ、bglA、cat、dapE、dat、ldh 和 lhkA)连锁序列,利用 MEGA 7.0构建Neighbor-Joining系统发育树。
2 结果与分析
2.1 抗生素敏感性结果分析
对945株食源性单核细胞增生李斯特菌进行抗生素敏感性分析发现,其中有6株(0.63%)对左氧氟沙星耐药,32株(3.39%)对环丙沙星耐药,30株(3.17%)对氧氟沙星耐药;另外,有 142株(15.03%)对左氧氟沙星中度耐药,367株(38.84%)对环丙沙星中度耐药,300株(31.75%)对氧氟沙星中度耐药。本实验选择32株喹诺酮类耐药的LM菌株进一步研究(图1)。
2.2 环丙沙星MIC实验结果分析
32株LM菌株对环丙沙星的药物敏感性结果,如表4。表4显示,8株LM对环丙沙星MIC值为4μg/mL,8株对环丙沙星MIC值为8μg/mL,9株对环丙沙星MIC值为16μg/mL,5株对环丙沙星MIC值为32μg/mL,2株对环丙沙星MIC值为256μg/mL。加入外排泵抑制剂利血平后,32株对喹诺酮耐药的LM的MIC值发生变化;其中,有7株的MIC值没有发生变化,有15株对环丙沙星的MIC值降低为原来的1/2,9株降低为原来的1/4,1株降低为原来的1/8。对32株喹诺酮类抗生素耐药的LM进行lde基因的PCR扩增,结果如图2。32株喹诺酮类抗生素耐药的LM菌株中皆含有lde基因。
2.3 QRDR基因突变和PMQR基因检测结果分析
对32株喹诺酮耐药LM菌株的gyrA、gyrB、parC、parE以及fepR基因QRDR进行测序,经过基因序列比对后发现,5个基因均有一定的碱基突变。其中,gyrA、gyrB、parC、parE 4个基因虽然发现碱基突变,但均属于同义突变,未引起氨基酸序列的变化。如表4所示,fepR基因发现了7个突变位点,且引起了氨基酸突变,但均非文献报道的关键突变位点(G61T)[18]。对32株喹诺酮耐药的LM进行PMQR基因的PCR扩增,均未检出qnr基因。
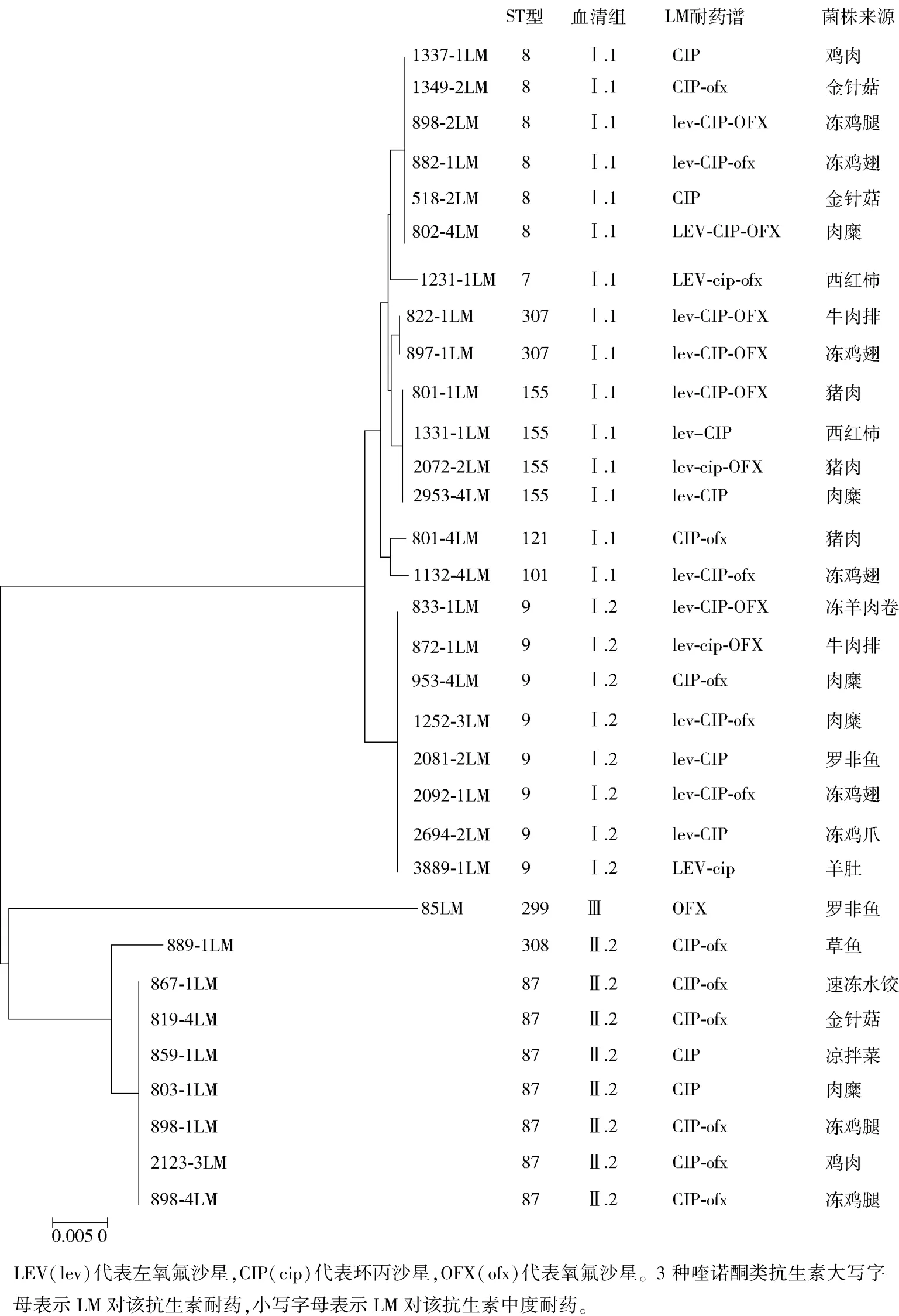
图1 32株喹诺酮耐药LM分离菌株特性分析Fig.1 Characteristics of 32 quinolone-resistant Listeria monocytogenes isolates
2.4 遗传多样性分析
如图1所示,采用多重PCR技术对32株喹诺酮类抗生素耐药的LM进行血清组分析,有15株属于血清组Ⅰ.1(血清型1/2a和3a),8株属于血清组Ⅰ.2(血清型1/2c和3c),1株属于血清组Ⅱ.1(血清型4b,4d和4e),7株属于血清组Ⅱ.2(血清型1/2b,3b和7),1株属于血清组Ⅲ血(清型4a和4c)。MLST分型技术将32株耐受喹诺酮类抗生素的LM 分属 10种 STs型,分别为 ST7、ST8、ST9、ST87、ST101、ST121、ST155、ST299、ST307 和 ST308。其中,ST87(7株)、ST9(8株)、ST8(6株)和 ST155(5株)为优势ST型。
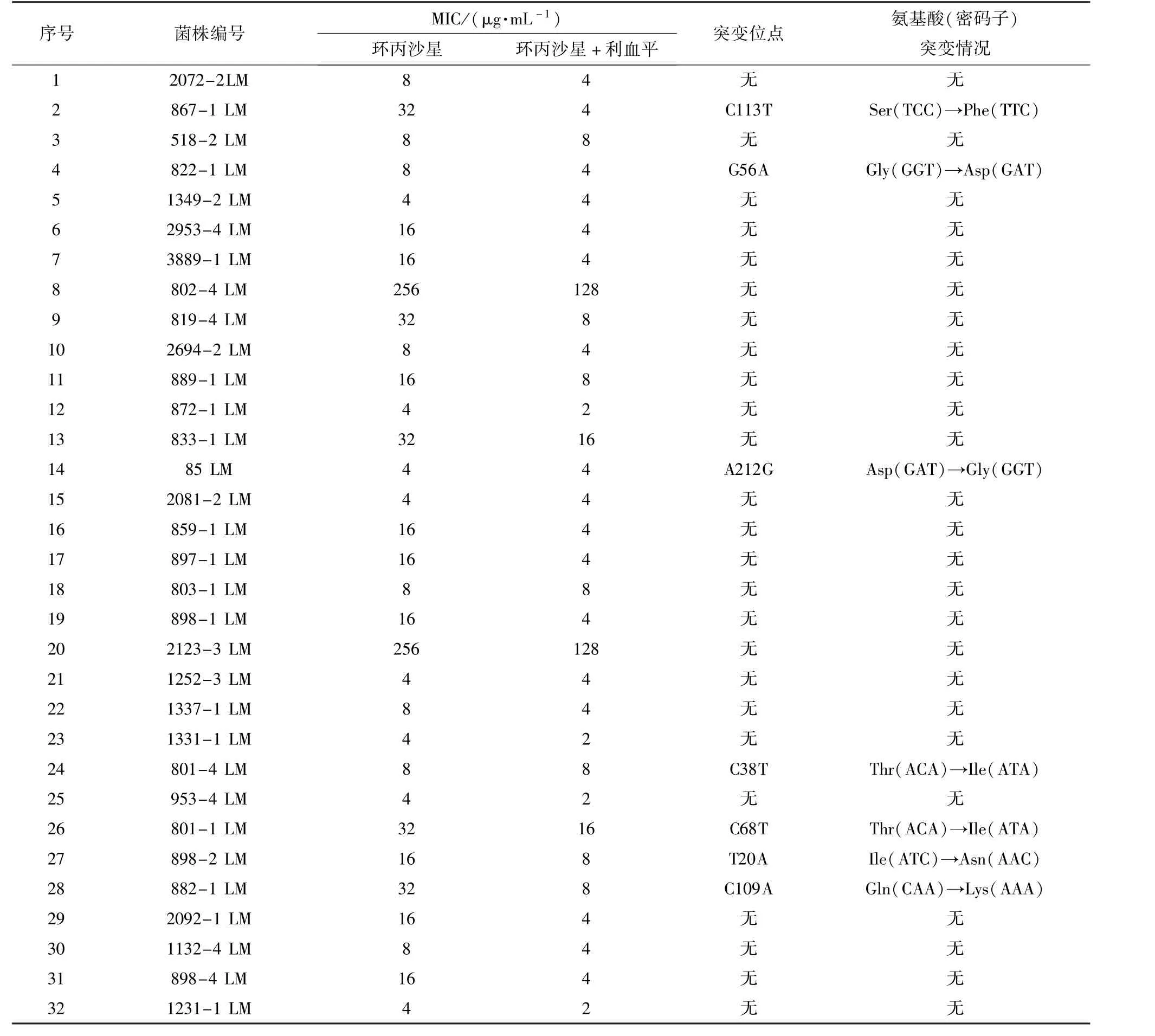
表4 加入利血平前后LM菌株对环丙沙星的MIC值以及fepR的突变情况Tab.4 MIC values of ciprofloxacin with or without addition of reserpine and mutation of fepR
3 讨 论
喹诺酮类药物的抗菌机制是通过在染色体DNA上形成DNA旋转酶和拓扑异构酶Ⅳ的三元复合物进行的[19],而细菌通过在靶基因DNA旋转酶和拓扑异构酶Ⅳ的关键氨基酸位点发生氨基酸置换,是一个产生喹诺酮类药物耐药的重要机制[20]。Friedman等[19]对喹诺酮类耐药的大肠杆菌进行研究,在GyrA的 QRDR 中发现 S83L、A51V、A67S、G81C、S83W、D87N、Q106H、D82A、A84P以及D87Y十个氨基酸突变位点;Zaki等[21]对喹诺酮类耐药的鲍曼不动杆菌进行研究,发现在gyrA的83号密码子以及parC的80号密码子发生了氨基酸突变。Zhang等[22]在喹诺酮类耐药的副猪嗜血杆菌中发现了gyrA(D87N)、parC(S73R)以及par(ET551A)几个喹诺酮相关氨基酸突变位点。本研究中,32株喹诺酮类耐药LM菌株的gyrA、gyrB、parC、parE四个基因的氨基酸序列与敏感株相比,并没有发生变化;而在fepR中,虽然有个别菌株发生了氨基酸序列变化,但并非文献报道的关键突变位点(G61T)[18],这些氨基酸突变位点是否导致喹诺酮类抗生素耐药仍需进一步研究。同时,本研究对5个PMQR进行了PCR检测,结果显示,这32株均未携带PMQR基因,说明喹诺酮耐药LM菌株均未携带含qnr系列基因的耐药质粒。QRDR和PMDR分析结果表明,外排泵有可能是介导LM菌株喹诺酮耐药的重要因素。
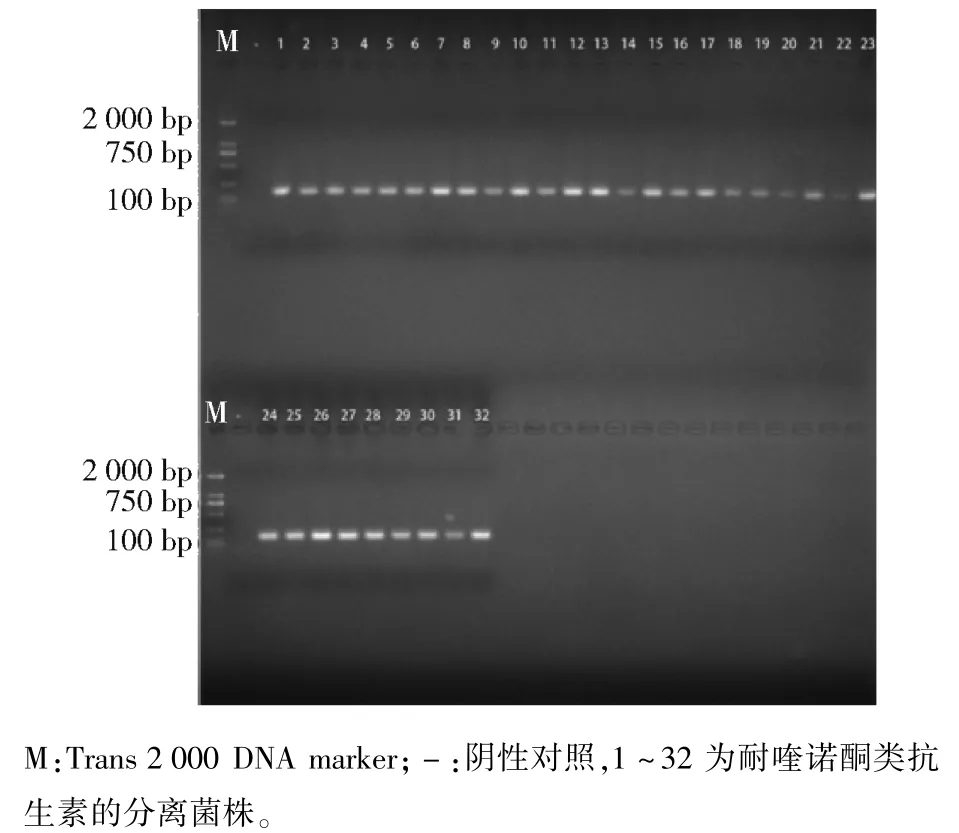
图2 喹诺酮类耐药LM分离菌株lde基因电泳结果Fig.2 Electrophoresis results of lde gene of quinoloneresistant Listeria monocytogenes isolates
细菌外排泵的功能主要将细胞内的抗生素排出细胞外,使得细菌细胞内保持低浓度的抗生素,从而导致细菌产生耐药性。Paltansing等[9]研究发现,外排泵基因的高水平表达可导致大肠杆菌产生喹诺酮类抗生素耐药。Shen等[23]对沙门氏菌的外排泵进行研究发现,外排泵AcrAB能介导沙门氏菌产生喹诺酮类抗生素耐药。Godreuil等[1]通过在LMCLIP 21369菌株基因组中插入失活lde基因,比较突变株与野生株的喹诺酮类抗生素耐药性发现,突变株的环丙沙星与诺氟沙星耐药性只有野生株的1/3~1/4,而且突变株的耐药性不会受到利血平的影响。可见外排泵是细菌产生耐药性的一种重要机制。利血平是一种吲哚型生物碱,能够抑制外排泵将喹诺酮类抗生素泵出细胞外。本研究对32株喹诺酮类抗生素耐药的LM进行lde基因的PCR扩增,发现这32株LM耐药株均检测出lde基因;但加入利血平后,10株分离株对环丙沙星的MIC值降低至原来的1/4以下,其中9株降低为原MIC的1/4,1株降低为原MIC的1/8,表明这10株菌外排泵是介导其对喹诺酮类抗生素耐药的关键因素。另外22株LM在加入利血平后对环丙沙星的MIC值下降不明显(降为原MIC的1/2)或是没发生变化,表明lde外排泵对这些菌株产生喹诺酮类抗生素耐药性影响不大。Jiang等[6]研究发现,外排泵基因lde在耐药株和敏感株中均有表达,且表达量相近;但在环丙沙星的作用下,lde基因在耐药株中的相对表达量显著增加,而在敏感株中的表达量基本没有变化,表明lde基因在菌株中的表达水平直接影响菌株对喹诺酮类抗生素的耐受性。
不同血清型的LM致病力具有差异,95%临床源分离株属于血清型1/2a,1/2b和4b。本研究32株喹诺酮类抗生素耐药菌株中,其中血清组Ⅰ.1(血清型1/2a和3a)15株,血清组Ⅰ.2(血清型1/2c和3c)8株,血清组Ⅱ.1(血清型4b,4d和4e)1株,血清组Ⅱ.2(血清型1/2b,3b和7)7株,血清组Ⅲ(血清型4a和4c)1株,表明血清组Ⅰ.1,Ⅱ.2和Ⅰ.2在喹诺酮类耐药菌株中占优势。同时,MLST分析发现 ST87、ST8、ST9和 ST155为优势 STs型。闫韶飞等[24]对2012年中国食源性LM耐药特征及多位点序列分型研究发现,ST155、ST9、ST705和ST87为我国LM耐药株常见型别。这些常见的耐药STs应该引起重视,其获得耐药的分子机制仍需进一步研究。Wang等[25]在四川省自贡市临床源分离株中发现,ST87、ST8和ST155是主要的致病ST型,说明32株耐喹诺酮类抗生素的LM分离株具有一定的致病能力,其对公众健康造成的潜在威胁值得重视。
4 结 论
本研究对从全国各地食品样品中分离的32株喹诺酮类抗生素耐药的LM进行研究,以期探讨LM喹诺酮类耐药的分子机制。结果表明,LM耐药株中的gyrA、gyrB、parC以及parE没有发生基因突变引起的氨基酸置换,在fepR中虽然发现有基因突变引起的氨基酸置换,但并非文献报道的突变位点,这些突变位点是否与喹诺酮类耐受性相关仍需进一步研究;同时这32株LM耐药株也未检出PMQR基因。通过外排泵抑制实验发现,外排泵lde可能是介导LM产生喹诺酮类抗生素耐药的重要因素,但lde基因表达水平是决定其耐药性强弱的关键因素。遗传多样性分析表明,32株喹诺酮耐药菌株具有潜在的致病性,应引起重视。
