米根霉α-淀粉酶热稳定性的理性设计
2018-07-30杨倩汤斌李松
杨倩,汤斌,李松
米根霉α-淀粉酶热稳定性的理性设计
杨倩,汤斌,李松
安徽工程大学 生物与化学工程学院,安徽 芜湖 241000

金城. 2018酶工程专刊序言. 生物工程学报, 2018, 34(7): 1021−1023.Jin C. Preface for special issue on enzyme engineering (2018). Chin J Biotech, 2018, 34(7): 1021−1023.
真菌α-淀粉酶被广泛应用于麦芽糖浆生产工业,但其热稳定性普遍较差,在制糖工艺中增加了由于酶活力损失而引起的追加生产成本。在充分研究了热稳定性对于真菌α-淀粉酶应用于工业生产的重要性的基础上,为提高米根霉α-淀粉酶 (ROAmy) 的热稳定性,基于酶蛋白B-factor分析和分子动力学模拟,利用重叠PCR技术分别对ROAmy中的3个氨基酸残基G128、K269和G393进行了单点突变及组合突变。结果表明,所获得的7个突变体均比原酶具有更好的热稳定性,其中效果最好的为组合突变体G128L/K269L/G393P,其在55 ℃下的热失活半衰期 (1/2) 约为原酶的5.63倍。同时,该突变体的最适温度由50 ℃提高到了65 ℃,最大反应速率 (max) 和催化效率 (cat/m) 分别提高了65.38%和99.86%。通过蛋白结构功能比较分析,发现氢键数目的增多或脯氨酸在特殊位置中的引入可能是突变体热稳定性得到提高的主要因素。
真菌α-淀粉酶,B-factor,定点突变,热稳定性,分子动力学模拟
真菌α-淀粉酶 (EC 3.2.1.1) 主要指一类真核微生物来源的α-淀粉酶,其不仅能够水解淀粉内部的α-1,4-糖苷键,也能水解麦芽三糖,终产物中的主要物质为麦芽糖、部分寡糖以及少量的葡萄糖[1-2]。由于真菌α-淀粉酶具有特殊的高麦芽糖生成能力,使其在很多工业上的应用逐年增加,例如高麦芽糖浆、烘焙和酿造工业等[3-4]。目前大部分商用真菌α-淀粉酶在麦芽糖浆生产中的最适作用温度在50–55 ℃之间[5],在该温度条件下,糖液在长时间的处理过程中容易被嗜温微生物污染而变质[6],而当反应温度超过60 ℃时酶的催化效率和活力单位会急剧下降。因此,提高真菌α-淀粉酶的耐热性将有助于改善制糖工艺以减少糖液感染嗜温细菌的几率并降低生产成本[7]。
由于蛋白质热稳定性的影响因素众多,目前还没有一种通用的蛋白质理性设计方法。随着生物信息学的快速发展,通常在对蛋白质结构进行分析的基础上利用B-factor、脯氨酸效应 (Proline theory)、折叠自由能或是同源比对等方法设计突变体以获得热稳定性提高的突变株。其中,B-factor (或B-value) 表示蛋白质晶体结构中原子的位移参数,可反映蛋白质分子在晶体中的构象,某一残基的B-factor越高,其相应部位的构象就越不稳定或柔性越强[8-9]。实验表明对蛋白质结构中B-factor较高的残基进行突变,部分突变体的温度稳定性得到了明显的提高[10-11]。同时,可以对B-factor较高的氨基酸进行饱和突变以获得更好的实验结果[12-13]。然而这种方法需要构建并筛选大量的突变体,将耗费大量的人力和时间。计算机辅助分子动力学模拟可在原子或分子水平上描述蛋白质结构中分子的运动过程,这种模拟技术已被广泛应用于蛋白质热稳定性的定向改造[14]。因此,在获取B-factor的基础上再根据计算机辅助分子动力学模拟结果进一步理性设计并快速选择合理的突变位点,是一种较为便捷、有效的蛋白质改造方法,例如:Huang等利用B-factor和折叠自由能计算方法,对一种转氨酶的柔性区域进行了突变,其中效果最好的突变体T130M/E133F在40 ℃下的热失活半衰期 (1/2) 是原酶的3.3倍[15];Li等利用多序列比对和PoPMuSiC算法辅助设计,构建了7个地衣芽孢杆菌α-淀粉酶突变体,其中最稳定的突变体在95 ℃下的1/2是原酶的9倍[16]。
为提高米根霉α-淀粉酶的热稳定性并揭示与耐热性相关的关键氨基酸结构,本研究基于B-factor和分子动力学模拟辅助设计,利用重叠PCR技术对3个潜在的关键氨基酸残基进行了单位点突变和组合突变,同时对各突变体的理化性质进行了分析。
1 材料与方法
1.1 材料
1.1.1 菌种和质粒
含有米根霉α-淀粉酶基因 () 的重组质粒pUC57-为本实验室在前期研究工作中构建。其中,基因(GenBank登录号HM234170) 克隆自菌株米根霉F0071。菌株大肠杆菌JM109和克鲁维乳酸酵母GG799以及质粒pUC57和pKLAC1为本实验室保藏。
1.1.2 工具酶、试剂盒及引物
实验中所使用的各种限制性核酸内切酶、DNA聚合酶、T4 DNA连接酶以及质粒抽提、PCR产物纯化和胶回收等试剂盒分别购自宝生物工程 (大连) 有限公司或生工生物工程 (上海) 股份有限公司。PCR引物由生工生物 (上海) 股份有限公司合成,具体信息如表1所示。
1.1.3 培养基
LB培养基 (g/L):蛋白胨10,酵母粉5,NaCl 10,其中固体LB培养基添加1.5%琼脂粉。YEPD培养基 (g/L):酵母粉10,胰蛋白胨20,葡萄糖20,自然pH。YEPG培养基 (g/L):酵母粉10,胰蛋白胨20,半乳糖20,自然pH。YCBA培养基 (g/L):酵母碳基10,乙酰胺0.3,琼脂粉15,自然pH。活性筛选培养基 (g/L):酵母粉10,胰蛋白胨20,葡萄糖20,可溶性淀粉2,自然pH。
1.1.4 试剂
酵母碳基 (YCB) 为英国OXOID公司产品,其他试剂均为国产或进口的生化或分析纯试剂。
1.2 方法
1.2.1 生物信息学分析
以米曲霉α-淀粉酶晶体结构 (PDB:2taa) 为模板,利用软件Swiss Model Server (https://swissmodel. expasy.org/) 建立米根霉α-淀粉酶 (ROAmy) 及其突变体的三维 (3-D) 结构模型。所构建模型的质量进一步利用Profile-3D、拉曼图和软件Molprobity (http://molprobity.biochem.duke.edu/) 进行评估。同时,通过B-FITTER软件计算ROAmy中所有氨基酸的B-factor[17]。利用生物学软件Discovery studio 2.5进行分子对接和分子动力学模拟。以ROAmy及其突变体为受体,以从NCBI (https://pubchem.ncbi.nlm.nih.gov/) 的PubChem Compound数据库中获得的麦芽三糖分子 (3D conformer,PubChem CID: 192826) 为配体进行分子对接。将对接好的蛋白质-配体复合物导入Discovery studio 2.5进行分子动力学模拟,赋予模拟体系CHARMm力场,然后模拟对复合物添加溶液环境,设置动力学模拟各项参数。在模拟结束后,对结果进行分析并根据分子动力学计算所得的轨迹计算蛋白质-配体复合物之间的相互作用能。
1.2.2 突变体的构建和筛选
在计算机辅助分析的基础上,利用定点突变技术构建了7个突变体。根据ROAmy成熟肽编码序列,设计了上游引物ROAmy-F和下游引物ROAmy-R,分别引入限制性酶切位点Ⅰ和Ⅰ。同时根据密码子使用,设计了引入突变位点的引物 (表1)。扩增得到的PCR产物纯化后使用Ⅰ和Ⅰ进行双酶切,酶切产物纯化后与经同样双酶切的pKLAC1相连接并转化JM109感受态细胞,将筛选得到的阳性转化子送至生工生物工程 (上海) 股份有限公司进行测序。验证正确后提取重组质粒并经Ⅱ线性化后通过电转化法导入GG799感受态细胞。重组酵母使用YCBA平板进行筛选并进行单菌落分离,将分离的单菌落分别点种至活性筛选培养基,在30 ℃静置培养48 h后使用稀碘液染色,筛选淀粉水解透明圈直径较大的转化子进行摇瓶发酵。
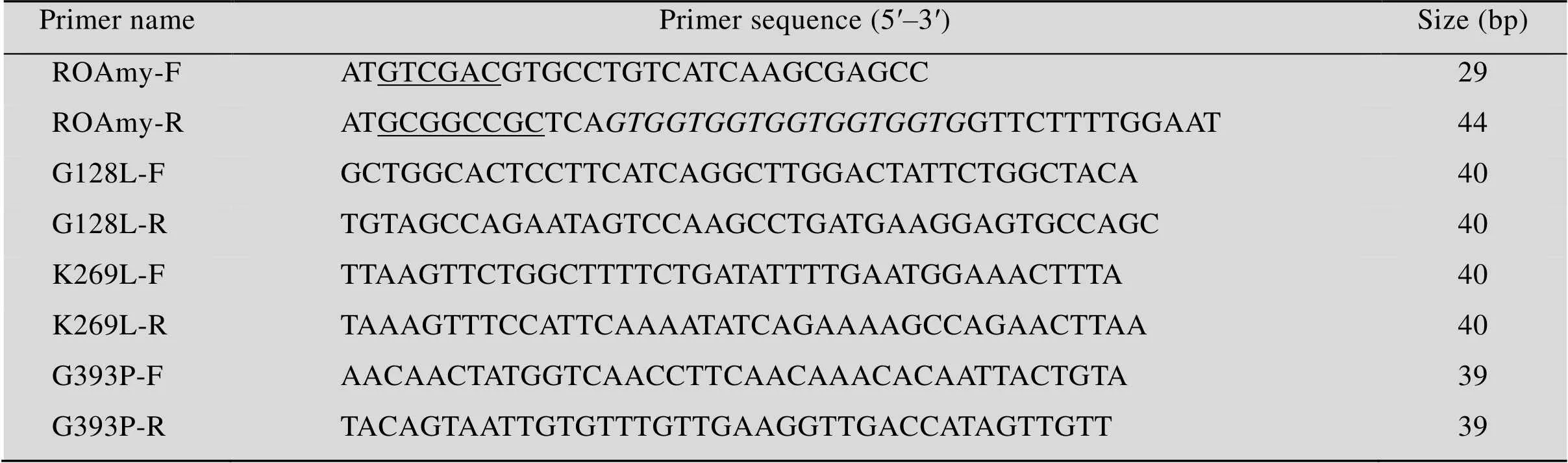
表1 本研究所用引物
Underlined letters are restriction enzyme cut sites and italic letters are His-tag coding sequence.
1.2.3 重组菌的发酵及酶液纯化
接种重组酵母单菌落于10 mL YEPD培养基中,在30 ℃、200 r/min条件下培养24 h后,取1 mL培养液接种于50 mL YEPG培养基中并在同等条件下继续培养36 h。发酵结束后通过离心 (8 000 r/min,5 min) 去除酵母细胞,上清液经硫酸铵盐析、金属亲和层析 (HisTrapTMHP 1 mL column,GE Healthcare)、脱盐 (HiTrapTM5 mL,GE Healthcare) 等步骤进行纯化,得到电泳纯的蛋白质之后将酶液进行真空冷冻干燥得到酶粉并贮存于4 ℃,使用时根据实验目的将酶粉溶解于不同pH值的柠檬酸-Na2HPO4缓冲液 (0.2 mol/L) 中。以牛血清蛋白为标准品,根据Bradford法测定蛋白质含量[18]。利用SDS-PAGE检测蛋白质的纯度。
1.2.4 重组酶酶学性质以及动力学参数测定
酶活测定按照文献[19]所述的淀粉-碘液法测定。具体为:取240 μL可溶性淀粉溶液 (2 g/L) 与240 μL酶液混合,于55 ℃下反应10 min后加入120 μL HCl溶液 (0.1 mol/L) 终止反应,再向体系中加入600 μL稀碘液 (5 mmol/L I2和5 mmol/L KI),混合均匀后在580 nm处测定吸光值以对反应液中残余淀粉进行定量。一个α-淀粉酶活力单位 (U) 定义为:在上述反应条件下,每分钟消耗1 mg可溶性淀粉所需要的酶量。反应中所使用的酶液指将一定量的酶粉溶解于柠檬酸-Na2HPO4缓冲液 (0.2 mol/L,pH 5.0) 而获得的淀粉酶溶液。
最适反应温度测定:在pH 5.0条件下,分别在35 ℃、40 ℃、45 ℃、50 ℃、55 ℃、60 ℃、65 ℃、70 ℃条件下测定酶活力,以最高酶活力为100%计算相对酶活力,以确定酶的最适反应温度。
温度稳定性测定:将酶液使用柠檬酸-Na2HPO4缓冲液 (0.2 mol/L,pH 5.0) 稀释至5 U/mL并于55 ℃条件下保温不同时间后取出,在冰上放置30 min后测定残余酶活力,并以未保温的样品所测定的酶活力为100%计算相对酶活力,以比较酶的热稳定性。热失活半衰期 (1/2) 利用公式1/2= ln2/d计算,其中d为一级动力学常数[20]。
最适反应pH测定:在55 ℃条件下,分别在pH值为3.0、3.5、4.0、4.5、5.0、5.5、6.0、6.5、7.0、7.5的缓冲体系中测定酶活力,以最高酶活力为100%计算相对酶活力,确定酶的最适反应pH。
pH稳定性测定:将酶粉溶解于不同pH值的柠檬酸-Na2HPO4缓冲液 (0.1 mol/L,pH 3.0–7.5) 中并于55 ℃下保温10 min后取出,在冰上放置30 min并使用柠檬酸-Na2HPO4缓冲液 (0.2 mol/L,pH 5.0),稀释10倍后测定剩余酶活力,以未保温的样品测定的酶活力为100%计算相对酶活力以确定酶的pH稳定性。
米氏常数的测定:使用不同浓度的可溶性淀粉溶液 (0.2–6.0 mg/mL) 为底物,在标准酶活力测定条件下测定α-淀粉酶活力,并依据Lineweaver- Burk双倒数作图法计算酶对可溶性淀粉的米氏常数m和max。
1.2.5 数据分析
实验中所有数据的测定均做3组平行,数据以平均值或平均值±标准差 (如有需要) 形式呈现。标准差计算以及显著性分析 (-test) 均利用软件OriginPro 2015 (OriginLab Corporation) 完成。
2 结果与分析
2.1 ROAmy的结构建模分析与突变体的设计
ROAmy的氨基酸序列与米曲霉α-淀粉酶 (PDB:2taa) 的氨基酸序列相似度高达45%,以其为模板,利用Swiss Model Server对ROAmy建立3D结构模型经过评估后用于后续的分析。为合理、快速地选择待突变位点,研究中首先利用软件B-FITTER计算ROAmy中所有氨基酸的B-factor,其中B-factor值较高的前10个氨基酸残基如表2所示。
研究中选择了B-factor值较高的前3个氨基酸残基G128、K269和G393进行分析并得到这3个残基在蛋白质结构中的位置 (图1),结构模拟表明这3个残基分别位于ROAmy中Domain B的无规则卷曲处、Domain A的 (α/β)8TIM桶状结构的第6个α-螺旋及Domain C处。
进一步利用Discovery studio 2.5软件将以上3个残基分别替换为其他19种氨基酸并进行分子动力学模拟,结果显示G128L、K269L和G393P的蛋白质-配体复合物之间的相互作用能最低 (表3)。为研究各残基突变的单独作用和累积效应,利用重叠PCR技术分别获得了3个单突变体(G128L、K269L和G393P) 以及4个组合突变体 (G128L/K269L、K269L/G393P、G128L/G393P和G128L/K269L/G393P)。
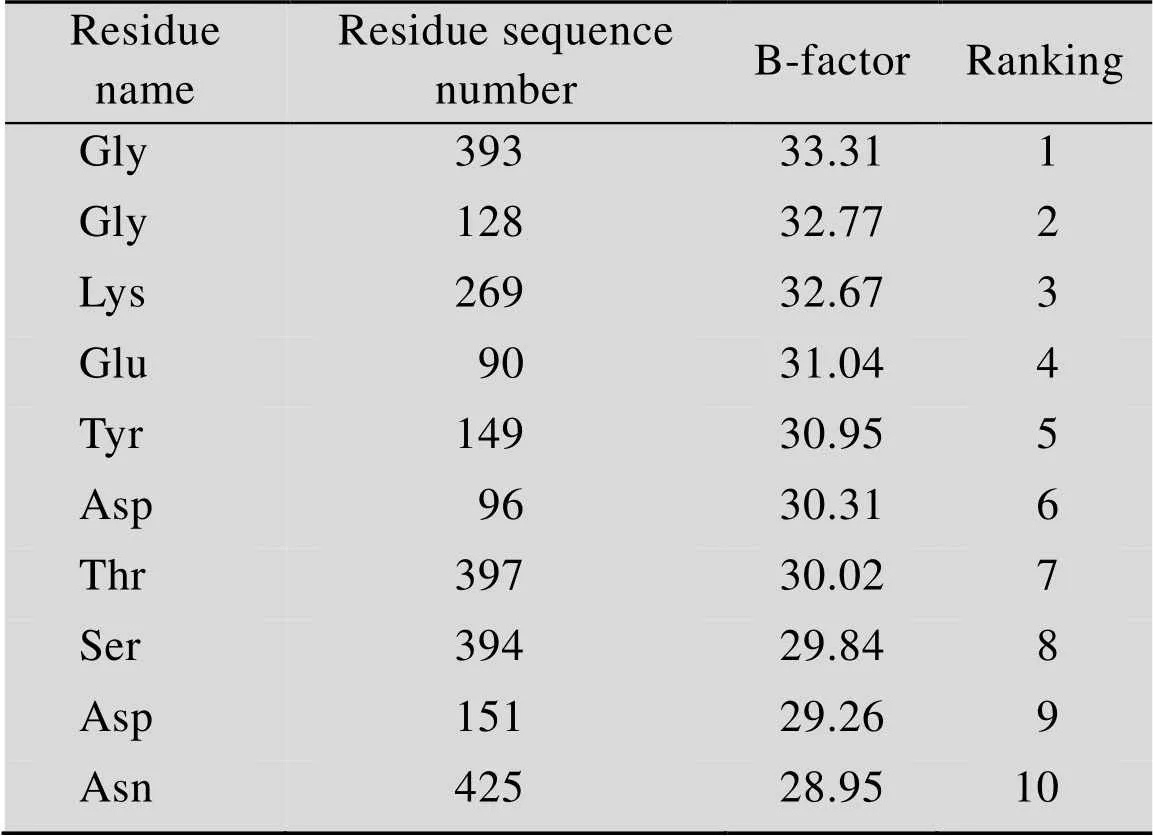
表2 ROAmy中B-factor值较高的前10个氨基酸残基

图1 ROAmy的3D结构模型
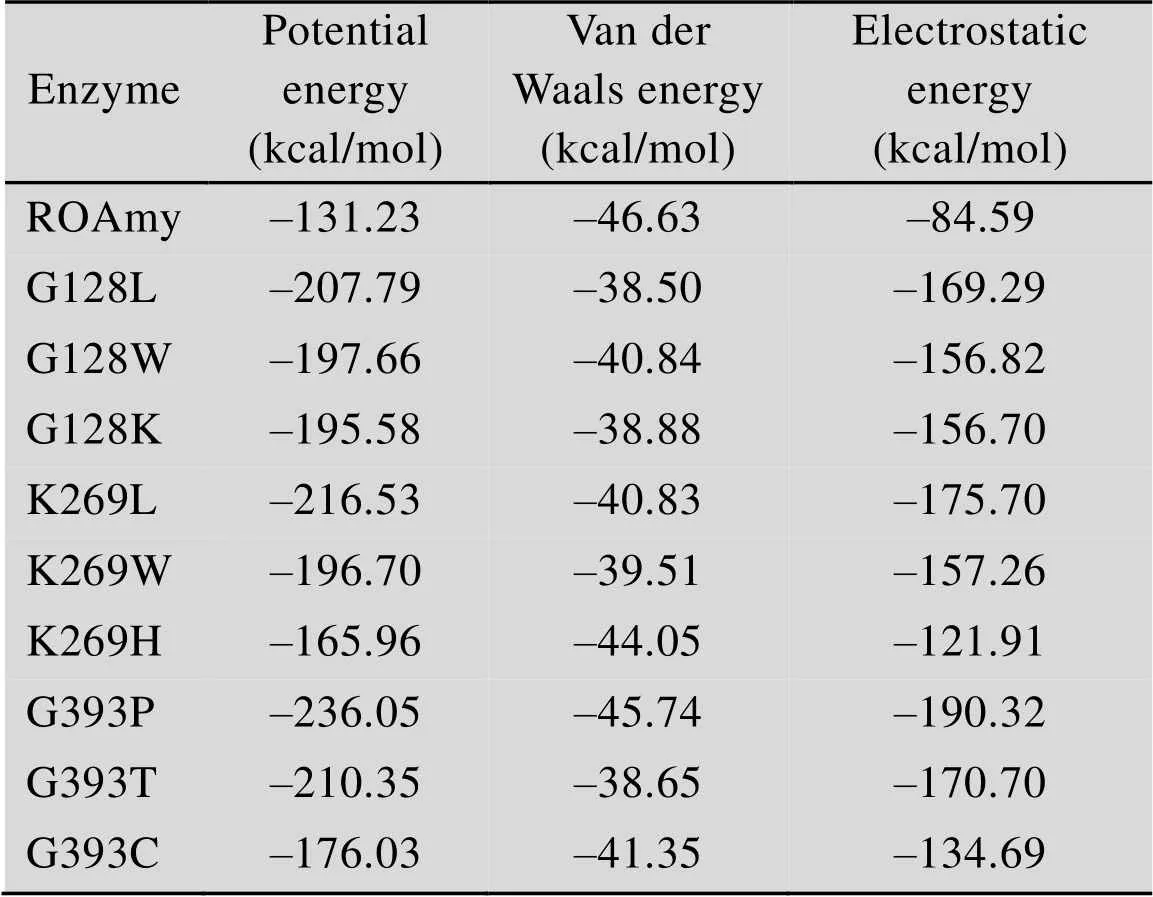
表3 ROAmy及其突变体蛋白-配体相互作用能
2.2 温度对酶活力及酶蛋白稳定性的影响
温度对酶活力影响研究结果显示,与原酶ROAmy的最适反应温度 (50 ℃) 相比,所有突变体的最适反应温度均产生了偏移。其中,突变体G393P的最适反应温度为65 ℃,其他6个突变体的最适反应温度为60 ℃ (图2A)。热稳定性研究表明,所有突变体的热失活半衰期均得到了不同程度的提升 (图2B),突变体G128L、K269L、G393P、G128L/K269L、K269L/G393P、G128L/ G393P和G128L/K269L/G393P的热失活半衰期分别约为原酶的1.20、2.56、5.20、2.79、3.07、3.35和5.63倍,表明突变体的热稳定性得到了显著提升,尤其是组合突变体G128L/K269L/G393P。
2.3 pH对酶活力及酶蛋白稳定性的影响
pH作用条件研究表明7个突变体的最适反应pH均与原酶相似,即在pH 5.5时具有最高催化活力 (图3A)。其中,在pH 4.0和pH 4.5条件下部分突变体的催化活力得到了提高,如组合突变体原酶的1.44、1.31、1.23、1.28倍。此外,与原酶相比,各突变体在更高的pH条件下亦表现出了较高的催化活力,突变体G128L、K269L和G128L/K269L/G393P在pH 7.0时的催化效率分别约为G128L/K269L、K269L/G393P、G128L/G393P和G128L/K269L/G393P在pH 4.5时的催化效率分别约为原酶的2.32、2.06、1.76倍 (图3A)。然而pH稳定性测定结果显示7个突变体在pH值低于5.0条件下的稳定性较原酶略有下降,仅在pH值高于5.5时表现出了较好的稳定性 (图3B)。
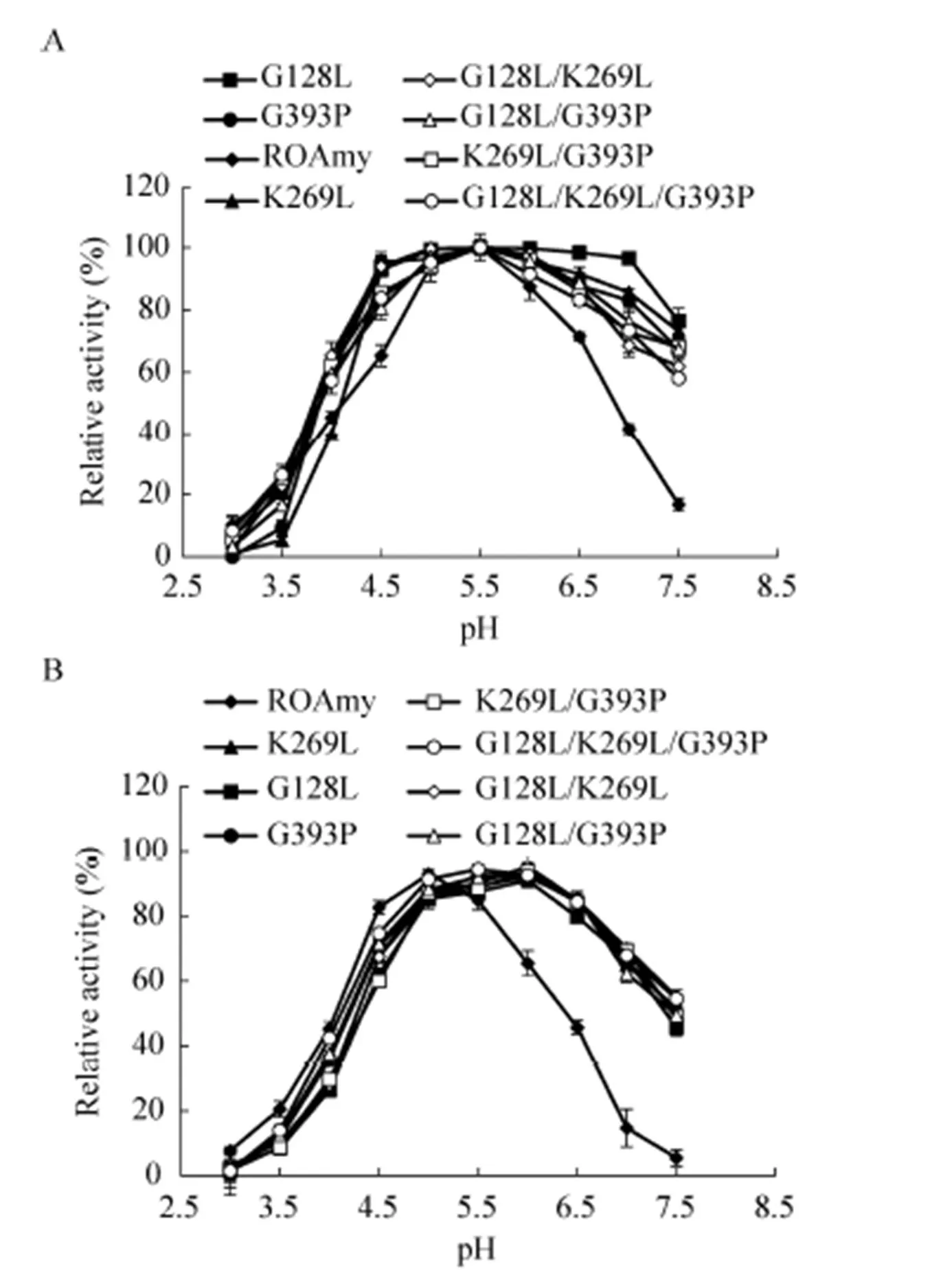
图3 pH对ROAmy及其突变体酶活力(A) 和稳定性(B) 的影响
Fig. 3 Effect of pH on the activity and stability of ROAmy and its mutants. (A) The optimum pH of ROAmy and mutants. (B) The pH stability of ROAmy and mutants.
通过与原酶相比较可以发现,各突变体在相对偏碱性的范围内 (pH 6.0–7.5) 表现出了更高的催化活力和稳定性。
2.4 动力学参数的测定
在热稳定性得到提高的同时,酶促动力学参数研究表明各突变体在底物结合能力和催化效率方面也分别得到了不同程度的提高 (表4),其中,突变体G128L、K269L和G128L/G393P对底物 (可溶性淀粉) 的亲和力得到了明显的提高,m值相比原酶分别降低了32.64%、29.17%和27.78%;同时,突变体G393P、K269L/G393P、G128L/G393P和G128L/K269L/G393P的max分别约是原酶的1.42、1.19、1.38、1.65倍;此外,突变体G128L、G393P、G128L/G393P和G128L/ K269L/G393P的cat/m分别约是原酶的1.72、1.83、2.40、2.00倍。
2.5 热稳定性的分子机制解析
为研究突变体热稳定性提高的分子机制,利用结构建模对以上突变体结构功能进行初步分析。通过对原始酶和突变体G128L蛋白质结构模型比较分析,可以看出当原始蛋白中位于蛋白质表面且柔性极强的G128 (图4A) 突变为疏水性较好的L128 (图4B) 之后,G122和T123分别与Y133形成了氢键 (图4B),氢键的总数目由原始的350个增加到352个。虽然蛋白质整体结构在突变前后没有发生显著变化,但是蛋白质的局部微观结构可能由于Leu对Gly的替换而发生了改变,从而可以促使在其他氨基酸之间形成更多的氢键并增强了该无规则卷曲区域结构的稳定性。因此,突变后蛋白质微观结构的调整和其他氨基酸之间新的氢键的形成可能是突变体G128L比原酶具有更好的热稳定性的主要原因。
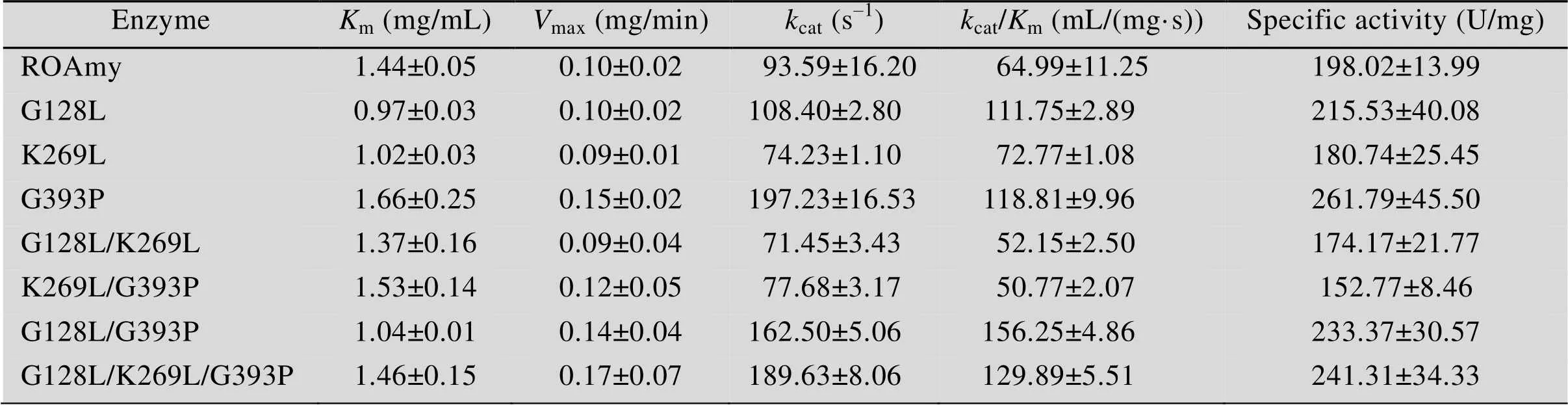
表4 ROAmy和突变体的比酶活以及动力学参数
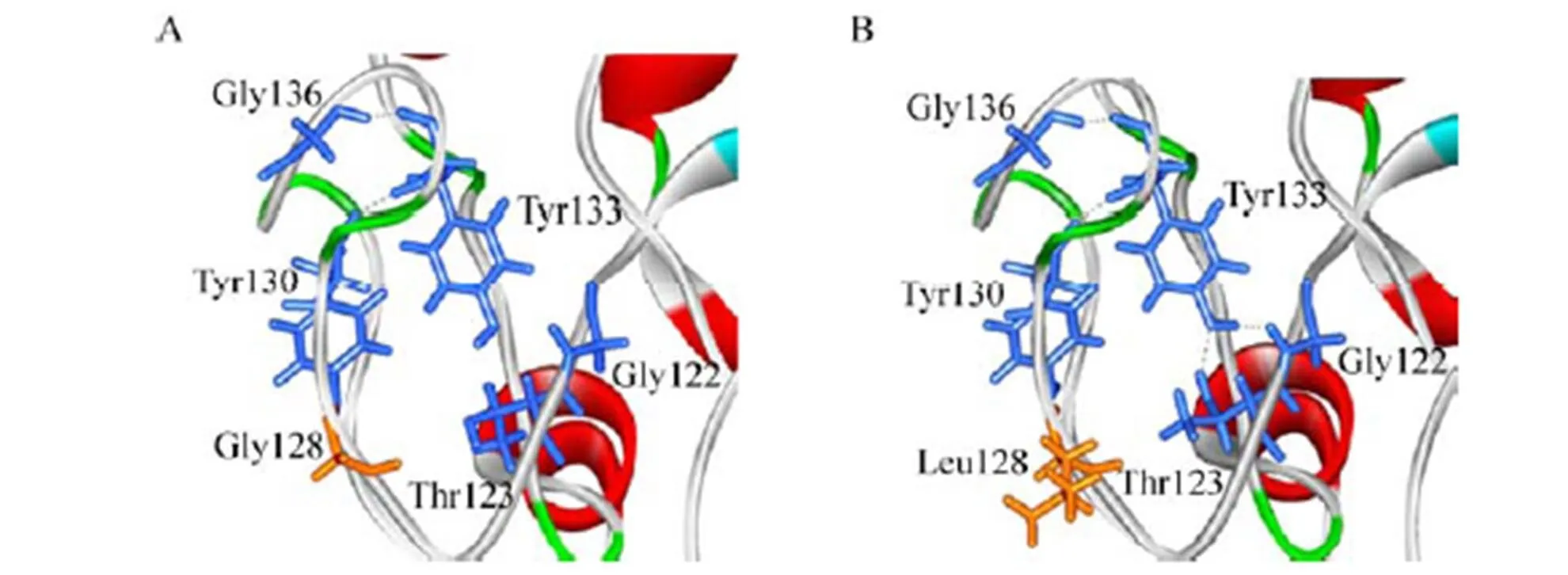
图4 ROAmy (A) 及其突变体G128L (B) 的局部3D结构模型
原酶蛋白质结构建模显示K269可分别与F265和N270形成一个氢键 (图5A),当K269突变为L269之后,L269与N270之间则没有氢键作用,同时在I276和L279之间形成新的氢键 (图5B)。此外,原酶蛋白质中位于无规则卷曲处的Y133仅与邻近的G136和Y130形成氢键 (图5C),而在突变体K269L中Y133可以同时与邻近的G136、Y130以及G122形成氢键 (图5D),因此使得氢键的总数目由原蛋白质中的350个增加到突变体K269L中的351个。这种由一个氨基酸残基突变而引起其他远距离氨基酸残基之间相互作用发生改变的现象在嗜碱单胞菌碱性α-淀粉酶的突变过程中也发现过[22]。有趣的是,G128L和K269L两个突变体中都在Y133和G122之间形成了新的氢键作用。由图5B可知L269位于该α-螺旋结构的外侧,而Leu属于疏水性氨基酸且含有Leu的侧链更倾向于包埋在蛋白质的内部从而使该局部结构更为紧密和稳定[23]。由此推测,疏水性氨基酸 (Leu) 对局部蛋白质结构的稳定作用以及由于微观结构改变而形成的更多的氢键作用可能是突变体K269L具有较高热稳定性的主要原因。
此外,在ROAmy中,G393位于该酶Domain C的一段柔性较强的β-折叠结构转角区域内 (如图1和图6A所示),而Gly作为柔性最强的氨基酸出现在柔性又相对较强的β-折叠结构中可能使得蛋白质处于一种易于变化的不稳定状态。同时,B-factor值的计算结果亦显示G393是ROAmy中最不稳定的一个氨基酸。当将ROAmy中393位的Gly突变为Pro之后,原酶中Q392与N374之间的氢键 (图6A) 消失,而在突变体G393P中重新于Q392和Y390之间形成了氢键。与原酶相比,虽然突变体G393P的氢键数目没有发生变化,但该突变体的热失活半衰期却是原酶的5.20倍,是3个单突变体中最稳定的一个。通过氨基酸结构分析可知Pro由于自身的吡咯烷环结构而具有较小的空间构象自由度,因此Pro属于一种刚性氨基酸并可能增强其所在区域结构的刚性。在氢键数目没有变化的情况下,这种在柔性结构中利用刚性氨基酸 (Pro) 替代柔性氨基酸 (Gly) 而提高蛋白质局部区域刚性的现象,可能是突变体G393P热稳定性得到显著增强的主要原因之一。
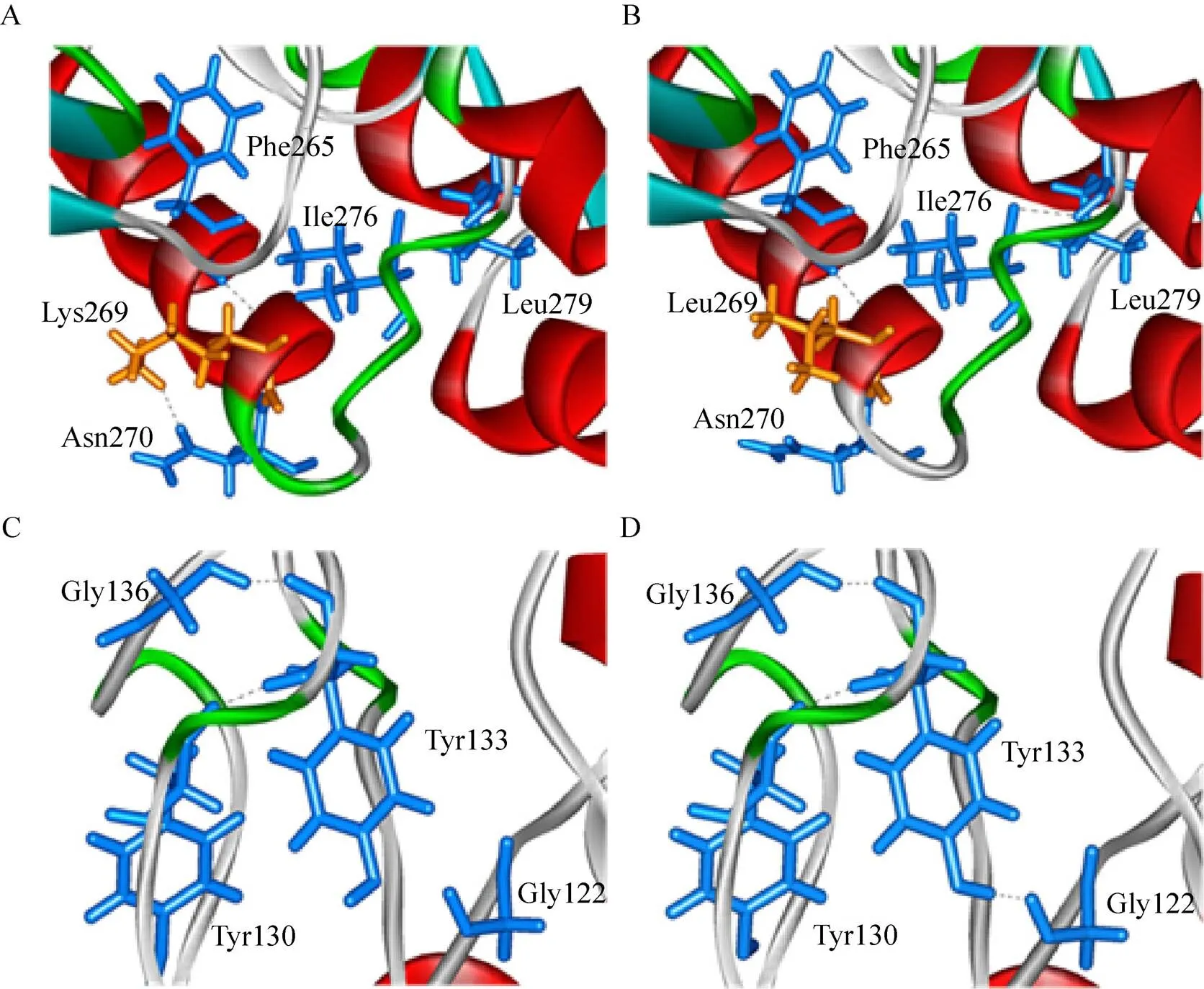
图5 ROAmy (A, C) 及其突变体K269L (B, D)的局部3D结构模型
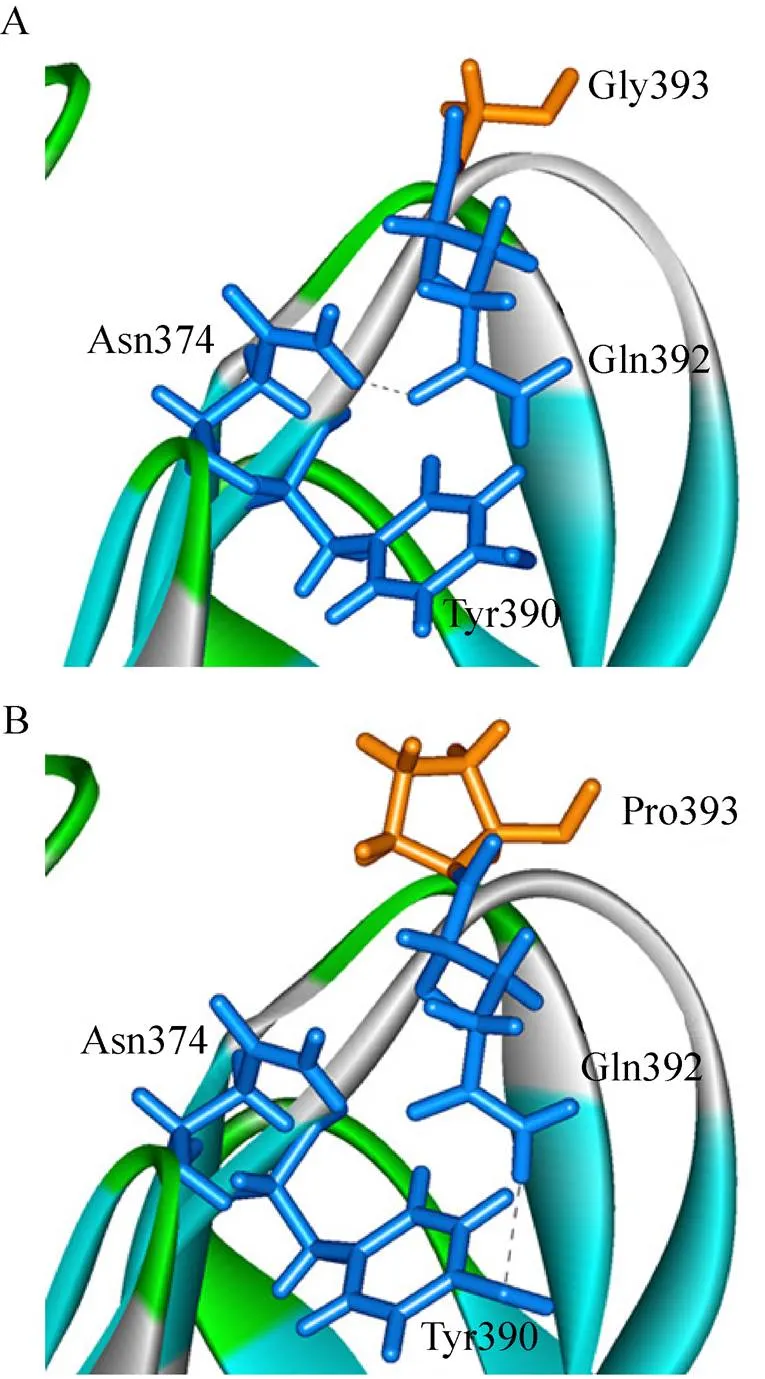
图6 ROAmy (A) 及其突变体G393P (B) 的局部3D结构模型
3 讨论
有关α-淀粉酶蛋白质结构与酶的热稳定性、pH耐受性或盐浓度耐受性等理化性质之间的关系的研究绝大部分都集中在细菌α-淀粉酶领域[24-26],而对真菌α-淀粉酶的相关研究则鲜有报道。目前已知米曲霉α-淀粉酶 (Taka-amylase) 中的Asp206、Glu230和Asp297是该酶的核心催化位点[21],同时上述3个氨基酸残基也是α-淀粉酶家族共有的主要催化位点[27],但是真菌α-淀粉酶中其他绝大部分氨基酸残基的功能均是未知的。基于此,在没有任何已知、可参考的数据前提下,本研究为了快速、有效地提升ROAmy的热稳定性,在设计中结合了B-factor值运算和分子动力学模拟两种优化设计方法并分别对ROAmy中B-factor值较高的前3个氨基酸残基进行了突变。结果显示,3个单突变体和4个组合突变体的热稳性均得到了不同程度的提高,其中单突变体G393P和组合突变体G128L/K269L/G393P的1/2分别是ROAmy的5.20倍和5.63倍。然而,组合突变体K269L/ G393P和G128L/G393P的1/2则小于单突变体G393P的1/2,并没有表现出一些研究在组合突变体中发现的累积效应或协同效果[22,28],可能是由于组合突变体并不是严格意义上单突变体效应的叠加且突变体与突变体之间也存在一定的拮抗效应[29]。
影响蛋白质热稳定性的因素主要有氢键、盐键、疏水相互作用和二硫键等,且不同类型的蛋白质具有不同的与其热稳定性相关的影响因素[30],如在葡聚糖内切酶CelA的β-转角和无规则卷曲处等构象不稳定区域引入脯氨酸后可以显著增强蛋白质的热稳定性[31],在α-淀粉酶催化区域中引入氢键和盐键可提高酶蛋白质在低pH下的稳定性和催化效率[32]。通过比较ROAmy及其单突变体蛋白质结构之间的差异,发现氢键和脯氨酸的引入可能是突变体热稳定性得到提高的主要因素。本文中所采用的设计方法和所获得的实验结果对其他真菌α-淀粉酶或工业用酶的热稳定性定向进化研究可能具有一定的参考价值。
[1] Hua HF, Luo HY, Bai YG, et al. A thermostable glucoamylase fromsp. MEY-1 with stability over a broad pH range and significant starch hydrolysis capacity. PLoS ONE, 2014, 9(11): e0113581.
[2] Li S, Zuo ZR, Niu DD, et al. Gene cloning, heterologous expression, and characterization of a high maltose-producing α-amylase of. Appl Biochem Biotechnol, 2011, 164(5): 581–592.
[3] Zeng J, Guo JJ, Yuan L, et al. Optimization of the thermal activity and stability of hyperthermophilic α-amylase ApkA. Biotechnol Bull, 2017, 33(8): 192–198 (in Chinese). 曾静, 郭建军, 袁林, 等. 极端嗜热α-淀粉酶ApkA的高温活性和热稳定性的优化研究. 生物技术通报, 2017, 33(8): 192–198.
[4] EI-Okki AAKEH, Gagaoua M, Bennamoun L, et al. Statistical optimization of thermostable α-amylase production by a newly isolatedstrain FSIS4using decommissioned dates. Waste Biomass Valori, 2017, 8(6): 2017–2027.
[5] Qian Y, Duan G. New acid-stable fungal α-amylase for maltose syrup production. Food Ferment Indust, 2008, 34(2): 87–89 (in Chinese). 钱莹, 段刚. 新型耐酸真菌淀粉酶在麦芽糖生产上的应用. 食品与发酵工业, 2008, 34(2): 87–89.
[6] Shen W, Lin LZ, Huang WW, et al. Heterologous expression of an acid-resistant fungal α-amylase and characterization of its recombinant enzyme. Food Ferment Indust, 2013, 39(8): 1–6 (in Chinese). 沈微, 林丽珍, 黄雯雯, 等. 一种酸性真菌α-淀粉酶的异源表达与重组酶性质. 食品与发酵工业, 2013, 39(8): 1–6.
[7] Li S, Wang ZX. Progress in research of fungal α-amylase. Biotechnol Bull, 2011, 224(3): 66–71 (in Chinese). 李松, 王正祥. 真菌α-淀粉酶的研究进展. 生物技术通报, 2011, 224(3): 66–71.
[8] Reetz MT, Carballeira JD, Vogel A. Iterative saturation mutagenesis on the basis of B factors as a strategy for increasing protein thermostability. Angew Chem Int Ed, 2006, 45(46): 7745–7751.
[9] Pang YP. Use of multiple picosecond high-mass molecular dynamics simulations to predict crystallographic B-factors of folded globular proteins. Heliyon, 2016, 2(9): e00161.
[10] Jochens H, Aerts D, Bornscheuer UT. Thermostabilization of an esterase by alignment-guided focussed directed evolution. Protein Eng Des Select, 2010, 23(12): 903–909.
[11] Duan XG, Chen S, Ai YX, et al. Enhancing the thermostability ofsucrose isomerase using B-factor-directed mutagenesis. PLoS ONE, 2016, 11(2): e0149208.
[12] Zhang JH, Lin Y, Sun YF, et al. High-throughput screening of B factor saturation mutatedLipase thermostability based on synthetic reaction. Enzyme Microb Technol, 2012, 50(6/7): 325–330.
[13] Liu XM, Zhang PP, Hu S, et al. Semi-rational molecular engineering to improve thermostability of cytochrome P450 BM-3. J Chem Eng Chin Univ, 2015, 29(5): 1138–1144 (in Chinese). 刘晓萌, 张彭湃, 胡升, 等. 细胞色素P450 BM-3热稳定性的半理性改造. 高校化学工程学报, 2015, 29(5): 1138–1144.
[14] Chen ZG, Fu Y, Xu WB. Insight into thermal stability of CGTase active site region by molecular dynamics simulation. Comput Appl Chem, 2013, 30(9): 967–972 (in Chinese). 陈志国, 傅毅, 须文波. 分子动力学模拟研究CGTase酶活性区域的热稳定性. 计算机与应用化学, 2013, 30(9): 967–972.
[15] Huang J, Xie DF, Feng Y. Engineering thermostable (R)-selective amine transaminase fromthrough in silico design employing B-factor and folding free energy calculations. Biochem Biophys Res Commun, 2017, 483(1): 397–402.
[16] Li Z, Duan XG, Chen S, et al. Improving the reversibility of thermal denaturation and catalytic efficiency ofα-amylase through stabilizing a long loop in domain B. PLoS ONE, 2017, 12(3): e0173187.
[17] Reetz MT, Carballeira JD. Iterative saturation mutagenesis(ISM) for rapid directed evolution of function enzymes. Nat Protoc, 2007, 2(4): 891–903.
[18] Bradford MM. A rapid and sensitive method for the quantitation of microgram quantitites of protein utilizing the principle of protein-dye binding. Anal Biochem, 1976, 72(1/2): 248–254.
[19] Xiao ZZ, Reginald S, Adrian T. A quantitative starch-iodine method for measuring alpha-amylase and glucoamylase activities. Anal Biochem, 2006, 351(1): 146–148.
[20] Ó’Fágáin C. Enzyme stabilization-recent experimental progress. Enzyme Microb Technol, 2003, 33(2/3): 137–149.
[21] Matsuura Y, Kusunoki M, Harada W, et al. Structure and possible catalytic residues of Taka-amylase A. J Biotechnol, 1984, 95(3): 697–702.
[22] Deng ZM, Yang HQ, Li JH, et al. Structure-based engineering of alkaline α-amylase from alkaliphilicfor improved thermostability. Appl Microbiol Biotechnol, 2014, 98(9): 3997–4007.
[23] Yang J, Li LZ, Xiao YZ, et al. Identification and thermoadaptation engineering of thermostability conferring residue of deep sea bacterial α-amylase AMY121. J Mol Catal B-Enzym, 2016, 126: 56–63.
[24] Singh S, Guruprasad L. Structure and sequence based analysis of alpha-amylase evolution. Protein Peptide Lett, 2014, 21(9): 948–956.
[25] Deng ZM, Yang HQ, Shin HD, et al. Structure-based rational design and introduction of arginines on the surface of an alkaline α-amylase fromfor improved thermostability. Appl Microbiol Biotechnol, 2014, 98(21): 8937–8945.
[26] Saxena RK, Dutt K, Agarwal L, et al. A highly thermostable and alkaline amylase from asp. PN5. Bioresource Technol, 2007, 98(2): 260–265.
[27] Sharma A, Satyanarayana T. Structural and biochemical features of acidic α-amylase of. Int J Biol Macromol, 2013, 61: 416–423.
[28] Li Z, Duan XG, Wu J. Improving the thermostability and enhancing the Ca2+binding of the maltohexaose-forming α-amylase from. J Biotechnol, 2016, 222: 65–72.
[29] Wen S, Tan TW, Zhao HM. Improving the thermostability of lipase Lip2 from. J Biotechnol, 2012, 164(2): 248–253.
[30] Yi Y, Wei YD, Deng C, et al. Research progress of amino acid residues in the protein thermal stability mechanism. J Guangxi Univ Sci Technol, 2015, 26(4): 1–5 (in Chinese). 易弋, 韦阳道, 邓春, 等. 氨基酸残基在蛋白质热稳定性机制中的研究进展. 广西科技大学学报, 2015, 26(4): 1–5.
[31] Yi ZL, Pei XQ, Wu ZL. Introduction of glycine and proline residues onto protein surface increases the thermostability of endoglucanase CelA from. Bioresource Technol, 2011, 102(3): 3636–3638.
[32] Yang HQ, Liu L, Shin HD, et al. Structure-based engineering of histidine residues in the catalytic domain of α-amylase fromfor improved protein stability and catalytic efficiency under acidic conditions. J Biotechnol, 2013, 164(1): 59–66.
(本文责编 郝丽芳)
Improving the thermostability of α-amylase fromby rational design
Qian Yang, Bin Tang, and Song Li
School of Biological and Chemical Engineering, Anhui Polytechnic University, Wuhu 241000, Anhui, China
Fungal α-amylases are widely used in the production of maltose syrup, while additional production costs may be required in the syrup production process due to the loss of enzyme activity, because of the poor thermostability exhibited in this type of enzyme. After deeply studying the importance of thermostability of fungal α-amylases applied in industrial production, with attempt to improve the thermostability ofα-amylase (ROAmy), single-point mutations and combined mutations that based on analysis of B-factor values and molecular dynamics simulations were carried out for amino acid residues G128, K269 and G393 of ROAmy by overlapping PCR. The results showed that all the 7 mutants obtained presented better thermostability than the wild-type enzyme, and the best mutant was G128L/K269L/G393P which showed a 5.63-fold increase in half-life at 55 ℃ compared with the wild-type enzyme. Meanwhile, its optimum temperature increased from 50 ℃ to 65 ℃, the maximum reaction rate (max) and catalytic efficiency (cat/m) increased by 65.38% and 99.86%. By comparing and analyzing the protein structure and function between the mutants and the wild-type enzyme, it was found that the increase of the number of hydrogen bonds or the introduction of proline in special position may be the main reasons for the improved thermostability that found in the mutants.
fungal α-amylase, B-factor, site-directed mutagenesis, thermostability, molecular dynamics simulation
December 12, 2017;
January 25, 2018
National Natural Science Foundation of China (No. 31401630), Natural Science Foundation for the Youth of Anhui Province (No. 1708085QC63).
10.13345/j.cjb.170492
BinTang . Tel: +86-553-2871254; E-mail: tangbin@ahpu.edu.cn
国家自然科学基金青年项目 (No. 31401630),安徽省自然科学基金青年项目 (No. 1708085QC63) 资助。
