基于重测序的晋汾52及其突变体抗性差异分析
2018-07-10韩彦卿郑洁武彩娟王慧娜韩渊怀
韩彦卿,郑洁,武彩娟,王慧娜,韩渊怀*
(1.山西农业大学 农学院,山西 太谷 030801;2.山西农业大学 农业生物工程研究所,山西 太谷 030801)
谷子(Setariaitalica(L.)Beauv)是我国非常重要的特色杂粮,具有营养丰富、抗旱耐瘠薄等特点,是“节水减肥”战略的理想作物。谷子因基因组较小(490 M)、生育期短、与其它主粮作物亲缘关系密切等特点,已逐渐成为禾本科基因组学研究的理想模式植物[1,2]。名优谷子品种晋谷21号是由晋汾52经钴60咖玛射线辐射诱变而来,经连续单株选择选育而成[3]。随着谷子产业的在我国的不断发展和日益壮大,人们更注重于谷子高产和品质的提升。然而,近年来在谷子上发生的病害越来越多,发病程度日趋严重。谷子白发病、谷子锈病、谷瘟病和红穗病等每年均有不同程度发生,大发生年份可造成减产60%以上。谷子病害的严重发生已经成为制约谷子产量和品质提升的重要限制因素。诱变的晋谷21与亲本晋汾52的相比,遗传背景高度相似,但是最大的缺点就是极易感病[4]。无数育种实践证明,明晰病原物与寄主之间的互作分子机理,挖掘抗病候选基因是实现抗病分子育种最有效的途径[5]。
随着测序技术的快速发展,全基因组重测序已成为物种进化分析、SNP鉴定以及突变基因鉴定的重要手段,并且应用于多种农作物和杂粮上[6~8]。施阳[9]利用高抗和高感黄瓜白粉病的2个品种通过全基因组测序分析,发现在基因组中主要存在单核苷酸多态性位点(SNP)和插入缺失位点(InDel)的差异,进一步分析发现5个NBS-LRR类的抗病基因, 其中2个基因中的非同义突变是导致黄瓜高抗白粉病的重要原因。Zhu等[10]采用基因组重测序技术对豫谷1号和张谷中的NBS基因分析,第8条染色体包含的NBS基因数量最多最大的基因簇也位于第8条染色体上,发现在豫谷1号和张谷中CC-NBS-LRR类型基因较多,TIR-NBS类型基因在2个品种中的数量最少。以上研究对我们借助全基因组测序技术研究谷子品种间抗感差异提供很好的借鉴。
本研究对晋谷21和晋汾52两个品种进行基因组重测序数据的生物信息学分析,主要针对全基因组变异位点及NBS基因家族内的基因进行鉴定、比对以及系统进化关系分析,旨在从基因组水平探寻晋汾52与晋谷21抗病性差异的原因,该研究结果可为确定NBS类抗病基因差异提供理论依据,并为进一步深入发掘晋谷21与其亲本的NBS基因抗性位点差异及培育抗病品种提供数据参考和理论依据。
1 材料和方法
1.1 试验材料
晋汾52号及其辐射诱变后的突变体品种晋谷21和豫谷1号(全基因组已测序)种子均由山西农业大学生物工程研究所提供,不同谷子品种于5月上旬种植于山西农业大学试验基地,常规田间管理,及时间苗除草,在谷子拔节期分别取长势一致的谷子新鲜叶片,迅速液氮冷冻后放入-70 ℃冰箱保存,用于基因组DNA的提取。
1.2 基因组DNA的重测序
采用基因组DNA提取试剂盒(柱式植物DNAout,天恩泽基因科技有限公司,北京)分别提取晋汾52与晋谷21基因组DNA。随后用1%琼脂糖凝胶电泳检测所提取DNA的质量纯度和完整性。随后将DNA送至百迈客公司进行基因组重测序。
1.3 晋谷21和晋汾52的重测序数据统计
通过全基因测序获得的晋汾52和晋谷21 两个品种的重测序数据,利用Varscan软件对SNP和InDel突变及变异进行识别分析。接着,利用Control-freec软件检测晋汾52和晋谷21两个品种之间的CNV位点进行分析,确定CNV数目;随后利用Breakdancer软件检测对2个品种的SV,根据SV在参考基因组上的位置信息,比对参考基因组的基因。
1.4 NBS类型基因的确定及定位分析
通过http://phytozome.jgi.doe.gov/pz/#!info?alias=Org_Sitalica检索下载得到谷子NBS类型基因的序列及相关信息。随后,利用BWA软件将NBS类型基因序列与参考基因组基因序列比对。通过PFAM鉴定NBS基因所编码的保守结构域。最后,结合利用谷子基因组数据库和参考基因组,利用数字化绘图软件AutoCAD 2010,将所有查找到的NBS类型抗病基因定位到染色体上的相应位置上。
1.5 NBS类型抗病基因系统进化树的构建
采用ClustalX软件对我们分别鉴定的2个品种中的NBS类型基因序列进行多序列比对,利用MEGA5.1软件分别构建2个品种NBS类型基因的系统进化树,并对其进行人工修饰,随后进行系统发育分析。
2 结果与分析
2.1 JF52和JG21重测序数据统计
晋汾52与晋谷21两个样品基因组重测序的测序数据见表1,所测序2个样品中的GC含量相当,分别为45.35%和45.70%。2个样品的测序质量(Q20碱基数量)高,均在90%以上,均达到了测序要求。随后将2个品种的测序数据,采用BWA软件与参考基因组进行比对分析发现,2个品种的映射率分别92.29%和94.55%,映射率均达到90%以上, 覆盖率分别为87.85%和90.21%,测序数据都能比到参考基因组(表2)可进一步进行后续分析。
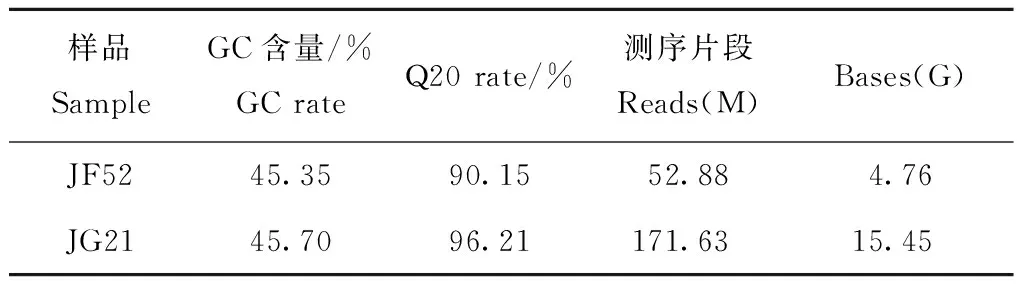
表1 两个品种测序数据比较Table 1 Comparison of two varieties of sequence data
2.2 两个谷子品在整个基因组水平SNP、InDel、CNV和SV分析
利用varscan软件对晋汾52和晋谷21基因组SNP分析发现,其主要变异主要发生在纯合突变(AA-aa),在谷子9条染色体上共发现530 181个纯合突变,占所有突变的76.81%,其次是杂合缺失(AB-A或AB-B)、杂合突变(AA-AB),其它类型的突变占比最少。在所有染色体上纯合突变>杂合缺失>杂合突变>其它类型突变(图1);通过插入缺失位点分析(InDel)分析, 共发现56 707个插入缺失位点,分布在各染色体上,其中8号染色体上的InDel位点最多,其次是3号和9号染色体,4号和1号染色体的InDel位点最少;使用control-freec软件分析拷贝数变异(CNV),共发现284个,其中扩增(gain)208个,缺失(loss)76个。这些变异在谷子的9条染色体上上均有发生,主要发生在1号和6号染色体上,2号和3号染色体上的发生变异数目最少;使用breakdancer软件在2个品种中共发现20 358个结构变异(SV),其中插入(INS)有9 000个,删除有(DEL)5 274个,移位有(ITX)3 260个,倒位有(INV)2 860个,易位(CTX)所占数目最少。由图1可知,发生在染色体上的结构变异数目以第8条染色体最多,9号染色体次之,4号染色体最少。
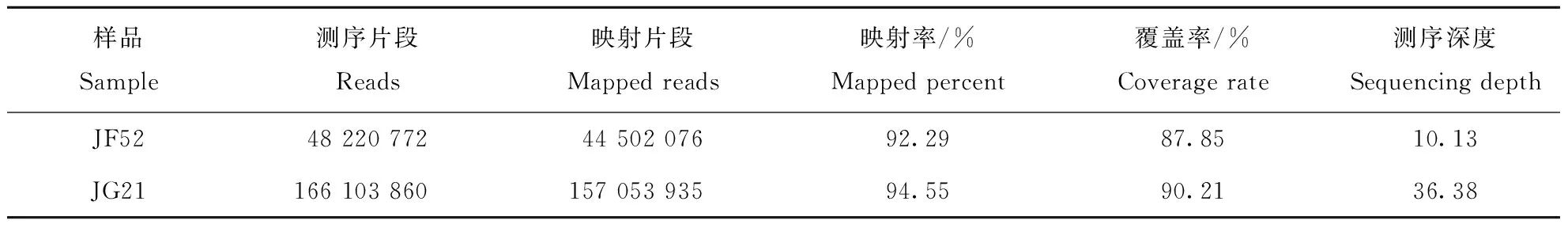
表2 两个品种测序数据与参考基因组比对Table 2 Comparison of two varieties of sequencing data and reference genome

图1 各染色体上SNP、Indel、CNV和SV的数目Fig.1 The number of SNP、InDel、CNV and SV on each chromosome
2.3 NBS类型基因在染色体上定位
将晋汾52和晋谷21与参考基因组比对后发现(表3),在晋汾52和晋谷21中分别有282和285个NBS类型基因(比晋汾52多3个基因:Si027 455m,Si028 382m,Si027 260m)。 各个品种除了6号和8号染色体上的NBS类型基因数目不一致外,在其他染色体上基因数目均没有差异。通过比较这些基因在染色体上的定位发现,多数基因都是在染色体相同的位置上,个别基因的位置是首尾相连的,如图2所示。综上所述,NBS类型基因主要分布在第8条染色体上,6号染色体上该类型基因最少。
表3NBS类型基因在谷子9条染色体上的分布
Table3Distribution of NBS genes on nine chromosomes in foxmail millet
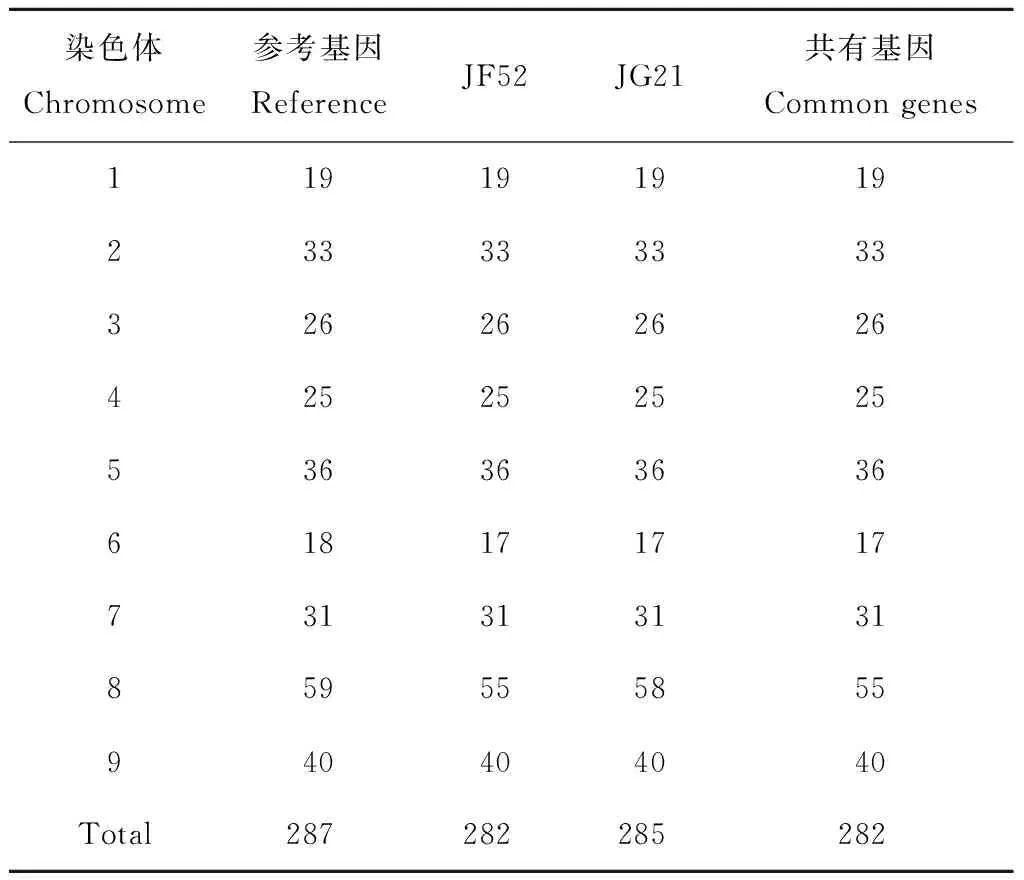
染色体Chromosome参考基因ReferenceJF52JG21共有基因Common genes119191919233333333326262626425252525536363636618171717731313131859555855940404040Total287282285282
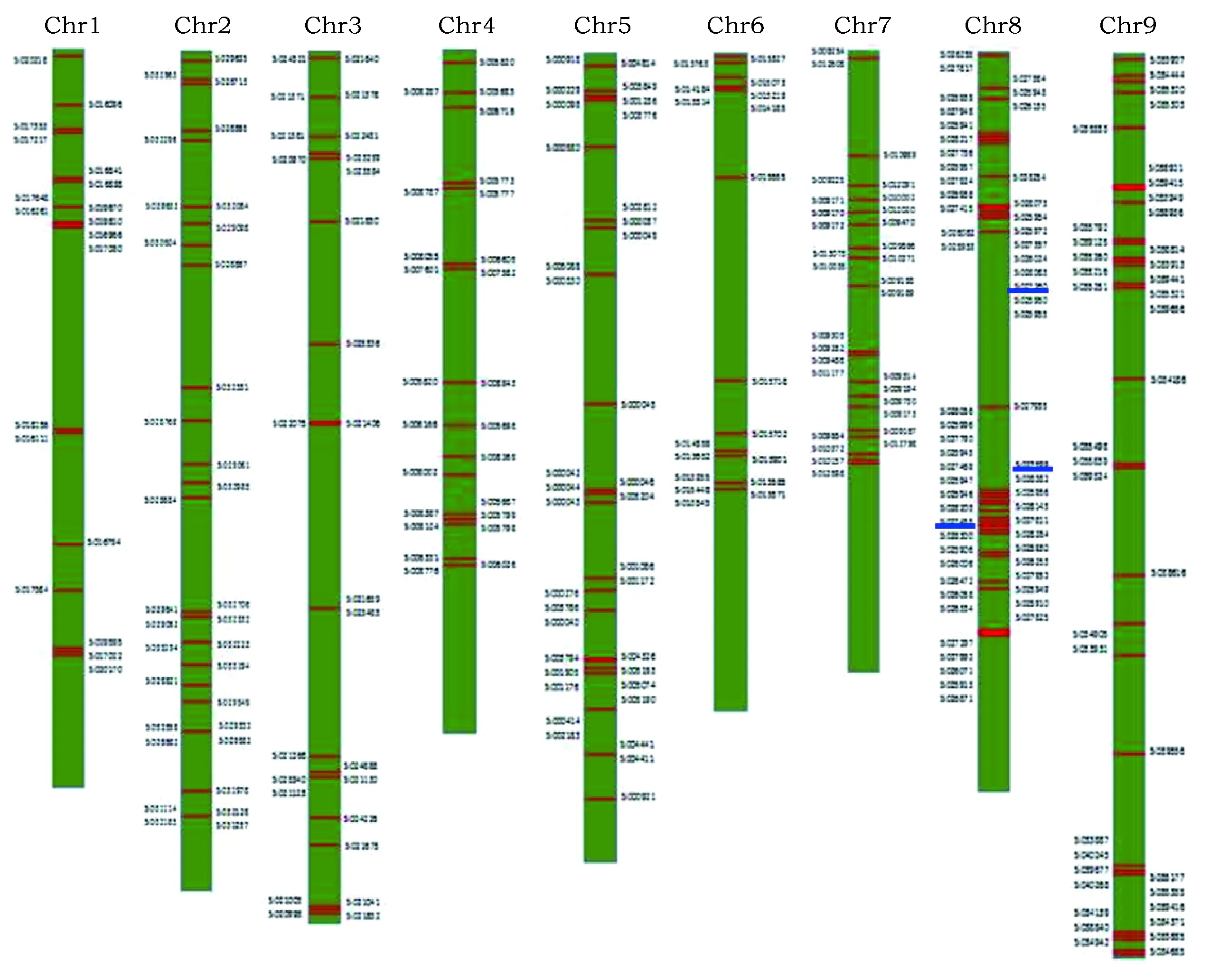
图2 晋谷21的NBS类型基因在染色体上的分布Fig.2 Chromosomal distribution of the NBS gene for JG21注:蓝色横线为晋谷21比晋汾52多出的3个基因。Note:Gene names with blue line represent three genes that presents in JG21 but not in JF52.
2.4 2个品种NBS类型基因保守结构域分析
把测序得到的reads利用blast与NBS类型基因的保守结构域进行比对。发现晋汾52的reads比对到了所有NBS类型基因的保守结构域(GGKTL、GLPLA、Kinase_2 a和MHDV),而晋谷21仅比对到一个保守结构域:GGKTL(表4)。推测其原因,可能是诱变后的晋谷21的抗病基因保守结构域的缺失,导致了晋汾52的抗病性比晋谷21强。

表4 2个品种NBS类型基因结构域Table 4 Domains of NBS genes in JG21 and JF52
2.5 NBS类型基因的同源性分析
通过对晋谷21的285个和晋汾52的282个NBS类型抗病基因系统发育分析(图3)发现,进化树明显地分为6大支,但是每一个分支中的所包含的NBS类型基因的数目是不一样的,晋谷21的6个分支相对亲本晋汾52来说显得比较杂乱(图3),由系统发育树可知,经辐射诱变后的晋谷21的NBS类型基因上许多位点发生突变,然而,这些突变位点的差异可能导致了2个品种抗病性差异。因此推测,抗病基因在演化过程中不是以单一的进化方式进行的,演化方式比较复杂。
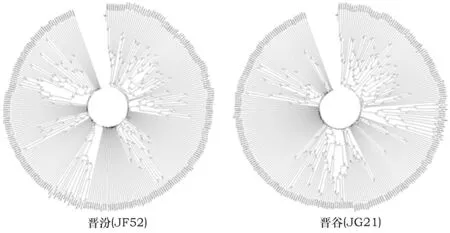
图3 JF52和JG21的NBS类型基因的系统树Fig.3 NBS type gene phylogenetic tree of JF52 and JG21
2.6 2个谷子品种NBS家族基因的SNP、InDel、CNV和SV分析
通过对晋汾52和晋谷21两个品种的中NBS家族基因进行分析,发现了2 229个SNP位点,包含LOH、Heter和Homo 3个主要突变类型,经统计,Homo类型突变数目最多,共1 431个,其次是 LOH类型突变, Heter类型突变最少(图4)。对3种类型的突变分析发现,在第8条染色体上LOH、Heter和Homo均是最多的,换而言之,第8条染色体上SNP变异微点最多;对2个品种NBS家族的插入缺失位点InDel位点分析,在2个品种中确定了70个基因共181个InDel位点,除了在第1条染色体没有发现外,在其他染色体上均有分布,我们发现,无论是NBS基因数目还是InDel位点,在第8号染色体也是最多的;对CNV比较,发现22个基因存在CNV,包括扩增(gain)和缺失(loss)。其中第 8号染色体上的基因数量和CNV也是最多的;通过对SV的分析,SV数目同样在第8号染色体占比最多。
3 讨论与结论
全基因组重测序技术是一种基于某一物种的全基因组信息已经公布,利用二代测序技术对某一植物病原菌的个体样本或群体样本进行测序,随后与已经公布的基因组序列比对分析,发掘样品中的单核苷酸多态性(SNP),插入缺失(InDel),拷贝数变异(CNV),结构变异(SV)等[11],对探究物种进化和重要功能基因挖掘具有非常重要的意义。
基于基因组重测序技术,首先对晋谷21及其亲本晋汾52的测序数据在全基因组水平进行了SNP,InDel,CNV 及SV分析,发现这4个类型的变异在谷子的第8条染色体上最多。随后,对2个品种中的NBS类型基因的SNP、InDel、CNV 及SV也进行分析,发现4个类型的变异位点也是以第8条染色体上最多。有研究发现,水稻的第11条染色体上的NBS类型基因数目比其他染色体上的都要多,该染色体与谷子的第8条染色体存在明显的共线性,2条染色体上的NBS类型基因数目和包含的基因簇的数量均是最多的[12],同时也验证了分析结果的可靠性。综合分析,由于晋汾52和晋谷21在遗传背景上高度相似,推测2个谷子品种抗病性的差异很可能与8号染色体上NBS类型基因位点差异关,其中还发现,单核苷酸变异数目较多,推测单核苷酸变异(SNP)也可能是造成2个谷子品种抗/感差异的原因之一。
对晋汾52和晋谷21的NBS类型基因的同源性分析,2个品种的NBS基因均6个分支中,但是每个分支中所包含基因的数目明显不同(图3)。对该类型的基因进行保守结构域分析发现,辐射诱变后的晋谷21 与其亲本相比,缺少了GLPLA、Kinase_2 a和MHDV 3个保守的结构域,可能也是引起聚类分支不一致的原因之一(表4)。并且分析发现NBS类型基因主要分布在第8条染色体上,而且这些抗病基因多数以基因簇的形式存在,这与Zhu等[10]研究分析结果一致。

图4 NBS家族基因各染色体上SNP、Indel、CNV和SV的数目Fig.4 The number of SNP、Indel、CNV and SV on each chromosome in NBS gene family
本研究通过利用全基因组重测序数据,对2个谷子品种在整个基因组水平和NBS类型基因存在的差异和变异位点分析。分析发现SNP的突变类型以纯合突变为主,InDel和SV突变数目在第8条染色是最多的; NBS类型基因分析,差异也主要存在于第8条染色体上。推测2个品种NBS类型基因结构的差异可能是造成抗感差异的原因,其研究结果可为我们进一步分析基因关键差异位点,克隆和验证抗病基因的功能提供理论依据。
