诺如病毒GII.6 P粒子原核表达与纯化
2018-07-04余明霞喻勇新潘迎捷王永杰
余明霞, 蔡 慧, 陈 豪, 喻勇新,2, 潘迎捷,2, 王永杰,2*
(1.上海海洋大学 食品学院,上海 201306;2.农业部 水产品贮藏保鲜质量安全风险评估实验室(上海),上海 201306)
诺如病毒(Norovirus, NoV)属于杯状病毒科(Caliciviridae)诺如病毒属(Norovirus),旧名“诺瓦克样病毒”或“小圆结构病毒”,可导致人和动物的非细菌性急性肠胃炎[1]NoV的“爆发”也是近年导致发达国家非细菌性腹泻发病率持续上升的重要原因之一[2],是人类健康的主要威胁[3]。研究表明,在牡蛎的组织中发现了NoV的特异性配体,并协助牡蛎特异性的富集NoV[4],进一步验证了牡蛎有可能是NoV在环境中传播的重要载体。由于NoV不能在体外高效增殖[5],表达带有与受体结合位点的蛋白成为目前NoV免疫学研究的主要途径。NoV是单股正链的RNA病毒,无囊膜,直径27~32 nm。基因组全长约7 500 nt,包含3个开放阅读框(Open Reading Frames, ORFs),即ORF1、ORF2(VP1)、ORF3(VP2)[6]。ORF1编码非结构蛋白,ORF2和ORF3分别编码主要衣壳蛋白和次要衣壳蛋白。ORF2能通过真核表达系统获得病毒样颗粒(Virus-like particle,VLP),VLP与NoV有相同的抗原类型和结合位点,在一定程度上可以代替NoV在体外进行病毒感染机制和免疫学研究[7-8]。相较于原核表达系统,真核表达系统耗时长,耗资大。然而ORF2难以在原核表达系统中表达。ORF2可分为S(Shell)区和P(Protruding)区,P区可在原核表达系统中表达获得P粒子。P粒子作为NoV的亚基粒子,与VLP显示相同的抗原类型,且该粒子与HBGAs受体(Histo-blood group antigens)结合的模式与VLP相同[7]。此外,P粒子具有高度稳定性和高免疫原性,能够在大肠埃希菌中高效表达[9-10]。本研究开展了NoV GII.6 P粒子的原核表达与纯化工作,为后续研究提供实验材料。
1 材料与方法
1.1 材料
1.1.1 样品 含GII.6 NoV的粪便样品,由中国疾病预防与控制中心提供。
1.1.2 仪器与设备 AKTA-pure蛋白纯化仪(GE),小型高速冷冻离心机(eppendorf,5417R),高速冷冻离心机(HITACHI,CR 21GIII),PCR仪(eppendorf,vapo.protect);GSTrap FF1×5 mL(GE,17-5131-01),0.22 μm滤器和0.45 μm滤器(Millipore),PVDF膜(Millipore)。
1.1.3 试剂 RNA提取试剂盒(捷瑞,GK3015),LB固体培养基和LB液体培养基(北京陆桥),T载体PCR产物克隆试剂盒(生工,SK2211),蛋白上样缓冲液(生工,C508319-0001),DNA分子标尺(天根),克隆菌株TOP10(天根,CB104),表达菌株BL21(microgene,CH5003A),表达菌株Rosetta(microgene,CH5003B),表达菌株OrigamiB(microgene,CH5003F),PCR Mix(TaKaRa,RR901A),非预染蛋白质分子标尺(TaKaRa,3597A),BamH I(Thermo,ER0057),SalI(Thermo,ER0641),T4连接酶(Thermo,EL0014),兔源GST-tag一抗(ABclonal,AE006),抗兔二抗(碱性磷酸酶标记)(ABclonal,AS009),预染蛋白质分子标尺(Thermo,26616),BCA法蛋白质浓度测定试剂盒(生工,C503021),考马斯亮蓝R-250(Ourchem,6104-59-2),还原型谷胱甘肽(amresco,70-18-8)。溶菌酶、过硫酸铵、β-巯基乙醇、TEMED、甘油、Tween-20、30%聚丙烯酰胺、苯甲基磺酰(PMSF)、BCIP/NBT显色液、氯化钠、氯化钾、十二烷基硫酸钠、脱脂奶粉均购自生工生物工程(上海)股份有限公司;N-N二甲基甲酰、氯化硝基四氮唑、5-溴-4-氯-3吲哚磷酸酯对甲苯胺盐、甲醇、冰乙酸、甘氨酸、尿素均购自上海国药集团。
1.2 方法
1.2.1 克隆 粪便样品按照10%(质量分数)重悬于1×PBS(pH 7.3)中,每管100 μL,-80 ℃保存。病毒RNA利用RNA提取试剂盒从100 μL粪便重悬的上清中提取,最终总RNA洗脱于35 μL 经DEPC处理的水中。利用嵌套式逆转录聚合酶链式反应(nest reverse transcription polymerase chain reaction, Nest RT-PCR)[11-12],从粪便样品中筛出GII.6阳性样品。从NCBI数据库中下载相关的NoV GII.6 VP1序列,根据这些参考序列设计GII.6 VP1的引物,采用逆转录聚合酶链式反应(RT-PCR)从GII.6阳性样品中获得GII.6 VP1的序列,经TA克隆至pUCm-T载体上进行测序验证,确保获得的序列是GII.6 的VP1序列。根据GII.6 VP1的序列,设计P区的表达引物,将BamH I和SalI这两种酶的酶切位点分别加在正反引物的5′端(斜体),在反向引物的5′端分别加上半胱氨酸(粗体)和终止密码子(下划线),并在正反引物的5′端酶切位点的前面分别加上对应的保护碱基(见表1)。通过PCR将P区序列从无突变的pUCm-T-GII.6 VP1扩增下来,经BamH I和SalI双酶切并纯化回收后,连接到同样经BamH I和SalI双酶切过的表达载体pGEX-4T-1上。转化到克隆菌株TOP10中,37 ℃,200 r/min过夜培养后提取重组质粒pGEX-4T-1-GII.6P,测序。将测序结果与pUCm-T-GII.6 VP1进行比对,确定所获得的P区表达序列没有突变。GII.6 P序列确认无误后,利用在线软件ExPASy(http://web.expasy.org/protparam/)预测P粒子的大小,作为验证表达的参考依据。并利用在线软件NCBI ORF finder (http://www.ncbi.nlm.nih.gov/orffinder/) 对表达载体pGEX-4T-1-GII.6 P进行ORF预测,判断目的蛋白表达断裂的可能性。
1.2.2 蛋白表达 将pGEX-4T-1-GII.6 P转化到表达菌株BL21中,挑取单克隆菌株置于LB(Amp+:100 mg/L)液体培养基中,37 ℃ 200 r/min过夜扩增培养后,按照1∶100的比例接种至LB(Amp+)液体培养基中,37 ℃、200 r/min培养3~4 h,至OD600值为0.5~0.7,加入1×10-3mol/L的 IPTG,在22 ℃、200 r/min下诱导培养22 h,12 000 r/min离心30 min收集菌体。将收集到的菌体重悬于含5%甘油的1×PBS(pH 7.3)中,加入蛋白酶抑制剂,置于-20 ℃冰箱中冷冻1.5 h后取出,置于室温融化后,加入溶菌酶(20 mg/mL)于37 ℃、200 r/min摇床培养30 min。取出置于冰上超声(350 W,超声时间为3 s,间隔4 s,循环80次)破碎,至菌液透亮。12 000 r/min、离心30 min,分离上清与沉淀。利用SDS-PAGE和Western blot分别对表达的蛋白进行定性和定量分析,验证目的蛋白是否表达,并确定目的蛋白的表达量。

表1 引物列表Table 1 Primers used in this study
注:“1”表示引物5′端在序列X86557上的位置;“2”表示引物5′端在序列KC576910上的位置
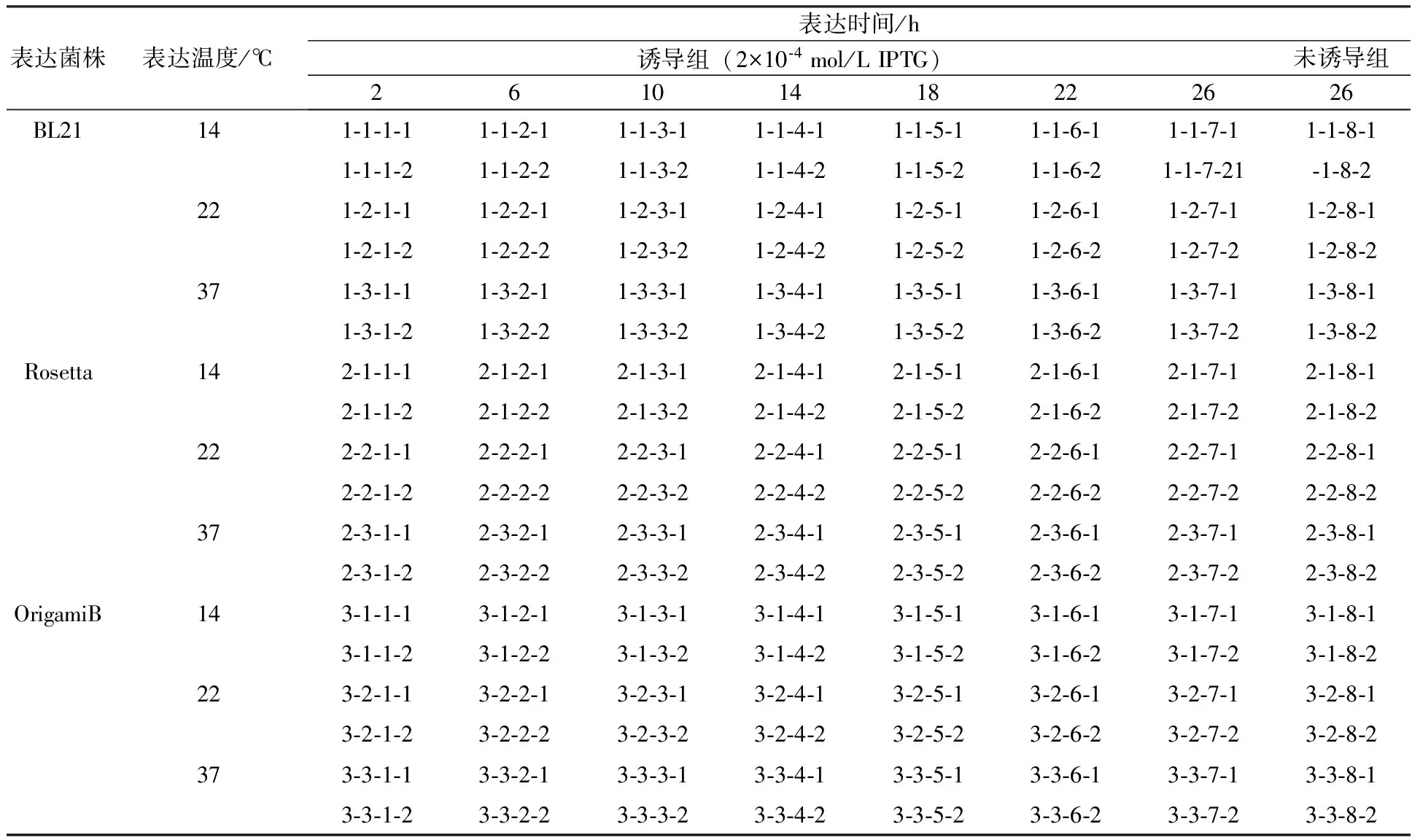
表2 表达优化方案Table 2 The strategy of expression optimization
1.2.3 蛋白表达的条件优化 具体优化方案如表2所示。根据文献中诱导剂IPTG的参考值,设计了对应的浓度梯度,再分别对表达菌株、表达时间和表达温度同时进行优化。每个实验组设置2个平行,未诱导组不加诱导剂,其他条件同诱导组。
1.2.4 蛋白纯化条件的优化 将上清置于冰上,用1×PBS(pH 7.3)将AKTA-pure系统平衡好后,上样并收集流穿液。然后用还原型谷胱甘肽进行梯度洗脱,收集洗脱产物。用SDS-PAGE验证流穿液和洗脱产物,确定目的蛋白是否挂柱,并确定能否被还原型谷胱甘肽洗脱下来以及还原型谷胱甘肽的具体洗脱浓度。根据SDS-PAGE和Western blot的结果,分别从清洗纯化柱的试剂、清洗浓度、洗脱浓度、上样速度和增加纯化步骤等方面,对纯化条件进行优化。
2 结果与分析
2.1 表达质粒的构建
通过RT-PCR,利用引物GII.6 VF和GII.6 VR(表1)从RNA样品中扩增出GII.6 VP1,电泳条带与目的产物大小吻合,如图1(a)中GII.6 VP1所示。由于需对GII.6 VP1测序验证,故选取克隆重组质粒pUCm-T-GII.6 VP1进行测序验证,pUCm-T-GII.6 VP1的电泳结果与预测大小一致,如图1(a)中GII.6 VP1重组质粒所示。根据获得的GII.6 VP1序列,设计了表达引物GII.6 PF和GII.6 PR,并利用表达引物通过PCR从pUCm-T-GII.6 VP1上扩增出带有酶切位点和半胱氨酸的插入序列,大小为993 bp(GII.6)+10 bp(GII.6 PF)+40 bp(GII.6 PR)=1 043 bp,与电泳结果一致,如图1(a)中表达片段所示。将表达质粒pGEX-4T-1(如图1(a)中表达载体所示)和GII.6 P表达序列经双酶切后,通过克隆,最后提取质粒测序,表达质粒预测大小与电泳结果一致,如图1(a)中表达质粒所示。通过比对最终的GII.6 P表达序列与最初的GII.6 VP1序列可知,二者的相似性为100%,表达载体pGEX-4T-1-GII.6 P构建成功。
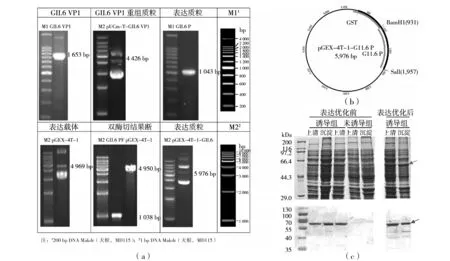
图1 GII.6 P粒子的表达Fig.1 GII.6 P particle expression(a)重组表达质粒pGEX-4T-1-GII.6 P的构建;(b)表达质粒pGEX-4T-1-GII.6 P的ORF预测结果:图中灰色标注部分分别是GST标签和GII.6 P,黑色加粗部分为ORF预测结果;(c)GII.6 P粒子的表达验证及表达条件的优化结果:图中箭头所指为目的蛋白条带位置(a) The recombination of expression plasmid pGEX-4T-1-GII.6 P; (b) ORF finder of pGEX-4T-1-GII.6 P: the grey marked part of GST tag and GII. 6 P, black bold marked ORF prediction result; (c) The results of expression checking and optimization: arrow in the figure presented the target protein bands
2.2 表达产物大小预测和ORF预测
在线蛋白大小预测软件ExPASy结果显示,GII.6 P表达产物的大小约为36.34 kDa。查阅文献可知,GST-tag蛋白的大小为26 kDa,所以融合蛋白的大小约为62 kDa。图1(b)中,灰色注释的序列分别是GST标签和插入GII.6 P片段,黑色加粗的序列是ORF预测的结果。ORF预测结果显示,表达质粒pGEX-4T-1-GII.6 P在表达过程中,融合蛋白出现表达断裂的机会比较小。
2.3 表达验证和表达优化的结果
图1(c)中,每孔上样量均为37.5 μL表达菌液的产物,所以每个点样孔之间都存在可比性,图1(c)中,上半部分是SDS的结果,下半部分是Western-blot的结果。对比诱导组和未诱导组的Western-blot结果,可以得出结论:GII.6 P粒子表达成功,并且是以可溶性蛋白的形式表达。比较上清和沉淀中目的蛋白的含量,发现沉淀中依然存在大量的目的蛋白,故分别从表达菌株、表达时间、表达载体、表达温度和诱导剂的浓度等项目,对表达条件进行优化,以期从上清中获得更多的目的蛋白。首先,根据SDS-PAGE胶图结果,优化诱导剂的诱导浓度,根据文献中诱导剂IPTG的参考值,设计的对应浓度梯度为1.5×10-3、1.0×10-3、8×10-4、5×10-4、4×10-4、2×10-4、5×10-5mol/L,最终确定的诱导剂IPTG的浓度为2×10-4mol/L。在此基础上,通过比较每一个表达产物的上清中目的蛋白的含量,确定最终的表达条件。其次,基于表达菌株的比较,待选表达菌株有BL21、Rosetta和OrigamiB,BL21和Rosetta表达的上清中目的蛋白含量均高于OrigamiB,这两者间没有明显的区别,上清中目的蛋白的含量基本相同,最终根据文献[19],选择BL21作为表达菌株。同时,将表达温度分别设定为16、22 和37 ℃,其中,最终通过比较表达上清中目的蛋白的含量决定最适表达温度,14 ℃ 表达产物的上清中目的蛋白总体低于22 ℃ 和37 ℃ 下的诱导产物,且14 ℃ 和37 ℃ 表达产物中目的蛋白主要存在于沉淀中,所以表达温度最终定为22 ℃ 。从诱导后2~26 h,4 h为一个梯度进行诱导表达,停止表达后需立即离心收集表达产物。SDS-PAGE胶图结果显示,上清中目的蛋白的含量随表达时间的延长而增加,但是诱导22 h后,随着时间的增长,上清中目的蛋白的含量有减少的趋势,所以表达时间最终定为22 h。优化后的表达条件为诱导剂浓度2×10-4mol/L,表达菌株为BL21,表达温度为22 ℃,表达时间为22 h。
2.4 蛋白纯化验证和纯化优化的结果
由图2结果显示,目的蛋白能挂柱,并且能被1×10-2mol/L还原型谷胱甘肽洗脱。在此基础上,对纯化条件做以下优化:降低上样速度(最大化样品挂柱效率)、上样后利用还原剂清洗亲和柱(在确保不影响目的蛋白的前提下,尽可能多地去掉杂蛋白)、结合多种纯化方法(获得纯度较高的目的蛋白)。
本研究中,对于500 mL培养液离心后的菌体裂解液,通过比较流穿液的Western-blot结果,发现上样流速不超过1 mL/min时,流穿液中没有目的蛋白。上样后,分别用DTT、尿素和1‰ Triton 100清洗标签柱中的杂蛋白,SDS-PAGE结果显示DTT清洗杂蛋白的能力最好。亲和纯化主要作用是最大限度地捕获目的蛋白。为了进一步得到纯度更高的蛋白,在亲和纯化的基础上,结合阴离子交换和凝胶过滤层析,最终获得纯度大于90%的目的蛋白,结果如图3所示。
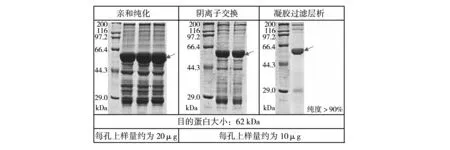
图3 蛋白纯化条件优化的结果Fig.3 Purification verification
3 讨 论
牡蛎是滤食性生物,组织中有NoV特异性的配体,富集NoV的能力较强,食用被污染的牡蛎是近年来NoV爆发的原因之一[20]。鉴于牡蛎作为NoV在环境中传播的主要载体[21-24],对于牡蛎富集NoV的机理研究显得尤为重要。最新研究结果表明[25-26],NoV能够在B细胞和肠道细胞中增殖,但不稳定,因此难以获得足够的病毒颗粒用于探索NoV的传播与富集。研究证实NoV P粒子具有与病毒相似的抗原特性,使得进一步的研究成为可能。
本研究对NoV P粒子表达的优化结果与前人研究基本相同[12-13,19,27],在选取表达菌株的时候,考虑到GII.6 P的序列中含有大肠埃希菌稀有密码子,Rosetta菌株能够补充大肠埃希菌缺乏的 6 种稀有密码子(AUA、AGG、AGA、CUA、CCC和GGA)对应的 tRNA,从而提高外源基因在原核系统中的表达水平;由于本研究最终是为了获得更多有活性的GII.6 P粒子,而OrigamiB菌株包含突变的硫氧还原蛋白酶和谷胱甘肽还原酶基因,可以表达主要还原途径的两个关键酶,有利于形成正确折叠的含有二硫键的蛋白,增强蛋白可溶性;至于BL21则是引自NoV P粒子表达的相关文献[19]。经过本研究的验证可以进一步证明,这个表达系统基本适用于NoV P粒子的表达,针对不同基因型P粒子的表达,仅需对IPTG的浓度进行优化即可。
本研究对NoV P粒子的纯化条件进行了系统优化,根据首次纯化产物的SDS-PAGE胶图结果,首先对上样速度进行了优化,从而保证尽可能多地捕获目的蛋白,最终上样速度确定为0.5 mL/min。在此基础上,尽可能多地去除杂蛋白。对于杂蛋白的去除,首先采取的方法是在亲和纯化的基础上引入还原剂,上样后,优化还原剂的浓度和类型来清洗亲和柱,根据亲和纯化的结果可知,仅仅基于亲和纯化是无法去除杂蛋白的。然而分析亲和纯化的结果可知,杂蛋白主要集中在25~50 kDa,与目的蛋白GII.6 P粒子(62 kDa)的大小差距不大,无法直接利用凝胶过滤层析除去。另外,由于未知蛋白GII.6 P粒子的等电点不详,无法根据GII.6 P粒子的特性来选择蛋白纯化工序,只能根据GST标签来间接纯化GII.6 P粒子。
由于GST标签可以与阴离子交换柱结合,因此在亲和纯化的下游选择了阴离子交换。经过对阴离子交换纯化条件的优化,最终成功除去了部分位于25~50 kDa区间的杂蛋白,通过分析阴离子交换蛋白纯化的结果可知,仅剩下三个主要杂蛋白带:近180 kDa、近44.3 kDa和近25 kDa,而目的蛋白GII.6 P粒子(62 kDa)的大小与近180 kDa和近25 kDa之间的差距较大(>25),故可尝试利用凝胶过滤层析去除这两个杂蛋白带。最终通过优化凝胶过滤层析的条件,获得了纯度较高的GII.6 P粒子。
本研究构建了NoV P粒子的原核表达及纯化体系,提供了获得NoV P粒子的详细步骤以及蛋白表达和纯化的优化方案。与此同时,通过原核表达得到了大批量、高纯度的GII.6 P粒子,为下一步的多克隆抗体制备提供充足的合格抗原,进而为后续牡蛎富集NoV的机理研究提供了实验材料。
参考文献:
[1] Blacklow NR, Greenberg HB. Viral gastroenteritis[J]. The New England journal of medicine, 1991, 325(4): 252-264.
[2] De wit MA, Koopmans MP, Kortbeek LM, et al. Sensor, a population-based cohort study on gastroenteritis in the Netherlands: incidence and etiology[J]. American journal of epidemiology, 2001, 154(7): 666-674.
[3] Van duynhoven YT, De jager CM, Kortbeek LM, et al. A one-year intensified study of outbreaks of gastroenteritis in The Netherlands[J]. Epidemiology and infection, 2005, 133(1): 9-21.
[4] Hale A, Mattick K, Lewis D, et al. Distinct epidemiological patterns of Norwalk-like virus infection[J]. Journal of medical virology, 2000, 62(1): 99-103.
[5] Wheeler JG, Sethi D, Cowden JM, et al. Study of infectious intestinal disease in England: rates in the community, presenting to general practice, and reported to national surveillance. The Infectious Intestinal Disease Study Executive[J]. Bmj, 1999, 318(7190): 1046-1050.
[6] Green KY. The role of human caliciviruses in epidemic gastroenteritis[J]. Archives of virology Supplementum, 1997, 13(153-165).
[7] Iturriza-gomara M, Elliot AJ, Dockery C, et al. Structured surveillance of infectious intestinal disease in pre-school children in the community: The Nappy Study[J]. Epidemiology and infection, 2009, 137(7): 922-931.
[8] 徐友富, 童贻刚, 茹志涛,等. 诺如病毒感染国内外研究概况[J]. 中国卫生检验杂志, 2008, 18(5): 949-951.
[9] Lees D. Viruses and bivalve shellfish[J]. International journal of food microbiology, 2000, 59(1-2): 81-116.
[10] Le Guyader FS, Atmar RL, Le Pend J. Transmission of viruses through shellfish: when specific ligands come into play[J]. Current opinion in virology, 2012, 2(1): 103-110.
[11] Lou F, Dicapeio E, Li X, et al. Variable High-Pressure-Processing Sensitivities for Genogroup II Human Noroviruses[J]. Applied and environmental microbiology, 2016, 82(19): 6037-6045.
[12] Jin M, Tan M, Xia M, et al. Strain-specific interaction of a GII.10 Norovirus with HBGAs[J]. Virology, 2015, 476:386-394.
[13] Tan M, Zhong W, Song D, et al.E.coli-expressed recombinant norovirus capsid proteins maintain authentic antigenicity and receptor binding capability[J]. Journal of medical virology, 2004, 74(4): 641-649.
[14] 吴琼, 何玉林. 诺如病毒的研究进展[J]. 中国人兽共患病学报, 2014, 30(12): 1245-1251.
[15] Tan M, Jiang X. Norovirus P particle: a subviral nanoparticle for vaccine development against norovirus, rotavirus and influenza virus [J]. Nanomedicine, 2012, 7(6): 889-897.
[16] 高建. 诺如病毒GⅡ.4型Hunter504D株P粒子的表达,表征及免疫原性研究[D]; 长春:吉林大学, 2013.
[17] Kojna S, Kageyana T, Fukushi S, et al. Genogroup-specific PCR primers for detection of Norwalk-like viruses[J]. Journal of virological methods, 2002, 100(1-2): 107-114.
[18] Kageyama T, Kojima S, Shinohara M, et al. Broadly reactive and highly sensitive assay for Norwalk-like viruses based on real-time quantitative reverse transcription-PCR[J]. Journal of clinical microbiology, 2003, 41(4): 1548-1557.
[19] Tan M, Jian X. The p domain of norovirus capsid protein forms a subviral particle that binds to histo-blood group antigen receptors[J]. Journal of virology, 2005, 79(22): 14017-14030.
[20] Le Guyader FS, Parnaudeau S, Schaegger J, et al. Detection and Quantification of Noroviruses in Shellfish[J]. Applied and environmental microbiology, 2009, 75(3): 618-624.
[21] 马丽萍, 苏来金, 赵峰, 等. 长牡蛎中类 HBGAs 的分型及与诺如病毒 P 粒子结合特性研究[J]. 食品安全质量检测学报, 2015, 10: 3970-3975.
[22] Le Guyader FS, Loisy F, Aatmar RL, et al. Norwalk virus-specific binding to oyster digestive tissues[J]. Emerging infectious diseases, 2006, 12(6): 931-936.
[23] Maalouf H, Zakhour M, Le Pendu J, et al. Distribution in Tissue and Seasonal Variation of Norovirus Genogroup I and II Ligands in Oysters[J]. Applied and environmental microbiology, 2010, 76(16): 5621-5630.
[24] Yu Y, Cai H, Hu L, et al. Molecular epidemiology of oyster-related human noroviruses and their global genetic diversity and temporal-geographical distribution from 1983 to 2014[J]. Applied and environmental microbiology, 2015, 81(21): 7615-7624.
[25] Jones MK, Watanabe M, Zhu S, et al. Enteric bacteria promote human and mouse norovirus infection of B cells[J]. Science, 2014, 346(6210): 755-759.
[26] Papafragkou E, Hewitt J, Park GW, et al. Challenges of culturing human norovirus in three-dimensional organoid intestinal cell culture models[J]. PloS one, 2014, 8(6): e63485.
[27] Jin M, He Y, Li H, et al. Two gastroenteritis outbreaks caused by GII Noroviruses: host susceptibility and HBGA phenotypes[J]. PloS one, 2013, 8(3): e58605.
