ApoE和Slco1B1基因多态性与缺血性脑卒中的相关性分析
2018-07-04罗宝昌张敬
罗宝昌 张敬
脑卒中是指由于脑循环障碍所致的局部或全面性脑功能缺损综合征,现已成为全球致死率及致残率较高的神经系统疾病[1]。脑卒中主要分为缺血性脑卒中和出血性脑卒中两大类,其中缺血性脑卒中患者占66.7%左右[2]。缺血性脑卒中的发病机制复杂,不仅与大量的传统危险因素相关,而且基因多态性也严重影响缺血性脑卒中的发生[3-4]。
APOE基因位于染色体上以三种等位基因ε2、ε3和ε4形式存在,分别编码三种不同的蛋白亚型E2、E3和E4[5]。三种蛋白亚型仅表现为112位和158位氨基酸的差异,从而导致脂质亲和力的差异而可能影响缺血性脑卒中发生[4]。APOE基因多态性与脑卒中的相关性目前尚未定论,部分学者认为APOE基因多态性与缺血性脑卒中具有相关性[6-7],而部分学者认为两者之间无明显相关性[8-10]。地区和种族可能是影响结果的一个因素,而汉川市汉族人群APOE基因多态性与缺血性脑卒中的发生是否相关,尚未见相关报道。
Slco1B1基因位于染色体上,具有高度遗传多态性,现已发现40个以上的非同义单核苷酸多态性位点。他汀类药物如匹伐他汀、新伐他汀、普伐他汀等是治疗缺血性脑卒中的常见药物。研究发现Slco1B1基因位点rs4149056(521T>C)多态性影响他汀类药物的吸收,从而对疗效产生影响[11-12]。而Slco1B1基因位点rs4149056多态性与缺血性脑卒中的发生是否相关,尚未见相关报道。
本研究通过直接测序法分析缺血性脑卒中患者和健康对照人群ApoE基因型及Slco1B1 rs4149056位点基因型,从而进一步研究ApoE基因和Slco1B1多态性与汉川市汉族人群缺血性脑卒中的相关性,为揭示缺血性脑卒中发病机制及缺血性脑卒中的预防及治疗提供一定的理论基础。
1 材料与方法
1.1 一般资料及纳入、排除标准
选取2015年6月-2017年6月本院收治450例缺血性脑卒中患者为实验组,同时选取同期于本院体检的450例正常人为对照组,研究对象均来自汉川市汉族人群。纳入标准:诊断参考全国脑血管病学术会议制定的诊断标准,并经脑CT或MRT检查证实。排除标准:严重感染、肝肾功能异常、恶性肿瘤、甲状腺和血液系统疾病等患者。
1.2 基因组DNA提取
清晨收集受试者空腹外周静脉血2 mL,加入抗凝剂后混匀。根据OMEGA公司试剂盒Blood DNA kit说明书提取基因组DNA后-20 ℃保存。
1.3 克隆及测序
根据Qiagen公司Hotstar Taq Master Mix试剂盒进行PCR反应(引物序列见表1)。PCR反应体系为总体积25 μL,其中模板6 μL(约100 ng),2×Taq PCR Master Mix 12.5 μL,上下游引物各1 μL,去离子水4.5 μL。 PCR反应条件为95 ℃预变性5 min, 95 ℃变性30 s,62 ℃退火30 s,72 ℃延伸1 min,共35个循环,最后72 ℃延伸5 min。
2%琼脂糖凝胶电泳检测PCR情况,选择阳性克隆送至华大基因进行测序,GeneMapper软件用于基因分型。
1.4 统计学处理
2 结 果
2.1 一般资料比较
2组性别、年龄、身高、体重均无显著性差异(P>0.05)。
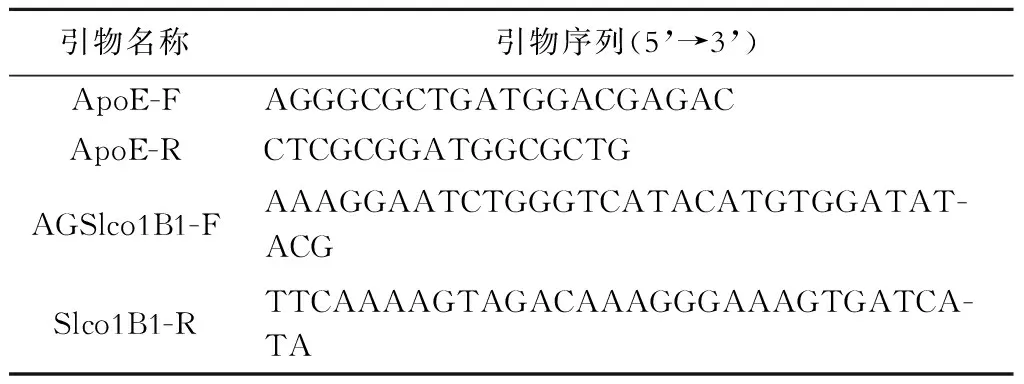
表1 PCR引物列表

表2 一般资料比较
2.2 ApoE基因多态性与缺血性脑卒中的相关性
对照组基因型频率都符合Hardy-weinberg平衡(P=0.982)(表3)。基因型分析发现2组中均含有ε2/ε2、ε2/ε3、ε2/ε4、ε3/ε3、ε3/ε4和ε4/ε4 6种基因型,2组基因型有显著性差异(P=0.034)(表4)。与ε3/ε3基因型比较,ε2/ε4、ε3/ε4和ε4/ε4基因型可能更易患缺血性脑卒中,分别为OR=3.219,95%CI=1.156~8.963;OR=1.391,95%CI=0.969~1.998和OR=3.219,95%CI=0.645~16.069。
2.3 Slco1B1基因多态性与缺血性脑卒中的相关性
对照组基因型频率都符合Hardy-weinberg平衡(P=0.469)(表5)。对照组T>C突变频率为11%,而实验组突变频率则达到15.2%,2组基因型有显著性差异(P=0.031)(表6)。与TT基因型比较,TC和CC基因型可能更易患缺血性脑卒中,分别为OR=1.290,95%CI=0.929~1.790;和OR=2.568,95%CI=1.109~5.945。

表3 对照组ApoE基因型Hardy-weinberg平衡

表4 实验组和对照组ApoE基因型分布

表5 对照组Slco1B1基因型Hardy-weinberg平衡
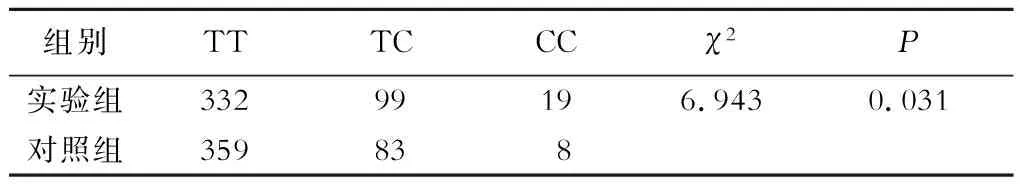
表6 实验组和对照组Slco1B1基因型分布
3 讨 论
载脂蛋白E(apolipoprotein E,ApoE)是一种富含精氨酸的碱性蛋白,可通过调节胆固醇及脂类物质的存储和再分布影响血浆蛋白代谢,同时参与组织修复、抑制血小板聚集等生理过程,并在神经系统的正常生长过程中及损伤后的修复过程中发挥作用[13]。截至目前,APOE基因多态性与缺血性脑卒中的相关性研究很多,但结论具有一些分歧[6-10]。本研究结果表明,汉川市汉族人群APOE基因多态性可能与缺血性脑卒中相关,ε2/ε4、ε3/ε4和ε4/ε4基因型可能更易患缺血性脑卒中,这与部分学者研究结果一致[6-7],但与Gu 等研究结果存在差异[8-10]。出现不同结果的原因可能是由于研究方法及纳入群体的差异所致。例如Gu等发现广西汉族APOE基因多态性与缺血性脑卒中无关,而通过Meta分析又发现汉族APOE基因多态性与缺血性脑卒中有关[8]。
Slco1B1基因编码蛋白质阴离子转运多肽(OATP1B1),阴离子转运多肽(OATP1B1)是一种特异性表达于肝细胞基底膜上转运蛋白,介导多种内源性或外源性物质从血液向肝细胞的跨膜转运[14-15]。SLCO1B1 rs4149056是常见的具有功能意义的SNP位点,其在不同种族人群中的分布存在很大的差异,在高加索人、非洲人和亚洲人中突变概率分别为15%~20%,1%~2%和10%~15%[16-18]。本研究对照组rs4149056突变频率为11%,而实验组突变频率为15.2%,这均与已报到的亚洲人群突变频率报道接近[18]。本研究发现Slco1B1基因rs4149056位点多态性可能与缺血性脑卒中有关,TC和CC基因型可能更易患缺血性脑卒中,等位基因C可能是脑卒中易感基因。
本研究结果显示,对照组APOE基因型和Slco1B1基因型都符合Hardy -weinberg平衡,这表明抽样具有代表性,适合于后续遗传分析(P=0.982和P=0.469)。通过病例-对照研究发现,汉川市汉族人群APOE基因多态性及Slco1B1基因rs4149056位点多态性可能与缺血性脑卒中有关。
本研究具有一定的局限性。一部分是样本数相对较少,可能会对结果产生影响,后期需扩大样本;另一部分是需多因素考虑,需排除个人生活习惯等造成的影响,后期需多因素综合分析,从而得出更准确的结论。
综上所述,汉川市汉族人群APOE基因多态性及Slco1B1基因rs4149056位点多态性可能与缺血性脑卒中有关。后期需加大样本量和多因素综合分析来进一步探索APOE基因多态性及Slco1B1基因多态性与缺血性脑卒中的相关性。
[1] Go AS,Mozaffarian D,Roger VL,et al.Heart disease and stroke statistics--2014 update: a report from the American Heart Association[J].Circulation,2014,129(3):e28-e292.
[2] Wang Y,Cui L,Ji X,et al.The China National stroke registry for patients with acute cerebrovascular events: design, rationale, and baseline patient characteristics[J].Int J Stroke,2011,6(4):355-361.
[3] Schulz UG,Flossmann E,Rothwell PM.Heritability of ischemic stroke in relation to age, vascular risk factors, and subtypes of incident stroke in population-based studies[J].Stroke,2004,35(4):819-824.
[4] Ribalta J VJ,Apolipoprotein RG.Blood lipids and disease[J].Curr Opin Clin Nutr Metab Care,2003,6(2):177-187.
[5] Utermann G. Isolation and partial characterization of an arginine-rich apolipoprotein from human plasma very-low-density lipoproteins: apolipoprotein E[J]. Hoppe Seylers Z Physiol Chem, 1975,356(2):1113-1121.
[6] Banerjee I,Gupta V,Ganesh S.Association of gene polymorphism with genetic susceptibility to stroke in Asian populations: a meta-analysis[J].J Hum Genet,2007,52(3):205-219.
[7] Wei LK,Au A,Menon S,et al.Clinical relevance of MTHFR, eNOS, ACE, and ApoE gene polymorphisms and serum vitamin profile among malay patients with ischemic stroke[J].J Stroke Cerebrovasc Dis,2015,24(9):2017-2025.
[8] Gu L,Su L,Chen Q,et al.Association between the apolipoprotein E gene polymorphism and ischemic stroke in Chinese populations: New data and meta-analysis[J].Exp Ther Med,2013,5(3):853-859.
[9] Lai CL,Liu CK,Lin RT,et al.Association of apolipoprotein E polymorphism with ischemic stroke subtypes in Taiwan[J].Kaohsiung J Med Sci,2007,23(10):491-497.
[10] Sturgeon JD,Folsom AR,Bray MS,et al.Apolipoprotein E genotype and incident ischemic stroke: the Atherosclerosis Risk in Communities Study[J].Stroke,2005,36(11):2484-2486.
[11] Takane H. Genetic polymorphisms of SLCO1B1 for drug pharmacokinetics and its clinical implications[J]. Yakugaku Zasshi Journal of the Pharmaceutical Society of Japan, 2011,131(11):1589.
[12] 隋双明,温金华,李新华,等.OATP1B1基因位点521T→C的突变对瑞舒伐他汀在中国健康人体内药代动力学特征的影响[J].药学学报,2011,46(6):695-700.
[13] Liu CC,Liu CC,Kanekiyo T,et al.Apolipoprotein E and alzheimer disease: risk, mechanisms and therapy[J].Nat Rev Neurol,2013,9(2):106-118.
[14] Abe T,Kakyo M,Tokui T,et al.Identification of a novel gene family encoding human liver-specific organic anion transporter LST-1[J].J Biol Chem,1999,274(24):17159-17163.
[15] K nig J,Cui Y,Nies AT,et al.A novel human organic anion transporting polypeptide localized to the basolateral hepatocyte membrane[J].Am J Physiol Gastrointest Liver Physiol,2000,278(1):G156-G164.
[16] Niemi M,Pasanen MK,Neuvonen PJ.Organic anion transporting polypeptide 1B1: a genetically polymorphic transporter of major importance for hepatic drug uptake[J].Pharmacol Rev,2011,63(1):157-181.
[17] Xu LY,He YJ,Zhang W,et al.Organic anion transporting polypeptide-1B1 haplotypes in Chinese patients[J].Acta Pharmacol Sin,2007,28(10):1693-1697.
[18] Pasanen MK,Neuvonen PJ,Niemi M.Global analysis of genetic variation in SLCO1B1[J].Pharmacogenomics,2008,9(1):19-33.
