耐碳青霉烯类肺炎克雷伯菌耐药基因检测与分子流行病学研究*
2018-06-05张湘燕
杨 雪,刘 琳,赵 丹,张湘燕△
(1.贵州医科大学,贵阳 550000;2.贵州省人民医院,贵阳 550000)
肺炎克雷伯菌是临床最常见的条件致病菌之一,可引起肺炎、尿路感染、败血症、伤口感染、脑膜炎等[1]。近年来随着抗菌药物的广泛应用,尤其是第3代头孢菌素及碳青霉烯类的不合理使用,导致细菌出现严重的耐药性。2015年我国耐药细菌监测网提供的数据显示,全国肺炎克雷伯菌对碳青霉烯类的耐药率为7.6%,各省为0.5%~20.0%,均较2014年有所增加[2]。耐碳青霉烯类肺炎克雷伯菌(carbapenemresistant Klebsiella pneumoniae,CRKP)的感染增加了临床治疗的难度和患者预后风险。本研究收集37株CRKP菌株,检测相关耐药基因,采用多位点序列分型(MLST)对其进行分子流行病学研究,以揭示其耐药克隆的特征,以期为防治CRKP的传播提供参考依据。
1 材料与方法
1.1材料
1.1.1菌株来源 随机选取2016年2月至2017年2月贵州省人民医院临床分离到的CRKP,排除同一患者重复菌株,共37株。标本来源包括痰液、血液、中段尿、腹腔积液等。菌株经美国BD公司Phoenixtm-100全自动微生物分析系统检测。
1.1.2主要试剂 药敏纸片及M-H平板均购自英国Oxoid公司,0.5 mol/L的EDTA溶液为本室所有;2×Taq Master Mix和离心柱型细菌基因组 DNA 提取试剂盒均购自北京康维世纪有限公司。所有引物均由上海生工生物工程公司合成。
1.1.3主要仪器 全自动微生物鉴定仪购自美国BD公司,PCR扩增仪购自美国ABI公司,电泳仪购自北京六一仪器厂,凝胶成像仪购自美国Alpha Innotech公司。
1.2方法
1.2.1细菌鉴定、药敏试验及表型确证 采用Phoenixtm-100全自动微生物分析系统进行细菌鉴定,用微量肉汤稀释法测定13种抗菌药物的最低抑菌浓度(MIC),结果判定按美国临床实验室标准化研究所(CLSI)2015年版标准执行;质控菌株:大肠埃希菌ATCC 25922及K.pneumoniae标准菌株ATCC700603均购自卫生部临床检验中心。碳青霉烯类检测参照CLSI介绍使用改良霍奇实验。金属β-内酰胺酶检测采用亚胺培南+EDTA复合纸片的双纸片协同法。
1.2.2耐药基因检测 PCR法检测A类(KPC-2、GES)、B类(IMP、VIM、NDM-1)和D类OXA-48耐药基因。参照GeneBank中目的基因的标准序列及相关文献设计引物[3-4]。PCR反应体系为50 μL,包括2×Taq Master Mix 25 μL,上、下游引物各2 μL,DNA模板2 μL,灭菌蒸馏水19 μL。PCR反应条件:94 ℃预变性4 min,94 ℃变性1 min,退火温度1 min,72 ℃延伸1 min,38个循环;最后72 ℃延伸10 min,见表1。扩增的产物经1.5%的琼脂糖凝胶中经110 V电泳1 h后,用凝胶成像系统观察结果并拍照分析。PCR产物经上海生工生物工程公司完成测序,结果用 BLAST软件与Genbank上已公布的基因序列进行比对。

表1 耐药基因引物扩增序列及退火温度
1.2.3MLST实验 根据MLST网站(http://bigsdb.pasteur.fr/klebsiella/primers_used.h-tml)提供的引物序列,扩增肺炎克雷伯菌的7个管家基因,包括gapA、infB、mdh、pgi、phoE、rpoB及tonB,引物序列及退火温度见表2。PCR产物经琼脂糖凝胶电泳后拍照并送至上海生工生物工程公司测序。将测序所得到的结果在肺炎克雷伯菌MLST数据库官方网站 (https://pubmlst.org/bigsdb?db=pubmlst_mlst_seqdef&set_id=13&page=sequ-enceQuery)中进行BLAST比对,得到每个管家基因的等位基因编号,和数据库中已有序列不能完全比对的,经再次PCR及测序后确认为新的等位基因型,将序列及测序文件递交至数据库以获得新的命名。将7个管家基因所得到的编号按照gapA-infB-mdh-pgi-phoE-rpoB-tonB组合,与MLST数据库中已有管家基因谱进行比对,得到每株细菌对应的ST型。
1.2.4MEGA软件构建进化树、eBURST软件分析亲缘关系 将本研究获得的等位基因序列数据及ST信息使用MEGA软件构建进化树、eBURST软件进行亲缘关系分析。
1.2.5临床资料的分析 对37株细菌的临床资料进行收集整理,包括感染者性别、年龄、基础疾病、感染途径、住院病房、侵入性操作、感染CRKP前抗菌药物使用情况、预后情况、住院时间等,将其分成ST11型组及非ST11型组。
1.3统计学处理 采用SPSS17.0统计软件进行分析。年龄、住院时间的组间比较采用t检验,其他项目的组间比较采用方差分析,以P<0.05为差异有统计学意义。
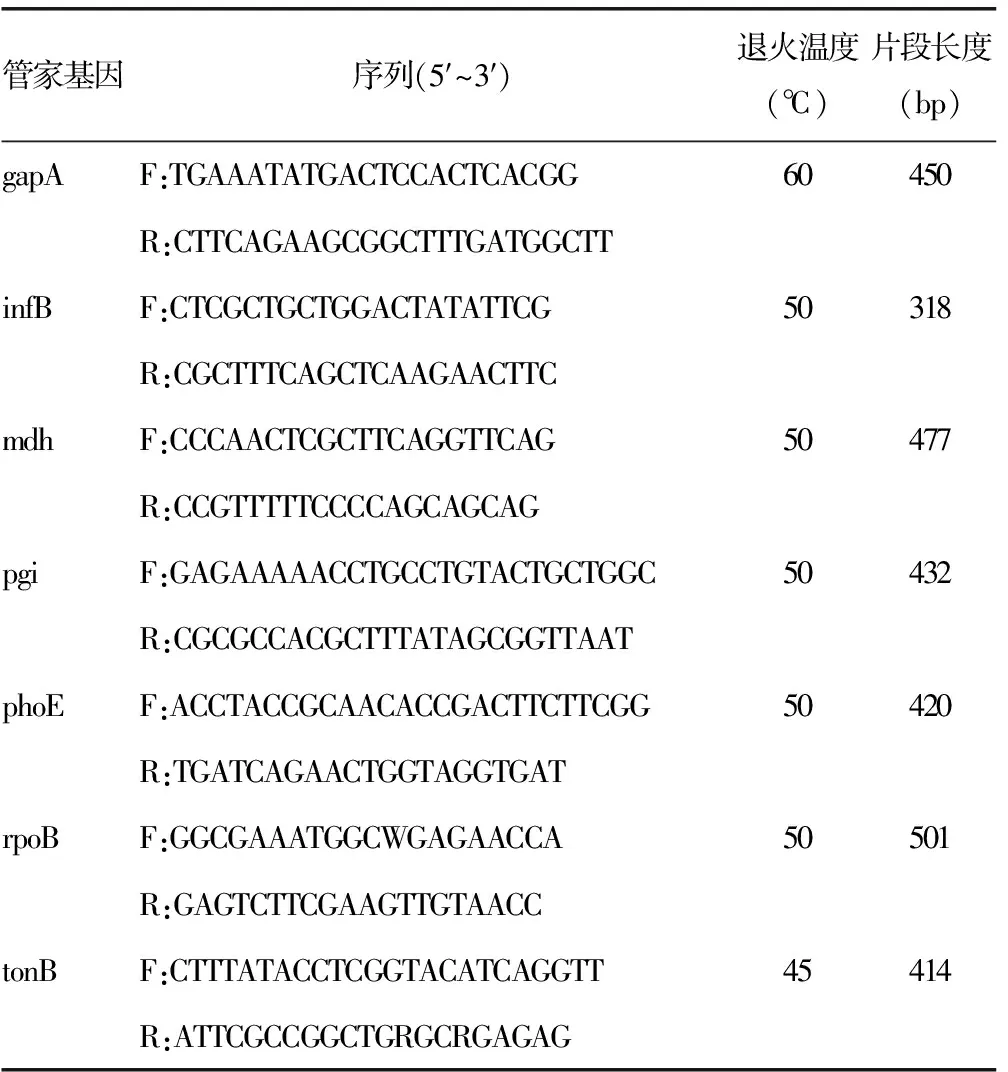
表2 管家基因引物扩增序列及退火温度
2 结 果
2.1药敏结果 37株CRKP对13种药物呈现不同程度的耐药,对碳青霉烯类药物(亚胺培南、美罗培南)呈现不同程度耐药,4~256 μg/mL。对氨基糖苷类的庆大霉素、第3代头孢菌素头孢曲松及临床上常用的抗菌药物联合β-内酰胺酶抑制剂(阿莫西林/克拉维酸、头孢哌酮/舒巴坦)均出现高水平耐药,较为特别的是,哌拉西林/他唑巴坦的耐药率为91.9%,32~256 μg/mL。而对阿米卡星耐药率稍低,为89.2%,1~256 μg/mL;对于阿奇霉素、左氧氟沙星、头孢西丁、头孢他啶耐药率均为97.3%。
2.2改良霍奇实验及金属酶检测结果 改良霍奇实验显示37株菌株均为阳性,阳性率100%,金属酶检测结果有3株阳性,阳性率为8.1%。
2.3耐药基因检测 PCR检测结果显示,37株菌株均含有KPC-2基因,检出率最高,有3株同时含有NDM-1基因,均未检出OXA-48、GES、IMP、VIM基因。所有阳性基因均经测序,BLAST软件与Genbank上已公布的基因序列进行比对,同源性均大于或等于99.0%,见图1、2。
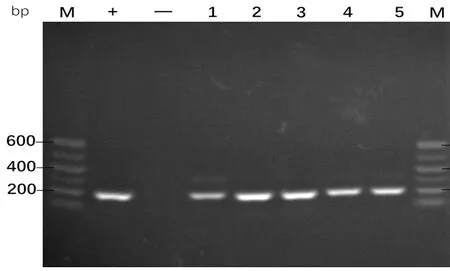
M:Marker600D;+:阳性对照;-:阴性对照;1~5:菌株编号
图1 KPC-2基因扩增产物电泳图
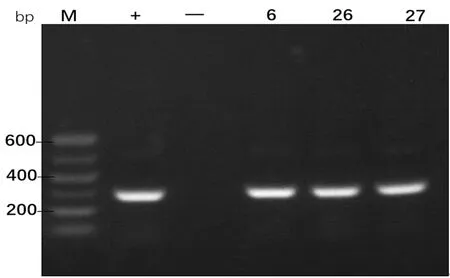
M:Marker600D;+:阳性对照;-:阴性对照;6、26、27:菌株编号
图2 NDM-1基因扩增产物电泳图
2.4MLST及分子遗传学结果分析 37株菌株经7个管家基因的PCR结果测序比对,可分为8个ST型,以ST11 25株为主,其次为ST789 4株、ST524 2株,ST35、ST29、ST1066及ST244各1株。有1个新的ST型已被PubMlst数据库确认收录并命名为ST2792(2株),见图3、4。
2.5临床资料分析 通过比较发现,ST11型组及非ST11型组在年龄、性别、感染途径、抗菌药物使用情况及住院时间等差异无统计学意义(P<0.05)。但总体而言,感染者年龄大、住院时间长、基础疾病多、入院后有较多的侵入性操作、使用多种抗菌药物治疗都是CRKP感染的潜在原因。

图中每一个点代表一个ST,蓝色代表各自克隆复合体的祖先,黄色代表各自克隆复合体亚群的原始型,标有红色圆圈切带有数字的点表示本次研究中所得到的ST型,其他黑色的点表示本次试验外数据库中的ST
图3 eBURST软件分析结果

图4 META分析构建37株菌株进化
3 讨 论
产生碳青霉烯酶是肺炎克雷伯菌对碳青霉烯类抗菌药物耐药的主要原因[5]。目前,碳青霉烯酶包括Ambler分类中的A、B、D类。A类主要水解除头霉素类以外的几乎所有β-内酰胺类抗菌药物,包括KPC、GES等;B类能水解青霉素类、头孢菌素类和碳青霉烯类抗菌药物,但不能水解氨曲南,主要包括IMP、VIM、NDM-1等;D类为苯唑西林水解酶,主要为OXA-48。本研究中37株菌株改良霍奇实验均为阳性,且经测序发现全部携带KPC-2基因,仅有3株同时携带NDM-1基因,未发现GES、IMP、VIM、OXA-48基因,说明KPC-2是CRKP对碳青霉烯类抗菌药物耐药最主要的原因,与国内外研究一致[6-8]。携带KPC-2基因的CRKP菌株对本研究中的13种抗菌药物均呈现了高水平耐药,耐药率基本达到90%以上,多耐药、高水平耐药情况严重;而同时携带KPC-2和NDM-1的菌株对包括氨曲南在内的13种抗生素全部耐药,但其耐药水平并未高于只携带KPC-2基因的耐药菌株,说明同时携带多种碳青霉烯酶基因可以扩大耐药范围,但并不一定提高耐药水平。目前已有研究证实,KPC-2编码的基因位于T4401的转座子上[9],毛彩萍等[10]在KPC-2基因阳性的菌株中检测到插入序列ISKpn6,且这两个基因连锁排列;这导致KPC-2基因可以通过转座子、插入序列等方式在不同菌株间传播[11],造成爆发流行。
本研究中37株菌株主要来源于重症监护室ICU(24.3%)、神经内科ICU(27.0%)、呼吸内科ICU(8.1%)、神经外科(10.8%)等;标本类型主要为痰(73%)和尿(11%),可能与患者基础疾病重,住院时间长,使用多种抗菌药物及进行侵入性操作有关[12]。利用MLST对37株菌株进行分型及建立进化树,得到包括ST11(25株)、ST524(2株)、ST35(1株)、ST1066(1株)、ST29(1株)、ST244(1株)、ST789(4株)及新发现的ST2792(2株)在内的8种ST型,其中ST11是最流行的克隆型,这也是我国CRKP最主要的ST型别[13]。本研究新发现的ST2792与最流行的ST11有高度的亲缘关系,仅有1个管家基因(infB)发生了变异,其携带的耐药基因也与ST11一致,且耐药性也无明显差别。将ST11型与非ST11型菌株的临床资料进行了对比发现,两组在年龄、基础疾病、使用抗生素种类及住院时间等方面差异无统计学意义(P>0.05),说明ST11型并没有感染CRKP的特有风险。这与DHAR等[14]的研究结果相似,可能由于这些ST型基本上同属一个克隆群,少数位点的变异并不会给临床感染带来特殊风险。
综上所述,本研究耐药基因检测发现KPC-2基因为最流行的耐药基因,同时含有NDM-1的菌株,也应引起重视。MLST结果显示ST11型为最流行的克隆型,新发现的ST2972与其为同一克隆系。在同一区域不同时间可能存在相同基因型或克隆群的CRKP的流行,临床及相关检验科室需加强监测,避免耐药菌株的播散。然而,本研究未对其他耐药机制进行检测,可能存在一定的局限性,因此还有待进一步研究和更多实验结果的支持。
[1]ZHANG W L,WANG X J,ZHAO X R,et al.Drug resistance analysis of Esche-richia coli and Klebsiella pneumoniae producing beta-ESBLs[J].Med Innov China,2015,12(30):105-108.
[2]CARSS.National report on bacterial drug resistance surveillance in 2015[EB/OL].[2015-12-12].http://news.medlive.cn/lab/in-foprogre-ss/show88818_246.Html.
[3]YAN J J,HSUEH P R,KO W C,et al.Metallo-beta-lactamases in clinical Pseudo-monas isolates in Taiwan and identification of VIM-3,a novel variant of the VIM-2 enzyme[J].Antimicrob Agents Chemother,2001,45(8):2224-2228.
[4]WALSH T R,WEEKS J,LIVERMORE D M,et al.Dissemination of NDM-l positive bacteria the New Delhi environment and its implications for human health:an envir-onmental point prevalence study[J].Lancet Infect Dis,2011,11(5):355-362.
[5]CHENG L,CAO X L,ZHANG Z F,et al.Clonal dissemination of KPC-2 produ-cing Klebsiella
pneumoniae ST11 clone with high prevalence of oqxAB and rmtB in a tertiary hospital in China:results from a 3-year period[J].Ann Clin Microbiol Antimicrob,2016,15(1):1-8.
[6]PITOUT J D,NORDMANN P,POIREL L.Carbapenemase-Producing Klebsiella pn-eumoniae,a Key Pathogen Set for Global Nosocomial Dominance[J].Antimicrob Agents Chemother,2015,59(10):5873-5884.
[7]WANG L H,WEI D D,WAN L G,et al.Diversity of the genetic environment of the blaKPC-2 gene among klebsiella pneumoniae clinical isolates in a chinese hospital[J].Microb Drug Resist,2016,22(1):15-21.
[8]LOGAN L K,WEINSTEIN R A.The epidemiology of carbapenem-resistant enterobacteriaceae:The impact and evolution of a global menace[J].Infect Dis,2017,215(1):S28-36.
[9]RIBEIRO V B,ANDRADE L N,LINHARES A R,et al.Molecular characterization of Klebsiella pneumoniae carbapenemase-producing isolates in southern Brazil[J].Med Microbiol,2013,62(23):1721-1727.
[10]毛彩萍,樊瑶,叶珍,等.耐药肺炎克雷伯菌β-内酰胺酶编码基因及KPC ISKpn6连锁检测[J].中华医院感染学杂志,2014,24(18):4424-4426.
[11]张冀霞,刘颖梅,陈宏斌,等.我国产碳青霉烯酶肠杆菌科细菌的基因型及流行病学研究[J].中华内科杂志,2014,53(2):116-120.
[12]豆清娅,邹明祥,李春辉,等.耐亚胺培南肺炎克雷伯菌的耐药机制研究[J].中华医院感染学杂志,2016,26(13):2906-2909.
[13]DAUTZENBERG M J,HAVERKATE M R,BONTEN M J,et al.Epidemic potential of Escherichia coli ST131 and Klebsiella pneumoniae ST258:a systematic re-view and meta-analysis[J].BMJ Open,2016,6(3):e009971.
[14]DHAR S,MARTIN E T,LEPHART P R,et al.Risk factors and outcomes for C-arbapenem resistant klebsiella pneumoniae isolation,stratified by its multilocus sequence typing:ST258 versus Non-ST258[J].Open Forum Infect Dis,2016,3(1):213-217.
