长链非编码RNA H19通过抑制CCND2促进膀胱癌细胞增殖的分子机制
2018-06-04程卓夫余淦王志华
程卓夫 余淦 王志华
膀胱癌的发病率在我国泌尿系统肿瘤中仍处于第一位,其研究和诊疗手段亦存在很大的局限性,迫切需要进一步研究其发病机制[1]。膀胱肿瘤的病理类型主要是尿路上皮癌,对于肌层浸润性膀胱癌一般选择根治性膀胱切除术,而非肌层浸润性膀胱肿瘤一般选择经尿道膀胱肿瘤电切术[2-3]。cyclin D2的编码基因位于12p13,称为细胞周期蛋白D2 (CCND2),在正常的二倍体细胞及Rb阳性肿瘤细胞中CCND 2的表达呈波动状态,其峰值在G1晚期。给G1期细胞注射微量CCND 2抗体,可使表达CCND 2的淋巴细胞停滞在G1期,说明CCND 2是细胞从G1期向S期转移所必须的,参与多种生理病理过程。miR-29c-3p作为小分子RNA可以与多种信使RNA及长链非编码RNA相互作用,进行转录后调控,参与多种肿瘤的病理生理进程。非编码RNA根据其转录本的长度或者大小的不同分为长链非编码RNA(long noncoding RNA,lncRNA)及短链非编码RNA,一般将长度大于200 nt的非编码RNA称作lncRNA,近些年来关于lncRNA在人类肿瘤中的研究越来越多[4],同时提出lncRNA可以作为内源性竞争性RNA调节肿瘤的进程。H19是其中研究较多的一种lncRNA,研究证实其参与了多种生理及病理过程,在人类疾病中发挥重要的调节作用,肿瘤性疾病包括膀胱癌、肾癌、胃肠道肿瘤、肺癌、乳腺癌及卵巢肿瘤[5-11]。本研究对H19作为内源性竞争性RNA促进膀胱癌细胞增殖的分子机制进行了相关探索。
材料与方法
一、基础材料及膀胱癌细胞系
常规细胞培养液、10%的胎牛血清(购自HYCLONE);膀胱癌细胞系(购自中科院上海细胞库);膀胱肿瘤组织(T)及肿瘤旁正常组织(N)(同济医院泌尿外科提供);实时定量PCR及转染相关试剂、miR-29c-3p类似物、抑制物以及相对应的对照(购自Invitrogen公司);其他相关试剂[分别购自Invitrogen公司、CST公司(抗体)以及Promega公司]。
二、实时定量逆转录PCR(RT-qPCR)
将膀胱癌细胞系T24和5637经实验处理后分为实验组(转染sh-H19质粒,sh-H19组)和对照组(转染阴性对照质粒,NC组),根据相关试剂使用方法提取RNA并进行后续PCR,分析相关目的基因的表达情况。
后述Western blot、细胞体外生长实验及流式细胞学检测中样本的实验分组处理方法同RT-qPCR。
三、荧光素酶报告系统
荧光素酶实验分为野生型组(Mutant)、阴性对照组(NC)、miR-29c-3p组及miR-29c-3p+H19组。将5637及T24细胞以1×105/孔密度接种于24孔板,第二天当细胞约70%融合时,利用Lipofectamine 2000共转染包含有CCND2的3′UTR的荧光素酶质粒和miR-29c-3p模拟物或抑制物,常规培养48 h后,按照双荧光素酶报告基因检测试剂盒说明在多功能亮度计上测定并分析。
四、Western blot
对样品进行分组处理后,用相对较弱的细胞裂解液NP-40裂解细胞,提取细胞总蛋白进行后续Western blot;制作丙烯酰胺凝胶,上蛋白样品进行电泳转膜封闭,CCND2抗体4 ℃孵育过夜,二抗37 ℃孵育1 h行HRE显色。
五、细胞体外生长实验
膀胱癌细胞系5637和T24正常培养后,经过实验分组进行预处理,分别种植于96孔板中,设计5个复孔,以24 h为1个检测时点,检测试剂应用MTS,按照无血清培养液和MTS 10∶1浓度正常孵育细胞2 h后检测吸光度进行数据分析,检测细胞增殖情况。
六、流式细胞学检测细胞周期
膀胱癌细胞系5637和T24正常培养后,经过实验分组进行预处理,于各组中取得50 000个细胞,先用冰乙醇4 ℃过夜固定细胞,磷酸盐缓冲液洗涤细胞后每个样品中加入0.5 ml磷酸盐缓冲液及50 μl溴化乙锭,之后再加入100 μg/ml的RNase A,0.2%的Triton X-100,4 ℃孵育30 min进行流式分析。
七、统计学方法
应用SPSS 16.0软件进行数据分析。两组之间符合正态分布的数据采用t检验,而组别较多情况下两两比较采用方差分析。以P<0.05为差异有统计学意义。
结 果
一、膀胱肿瘤组织中H19和miR-29c-3p的表达
RT-qPCR检测结果显示膀胱肿瘤新鲜组织中lncRNA H19呈高表达, miR-29c-3p呈低表达,二者在肿瘤组织(T)及癌旁组织(N)中的表达差异均具有统计学意义(P<0.01)。见图1。

A:H19;B:miR-29c-3p
图1 膀胱肿瘤及癌旁组织中H19和miR-29c-3p的相对表达水平
二、抑制细胞中H19的表达后膀胱癌细胞系5637和T24细胞生物学行为的变化
5637和T24细胞中转染sh-H19后,相对于NC组,实验组细胞的增殖受到了明显的抑制,抑制率分别为40%和30%左右。见图2A、2B。转染sh-H19后,相对于NC组G1期细胞比例降低,同时S期细胞增加,G2/M期细胞比例降低,余2期细胞基本没有明显变化。见图2C、2D。
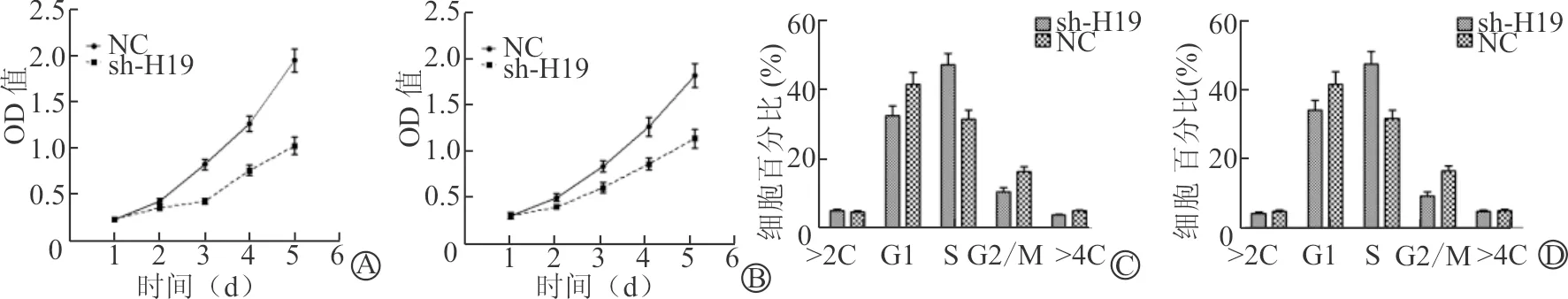
A:下调H19后5637细胞的增殖曲线;B:下调H19后T24细胞的增殖曲线;C:下调H19后5637细胞周期的变化;D:下调H19后T24细胞周期的变化
图2 抑制细胞中H19的表达后膀胱癌细胞系5637和T24细胞生物学行为的变化
三、荧光素酶报告靶标验证结果
miR-29c-3p种子序列及与H19按照碱基互补配对原则的配对序列见图3A。sh-H19可以部分回复miR-29c-3p的荧光素酶信号抑制作用(P<0.01),表明可以靶向CCND2的miR-29c-3p可能同时靶向H19。通过共转染降低H19的表达的荧光素酶报告系统还提示外源降低H19的表达可以部分回复miR-29c-3p的荧光素酶信号抑制作用,而在突变型3′UTR中则无影响。在膀胱癌两株细胞系中得到相同的结果。见图3B。在膀胱癌5637和T24细胞中过表达miR-29c-3p后,H19表达下降(P<0.01)。见图3C。膀胱癌细胞系5637和T24中转染sh-H19后,相较NC组,实验组H19的表达显著下降(P<0.01)。见图3D。
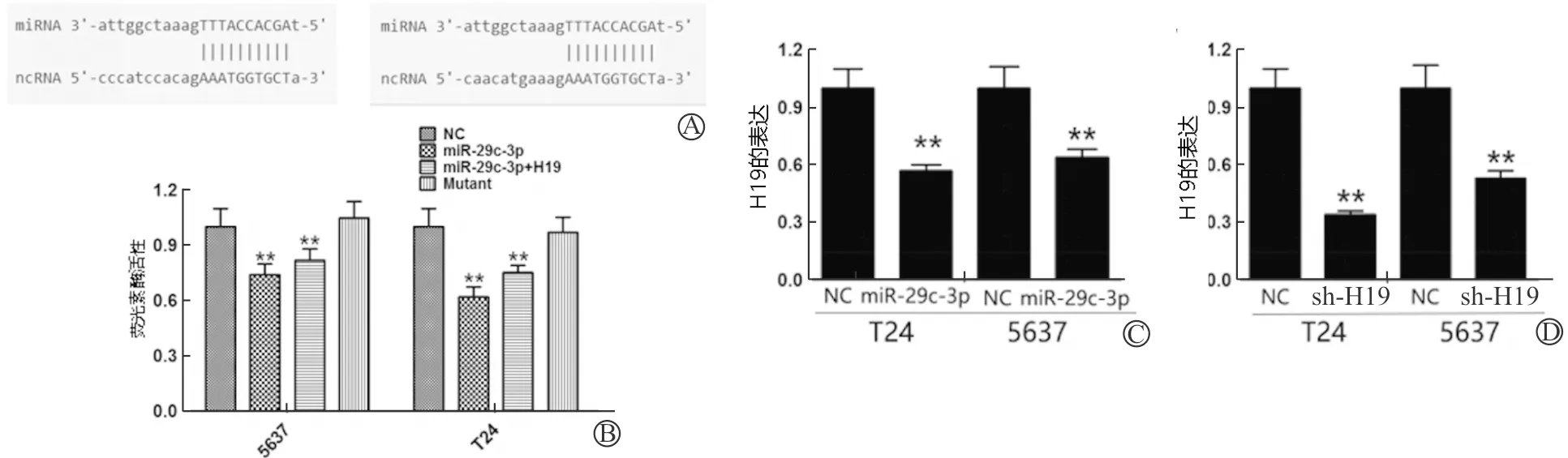
A:配对的种子序列及两处可以相互结合作用的位点;B:荧光素酶报告实验;C:膀胱癌细胞系中转染miR-29c-3p后H19的表达变化;D:膀胱癌细胞系中转染sh-H19后H19的表达变化
图3 荧光素酶报告靶标验证结果
四、膀胱癌组织中CCND2、H19和miR-29c-3p表达关联分析
膀胱癌组织中CCND2、H19和miR-29c-3p表达关联分析显示,H19与miR-29c-3p的基础表达呈负相关关系(r2=0.331 1,P=0.019 7),见图4A; CCND2与miR-29c-3p的基础表达呈负相关关系(r2=0.384 2,P=0.010 4),见图4B;CCND2和H19的基础表达呈正相关关系(r2=0.786 7,P<0.01),见图4C;抑制H19的表达后,膀胱癌细胞系T24和5637中CCND2的表达减少,见图4D。

A:H19与miR-29c-3p的表达相关性;B:CCND2与miR-29c-3p的表达相关性;C:CCND2与H19的表达相关性;D:抑制H19的表达后CCND2的表达
图4 膀胱癌中CCND2、H19和miR-29c-3p表达关联分析
讨 论
肿瘤的发生通常是多个分子事件共同作用的结果,多年来,关于肿瘤的研究(包括膀胱肿瘤)取得了一些重大进展,但是进展期膀胱癌的治疗效果还远不及人们的要求,这就需要开展进一步更深层次的研究[12-13]。基因组学时代最意想不到的发现之一是基因组的非蛋白质编码区域能广泛转录RNA[14]。lncRNA被定义为长度大于200 nt且无蛋白编码潜力的转录物,它不具有开放阅读框,无法翻译出有功能的蛋白质[15]。许多lncRNA在独特的组织或癌症中特征性表达。尽管总体序列相似性较小,lncRNA常有进化上保守的功能、二级结构和微同源区段[16]。某些lncRNA在肿瘤中特异性表达,这给lncRNA研究提供了必要前提,且这些新的转录分子可能在肿瘤发生发展中发挥重要的调控作用。不同的lncRNA其序列长度及结构迥异,并可在表观遗传水平、转录水平、转录后水平和翻译水平等多个层次调控靶标基因的表达水平及功能状态。lncRNA可以与蛋白质相互作用以调节蛋白质功能、调节蛋白-蛋白相互作用或指导蛋白定位于细胞区室[17]。lncRNA是近年来肿瘤研究的重点,已经取得了大量的研究成果,lncRNA H19是其中研究较多的一种lncRNA,H19参与了多种人类肿瘤的发生、进展和转移,可以作为肿瘤进展预测分子,甚至可以作为新的肿瘤标志物[18-20]。关于竞争性内源性RNA此前已有相关报道[21]。但H19作为内源性竞争性RNA在膀胱癌中的研究尚未见报道,本研究初步探讨了lncRNA H19在膀胱癌发生发展中的分子机制。首先我们在膀胱癌中分别检测了H19和miR-29c-3p的表达,发现H19在膀胱癌中上调,miR-29c-3p 在膀胱癌中下调;其后在膀胱癌细胞系5637和T24中进行了相关功能实验,验证了H19在膀胱癌中的肿瘤促进作用;通过荧光素酶报告实验验证了miR-29c-3p可能作为一个重要的相互作用小分子RNA,共同调节H19和CCND2的表达从而参与膀胱癌的发生与发展;最后,基础表达相关分析提示H19可以通过miR-29c-3p途径促进CCND2的表达,参与膀胱癌的发生发展。本项目尚处于实验阶段,无体内研究数据,研究内容相对粗放,后续研究会涉及到动物实验内容,同时完善更多H19/miR-29c-3p/CCND2途径相关内容的研究,并进一步研究H19是否可以通过其他生物学机制参与膀胱肿瘤的发生与发展。
综上,H19不仅可以作为癌基因还可以作为内源性竞争性RNA参与膀胱肿瘤的进展,这给临床膀胱肿瘤的诊疗提供了新的可能途径。
[1] Ferlay J, Shin HR, Bray F, et al. Estimates of worldwide burden of cancer in 2008: GLOBOCAN 2008[J]. Int J Cancer,2010,127(12):2893-2917.
[2] Stenzl A, Cowan NC, De Santis M, et al. Treatment of muscle-invasive and metastatic bladder cancer: update of the EAU guidelines[J]. Eur Urol,2011,59(6):1009-1018.
[3] Bertz S, Otto W, Denzinger S, et al. Combination of CK20 and Ki-67 immunostaining analysis predicts recurrence, progression, and cancer-specific survival in pT1 urothelial bladder cancer[J]. Eur Urol,2014,65(1):218-226.
[4] Akhtar A, Fuchs E, Mitchison T, et al. A decade of molecular cell biology: achievements and challenges[J]. Nat Rev Mol Cell Biol,2011,12(10):669-674.
[5] Atala A. Re: Long non-coding RNA H19 increases bladder cancer metastasis by associating with EZH2 and inhibiting E-cadherin expression[J]. J Urol,2013,190(6):2306.
[6] Luo M, Li Z, Wang W, et al. Upregulated H19 contributes to bladder cancer cell proliferation by regulating ID2 expression[J]. FEBS J,2013,280(7):1709-1716.
[7] Hu Q, Wang YB, Zeng P, et al. Expression of long non-coding RNA (lncRNA) H19 in immunodeficient mice induced with human colon cancer cells[J]. Eur Rev Med Pharmacol Sci,2016,20(23):4880-4884.
[8] Wu KF, Liang WC, Feng L, et al. H19 mediates methotrexate resistance in colorectal cancer through activating Wnt/beta-catenin pathway[J]. Exp Cell Res,2017,350(2):312-317.
[9] Wang Q, Cheng N, Li X, et al. Correlation of long non-coding RNA H19 expression with cisplatin-resistance and clinical outcome in lung adenocarcinoma[J]. Oncotarget,2017,8(2):2558-2567.
[10] Si X, Zang R, Zhang E, et al. LncRNA H19 confers chemoresistance in ERalpha-positive breast cancer through epigenetic silencing of the pro-apoptotic gene BIK[J]. Oncotarget,2016,7(49):81452-81462 .
[11] Sun D, Li X, He Y, et al. YAP1 enhances cell proliferation, migration, and invasion of gastric cancer in vitro and in vivo[J]. Oncotarget,2016,7(49):81062-81076.
[12] Li HT, Duymich CE, Weisenberger DJ, et al. Genetic and Epigenetic Alterations in Bladder Cancer[J]. Int Neurourol J,2016,20(Suppl 2):S84-94.
[13] Bidnur S, Savdie R, Black PC. Inhibiting Immune Checkpoints for the Treatment of Bladder Cancer[J]. Bladder Cancer,2016,2(1):15-25.
[14] Iyer MK, Niknafs YS, Malik R, et al. The landscape of long noncoding RNAs in the human transcriptome[J]. Nat Genet,2015,47(3):199-208.
[15] Huarte M. The emerging role of lncRNAs in cancer[J]. Nat Med,2015,21(11):1253-1261.
[16] Quinn JJ, Zhang QC, Georgiev P, et al. Rapid evolutionary turnover underlies conserved lncRNA-genome interactions[J]. Genes Dev,2016,30(2):191-207.
[17] Wang KC, Chang HY. Molecular mechanisms of long noncoding RNAs[J]. Mol Cell,2011,43(6):904-914.
[18] Jing W, Zhu M, Zhang XW, et al. The Significance of Long Noncoding RNA H19 in Predicting Progression and Metastasis of Cancers: A Meta-Analysis[J]. Biomed Res Int,2016:5902678.
[19] Li T, Mo X, Fu L, et al. Molecular mechanisms of long noncoding RNAs on gastric cancer[J]. Oncotarget,2016,7(8):8601-8612.
[20] Guo LL, Song CH, Wang P, et al. Competing endogenous RNA networks and gastric cancer[J]. World J Gastroenterol,2015,21(41):11680-11687.
[21] Tay Y, Rinn J, Pandolfi PP. The multilayered complexity of ceRNA crosstalk and competition[J]. Nature,2014,505 (7483):344-352.
