茶树CsLhcb4基因的电子克隆与生物信息学分析
2018-05-30梅菊芬徐德良汤茶琴周静峰邵元海
梅菊芬, 徐德良, 汤茶琴, 周静峰, 邵元海
(无锡市茶叶品种研究所/江苏省茶树种质改良与推广工程技术中心,江苏无锡 214125)
光合作用产物是人类赖以生存和发展的基础,光捕获是植物光合作用的重要过程。在高等植物中,捕光叶绿素a/b结合蛋白(light harvesting chlorophyll a/b binding protein,LHC)是光捕获中的重要功能蛋白,其与叶绿素a/b形成复合体,将光能迅速传到光系统Ⅰ(PSⅠ)和光系统Ⅱ(PSⅡ)的反应中心,使光能转化为化学能,促进光合反应的进行[1]。PSⅠ和PSⅡ都含有各自的LHC,即LHCⅠ和LHCⅡ。其中,LHCⅡ在类囊体膜上的含量最为丰富,它所结合的叶绿素约占类囊体膜上色素量的50%,目前研究较多,也是结构较清楚的一类膜蛋白[1-3]。LHCⅡ被认为是一类结构相似、进化相关、由核基因家族(Lhcb)编码的蛋白,与色素所形成的色素蛋白复合体家族,含有保守的叶绿素结合(chlorophyllbinding,CB)结构域,它们除进行光能的捕获与传递外,还广泛参与了激发能在PSⅠ和PSⅡ之间的调节与分配、类囊体膜结构的维持、光保护以及对各种环境的应答等[4]。LHCⅡ主要由6种色素蛋白复合体组成,分别由Lhcb1、Lhcb2、Lhcb3、Lhcb4、Lhcb5和Lhcb6编码,其组成了4种蛋白亚复合物,LHCⅡa、LHCⅡb、LHCⅡc、LHCⅡd。其中,LHCⅡb为主体LHCⅡ,包括LHCB1、LHCB2、LHCB3;其他3种合称为微量LHCⅡ,分别为LHCⅡa(LHCB4)、LHCⅡc(LHCB5)、LHCⅡd(LHCB6)[1]。
LHCB4别称CP29,是PSⅡ内部天线叶绿素a/b结合亚复合体,其为次要捕光色素蛋白,较其他蛋白复合体更接近PSⅡ核心复合物,其编码基因为Lhcb4[1]。目前已从拟南芥、籼稻、杨树、菠菜、银杏等多种高等植物中克隆了Lhcb4基因,并研究了其在各种生态环境下的表达情况[5-10]。
近年,茶树育种目标倾向叶色多样化,白化或紫化等特异叶色品种的选育成为茶树新品种选育的热点。研究表明,光是诱导茶树花青素形成的主要原因[11],光合作用与花青素的合成之间是否存在一定关系有待于研究。本研究从紫芽中筛选到下调表达基因片段,Blastx比对为Lhcb4的部分基因片段。以该基因片段为探针,在NCBI中Blast检索茶树中与探针序列相似性较高的ESTs,然后利用DNAstar进行序列拼接,得到茶树叶绿素a/b结合蛋白CP29基因(CsLhcb4),利用生物信息学技术预测该基因编码蛋白的理化性质和结构分析,为后续研究CsLhcb4与花青素代谢的关系提供参考。
1 材料与方法
1.1 材料
笔者所在研究组从紫芽中筛选的差异表达基因片段(D-ZY-5),经Blastx比对为Lhcb4的部分基因片段。
1.2 方法
1.2.1 茶树CsLhcb4基因的电子克隆 以差异筛选的EST基因片段为查询探针,利用NCBI中的Blastn工具,搜索茶树EST数据库(taxid:4442),得到具有高度同源性(选择相似性大于等于90%,覆盖率大于100 bp)的EST序列,再利用DNAStar进行重叠区域拼接和组装,构建重叠序列群。以重叠序列群为信息标签,进一步检索比对,搜索高度同源序列,并拼接组装,直至没有发现高度同源序列。在非冗余数据库中进行搜索,确认新基因序列,获得茶树CsLhcb4的cDNA序列。
1.2.2 茶树CsLhcb4基因及蛋白质序列的生物信息学鉴定 以NCBI的ORFfinder和Conserved Domain Database功能对扩增的基因序列进行蛋白质翻译及其保守结构域预测。以Expasy的在线分析软件对蛋白序列进行理化性质(ProtParam tool)、疏水性(ProtScale)、二级结构(SOPMA)和三维结构分析预测(SWISS-MODEL),同时利用在线分析工具分析无序区域(foldindex)、亚细胞定位(TargetP 1.1 Server)及磷酸化位点预测(NetPhos 3.1 Server)。利用NCBI的PSI-BLAST进行蛋白序列同源比对,利用DNAman软件进行同源蛋白的氨基酸多序列比对并用Mega构建系统进化树。
2 结果与分析
2.1 茶树CsLhcb4基因克隆及序列分析
以筛选的EST基因片段为查询探针,利用Blastn对茶树EST数据库进行搜索,得到2个高度同源的EST序列(FS954792.1和JK993206.1),利用DNAStar对序列进行重叠区域拼接和组装,在以拼接的重叠序列群进行检索,得到高度同源的EST序列(FS960161.1),重复拼接得到茶树CsLhcb4基因cDNA序列,长度为1 085 bp。经ORFfinder分析,该基因包含1个858 bp的ORF,编码1个含285个氨基酸的蛋白质。
2.2 茶树CP29蛋白保守结构域预测
利用NCBI在线分析软件Conserved Domain Database(CDD)对蛋白进行结构域分析,结果(图1)显示,在茶树叶绿素a/b连接蛋白CP29的氨基酸序列第13个和第285个之间存在光系统Ⅱ光捕获复合体结构域(PLN00187),编码蛋白属于叶绿素a/b连接蛋白超家族成员。

2.3 茶树CP29蛋白同源比对及序列进化分析
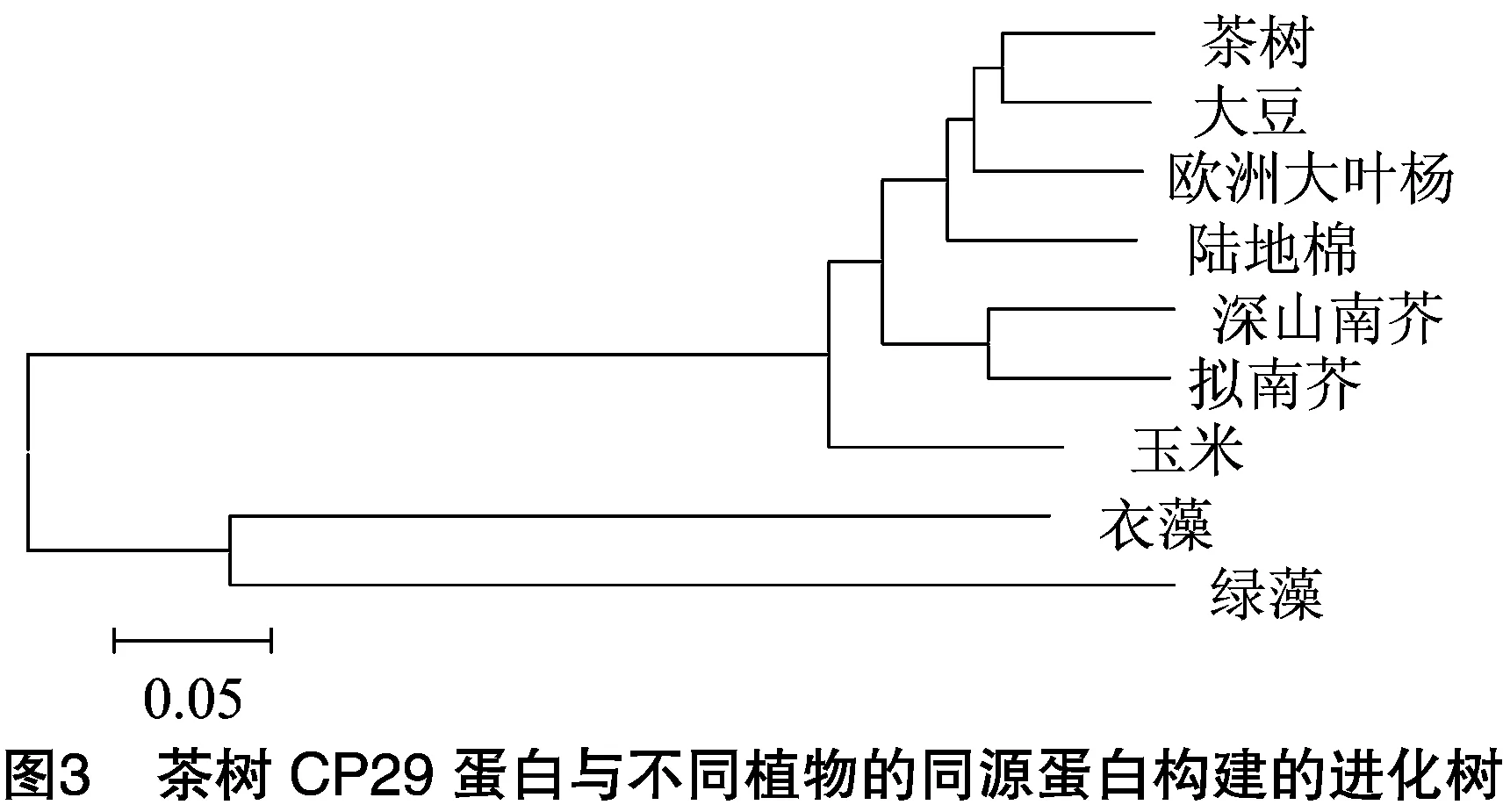
在NCBI中的Blastp中采用PSI-Blast方法在refseq数据库中进行氨基酸序列同源比对,结果显示,有478个蛋白与茶树CP29蛋白同源,一致性最大为89%,最小为32%,这些蛋白包括假定未知的蛋白、预测的蛋白、功能已鉴定的蛋白,它们都有叶绿素a/b结合蛋白超家族结构域(PLN00187),相对保守。在功能已鉴定的蛋白中,茶树CP29蛋白与陆地棉CP29.1(NP001314598.1)、欧洲大叶杨CP29(XP002323575.1)、大豆CP29(NP001304343.1)、拟南芥(NP187506.1)、深山南芥(XP002873005.1)、玉米(NP001105502.1)、衣藻(XP001697193.1)、绿藻(XP005646225.1)一致性分别是87%、87%、87%、83%、83%、82%、51%、47%,这表明植物叶绿素a/b连接蛋白CP29序列在不同植物之间具有一定程度的序列分化。利用DNAman软件将茶树与这8个植物的CP29蛋白进行多重比较分析,序列一致性为74.19%(图2)。Mega构建系统发育树表明,高等植物的CP29区别于藻类,在进化距离上较近;茶树与大豆、欧洲大叶杨和陆地棉进化距离最近,聚为一类(图3)。

2.4 茶树CP29蛋白理化性质分析
CsLhcb4编码的蛋白质分子式为C1 427H2 180N366O407S4,分子量为31.1 ku,理论等电位点5.79。亮氨酸(Leu)和丙氨酸(Ala)占比最大,分别为11.2%和10.9%,芳香族氨基酸[苯丙氨酸(Phe)、酪氨酸(Tyr)、色氨酸(Trp)]33个,占比11.6%。负电荷残基[天冬氨酸(Asp)+谷氨酸(Glu)]数为31个,正电荷残基[精氨酸(Arg)+赖氨酸(Lys)]数为27个,脂溶系数为84,总平均疏水性为-0.099。不稳定系数31.69,该蛋白为稳定蛋白。无序区域预测结果表明,其存在1个无序区域1~50位氨基酸处,不可折叠性0.194,对蛋白折叠、表达水平干扰较小。
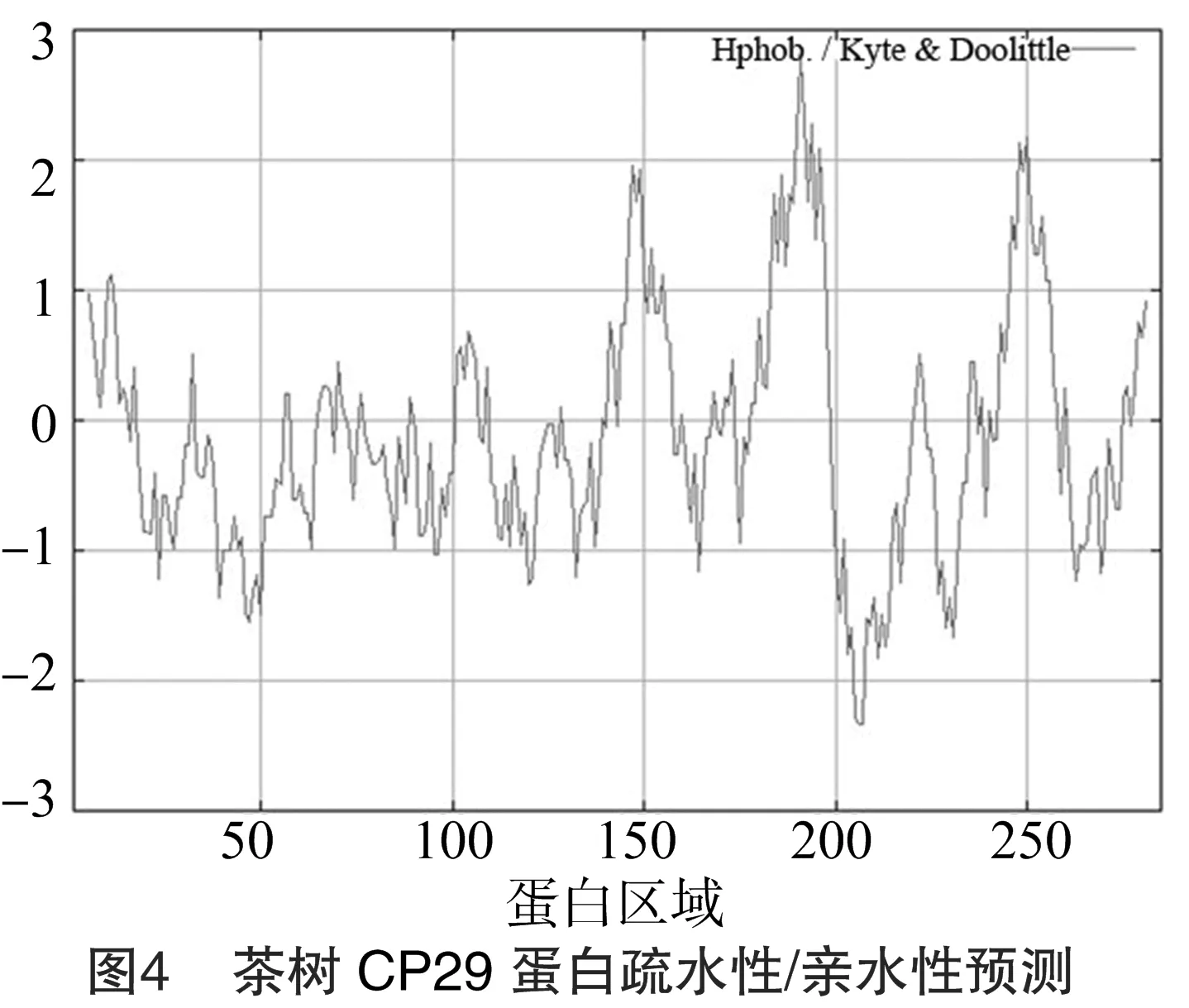
2.5 茶树CP29蛋白疏水性分析
用ProtScale软件对蛋白质进行疏水性/亲水性分析(图4),正值越大表示越疏水,负值越大表示越亲水,介于+0.5~-0.5之间的主要为两性氨基酸。结果表明,Glu191疏水性最强,疏水性参数为2.789;Leu206和Asp207亲水性最强,疏水性参数为-2.333。蛋白中约在183~197区域疏水性最强,其次是245~256、144~157区域具有较强的疏水性;在199~219区域亲水性最强,其次在38~54、227~232区域具有较强的亲水性。该蛋白的亲水区域大于疏水区域,预测该蛋白为亲水蛋白,与ProtParam分析结果一致。
2.6 茶树CP29蛋白亚细胞定位预测
结合TargetP亚细胞定位预测(表1),蛋白定位于叶绿体。CP29为植物PSⅡ3种次要捕光色素蛋白之一,蛋白亚细胞定位结果符合其功能预测。

表1 茶树CP29蛋白亚细胞定位
2.7 茶树CP29蛋白磷酸化修饰位点及分析
磷酸化是蛋白翻译前修饰的最常见的形式之一,在调节从基因表达到信号和代谢调控等所有细胞功能中都起作用[4]。CP29的磷酸化可能与植物的抗逆有关[12-13],其磷酸化可能是调节光能的捕获及激发能向反应中心传递的一种方式,也是一种新的光保护机制。利用NetPhos 3.1预测茶树CP29蛋白磷酸化位点,结果(表2)显示,CP29蛋白磷酸化位点有23个,其中Thr 9个,丝氨酸(Ser)10个,Tyr 4个。
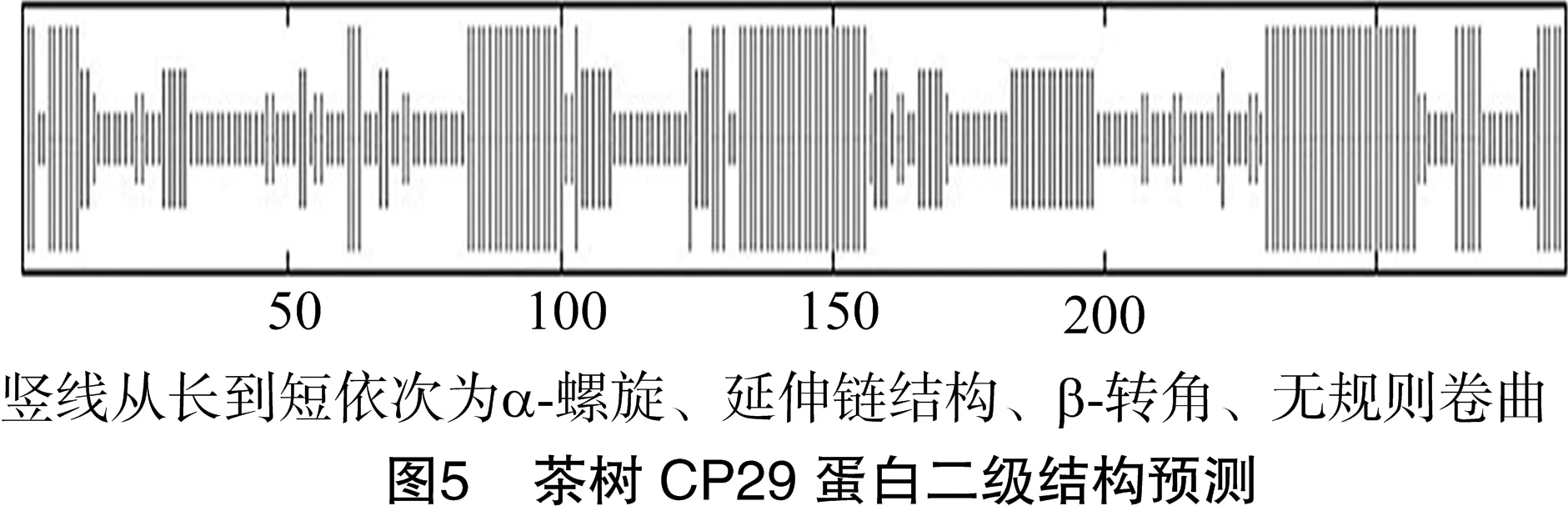
2.8 茶树CP29蛋白二级结构预测和分析
利用SOPMA在线预测蛋白的二级结构(图5),α-螺旋和无规则卷曲是其主要二级结构,其中115个氨基酸可能形成无规则卷曲,占40.35%,97个氨基酸可能形成α-螺旋,占34.04%,49个氨基酸可能形成延伸链,占17.19%,24个氨基酸可能形成β-转角,占8.42%,不含其他二级结构。
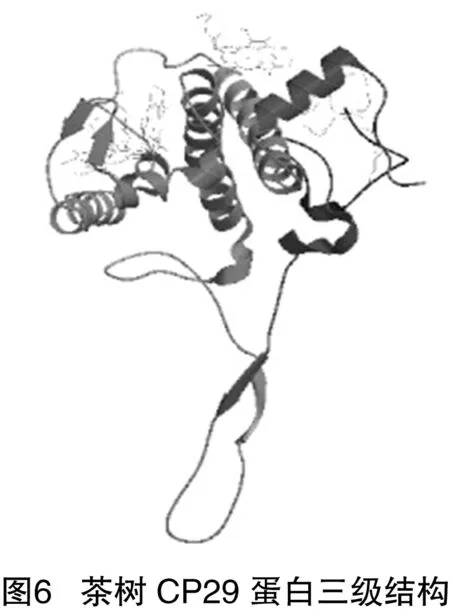
2.9 茶树CP29蛋白三级结构建模及分析
利用swiss-model在线对其进行建模,从PDB(蛋白质结构数据库)中获得63个同源蛋白序列,选择其中的3jcu.1菠菜PSⅡ-LHCⅡ超分子复合物,与靶标序列比对结果作模板,其序列一致性为88.51%,获得其三级结构(图6)。三级结构从N端开始形成8端螺旋结构,至C端形成较长的延伸链。建模结果估值(OMQE)为0.75,介于0~1之间,接近1,建模结果处于接受水平。但是QMEAN4值为-3.95,其理想值为1,因此建模结果并不十分理想。
3 讨论与结论
LHC蛋白是高等植物光合作用过程中的重要功能蛋白,本研究以在紫芽中下调表达的基因片段(D-ZY-5)为查询探针,通过电子克隆的方式得到编码茶树CP29蛋白基因的cDNA序列,命名为CsLhcb4,长度为1 085 bp,ORF为 858 bp,编码285个氨基酸,分子量为31.1 ku。蛋白保守结构域分析表明其编码氨基酸序列第13个和第285个之间存在光系统Ⅱ光捕获复合体结构域(编号PLN00187),属于叶绿素a/b连接蛋白超家族成员。亚细胞定位于叶绿体上,与蛋白预测的功能相吻合。
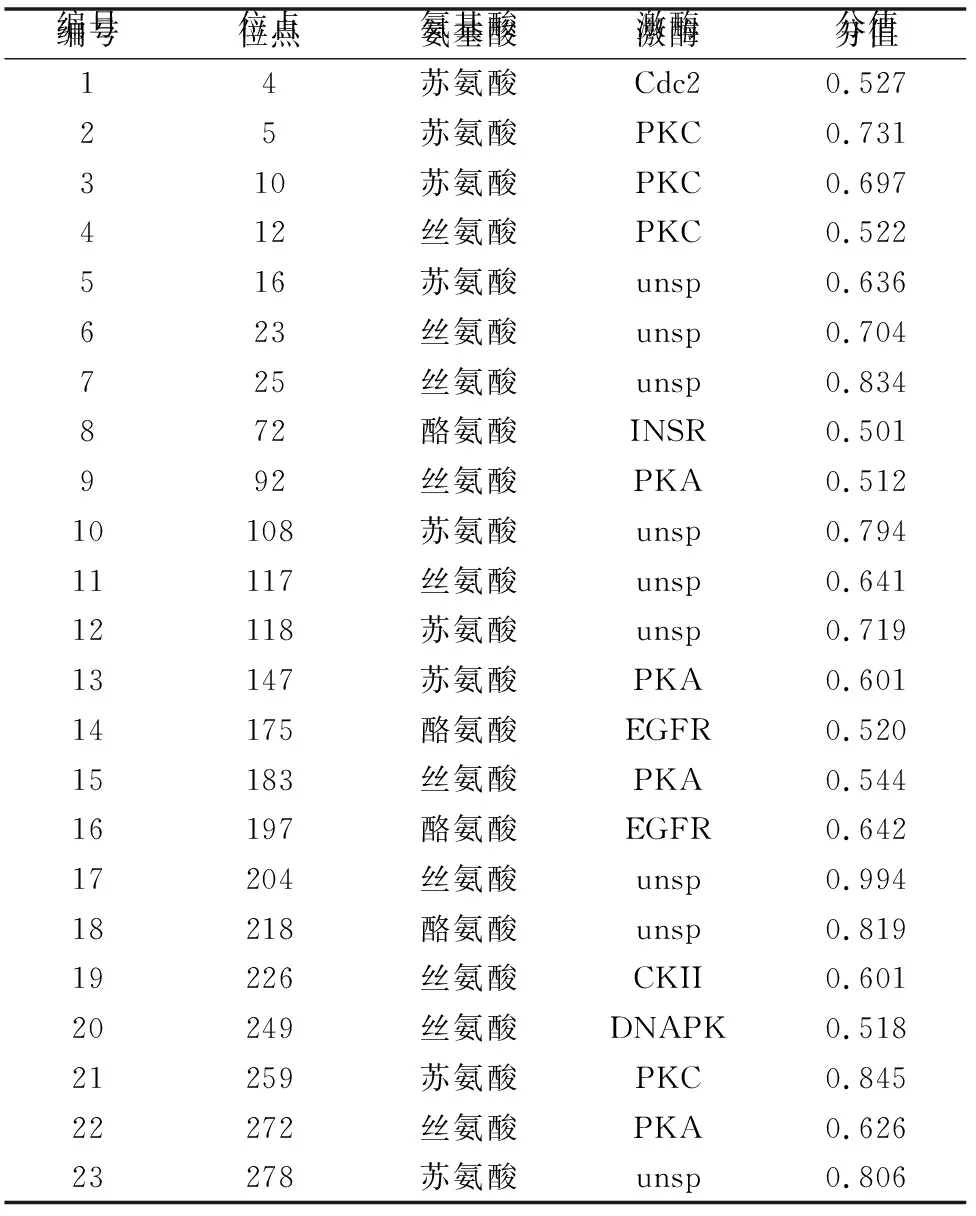
表2 茶树CP29蛋白磷酸化修饰位点预测
氨基酸同源序列分析表明,克隆的茶树CsLhcb4编码氨基酸与这些序列相似性为32%~89%,总体上相对保守,属于叶绿素a/b结合蛋白CP29。构建进化树表明,茶树与高等植物陆地棉、绿豆和烟草进化距离最近,聚为一类。
二级结构分析表明,α-螺旋和无规则卷曲是茶树CP29的主要二级结构,α-螺旋是一种稳定结构,能维持蛋白的结构稳定性。三维结构分析表明,基于茶树CP29与菠菜PSⅡ-LHCⅡ超分子复合物(3jcu.1)序列同源性为88.51%,利用Swiss-Model中的同源建模法构建茶树CP29的三维结构,构建模型为单体形式。
氨基酸序列亲水性/疏水性分析结果显示,该蛋白的亲水区域大于疏水区域,预测该蛋白为亲水蛋白。不稳定系数为31.69,该蛋白为稳定蛋白,能稳定保持其与叶绿素a/b形成的复合体,传输光能,促进光合作用。
在高等植物中,CP29在LHCⅡ中分子量最大,结构最简单,光谱值相对简单,结合6个叶绿素a、2个叶绿素b和2个类胡萝卜素分子,其结构及功能研究相对明确[14-15]。研究表明,CP29是植物光系统Ⅱ天线蛋白不可或缺的结构组成,在非光化学淬灭(NPQ)作用中至关重要,其缺失会造成光系统Ⅱ结构变化,并影响光保护作用[16-18]。CP29的磷酸化可能与植物的抗逆性有一定关系,低温、强光等逆境能诱导CP29的磷酸化,其磷酸化可在多个位点发生,并通过可逆磷酸化调节和决定LHCⅡ在2个光系统之间的亲和性和LHCⅡ的状态迁移[19]。
本研究通过电子克隆方式得到在高花青素茶树中下调表达的叶绿素a/b结合蛋白CP29基因,通过生物信息学分析预测其理化性质、二级结构和三维结构,为进一步研究CP29在高花青素茶树代谢中的作用奠定了基础。CP29是否通过光保护作用或磷酸化等来影响花青素的代谢有待于进一步研究。
捕光色素蛋白有14类,最初序列上的相似性却表明这些色素蛋白都起始于1个相同的原始基因,原始基因能够编码1个含跨膜螺旋的蛋白[4]。高等植物的PSⅡ外周光捕获蛋白都在非光化学淬灭(NPQ)下发挥其作用,起到光保护作用[17]。因此,在CP29的研究基础上继续对高花青素茶树捕光色素蛋白的表达及功能进行研究具有一定的意义。
:
[1]孙钦秒,冷 静,李良璧,等. 高等植物光系统Ⅱ捕光色素蛋白复合体结构与功能研究的新进展[J]. 植物学通报,2000,17(4):289.
[2]Labate M T,Ko K,Ko Z W,et al. Constitutive expression of peaLhcb1-2 in tobacco affects plant development,morphology and photosynthetic capacity[J]. Plant Molecular Biology,2004,55(5):701-714.
[3]李晓鹏. 杜林方和梁厚果.菠菜光系统Ⅱ天线组分CP29的分离及其性质[J]. 生物化学与生物物理学报,1999,31(6):631-636.
[4]罗 玲. 捕光色素蛋白复合物的研究进展[J]. 现代农业科技,2008(22):270-273,276.
[5]Xu Y H,Liu R,Yan L,et al. Light-harvesting chlorophyll a/b-binding proteins are required for stomatal response to abscisic acid inArabidopsis[J]. Journal of Experimental Botany,2012,63(3):1095-1106.
[6]袁定阳,余 东,谭炎宁,等. RT-PCR克隆籼稻叶绿素a/b结合蛋白基因全长cDNA及序列的in silico分析[J]. 基因组学与应用生物学,2012,31(2):173-177.
[7]王欢利,刘新亮,郁万文,等. 银杏叶绿素a/b结合蛋白基因(GbLhcb4)及其启动子克隆[J]. 中南林业科技大学学报,2015,35(5):114-121.
[8]Klimmek F,Sjödin A,Noutsos C,et al. Abundantly and rarely expressed Lhc protein genes exhibit distinct regulation patterns in plants[J]. Plant Physiology,2006,140(3):793-804.
[9]Pan X,Li M,Wan T,et al. Structural insights into energy regulation of light-harvesting complex CP29 from spinach[J]. Nature Structural & Molecular Biology,2011,18(3):309-315.
[10]邹 智,安 锋,杨礼富,等. 大戟科Lhcb基因家族的全基因组鉴定、分类与进化分析[J]. 中南林业科技大学学报,2013,33(12):46-52.
[11]张泽岑,王能彬. 光质对茶树花青素含量的影响[J]. 四川农业大学学报,2002,20(4):337-339.
[12]刘文娟,袁 澎,林宏辉. 高等植物的光系统Ⅱ蛋白磷酸化机制及其对环境胁迫的响应[J]. 植物生理学通讯,2007,43(6):995-1001.
[13]Liu W J,Chen Y E,Tian W J,et al. Dephosphorylation of photosystem Ⅱ proteins and phosphorylation of CP29 in barley photosynthetic membranes as a response to water stress[J]. Biochimica et Biophysica acta,2009,1787(10):1238-1245.
[14]Pascal A,Peterman E,Gradinaru C,et al. Structure and interactions of the chlorophyll a molecules in the higher plantLhcb4 antenna protein[J]. Journal of Physical Chemistry B,2000,104(39):9317-9321.
[15]Gastaldelli M,Canino G,Croce R,et al. Xanthophyll binding sites of the CP29 (Lhcb4) subunit of higher plant photosystem Ⅱ investigated by domain swapping and mutation analysis[J]. Journal of Biological Chemistry,2003,278(21):19190-19198.
[16]de Bianchi S,Betterle N,Kouril R,et al.Arabidopsismutants deleted in the light-harvesting proteinLhcb4 have a disrupted photosystem Ⅱ macrostructure and are defective in photoprotection[J]. The Plant Cell,2011,23(7):2659-2679.
[17]Mozzo M,Passarini F,Bassi R,et al. Photoprotection in higher plants:the putative quenching site is conserved in all outer light-harvesting complexes of photosystem Ⅱ[J]. Biochimica et Biophysica Acta,2008,1777(10):1263-1267.
[18]Miloslavina Y,de Bianchi S,Dall’osto L,et al. Quenching inArabidopsisthalianamutants lacking monomeric antenna proteins of photosystem Ⅱ[J]. The Journal of Biological Chemistry,2011,286(42):36830-36840.
[19]Kargul J,Turkina M V,Nield J,et al. Light-harvesting complex Ⅱ protein CP29 binds to photosystem Ⅰ ofChlamydomonasreinhardtiiunder state 2 conditions[J]. FEBS Journal,2005,272(18):4797-4806.
