普通烟草PMEI家族的鉴定与表达分析
2018-05-30韩林贺丁安明孔英珍
韩林贺, 丁安明, 孔英珍
(1.中国农业科学院烟草研究所/中国农业科学院烟草遗传改良与生物技术重点开放实验室,山东青岛 266101;2.中国农业科学院研究生院,北京 100081)
果胶是存在于植物细胞胞间层以及初生细胞壁和次生细胞壁中一类成分复杂的多糖,是植物中一大类主要以半乳糖醛酸组成骨架结构的酸性多糖所形成的复合体的统称,也是双子叶植物和除玉米外的单子叶植物细胞壁的主要组成成分,约占细胞壁组成的30%~40%[1]。根据果胶多糖大分子的成分与构成的不同,主要可以分为以下3类:同聚半乳糖醛酸聚糖(homogalacturonan,简称HG),Ⅰ型鼠李糖半乳糖醛酸聚糖(rhamnogalacturonan Ⅰ,简称RG Ⅰ)和Ⅱ型鼠李糖半乳糖醛酸聚糖(rhamnogalacturonan Ⅱ,简称RG Ⅱ),这3类聚糖是果胶多糖组分的主要多糖[2-4]。细胞壁中的果胶对维持细胞壁稳定和抗压能力有一定的作用,也是植物应对病原体侵害的重要屏障。果胶的甲酯化和去酯化能够引起植物细胞壁结构和功能特性的改变,在果实成熟软化、器官脱落和衰老[5-7]、花粉发育、花粉管生长[8-10]和抗逆性[11-12]等方面具有重要作用。果胶去甲酯化形成的果胶酸,会与胞外的Ca2+结合形成果胶酸盐,使植物细胞壁硬化,导致植物细胞生长缓慢。同时,去甲酯化形成的甲醇和氢离子,会影响其他果胶水解酶的活性[13-14]。果胶去甲酯化的过程是由果胶甲酯酶(pectin methyl ester enzyme,简称PME)催化完成的,PME的活性同时又受果胶甲酯酶抑制剂(pectin methyl ester enzyme inhibitor,简称PMEI)的调控。Sénéchal等通过对PME17、PMEI4在拟南芥中的共表达分析和PMEI4突变体的特征分析,发现PMEI17和PMEI4相互作用;后来通过对体外PME/PMEI复合体生物化学特征进行详细分析,发现在一定的pH值范围内,PME3的酶活性直接受PMEI7调控[15-16]。PMEI广泛存在于高等植物以及一些能降解植物细胞壁的微生物如细菌、真菌中[17]。目前人们仅对拟南芥、石松类以及桃、猕猴桃等高等植物的PMEI基因家族进行了相关结构分析和功能研究。Hothorn等于2004年获得了拟南芥PMEI的三维结构,该结构由4条反向平行的α螺旋组成,分别为α1、α2、α3、α4,构成PMEI的活性区域;在氮末端区域有3条短的弯曲的α螺旋,分别为αa、αb、αc,维持PMEI结构的稳定性。PMEI蛋白序列中有4个极为保守的半胱氨酸,分别位于α2、α3、αa、αb上[18]。Di Matteo等根据已获得的猕猴桃果胶甲酯酶抑制剂氨基酸序列,人工合成其基因序列,在毕赤酵母中进行表达获得纯化的PMEI蛋白,将其与从番茄中提取的PME组合,获得二者复合物的三维结构[19]。梅晓宏等发现,在新鲜果蔬汁中加入PMEI可以阻止果胶形成果胶酸盐吸附果肉,防止果蔬汁变质[20]。根据Juge等的研究,PMEI成员参与果胶新陈代谢,影响谷类植物细胞壁多糖的形成[21-22],从而影响韧皮纤维的伸长。相关研究表明,PMEI参与果实的形成过程,番茄中的SolyPMEI在果实的发育过程中特异表达[23],葡萄中的VvPMEI1也参与果实的成熟过程[24]。用水杨酸(SA)处理辣椒6 h后,辣椒细胞内的PMEI含量显著升高,表明PMEI与水杨酸信号调节有关[25-26]。在用双氧水处理水稻幼苗 48 h 后,辣椒幼苗体内PMEI含量显著升高,表明其与植物体抗氧化作用有关[27]。因此,对植物中PMEI的特性及生物学功能进行研究具有重要的理论和应用价值。
烟草是我国重要的经济作物,同时也是进行生物学研究的重要模式植物,但是对烟草PMEI基因家族的研究鲜见报道。烟草的主要收获器官是叶片,叶片细胞壁中的多糖聚合物不仅维持烟叶在生长发育过程中的形态,还影响烟叶的物理特性、加工特性以及烟叶的吸食质量。烟叶细胞壁中的果胶含量与烟叶平衡含水率、填充力、燃烧性呈正相关,与烟叶厚度、叶质重及香气、余味、杂气得分、评吸总分呈负相关[28-30]。因此,研究烟草PMEI家族基因对果胶甲酯化过程的调控作用及其对细胞壁结构的影响具有重要意义。本研究以普通烟草PMEI家族(NtPMEI)为研究对象,采用生物信息学手段对其基因家族各成员之间的进化关系及其基因结构、编码蛋白的物理化学性质、保守结构域、亚细胞定位、基因本体(gene ontology,简称GO)分析、组织表达等进行研究,以期为研究烟草中PMEI家族的功能提供参考。
1 材料与方法
1.1 植物材料
普通烟草品种红花大金元(简称HD),于温室种植,自然条件下培养至成熟期。取幼苗期的根、茎、叶、叶脉和成熟期的根、茎、叶、叶脉及花芽,经处理后于液氮中速冻,-80 ℃保存备用。
1.2 NtPMEI家族序列鉴定与理化性质分析
基于Wang等报道的拟南芥71个PMEI家族成员的序列[31],利用序列相似性分析,预测烟草基因组PMEI家族。从TAIR数据库(http://www.arabidopsis.org/)下载拟南芥PMEI家族蛋白全长序列,从Pfam数据库(http://pfam.xfam.org/)下载PMEI家族蛋白结构域序列(Pfam ID:PF04043)。将两类序列在烟草基因组数据库(http://218.28.140.17/)和NCBI数据库(http://www.ncbi.nlm.nih.gov/)中进行Blastp检索(e值≤10-15),获得普通烟草的PMEI类似序列,人工去除冗余序列,作为NtPMEI家族的候选序列。结合Pfam数据库[32]中PMEI结构域保守序列(序列号PF04043)和SMART在线分析软件(http://smart.embl-heidelberg.de/)[33]对候选序列进行PMEI结构域分析,剔除不含有该结构域的蛋白质序列,最终鉴定出90条NtPMEI家族蛋白序列。利用ExPASyProtParam(http://web.expasy.org/protparam/)对所得烟草PMEI家族蛋白序列进行理化性质分析[34]。
1.3 NtPMEI蛋白序列保守域与进化分析
利用Muscle程序[35]对拟南芥和普通烟草PMEI家族的蛋白序列进行多序列比对,参数选为默认值。基于比对结果,利用MEGA6.0软件[36],采用邻接法(neighbor-joining,简称NJ)构建拟南芥和普通烟草PMEI家族进化树,Bootstrap检验参数设置为1 000。利用MEME4.11.2(Multiple Expectation Maximization for Motif Elicitation)[37]在线平台(http://meme.nbcr.net/meme/cgi-bin/meme.cgi)对90个NtPMEI家族蛋白进行保守基序分析,最大motif检索值定为20。利用Multiple Sequence Alignment by CLUSTALW在线平台(http://www.genome.jp/tools/clustalw/)进行多序列比对,Output Format格式设置为FASTA格式。利用BoxShade Server在线平台(http://www.ch.embnet.org/software/BOX_form.html),将各蛋白保守区域进行可视化。选取结构域保守的蛋白序列为目标序列,在SWISS-MODEL网站(http://swissmodel.expasy.org/interactive)上查找三维结构模板,根据覆盖率、一致性百分比、试验可靠性选择最适的三维结构模板。
1.4 NtPMEI蛋白编码基因的表达模式分析与验证
为分析所预测的候选NtPMEI基因的表达模式,在烟草基因组数据库(http://218.28.140.17/)中下载烟草基因组组织表达数据,利用R语言Bioconductor程序对转录数据进行分析,使数据均一化,利用MeV本地软件对结果进行可视化分析。选择部分候选NtPMEI基因进行组织特异性表达验证,使用TRANS Kit提取总RNA,参照说明书步骤,利用反转录试剂盒TransScript One-Step gDNA Removal and cDNA Synthesis SuperMix Kit合成第一链cDNA,作为PCR扩增模板。以烟草管家基因actin(GenBank登录号为EU938079.1)作为内参基因,反应体系:10 μL 2×FastStart Universal SYBR Green Master(ROX)、0.6 μL正反向引物(10 μmol/L)、1 μL模板cDNA、7.8 μL ddH2O(RNase free)。在荧光定量PCR仪Applied Biosystem 7500(ABI7500)上进行qRT-PCR反应,程序如下:50 ℃ 2 min,95 ℃ 2 min,95 ℃ 15 s,60 ℃ 34 s,共40个循环,加溶解曲线;每个样品重复3次。用7500 software v2.0.1对结果进行分析,并对相对表达量进行作图。
2 结果与分析
2.1 NtPMEI家族蛋白序列鉴定及进化分析
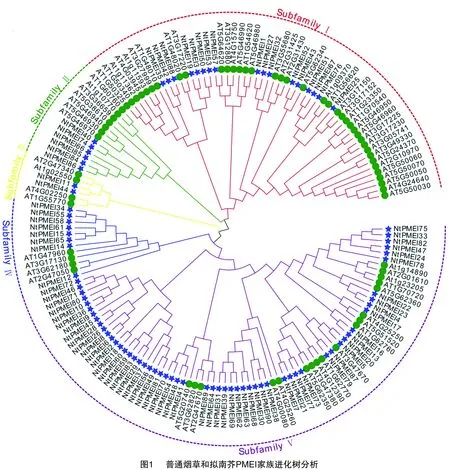
利用拟南芥PMEI家族71条蛋白全长序列和保守结构域序列,在烟草基因组数据库和NCBI数据库中进行Blastp比对,获得105条烟草PMEI家族候选序列。结合Pfam数据库中PMEI结构域保守序列(序列号PF04043)和SMART在线程序,对候选序列进行保守结构域分析,去除不含有PMEI结构域的序列,在普通烟草基因组中共鉴定出90条PMEI蛋白序列。根据烟草注释信息中基因在染色体上的位置,对90个成员进行重新编号,以便后续研究(表1)。
将得到的90个NtPMEI家族成员全长蛋白序列与拟南芥PMEI家族71个成员进行系统发生分析(图1),根据拟南芥的分类方式将NtPMEI家族成员分为5个亚家族(Ⅰ、Ⅱ、Ⅲ、Ⅳ、Ⅴ),各个亚家族分别含有17、6、2、8、57个NtPMEI成员(图1、表1)。通过分析NtPMEI蛋白编码基因结构发现,大多数基因不含有内含子。其中,第Ⅱ、Ⅲ亚家族成员均不含有内含子,第Ⅰ亚家族中的NtPMEI1、NtPMEI42分别含有6、5个内含子,第Ⅳ亚家族中有3个成员各含有1个内含子,第V亚家族中有1/2的成员含有内含子(表1)。
2.2 NtPMEI家族蛋白理化性质分析
对获得的NtPMEI家族成员蛋白序列进行理化特征分析,表1结果显示,NtPMEI蛋白序列平均长度在284个氨基酸左右,其中序列最长的NtPMEI37和最短的NtPMEI17都属于第Ⅴ亚家族,分别由685、108个氨基酸残基组成。不同亚家族之间的氨基酸序列长度变化呈现不同规律,第Ⅱ亚家族蛋白序列长度大多在200个氨基酸左右;第Ⅴ亚家族有30%以上成员的蛋白序列长度在500个氨基酸以上。NtPMEI家族成员蛋白分子量变化范围在11.72~75.63ku之间,变化范围较大。NtPMEI家族成员理论等电点变化范围较大,介于 3.99~10.55 之间。第Ⅰ亚家族成员理论等电点大多介于 4.45~6.99,只有1个蛋白的理论等电点为8.64,说明该家族成员蛋白序列中多为酸性氨基酸;第Ⅲ亚家族的2个成员理论等电点均为4.26,在酸性范围内,说明该亚家族成员蛋白序列中含有较多的酸性氨基酸。

表1 NtPMEI家族理化性质分析
续表1
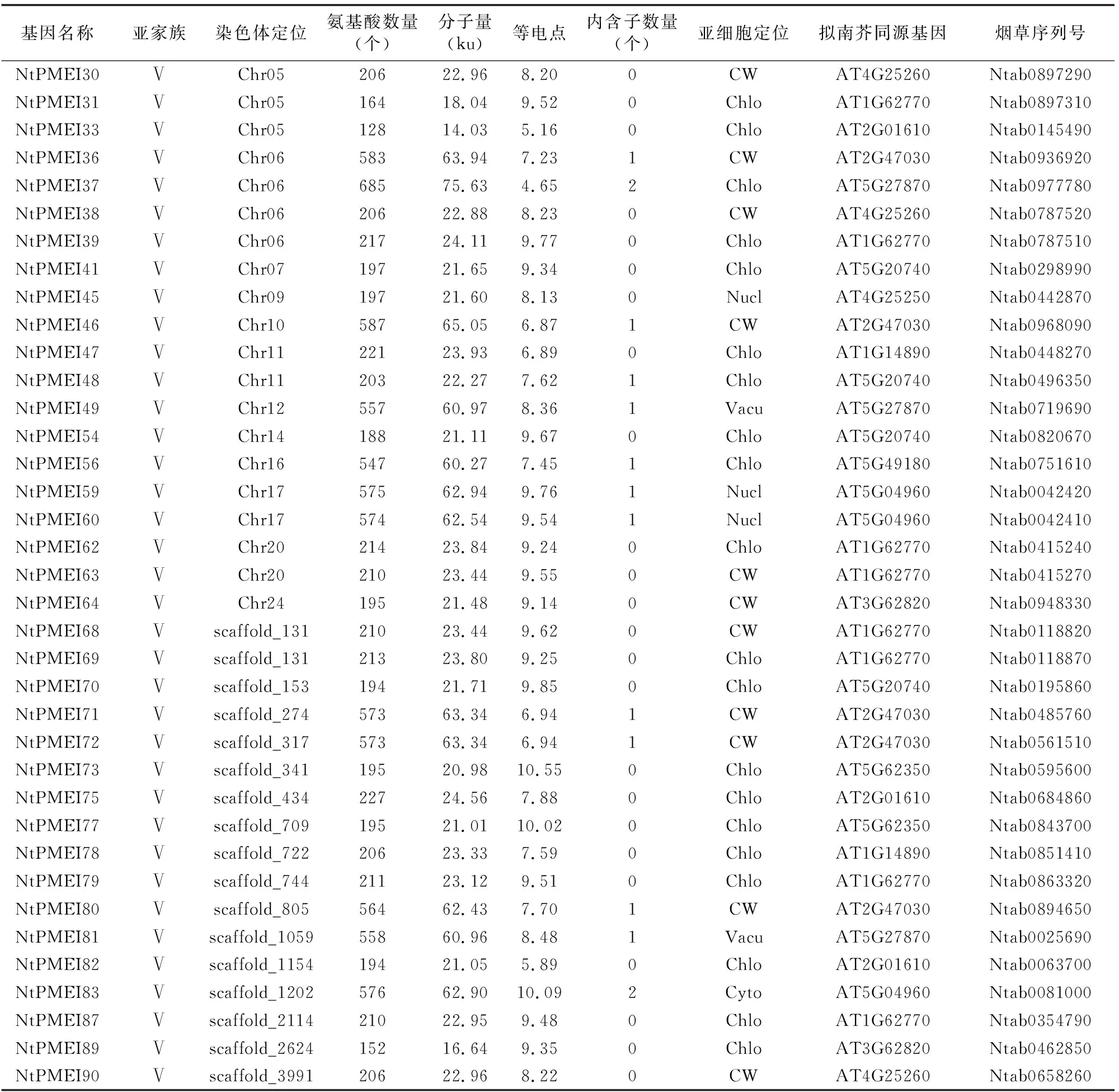
基因名称亚家族染色体定位氨基酸数量(个)分子量(ku)等电点内含子数量(个)亚细胞定位拟南芥同源基因烟草序列号NtPMEI30ⅤChr0520622.968.200CWAT4G25260Ntab0897290NtPMEI31ⅤChr0516418.049.520ChloAT1G62770Ntab0897310NtPMEI33ⅤChr0512814.035.160ChloAT2G01610Ntab0145490NtPMEI36ⅤChr0658363.947.231CWAT2G47030Ntab0936920NtPMEI37ⅤChr0668575.634.652ChloAT5G27870Ntab0977780NtPMEI38ⅤChr0620622.888.230CWAT4G25260Ntab0787520NtPMEI39ⅤChr0621724.119.770ChloAT1G62770Ntab0787510NtPMEI41ⅤChr0719721.659.340ChloAT5G20740Ntab0298990NtPMEI45ⅤChr0919721.608.130NuclAT4G25250Ntab0442870NtPMEI46ⅤChr1058765.056.871CWAT2G47030Ntab0968090NtPMEI47ⅤChr1122123.936.890ChloAT1G14890Ntab0448270NtPMEI48ⅤChr1120322.277.621ChloAT5G20740Ntab0496350NtPMEI49ⅤChr1255760.978.361VacuAT5G27870Ntab0719690NtPMEI54ⅤChr1418821.119.670ChloAT5G20740Ntab0820670NtPMEI56ⅤChr1654760.277.451ChloAT5G49180Ntab0751610NtPMEI59ⅤChr1757562.949.761NuclAT5G04960Ntab0042420NtPMEI60ⅤChr1757462.549.541NuclAT5G04960Ntab0042410NtPMEI62ⅤChr2021423.849.240ChloAT1G62770Ntab0415240NtPMEI63ⅤChr2021023.449.550CWAT1G62770Ntab0415270NtPMEI64ⅤChr2419521.489.140CWAT3G62820Ntab0948330NtPMEI68Ⅴscaffold_13121023.449.620CWAT1G62770Ntab0118820NtPMEI69Ⅴscaffold_13121323.809.250ChloAT1G62770Ntab0118870NtPMEI70Ⅴscaffold_15319421.719.850ChloAT5G20740Ntab0195860NtPMEI71Ⅴscaffold_27457363.346.941CWAT2G47030Ntab0485760NtPMEI72Ⅴscaffold_31757363.346.941CWAT2G47030Ntab0561510NtPMEI73Ⅴscaffold_34119520.9810.550ChloAT5G62350Ntab0595600NtPMEI75Ⅴscaffold_43422724.567.880ChloAT2G01610Ntab0684860NtPMEI77Ⅴscaffold_70919521.0110.020ChloAT5G62350Ntab0843700NtPMEI78Ⅴscaffold_72220623.337.590ChloAT1G14890Ntab0851410NtPMEI79Ⅴscaffold_74421123.129.510ChloAT1G62770Ntab0863320NtPMEI80Ⅴscaffold_80556462.437.701CWAT2G47030Ntab0894650NtPMEI81Ⅴscaffold_105955860.968.481VacuAT5G27870Ntab0025690NtPMEI82Ⅴscaffold_115419421.055.890ChloAT2G01610Ntab0063700NtPMEI83Ⅴscaffold_120257662.9010.092CytoAT5G04960Ntab0081000NtPMEI87Ⅴscaffold_211421022.959.480ChloAT1G62770Ntab0354790NtPMEI89Ⅴscaffold_262415216.649.350ChloAT3G62820Ntab0462850NtPMEI90Ⅴscaffold_399120622.968.220CWAT4G25260Ntab0658260
注:CW表示细胞壁,Nucl表示细胞核,Mito表示线粒体,Chlo表示叶绿体,Cyto表示细胞质,E.R.表示内质网,Vacu表示液泡,Plas表示质粒。
从上述分析结果可以看出,NtPMEI同一亚家族中蛋白成员及其编码基因在结构和理化特征上均比较相似,从侧面反映了系统发育分析的可靠性。
2.3 NtPMEI家族保守基序分析及PMEI结构域三维结构预测
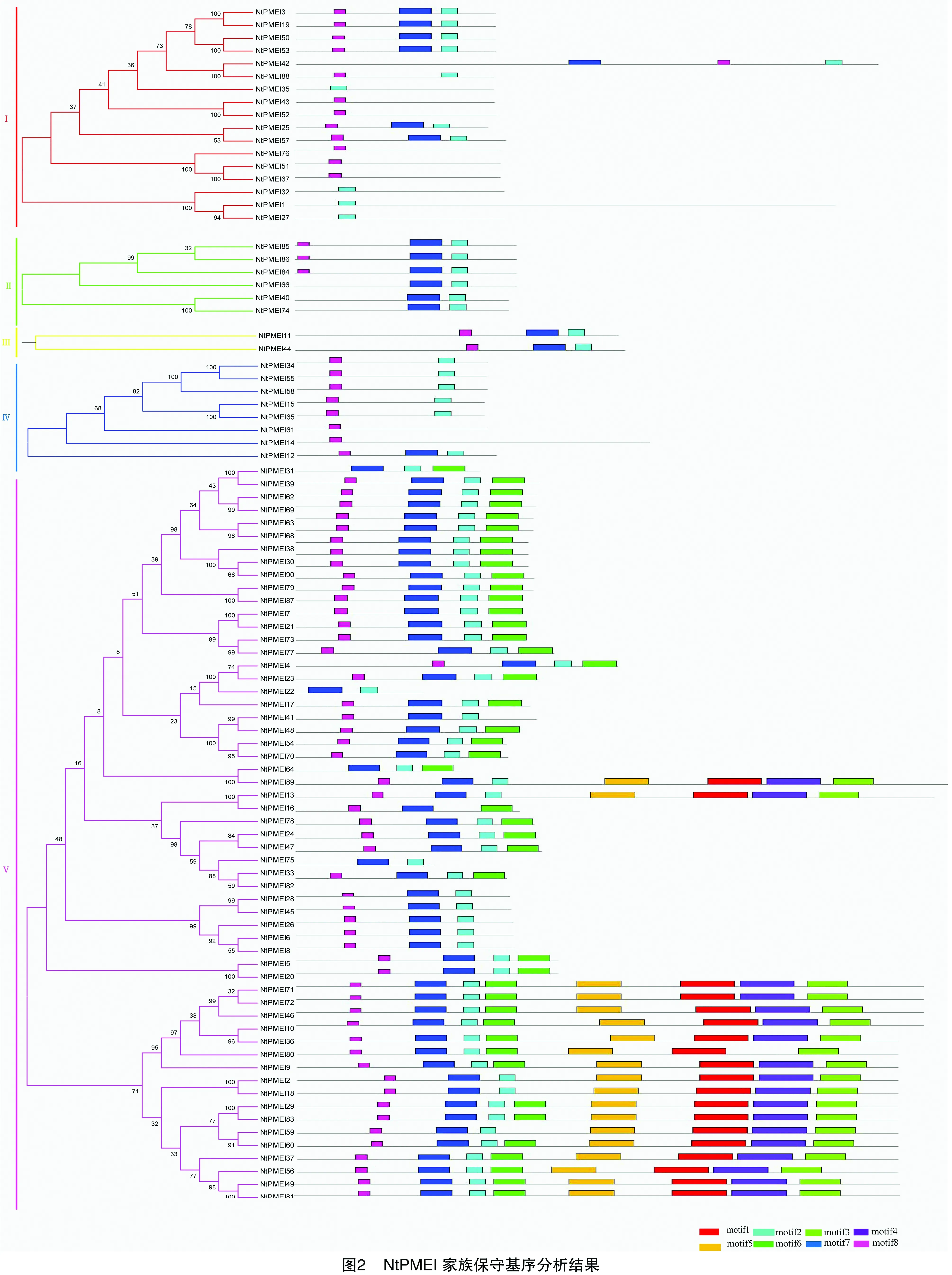
利用MEME4.11.2在线程序对NtPMEI家族90个成员蛋白序列进行保守基序(motif)分析,以便进一步了解NtPMEI家族中蛋白氨基酸序列的保守性,共鉴定出8个保守基序(图2)。
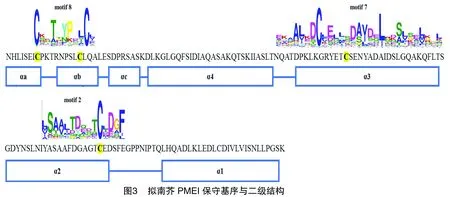
使用Pfam和SMART对每个保守基序进行注释分析,发现有3个保守基序与PMEI结构域重合,分别为motif2、motif7、motif8。对拟南芥PMEI蛋白序列进行保守基序分析,同样鉴定出PMEI结构域序列对应3个相同的保守基序,分别为motif2、motif7、motif8(图3),与烟草PMEI的保守基序相同,证明了对烟草序列进行保守基序分析结果的可靠性。与二级结构相比较,这3个保守基序分别对应PMEI的活性区域和氮末端螺旋区域。因此,推测烟草中moti2、moti7组成NtPMEI的活性区域,motif8维持NtPMEI结构的稳定性。
以第Ⅳ亚家族为例,将其成员NtPMEI15、NtPMEI58、NtPMEI65与拟南芥AtPMEI1进行多序列比对,发现烟草和拟南芥的PMEI结构域序列相似,都有4个极保守的半胱氨酸残基(图4-A)。选择PMEI结构保守的NtPMEI65进行保守结构域的三维结构预测,与拟南芥PMEI的三维结构(图4-B)进行比较,发现烟草的PMEI结构同样由4条长的反向平行的α螺旋和3条短的α螺旋组成(图4-C)。
2.4 NtPMEI家族的表达模式分析
从普通烟草高通量转录组测序数据库中提取PMEI基因家族在烟草10个组织的表达量数据,包括播种后种子、叶脉、叶片(离体24 h)、愈伤组织、幼根、幼叶、成熟根、成熟叶、花芽、茎。对获得的数据进行分析,利用R语言将数据进行可视化,绘制热图。
由图5可以看出,有28个NtPMEI成员在所有组织中均没有表达,其余62个成员分别在不同时期、不同组织中有所表达,约占整个亚家族的68.9%,表明大部分NtPMEI成员参与烟草的生长发育过程。其中有11个成员在所有组织中都有表达,但在不同组织中的表达量有明显差异。进一步分析发现,不同亚家族成员在不同组织中的表达情况有较大差异,具有一定的组织特异性。第Ⅱ、Ⅲ亚家族成员在各组织中的表达量很低;第Ⅰ亚家族中有3个成员在所选取的10个组织中未有表达,其他成员在不同组织中均有表达,在幼根、花芽和茎中表达数较多;第Ⅳ亚家族成员在叶脉、茎中的表达量较高;第Ⅴ亚家族成员广泛参与植物各阶段的发育,其中在叶脉、幼叶、幼根和茎中的表达量较高。分析在幼根、成熟根中表达的基因发现,在幼根中表达量较高的基因在成熟根中绝大多数也有表达而且表达量相对较高,但相比在成熟根中的表达量较低。结果分析表明,NtPMEI基因家族在不同组织中的表达存在差异,这可能与基因的表达模式以及基因在组织中特异性表达和环境因素有关。
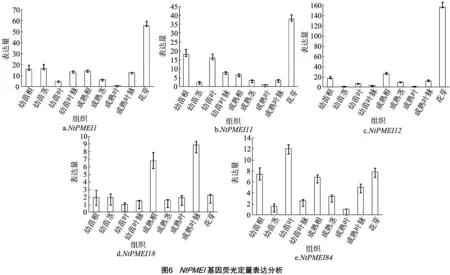
为验证基因芯片数据分析的可靠性,本研究选取烟草8个不同时期的组织提取RNA并反转为cDNA,进行5个NtPMEI基因在其中的表达量验证。图6显示NtPMEI1、NtPMEI11、NtPMEI12、NtPMEI18和NtPMEI84在烟草(HD)幼苗期根、茎、叶、叶脉和成熟期根、茎、叶、叶脉及花芽中的表达情况。总体上看出,这5个基因在所选取的组织中均有表达,在不同组织中的表达量存在明显差异,但表达趋势和芯片数据大体一致。NtPMEI1、NtPMEI11和NtPMEI12 在花芽中的表达量相对较高,而NtPMEI1、NtPMEI11在成熟期的叶中几乎不表达;NtPMEI12在幼苗期的茎和成熟期的叶中几乎不表达,在幼苗期的叶脉中表达量也很低;NtPMEI18在成熟期的根、叶脉中表达量相对较高;NtPMEI84在幼苗期的叶中表达量相对较高。比较各基因在幼苗根、成熟根中的表达量发现,除NtPMEI12外,其他4个基因在成熟根中的表达量均呈下调趋势。

3 讨论
前人研究表明,PMEI家族在植物果实成熟软化、器官脱落和衰老、花粉发育、花粉管生长和抗逆性等多个生长发育阶段均发挥重要作用。烟草是分子生物学研究的模式植物,中国烟草基因组计划的完成,为烟草基因组PMEI家族的鉴定和进一步的功能研究提供了便利。本研究采用生物信息学方法,从普通烟草基因组中鉴定出90条NtPMEI基因家族成员。系统发生分析结果表明,烟草PMEI家族分成5个亚家族,同一亚家族成员都能较好地聚为一类,并且相应的结构域类型与前人研究的基本一致。对基因中内含子和外显子的组成进行分析,可为基因在进化分析研究中提供重要证据[38]。本研究对候选NtPMEI基因家族各成员的内含子数量进行分析发现,大多数NtPMEI基因家族各成员的基因结构中都不包含内含子,部分基因包含1~2个内含子,只有NtPMEI1、NtPMEI42分别含有6、5个内含子,表明NtPMEI基因家族各基因间保守性较高,该基因家族进化程度比较高。通过将烟草PMEI结构域保守序列与拟南芥PMEI序列作多序列比对分析,发现烟草PMEI序列中也含有4个极保守的半胱氨酸残基,形成2个二硫键,维持PMEI的三维结构[39]。通过以第Ⅳ亚家族中的NtPMEI65为例作三维结构预测,发现烟草PMEI的三级结构和拟南芥极为相似。
组织表达分析发现,在不同组织中均有NtPMEI成员进行表达,说明该基因家族成员广泛参与了烟草的生长发育过程。Wolf等的研究发现,AtPMEI1(AT1G48020)和AtPMEI2(AT3G17220)对促进花粉及花粉管的形成具有一定的作用[40-41]。在系统进化树中,烟草中的NtPMEI3、NtPMEI19、NtPMEI50和NtPMEI53与AtPMEI1、AtPMEI2基因高度同源,推测这4个基因在烟草中也能够影响花粉和花粉管的形成与发育。部分NtPMEI基因的验证结果也同样表明,所选取的5个NtPMEIs在普通烟草的花都中有表达,表明这几个基因参与花的发育过程。相关研究表明,PMEI基因的敲除和过表达都会对拟南芥的生理特性产生影响。Peaucelle等通过对AtPMEI3转基因株系进行研究,发现AtPMEI3过表达会引起分生组织中同聚半乳糖醛酸(HGA)高度甲酯化, 并且影响叶序的发育[42]。烟草中的NtPMEI41、NtPMEI48、NtPMEI54和NtPMEI70与AtPMEI3基因高度同源,推测这4个基因在烟草中过表达也会影响分生组织中HGA的甲酯化程度和叶序的发育。Müller等发现,AtPMEI5在拟南芥中过表达会引起不正常的发育形态,如发芽率升高、茎卷曲和器官融合[43-44]。通过系统发育分析发现,NtPMEI基因家族中NtPMEI14、NtPMEI34、NtPMEI35、NtPMEI61和NtPMEI65与AtPMEI5高度同源,推测这5个基因在烟草中过表达也会导致植株的不正常发育。An等发现,辣椒中的CaPMEI1(A5X5I9)基因与水杨酸(SA)信号调节有关,能够响应逆境胁迫[26]。通过序列相似性比对分析,发现烟草中的NtPMEI4、NtPMEI22和NtPMEI23与A5X5I9基因高度同源,推测这3个基因在烟草中与抗非生物胁迫有关。Saez-Aguayo等研究发现,拟南芥种皮表皮细胞中的AtPMEI6(AT2G47670)通过抑制同聚半乳糖醛酸的甲酯化,从而提高种皮黏液质的分泌[45],通过序列相似性比对分析发现,烟草中的NtPMEI64、NtPMEI89与AtPMEI6基因高度同源,推测这2个基因在烟草中可能行使相似的功能。

综上,本研究对普通烟草基因组中的90个PMEI家族成员进行了进化分析、蛋白理化性质和功能域分析,确定了其在染色体上的位置,并通过分析芯片数据和荧光定量试验验证了部分成员的组织表达模式。通过本研究,对烟草PMEI家族成员有了初步了解,为今后PMEI家族成员的功能研究奠定了基础。为了更好地了解烟草PMEI家族成员的结构和功能,下一步将对相关基因进行克隆、表达模式分析、功能验证等,探索其在烟草生长发育中的作用。
:
[1]Yu L,Shi D,Li J,et al. CELLULOSE SYNTHASE-LIKE A2,a glucomannan synthase,is involved in maintaining adherent mucilage structure inArabidopsisseed[J]. Plant Physiology,2014,164(4):1842-1856.
[2]Ridley B L,O’Neill M A,Mohnen D A. Pectins:structure,biosynthesis,and oligogalacturonide-related signaling[J]. Phytochemistry,2001,57(6):929-967.
[3]Wong D. Enzymatic deconstruction of backbone structures of the ramified regions in pectins[J]. Protein Journal,2008,27(1):30-42.
[4]Zandleven J,Sørensen S O,Harholt J,et al. Xylogalacturonan exists in cell walls from various tissues ofArabidopsisthaliana[J]. Phytochemistry,2007,68(8):1219-1226.
[5]Castillejo C,de La Fuente J I,Iannetta P,et al. Pectin esterase gene family in strawberry fruit:study of FaPE1,a ripening-specific isoform[J]. Journal of Experimental Botany,2004,55(398):909-918.
[6]Louvet R,Cavel E,Gutierrez L,et al. Comprehensive expression profiling of the pectin methylesterase gene family during silique development inArabidopsisthaliana[J]. Planta,2006,224(4):782-791.
[7]Zhu S H,Zhou J. Effects of nitric oxide on fatty acid composition in peach fruits during storage[J]. Journal of Agricultural and Food Chemistry,2006,54(25):9447-9452.
[8]Bosch M,Cheung A Y,Hepler P K. Pectin methylesterase,a regulator of pollen tube growth[J]. Plant Physiology,2005,138(3):1334-1346.
[9]Francis K E,Lam S Y,Copenhaver G P. Separation ofArabidopsispollen tetrads is regulated by QUARTET1,a pectin methylesterase gene[J]. Plant Physiology,2006,142(3):1004-1013.
[10]Tian G W,Chen M H,Zaltsman A,et al. Pollen-specific pectin methylesterase involved in pollen tube growth[J]. Developmental Biology,2006,294(1):83-91.
[11]Collmer A C,Keen N T. The role of pectic enzymes in plant pathogenesis[J]. Annual Review of Phytopathology,2003,24(1):383-409.
[12]Schmohl N,Pilling J,Fisahn J,et al. Pectin methylesterase modulates aluminium sensitivity inZeamaysandSolanumtuberosum[J]. Physiologia Plantarum,2001,109(4):419-427.
[13]于海伟,陈宝刚. 白桦果胶甲酯酶抑制剂(PMEI)基因克隆及序列分析[J]. 防护林科技,2012(1):64-67.
[14]霍如雪,刘振宁,杨 青,等. 桃PME基因家族的鉴定与分析[J]. 江苏农业科学,2016,44(5):24-30.
[15]Sénéchal F,L’Enfant M,Domon J M,et al. Tuning of pectin methylesterification:PECTIN METHYLESTERASE INHIBITOR 7 modulates the processive activity of co-expressed PECTIN METHYLESTERASE 3 in a pH-dependent manner[J]. Journal of Biological Chemistry,2015,290(38):23320-23335.
[16]Senechal F,Mareck A,Marcelo P,et al.ArabidopsisPME17 activity can be controlled by pectin methylesterase inhibitor4[J]. Plant Signaling & Behavior,2015,10(2):e983351.
[17]Markovic O,Janecek S. Pectin methylesterases:sequence-structural features and phylogenetic relationships[J]. Carbohydrate Research,2004,339(13):2281-2295.
[18]Hothorn M,Wolf S,Aloy P,et al. Structural insights into the target specificity of plant invertase and pectin methylesterase inhibitory proteins[J]. Plant Cell,2004,16(12):3437-3447.
[19]Di Matteo A,Giovane A,Raiola A,et al. Structural basis for the interaction between pectin methylesterase and a specific inhibitor protein[J]. Plant Cell,2005,17(3):849-858.
[20]梅晓宏,陈燕卉,高红岩,等. 果胶甲酯酶抑制剂的研究进展[J]. 食品科技,2008,33(6):64-68.
[21]Juge N. Plant protein inhibitors of cell wall degrading enzymes[J]. Trends in Plant Science,2006,11(7):359-367.
[22]Juge N,Svensson B. Proteinaceous inhibitors of carbohydrate‐active enzymes in cereals:implication in agriculture,cereal processing and nutrition[J]. Journal of the Science of Food and Agriculture,2006,86(11):1573-1586.
[23]Reca I B,Lionetti V,Camardella L,et al. A functional pectin methylesterase inhibitor protein (SolyPMEI) is expressed during tomato fruit ripening and interacts with PME-1[J]. Plant Molecular Biology,2012,79(4/5):429-442.
[24]Lionetti V,Raiola A,Mattei B,et al. The grapevineVvPMEI1 gene encodes a novel functional pectin methylesterase inhibitor associated to grape berry development[J]. PLoS One,2015,10(7):e0133810.
[25]Loake G,Grant M. Salicylic acid in plant defence—the players and protagonists[J]. Current Opinion in Plant Biology,2007,10(5):466-472.
[26]An S H,Sohn K H,Choi H W,et al. Pepper pectin methylesterase inhibitor protein CaPMEI1 is required for antifungal activity,basal disease resistance and abiotic stress tolerance[J]. Planta,2008,228(1):61-78.
[27]Tsai Y C,Hong C Y,Liu L F,et al. Expression of ascorbate peroxidase and glutathione reductase in roots of rice seedlings in response to NaCl and H2O2[J]. Journal of Plant Physiology,2005,162(3):291-299.
[28]任晓红,陈 刚,马海燕,等. 烤烟细胞壁物质对烟叶质量影响研究[J]. 中国农学通报,2010,26(4):113-116.
[29]闫克玉,闫洪洋,李兴波,等. 烤烟烟叶细胞壁物质的对比分析[J]. 烟草科技,2005(10):6-11.
[30]李兴波,阎克玉,阎洪洋,等. 河南烤烟(40级)细胞壁物质含量及规律性研究[J]. 郑州轻工业学院学报(自然科学版),1999,14(3):27-30.
[31]Wang M J,Yuan D J,Gao W H,et al. A comparative genome analysis of PME and PMEI families reveals the evolution of pectin metabolism in plant cell walls[J]. PLoS One,2013,8(8):e72082.
[32]Nakano T,Suzuki K,Fujimura T,et al. Genome-wide analysis of theERFgene family in Arabidopsis and rice[J]. Plant Physiology,2006,140(2):411-432.
[33]Kim S,Park M,Yeom S I,et al. Genome sequence of the hot pepper provides insights into the evolution of pungency inCapsicumspecies[J]. Nature Genetics,2014,46(3):270-278.
[34]Artimo P,Jonnalagedda M,Arnold K A,et al. ExPASy:SIB bioinformatics resource portal[J]. Nucleic Acids Research,2012,40:W597-W603.
[35]Tamura K,Peterson D,Peterson N,et al. MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J]. Molecular Biology and Evolution,2011,28(10):2731-2739.
[36]Tamura K,Stecher G,Peterson D,et al. MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution,2013,30(12):2725-2729.
[37]Gu Y B,Ji Z R,Chi F M,et al. Bioinformatics and expression analysis of theWRKYgene family in apple[J]. Scientia Agricultura Sinica,2015,48(16):229-230.
[38]吴洁云. 灰葡萄孢胞壁降解酶,角质酶及其对番茄植株的致病作用[D]. 扬州:扬州大学,2007.
[39]杨模坤. 叶绿体的结构和化学[J]. 植物生理学报,1963(3):40-47.
[40]Wolf S,Grsic-Rausch S,Rausch T,et al. Identification of pollen-expressed pectin methylesterase inhibitors inArabidopsis[J]. FEBS Letters,2003,555(3):551-555.
[41]Roeckel N,Wolf S,Kost B,et al. Elaborate spatial patterning of cell-wall PME and PMEI at the pollen tube tip involves PMEI endocytosis,and reflects the distribution of esterified and de-esterified pectins[J]. Plant Journal,2008,53(1):133-143.
[42]Peaucelle A,Louvet R,Johansen J N,et al.Arabidopsisphyllotaxis is controlled by the methyl-esterification status of cell-wall pectins[J]. Current Biology,2008,18(24):1943-1948.
[43]Müller K,Levesque-Tremblay G,Bartels S,et al. Demethylesterification of cell wall pectins inArabidopsisplays a role in Seed germination[J]. Plant Physiology,2013,161(1):305-316.
[44]Müller K,Levesque-Tremblay G,Fernandes A,et al. Overexpression of a pectin methylesterase inhibitor inArabidopsisthalianaleads to altered growth morphology of the stem and defective organ separation[J]. Plant Signaling & Behavior,2013,8(12):e26464.
[45]Saez-Aguayo S,Ralet M C,Berger A A,et al. PECTIN METHYLESTERASE INHIBITOR6 promotesArabidopsismucilage release by limiting methylesterification of homogalacturonan in seed coat epidermal cells[J]. Plant Cell,2013,25(1):308-323.
