肺炎克雷伯菌临床株喹诺酮类耐药性及ST494型菌株耐药机制分析
2018-05-30袁瑾懿徐晓刚胡付品徐庆庆丁百兴王明贵
袁瑾懿, 徐晓刚, 胡付品, 郭 燕, 杨 洋, 董 栋, 徐庆庆, 丁百兴, 王明贵
肺炎克雷伯菌是临床最重要的条件致病菌之一,可导致肺部感染、尿路感染、血流感染、化脓性肝脓肿、眼内炎、脑膜炎等多种感染性疾病,重则威胁生命[1-2]。其耐药率逐年升高,尤其多重耐药菌株日益增多,已成为全球关注焦点[3]。
多位点序列分型(multilocus sequence typing,MLST)是最常用的细菌基因分型方法之一,肺炎克雷伯菌的MLST方法最初建立于2005年[4],后被广泛用于对肺炎克雷伯菌临床菌株耐药性、毒力等的特征分析和分子流行病学研究[5]。
ST11为我国肺炎克雷伯菌临床株中最主要型别,ST15、23和29型等亦多见[6-7]。ST11型肺炎克雷伯菌为常见耐药克隆,已有产碳青霉烯酶(KPC-2和VIM-1等)、超广谱β内酰胺酶(CTX-M和SHV等)和16S rRNA甲基化酶的ST11型肺炎克雷伯菌在世界各地流行[8-10]。除β内酰胺酶和16S rRNA甲基化酶外,ST11型肺炎克雷伯菌还常同时存在喹诺酮耐药决定区(QRDR)突变、并携带质粒介导喹诺酮耐药基因,故常呈多重耐药[11]。ST11型多重耐药肺炎克雷伯菌近年在中国广泛传播[12-14]。不同于欧美国家以ST258型为主[15],我国碳青霉烯类耐药肺炎克雷伯菌主要为ST11型、其次为ST15型[16-17]。我国替加环素耐药肺炎克雷伯菌株也是以ST11和ST15型为主[18-19]。
为了解复旦大学附属华山医院临床分离肺炎克雷伯菌基本情况,本研究收集2010年本院临床分离肺炎克雷伯菌112株进行耐药性及分子流行病学分析,以期对本院肺炎克雷伯菌耐药克隆传播的防控及抗菌治疗提供理论依据。
1 材料与方法
1.1 材料
1.1.1 菌株来源 连续收集2010年3-5月我院临床分离的肺炎克雷伯菌,按常规方法进行菌种鉴定,剔除同一患者同一部位重复分离菌株,共获得112株。质控菌株大肠埃希菌ATCC 25922为本研究所临床微生物室保存菌株。
1.1.2 病史资料 采集患者住院期间的病史资料,包括性别、年龄、科室、标本来源和菌株分离时间等资料。
1.1.3 主要试剂 药敏试验所用萘啶酸为Sigma-Alorich公司产品(纯度≥98%),余抗菌药物粉剂来自中国药品生物制品检定所标准品或对照品,PCR相关试剂均为大连TaKaRa公司产品。
1.2 方法
1.2.1 琼脂稀释法测定抗菌药敏感性 按照美国临床与实验室标准化协会(CLSI)文件[20]推荐的琼脂稀释法测定抗菌药物对受试菌株的MIC。替加环素按照美国FDA规定的折点(http://www.pfizerpro.com/hcp/tygacil)判定耐药、中介和敏感,其他抗菌药物按照2017年CLSI规定的折点判定(其中萘啶酸和诺氟沙星采用尿液分离菌标准)。
1.2.2 MLST 分型根据http://bigsdb.pasteur.fr/klebsiella/klebsiella.html网站提示设计引物并对部分菌株进行PCR扩增,产物送杰里生物科技公司测序,测序结果提交该网站进行分析,并使用eBURST V3在线软件(http://eburst.mlst.net/)进行聚类分析。
1.2.3 耐药基因检测 参考文献设计引物并对部分菌株PCR扩增检测16S rRNA甲基化酶armA、rmtB、rmtD基因[21]、gyrA和parCQRDR[22]、多重耐药外排泵oqxAB基因[23]、质粒介导喹诺酮耐药元件qnrA、qnrB、qnrC、qnrD、qnrS、aac(6')-lbcr和qepA基因[24-29]、β内酰胺酶基因blaCTX-M1组、2组、9组和8/25/26组[30-31]、blaSHV和blaTEM和碳青霉烯酶blaKPC[32]。
1.2.4 统计学方法 采用IBM SPSS statistics 20软件进行统计分析,计数资料组间比较采用χ2检验或Fisher's exact test,P<0.05为差异有统计学意义。
2 结果
2.1 肺炎克雷伯菌临床株来源
112株肺炎克雷伯菌临床株主要来源于痰标本(76株,67.9%),19.6%来源于尿标本(22株),血培养(5株)和其他无菌体液(4株)标本共占8.0%。
94.6%(106株)的受试菌株分离自住院患者,共涉及11个科室,其中老年科(25/106,23.6%)、脑外科(23/106,21.7%)、康复科(19/106,17.9%)和ICU(12/106,11.3%)为主要科室来源,综合病房、普外科、感染科、神经内科、内分泌科、肾内科、消化科、骨科等亦有分布。
2.2 肺炎克雷伯菌临床株药敏试验结果
受试肺炎克雷伯菌对喹诺酮类耐药程度高(表 1)。
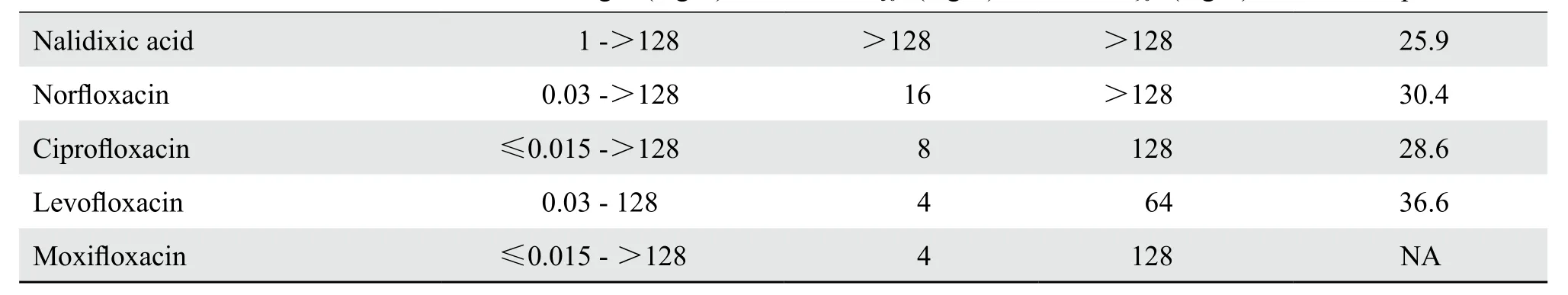
表1 112株肺炎克雷伯菌临床株对常用喹诺酮类的药敏试验结果Table 1 Susceptibility profile of 112 K. pneumoniae strains to the commonly used quinolones
2.3 MLST
受试菌株在环丙沙星各MIC层均有分布,故根据该药MIC分层,选取代表性菌株48株进行MLST。选择菌株包括全部15株环丙沙星MIC≥128 mg/L菌株、13株MIC≤0.015 mg/L菌株中的9株、以及其间各MIC梯度分别1~4株。
48株肺炎克雷伯菌临床株进行MLST分析,结果归为4个克隆复合体(CC)、22个ST型:最常见的为CC1型(均为ST11),CC2~CC4型各含2~3株临床株,余菌株不属于任何CC型。值得注意的是,仅次于ST11型(15株),18.8%受试肺炎克雷伯菌为ST494型(9株),余ST型各含1~2株受试株。
环丙沙星敏感率下降菌株中存在克隆传播趋势:CC1(ST 11)、CC4(ST15和ST655)和ST494菌株均对环丙沙星不敏感(MIC分别为16~>128 mg/L、4~128 mg/L和2~8 mg/L),以上各ST型菌株共占环丙沙星不敏感菌株86.2%(25/29)。而19株敏感菌株分属17个ST分型,未呈现明显克隆集中现象。
2.4 ST494型肺炎克雷伯菌耐药性及耐药机制
2.4.1 临床背景 ST分型共发现9株ST494型菌株,均分离自不同患者;标本来源于痰(7株)、尿和伤口分泌物;3株分离自同一病区,3株分离自脑外科3个不同病区,ICU、骨科和肾内科各分离1株。
2.4.2 耐药表型 ST494型菌株对庆大霉素、萘啶酸、头孢他啶、头孢噻肟均耐药;但对四环素、替加环素均敏感。比较ST494型、ST11型和非ST494/11型3组菌株对受试常用抗菌药物的敏感率,结果有显著差异:ST11型菌株对氟喹诺酮类的耐药率显著高于ST494型和非ST494/11型菌株;ST11型菌株对阿米卡星和β内酰胺类的耐药率也高于非ST494/11型菌株、而与ST494型菌株无明显差异;ST11型菌株对替加环素和四环素的耐药率与非ST494/11型无显著差异、而对四环素的耐药率高于ST494型菌株(P=0.006);ST494型菌株对受试氨基糖苷类、喹诺酮类、β内酰胺类药物敏感率均低于非ST494/11菌株(表2)。
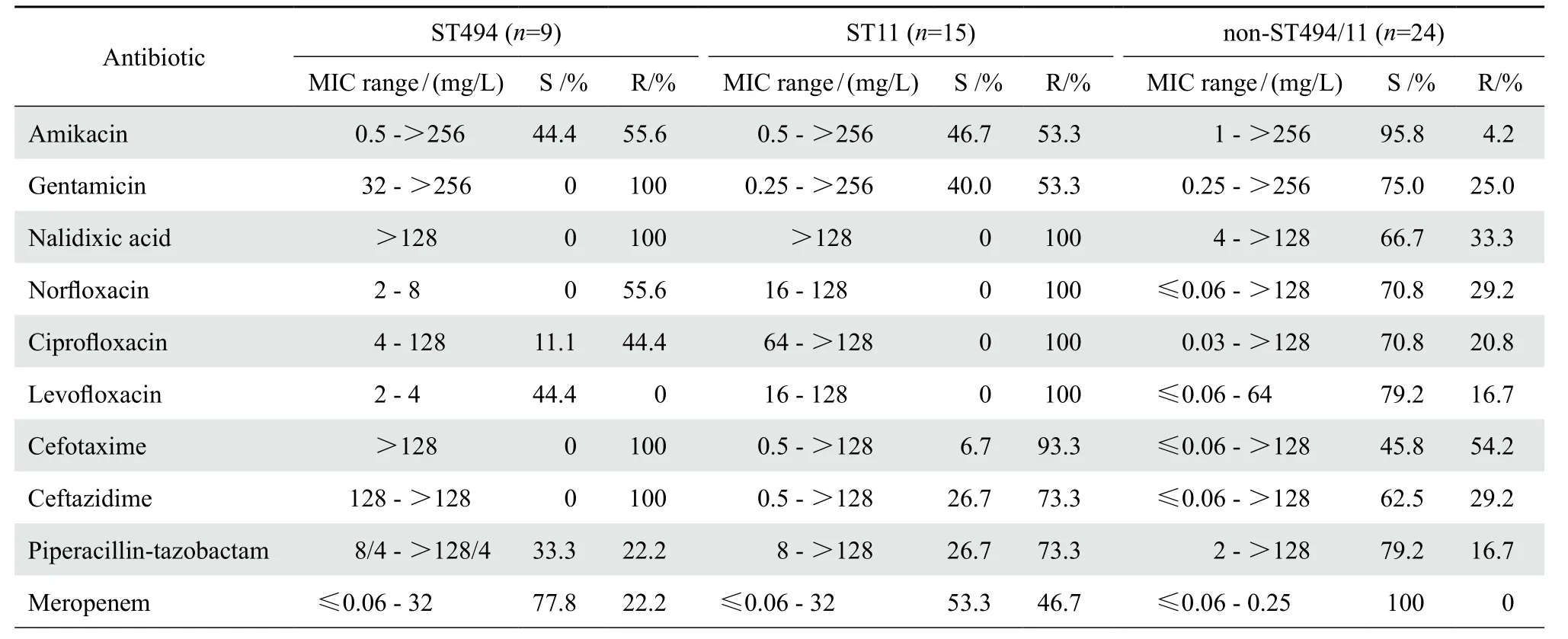
表2 ST494型、ST11型与非ST494/11型肺炎克雷伯菌临床株对抗菌药物敏感性Table 2 Susceptibility of K. pneumoniae strains - antimicrobial agents in terms of ST494, ST11 and non-ST494/11
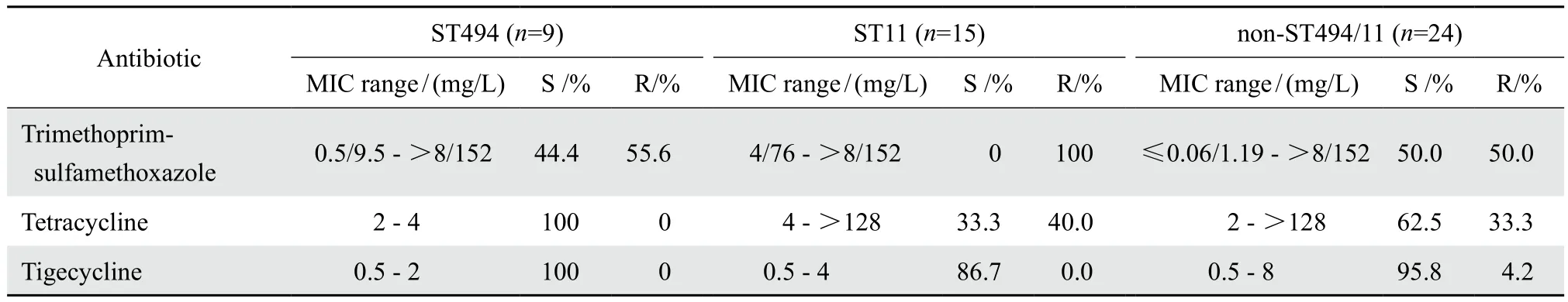
表2(续)Table 2(continued)
2.4.3 耐药基因 5株对阿米卡星、庆大霉素高度耐药(MIC均>256 mg/L)的菌株均携带armA基因,9株ST494型菌株均未检出rmtB、rmtD基因。
9株ST494型肺炎克雷伯菌临床株gyrA的QRDR均存在单位点突变,其中8株为S83Y、1株为S83I,praC的QRDR未发现有意义点突变。受检肺炎克雷伯菌均携带多药外排泵基因oqxAB,根据既往文献[23],考虑为染色体携带。对9株ST494型肺炎克雷伯菌进行质粒介导喹诺酮耐药基因筛查,其中3株同时携带qnrD和aac(6')-lb-cr基因,1株携带aac(6')-lb-cr基因,未检出qnrA、qnrS、qnrB、qnrC或qepA基因。
9株均同时携带blaCTX-M1组、blaSHV和blaTEM基因,其中2株同时产blaKPC酶,这2株菌对哌拉西林-他唑巴坦、美罗培南耐药。
3 讨论
肺炎克雷伯菌临床株对常用抗菌药耐药物日益严重,使临床抗感染治疗面临极大挑战。我院分离肺炎克雷伯菌临床株对环丙沙星敏感率为28.6%,远低于同期CHINET数据[33]。经MLST分析提示,我院分离环丙沙星不敏感肺炎克雷伯菌临床株中存在克隆传播趋势:CC1(ST11)、CC4(ST15和ST655)以及ST494菌株共占环丙沙星不敏感菌株的86.2%。既往报道ST494型菌株仅在产KPC酶肺炎克雷伯菌临床株被发现,占受试菌株0.5%(2/378)~5.7%(2/35)[34-35]。而本研究发现ST494型菌株占受试临床株18.9%,居于第2位。ST494型成为临床分离肺炎克雷伯菌主要克隆之一,这一现象为我院特有。这或可解释我院肺炎克雷伯菌菌株对氟喹诺酮类敏感率甚低这一现象。当然,本研究菌株仅为短期内集中收集,可能存在选择偏倚,有待更大规模研究佐证。
为了解ST494型菌株对我院肺炎克雷伯菌临床株耐药性影响,对其进行进一步分析,发现除对替加环素和四环素100%敏感、美罗培南敏感率78%外,其对常用抗菌药物敏感率均低于50%。ST494型菌株均携带blaCTX-M1组、blaSHV和blaTEM等多种β内酰胺酶基因,因而对所有头孢菌素均耐药;其中22.2%菌株同时产KPC酶,导致对碳青霉烯类耐药;ST494型菌株均存在gyrA的QRDR单位点突变,均携带oqxAB基因,部分携带qnrD和aac-(6')-lb-cr,导致对喹诺酮类敏感率下降;对氨基糖苷类耐药率高达56%~100%,产16S rRNA甲基化酶armA是其主因。有意思的是,ST494型肺炎克雷伯菌不仅对替加环素均敏感,而且对四环素也有很高敏感率,MIC仅2~4 mg/L。
qnrD基因首先在我国临床分离沙门菌中被发现[26],接合转入该基因能使受体菌环丙沙星MIC提高32倍(从0.002到0.06 mg/L)。qnrD基因多见于沙门菌属、变形杆菌属和大肠埃希菌中,检出率低于qnrA、qnrB或qnrS。检索PubMed和EMBASE数据库发现仅报道临床分离肺炎克雷伯菌携带qnrD基因2株:其一来自我国某医院分离人类尿标本菌株,同时携带aac(6')-lb-cr基因[36];另一来自匈牙利某大学医院血培养标本,同时产ESBL[37]。而本研究在ST494型肺炎克雷伯菌中检出携带qnrD肺炎克雷伯菌临床株3株,阳性率高达33.3%,或为该ST型肺炎克雷伯菌基因特征之一。
ST494型肺炎克雷伯菌临床株在我院为常见克隆之一,其对喹诺酮类、头孢菌素类、氨基糖苷类、碳青霉烯类等多种常用抗菌药物均有较高耐药率,或为我院肺炎克雷伯菌临床株对喹诺酮类高度耐药的原因之一;但ST494型菌株对四环素、替加环素均敏感,在替加环素价格昂贵且无口服制剂的现状下,价格低廉、生物利用度高、可静脉/口服序贯使用的四环素类药物,如多西环素、米诺环素,不失为治疗肺炎克雷伯菌感染的良好选择之一。
[1] BROBERG CA, PALACIOS M, MILLER VL.Klebsiella: a long way to go towards understanding this enigmatic jet-setter[J].F1000 Prime Rep, 2014, 6 :64.
[2] SIU LK, YEH KM, LIN JC, et al.Klebsiella pneumoniaeliver abscess: a new invasive syndrome[J]. Lancet Infect Dis, 2012,12(11):881-887.
[3] TZOUVELEKIS LS, MARKOGIANNAKIS A, PSICHOGIOU M, et al. Carbapenemases inKlebsiella pneumoniaeand otherEnterobacteriaceae: an evolving crisis of global dimensions[J].Clin Microbiol Rev, 2012, 25(4):682-707.
[4] DIANCOURT L, PASSET V, VERHOEF J,et al. Multilocus sequence typing ofKlebsiella pneumoniaenosocomial isolates[J].J Clin Microbiol,2005, 43(8):4178-4182.
[5] GUO C, YANG X, WU Y, et al. MLST-based inference of genetic diversity and population structure of clinicalKlebsiella pneumoniae, China[J]. Sci Rep, 2015, 5 : 7612.
[6] XU X,LI X,LUO M, et al. Molecular characterisations of integrons in clinical isolates ofKlebsiella pneumoniaein a Chinese tertiary hospital[J]. Microb Pathog,2017, 104 : 164-170.
[7] XIAO SZ, WANG S, WU WM, et al. The Resistance phenotype and molecular epidemiology ofKlebsiella pneumoniaein bloodstream infections in Shanghai, China, 2012-2015[J]. Front Microbiol, 2017, 8 :250.
[8] HRABÁK J, PAPAGIANNITSIS CC, ŠTUDENTOVÁ V,et al. Carbapenemase-producingKlebsiella pneumoniaein the Czech Republic in 2011[J]. Eur Surveill, 2013, 18(45):20626.
[9] HU F, MUNOZ-PRICE LS, DEPASCALE D, et al.Klebsiella pneumoniaesequence type 11 isolate producing RmtG 16S rRNA methyltransferase from a patient in Miami, Florida[J].Antimicrob Agents Chemother, 2014, 58(8):4980-4981.
[10] LI JJ, SHENG ZK, DENG M, et al. Epidemic ofKlebsiella pneumoniaeST11 clone coproducing KPC-2 and 16S rRNA methylase RmtB in a Chinese University Hospital[J]. BMC Infect Dis, 2012, 12 :373.
[11] NAGASAKA Y, KIMURA K, YAMADA K,et al. Genetic profiles of fluoroquinolone-nonsusceptibleKlebsiella pneumoniaeamong cephalosporin-resistantK. pneumonia[J]. Microb Drug Resist, 2015, 21(2):224-233.
[12] BI W, LIU H, DUNSTAN RA, et al. Extensively drugresistantKlebsiella pneumoniaecausing nosocomial bloodstream infections in China: molecular investigation of antibiotic resistance determinants, informing therapy, and clinical outcomes[J]. Front Microbiol, 2017, 8 :1230.
[13] SUN K, CHEN X, LI C, et al. Clonal dissemination of multilocus sequence type 11Klebsiella pneumoniaecarbapenemase-producingK. pneumoniaein a Chinese teaching hospital[J]. Apmis,2015, 123(2):123-127.
[14] JIAO Y, QIN Y, LIU J, et al. Risk factors for carbapenemresistantKlebsiella pneumoniaeinfection/colonization and predictors of mortality: a retrospective study[J]. Pathog Glob Health, 2015, 109(2):68-74.
[15] MUNOZ-PRICE LS1, POIREL L, BONOMO RA, et al.Clinical epidemiology of the global expansion ofKlebsiella pneumoniaecarbapenemases[J]. Lancet Infect Dis, 2013, 13(9):785-796.
[16] QI YAN,WEI ZEQING, JI SHUJUAN, et al. ST11, the dominant clone of KPC-producingKlebsiella pneumoniaein China[J]. J Antimicrob Chemother, 2011, 66(2):307-312.
[17] CHENG L, CAO XL, ZHANG ZF, et al. Clonal dissemination of KPC-2 producingKlebsiella pneumoniaeST11 clone with high prevalence of oqxAB and rmtB in a tertiary hospital in China: results from a 3-year period[J]. Ann Clin Microbiol Antimicrob, 2016,15:1.
[18] ZHONG X, XU H, CHEN D, et al. First emergence of acrAB and oqxAB mediated tigecycline resistance in clinical isolates ofKlebsiella pneumoniaepre-dating the use of tigecycline in a Chinese hospital[J]. PLoS One, 2014, 9(12):e115185.
[19] CHIU SK, CHAN MC, HUANG LY, et al. Tigecycline resistance among carbapenem-resistantKlebsiella Pneumoniae:clinical characteristics and expression levels of efflux pump genes[J]. PLoS One,2017, 12(4): e0175140.
[20] Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing[S]. Twentyseventh informational supplement. CLSI document M100-S25.Wayne, PA : 2017.
[21] ZHOU Y, YU H, GUO Q, et al. Distribution of 16S rRNA methylases among different species of Gram-negative bacilli with high-level resistance to aminoglycosides[J]. Eur J Clin Microbiol Infect Dis, 2010, 29(11):1349-1353.
[22] RODRIGUEZ-MARTINEZ JM, VELASCO C, PASCUAL A,et al. Correlation of quinolone resistance levels and differences in basal and quinolone-induced expression from three qnrA-containing plasmids[J]. Clin Microbiol Infect, 2006, 12(5):440-445.
[23] YUAN J, XU X, GUO Q,et al. Prevalence of the oqxAB gene complex inKlebsiella pneumoniaeandEscherichia coliclinical isolates[J]. J Antimicrob Chemother, 2012, 67(7):1655-1659.
[24] ROBICSEK A, STRAHILEVITZ J, SAHM DF, et al. qnr prevalence in ceftazidime-resistantEnterobacteriaceaeisolates from the United States[J]. Antimicrob Agents Chemother, 2006,50(8):2872-2874.
[25] NOVAIS A, CANTÓN R, VALVERDE A, et al. Dissemination and persistence ofblaCTX-M-9 are linked to class 1 integrons containing CR1 associated with defective transposon derivatives from tn402 located in early antibiotic resistance plasmids of IncHI2, IncP1-α, and IncFI groups[J]. Antimicrob Agents Chemother, 2006, 50(8): 2741-2750.
[26] CAVACO LM, HASMAN H, XIA S, et al.qnrD, a novel gene conferring transferable quinolone resistance in salmonella enterica serovar kentucky and bovismorbificans strains of human origin[J]. Antimicrob Agents Chemother, 2009, 53(2): 603-608.
[27] CATTOIR V, POIREL L, ROTIMI V, et al. Multiplex PCR for detection of plasmid-mediated quinolone resistanceqnrgenes in ESBL-producing enterobacterial isolates[J]. J Antimicrob Chemother, 2007, 60(2):394-397.
[28] PARK CH, ROBICSEK A, JACOBY GA, et al. Prevalence in the United States ofaac(6')-Ib-crencoding a ciprofloxacinmodifying enzyme[J]. Antimicrob Agents Chemother, 2006, 50(11):3953-3955.
[29] WANG M, SAHM DF, JACOBY GA, et al. Emerging plasmidmediated quinolone resistance associated with the qnr gene inKlebsiella pneumoniaeclinical isolates in the United States[J].Antimicrob Agents Chemother, 2004, 48(4):1295-1299.
[30] WOODFORD N, FAGAN EJ, ELLINGTON MJ. Multiplex PCR for rapid detection of genes encoding CTX-M extendedspectrum (beta)-lactamases[J]. J Antimicrob Chemother,2006, 57(1):154-155.
[31] PATRICK RM, ELLEN JB, JAMES HJ, et al. Detection and characterization of antimicrobial resistance genes in pathogenic bacteria[M]//KAMILE RASHEED J, FRANKLIN C, FRED TC. Manual of Clinical Microbiology, 9th ed. American Society for Microbiology, Washington DC, 2007 : 1250-1256.
[32] WANG P, CHEN S, GUO Y, et al. Occurrence of false positive results for the detection of carbapenemases in carbapenemasenegativeEscherichia coliandKlebsiella pneumoniaeisolates[J].PLoS One, 2011, 6(10):e26356.
[33] 胡付品,朱德妹,汪复,等. 2010年中国CHINET细菌耐药性监测[J]. 中国感染与化疗杂志,2011,11(5):321-329.
[34] GIAKKOUPI P, PAPAGIANNITSIS CC, MIRIAGOU V,et al. An update of the evolving epidemic ofblaKPC-2-carryingKlebsiella pneumoniaein Greece (2009-10)[J]. J Antimicrob Chemother, 2011, 66(7):1510-1513.
[35] WANG LH, WEI DD, WAN LG, et al. Diversity of the genetic environment of theblaKPC-2 gene amongKlebsiella pneumoniaeclinical isolates in a Chinese hospital[J]. Microb Drug Resist, 2016, 22(1):15-21.
[36] ZHANG S, SUN J, LIAO XP, et al. Prevalence and plasmid characterization of the qnrD determinant inEnterobacteriaceaeisolated from animals, retail meat products, and humans[J].Microb Drug Resist, 2013, 19(4):331-335.
[37] DOMOKOS J, SZABO D, KOCSIS B, et al. Detection of plasmid-mediated quinolone resistance determinants in ESBL producing enterobacteriaceae strains. Acta Microbiologica et Immunologica Hungarica[C]. Conference: 17th International Congress of the Hungarian Society for Microbiology.Budapest Hungary. Conference Publication: (var. pagings),2015:145.
