鸡源大肠杆菌CRISPR特征及其与耐药性的关系
2018-05-24王文静张瑞良代兴杨杨艳菲徐园园
李 琳,赵 霞,王 磊,王文静,张瑞良,代兴杨,杨艳菲,徐园园
(安徽农业大学 动物科技学院,安徽 合肥 230036)
大肠杆菌(Escherichiacoli)是肠道内的共生菌,也是导致畜牧业细菌性疾病最常见的病原菌,大肠杆菌在抗菌药物压力下易产生耐药性,且可通过菌毛传播耐药质粒,这给大肠杆菌病的免疫预防带来了极大挑战。
近年来研究发现,成簇间隔短回文重复序列(Clustered regularly interspaced short palindromicrepeats,CRISPR)广泛分布于40%细菌和90%古细菌的基因组中[1-3]。CRISPR位点由一个前导区(Leader)、多个长度为22~47 bp的重复序列(Repeat sequence)及数个与重复序列长度相近的间隔序列(Spacers sequence)组成,这些重复序列与间隔序列相互交替串联形成一个完整的CRISPR位点[4]。富含AT的前导序列由相邻的蛋白基因(CRISPR associated,CAS)组成,与第1个重复序列连接,在种内比较保守,在种间存在差异[5]。CRISPR与其CRISPR/CAS系统具有识别和处理质粒等外源基因并产生获得性免疫的能力,能够有效地抵御外源基因的水平转移[6]。当外源噬菌体、质粒首次侵袭原核生物时,CRISPR系统会选择性地截取并保留某些外源基因片段,并将外源基因片段整合到CRISPR中形成新的间隔序列,使宿主获得抵抗相应噬菌体、质粒的能力[5]。不同菌种中CRISPR与耐药性的关系并不一致。目前CRISPR系统的应用主要有细菌分型[7]、细菌进化分析[8]、抵御噬菌体[9]和CRISPR/CAS9介导的基因组定点编辑技术[10-11]等,尚未见对鸡源大肠杆菌CRISPR与耐药性关系的相关研究。为此,本研究检测了合肥地区鸡源大肠杆菌中的CRISPR,分析其与耐药性的关系,并对CRISPR的结构和功能进行生物信息学分析,旨在为进一步研究CPISPR的功能及其对细菌耐药的影响奠定基础。
1 材料与方法
1.1 材 料
1.1.1 菌 株 2012年3-5月,从合肥地区4个养鸡场鸡肛拭子中分离大肠杆菌,经大肠杆菌显色培养基和鸡肠杆菌科细菌生化鉴定,共鉴定出130株大肠杆菌(编号1~130);大肠杆菌ATCC25922由安徽农业大学兽医药理与毒理研究室保存。
1.1.2 药 品 阿莫西林、土霉素、氧氟沙星、头孢噻呋、头孢曲松、庆大霉素、安普霉素、沙拉沙星、阿米卡星、氟苯尼考、恩诺沙星、头孢喹诺、强力霉素、洛美沙星、利福平和磺胺-6-甲氧嘧啶等16种抗菌药物,购自中国药品生物制品鉴定所。
1.1.3 试 剂 PCR相关试剂(PremixTaq、Loading Buffer、DNA Marker),购自日本TaKaRa公司;沙门菌基因组DNA提取试剂盒,购自OMEGA Bio-tek公司;脑心浸液(BHI)培养基,购自杭州微生物试剂有限公司;琼脂粉,购自BIOWEST公司;50×TAE,购自上海生工生物工程股份有限公司。其余常规试剂均为国产分析纯级试剂。
1.2 耐药性测定
根据CLSI(2015 版)指导原则和执行标准,用微量肉汤稀释法,测定130株分离菌对16种抗菌药物的敏感性,以大肠杆菌ATCC25922作为质控菌株。
1.3 CRISPR序列的PCR扩增
1.3.1 引物设计 根据CRISPR database公布的大肠杆菌CRISPR结构序列,运用Oligo软件设计引物序列[12],交由上海生工生物工程股份有限公司进行合成。CRISPR基因的上游引物序列为:5′-TACCGTTGGTGAAGGAGCTG-3′,下游引物序列为:5′-TTCCGGTGGATTTGGATGGG-3′,引物退火温度为53 ℃,预期扩增片段为1 000 bp左右。
1.3.2 PCR扩增 以大肠杆菌菌液为模板,进行CRISPR扩增。扩增体系为25 μL,其中ddH2O 11 μL,上、下游引物各0.5 μL,菌液0.5 μL,2×TaqPCR Premix 12.5 μL。反应条件为:95 ℃预变性10 min;94 ℃变性45 s,53 ℃退火45 s,72 ℃延伸45 s,35个循环;72 ℃延伸10 min。取5 μL PCR扩增产物进行1%琼脂糖凝胶电泳,凝胶电泳的反应条件为:电压100 V、电流80 mA、时间35 min,用凝胶成像仪观察并记录结果。根据PCR结果,挑选扩增片段长度不同的12株大肠杆菌(即3,11,27,31,40,65,78,87,105,117,128,129号菌株),切胶回收CRISPR扩增产物,送上海英潍捷基贸易有限公司测序。
1.4 CRISPR序列分析
将12株大肠杆菌CRISPR的测序结果按片段长度相同或相近分成4组,分别为:片段长度约850 bp(40号菌(874 bp),87号菌(851 bp),129号菌(822 bp))、910 bp(27号菌(913 bp),105号菌(913b p),117号菌(913 bp),128号菌(917 bp))、1 150 bp(3号菌(1 121 bp),11号菌(1 243 bp))和1 650 bp(31号菌(1 760 bp),78号菌(1 610 bp),65号菌(1 614 bp))。运用CRISPR web server网站中的CRISPR Finder工具查找每个菌株的重复序列和间隔序列并进行序列比对[13]。采用RNAfold web server (http://rna.tbi.univie.ac.at/cgi-bin/RNAfold.cgi)预测序列的二级结构[14],通过CRISPRTarget分析间隔序列的同源性[15]。
2 结果与分析
2.1 大肠杆菌的耐药性
由表1可知,130株大肠杆菌菌株对16种抗菌药物的耐药率在2.3%~97.7%,130株大肠杆菌中有118株是3耐及3耐以上菌株(以下称多重耐药菌株)。其中对土霉素和磺胺-6-甲氧嘧啶的耐药率在95.0%以上,对强力霉素、阿莫西林、头孢曲松、氧氟沙星、洛美沙星、利福平、氟苯尼考和恩诺沙星这8种药的耐药率均在50.0%以上,对庆大霉素、安普霉素、头孢噻呋、阿米卡星、头孢喹诺和沙拉沙星这6种药的耐药率较低。
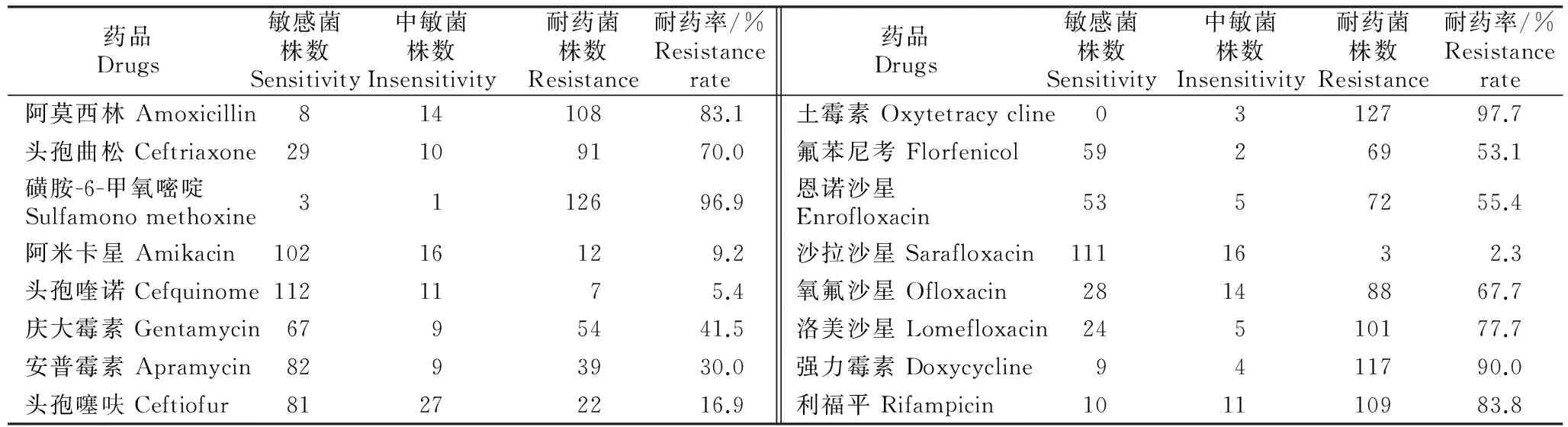
表1 130株鸡源大肠杆菌耐药性的测定结果Table 1 Drug resistance test of 130 isolates
2.2 CRISPR的PCR扩增
试验发现,130株菌中有39株能够扩增出CRISPR,阳性检出率为30%,部分凝胶成像结果如图1所示。对39株阳性菌株的CRISPR再次进行扩增,结果如图2所示。
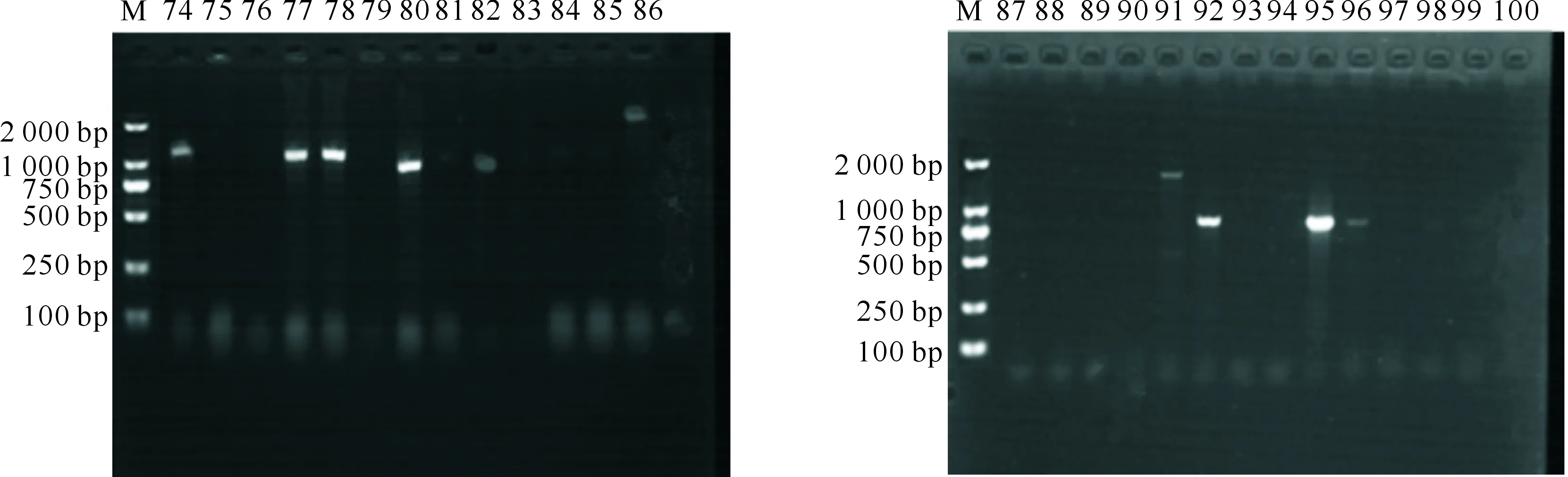
M.DNA Marker;74~100.分离菌株编号,其中74,77,78,80,82,86,91,92,95,96号菌为阳性菌,其余菌株为阴性菌M.DNA Marker;74-100.Isolates,74,77,78,80,82,86,91,92,95 and 96 bacteria are positive and the other strains are negative图1 部分鸡源大肠杆菌CRISPR的PCR扩增Fig.1 PCR results of some chicken-derived Escherichia coli CRISPR

M.DNA Marker; 泳道上数据为阳性菌株编号M.DNA Marker;Data on the lane are numbers of positive strains图2 39株CRISPR阳性菌株的PCR扩增Fig.2 PCR results of 39 CRISPR positive strains
2.3 CRISPR与耐药性的关系分析
由表2可知,CRISPR阳性菌株的多重耐药率显著高于CRISPR阴性菌株,推测大肠杆菌的多重耐药性可能与CRISPR具有相关性。
由CRISPR比对结果可知,27,105,117,128号菌株CRISPR的同源性较高(99.6%),其余菌株CRISPR之间的同源性都较低,有12株多重耐药大肠杆菌CRISPR之间的同源性仅有42.49%(图3),提示这12株多重耐药大肠杆菌CRISPR的差异性与耐药性之间无直接相关性。

表2 CRISPR与鸡源大肠杆菌多重耐药性的相关性分析Table 2 Correlation analysis of CRISPR and multi-drug resistance
注:同列数据后标不同小写字母表示χ2分析差异显著(P<0.05)。
Note:Differernt lowercase superscripts in each column mean significant by chi-squared test (P<0.05).
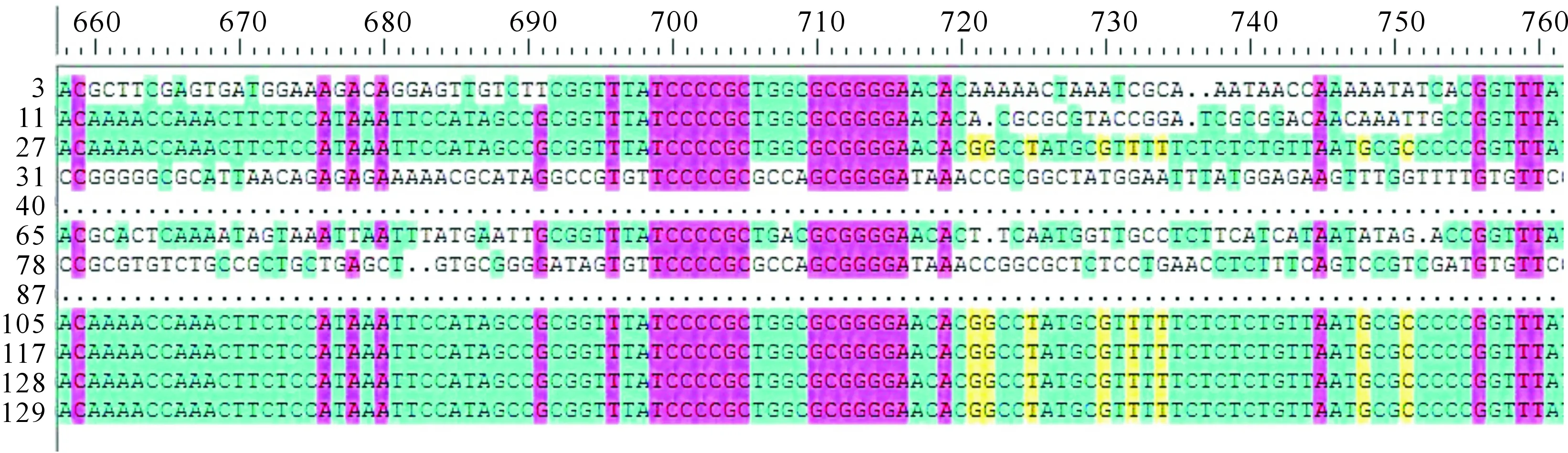
图3 12株鸡源大肠杆菌的CRISPR比对(部分结果)Fig.3 CRISPR sequence alignment of 12 strains of chicken-derived Escherichia coli (partial result)
2.4 大肠杆菌CRISPR的特征分析
2.4.1 重复序列 同源性较低的12株大肠杆菌均包含2条(11号菌株除外)长度为29 bp的重复序列,且这2条序列反向互补形成短回文序列,分为a、b、c、d、e 5类,具体见表3。其中,a和b为31,78,87,40号菌共有重复序列;c为65,128,27,105,117,129号菌共有重复序列;d为65,128,27,105,117,129,11和3号菌共有重复序列;e为3号大肠杆菌重复序列。a和b这2类重复序列从5′到3′端的第13个碱基不同;c和d这2类重复序列从5′到3′端的第17个碱基不同;d和e这2类重复序列从5′端到3′端的前2个碱基和最后1个碱基不同。
在古细菌中的试验证实,CRISPR结构中的重复序列可以转录为RNA并形成茎环结构,这种结构在CRISPR发挥免疫功能的过程中是一种结构标识物,CAS蛋白通过识别该茎环结构而发挥剪切作用,并使得长链的pro-crRNA形成短链的crRNA,从而发挥免疫功能[16]。本研究对12株大肠杆菌CRISPR重复序列的二级结构进行预测,结果(图4)显示,根据茎环特征,12株菌株的二级结构可以分为2类:一类以图4中a、b、c、d序列为代表,在整个结构中间含有唯一的茎部分,一大一小2个环分布在茎的两端,该类结构以“环”为主;另一类以图4中的e序列为代表,形成多个茎、环结构,该结构特征是茎环依次形成。图4中a、b、c、d序列茎环结构的头部环都由6个碱基组成,茎区都是5个碱基长度,a、b序列中间部分还有9个碱基组成的环;图4中e序列头部环由6个碱基组成,中间的环由7个碱基组成,两环间的茎区是5个碱基长度,尾部茎长3个碱基。各重复序列茎环结构的尾部差异较大,图4中a、b序列尾部较短,仅2个碱基,而c、d序列尾部长达13碱基, e序列按照最小自由能原则,尾部没有游离的碱基。a和b序列的最小自由能都为-15.20 kJ/mol,c和d序列的最小自由能都为-14.20 kJ/mol,e序列的最小自由能为-14.90 kJ/mol。
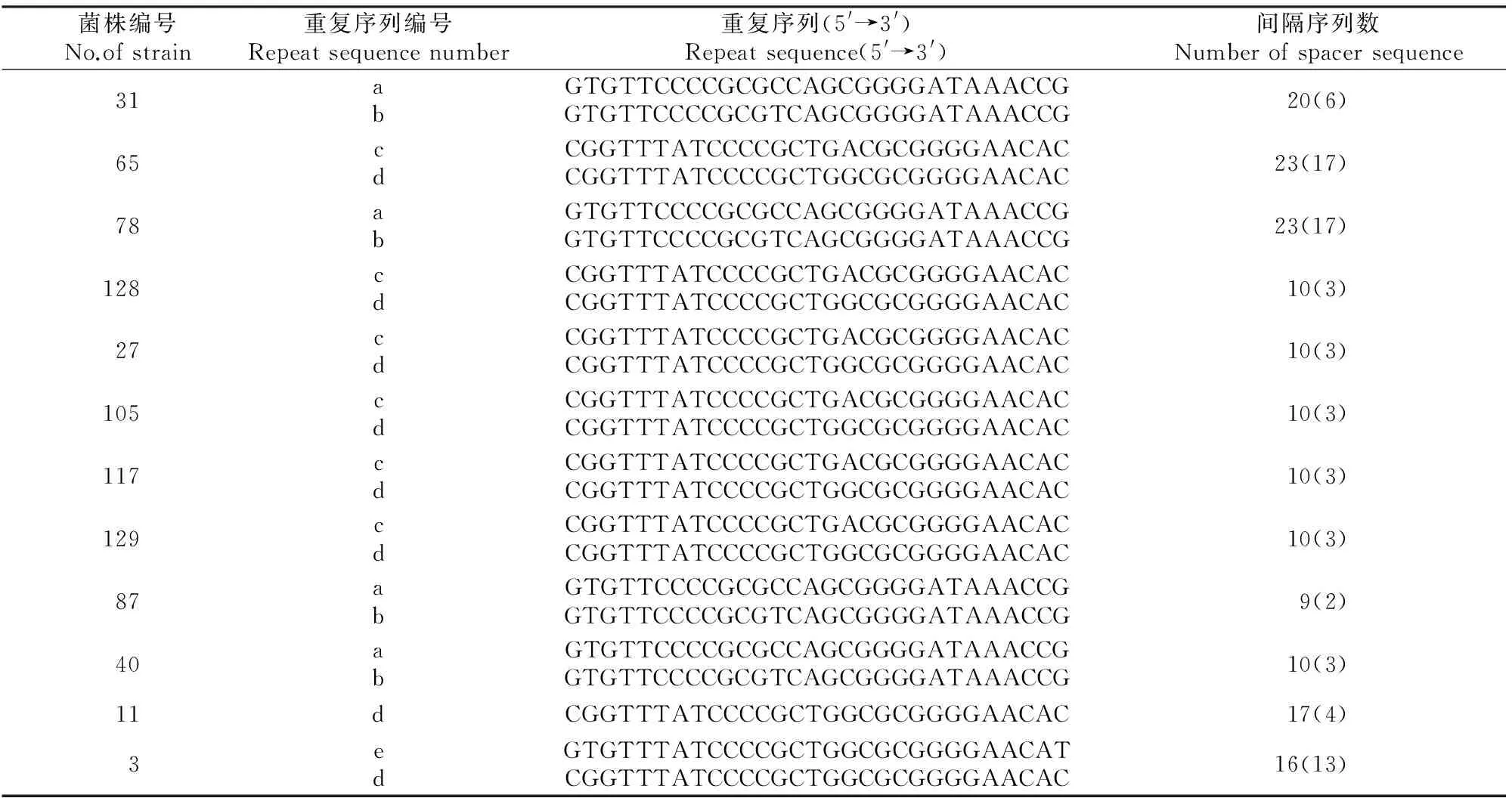
表3 鸡源大肠杆菌CRISPR序列分析Table 3 Information of chicken-derived E.coli CRISPR sequences
注:括号中数据为新的间隔序列。
Note:Data in parentheses represent new spacers.

图4 鸡源大肠杆菌CRISPR重复序列的二级结构预测Fig.4 Prediction of secondary structure of chicken-derived E.coli CRISPR repeat sequence
以5类重复序列构建系统进化树,结果(图5)显示共形成了2大聚类分支,但同一种属大肠杆菌的重复序列并不完全聚集于同一聚类分支中。
2.4.2 间隔序列 CRISPR Finder软件从同源性较低的12株大肠杆菌CRISPR中共识别出间隔序列168条,将其与CRISPR数据库中大肠杆菌的间隔序列进行比对发现,其中91条为CRISPR数据库中已有序列,77条为新发现间隔序列(表3)。本研究对12株大肠杆菌的间隔序列进行BLAST同源性比对(比对数据库包括GenBank-Phage、RefSeq-Plasmid),结果发现有56条无比对结果,有比对结果的112条间隔序列中,72.3%来源于质粒,25.0%来源于噬菌体,2.7%来源于细菌和病毒。

图5 鸡源大肠杆菌CRISPR重复序列系统进化树Fig.5 Phylogenetic tree of chicken-derived E.coli CRISPR repeat sequences
3 讨 论
3.1 CRISPR与大肠杆菌的耐药性
CRISPR在原核生物中多应用于基因定点编辑技术、细菌分型、细菌进化等方面的研究,而有关CRISPR与耐药关系的研究较少。Burley等[17]检测了粪肠球菌的CRISPR相关蛋白基因,发现其与多重耐药呈负相关,Palmer等[18]分析粪肠球菌的CRISPR/CAS,发现CRISPR/CAS与获得性耐药呈负相关。但有研究表明,大肠杆菌的CAS基因表达量在敏感株和耐药株间无差异,并且耐药质粒可以在CRISPR阳性大肠杆菌之间传播[19]。王琳琳等[20]对志贺菌CRISPR与耐药性关系进行研究发现,CRISPR重复序列相对保守,而间隔序列多变且与多重耐药性显著相关。
本研究对合肥地区鸡源大肠杆菌CRISPR流行分布情况进行调查,结果发现130株大肠杆菌中有39株携带CRISPR,检出率为30%,这与王琳琳等[20]“志贺菌CRISPR的检出率为93%”相比较低,这可能是由于菌种差异所导致。本研究通过对大肠杆菌CRISPR阳性和阴性菌株的多重耐药率进行比较发现,CRISPR阳性菌株的多重耐药率显著高于CRISPR阴性菌株,可见CRISPR阳性菌株比阴性菌株更容易产生多重耐药性,说明合肥地区鸡源大肠杆菌中存在CRISPR的流行,且与耐药表型具有相关性。但本研究发现,CRISPR序列差异性与耐药性之间无相关性,其原因尚需进一步研究。
3.2 CRISPR的特征
CRISPR结构是一种抵抗外来遗传物质入侵的防御系统,近年来对其研究逐步深入。本研究对大肠杆菌CRISPR的重复序列和间隔序列进行了系统的生物信息学分析。
有研究指出,大多数CRISPR重复序列存在保守序列,如5′端的GTT和3′端的GAAA,且重复序列具有回文序列,可以转录并形成RNA二级结构,形成茎环结构[21-22]。茎环结构已经被证明在CRISPR系统中发挥着识别功能[23],本研究对重复序列RNA二级结构进行预测,发现5类重复序列都能形成回文结构,但茎环大小有差异,表明正向重复序列可能介导外源DNA或RNA与CAS编码蛋白的相互作用。以重复序列构建系统进化树,结果显示共形成了2大聚类分支,但同一种属大肠杆菌的重复序列并不一定在同一聚类分支中。
间隔序列是CRISPR系统中最重要的部分,现有研究表明,间隔序列与噬菌体、质粒等外源基因有同源性[24]。间隔序列可靶向定位入侵的外源基因[25],CRISPR系统会选择性地截取并保留某些外源基因片段,并将外源基因片段整合到CRISPR系统中形成新的间隔序列,从而抵御外源基因的再次侵入。本研究对同源性较低的12株大肠杆菌间隔序列进行BLAST同源性比对,发现112条有比对结果的间隔序列中,72.3%来源于质粒,25.0%来源于噬菌体,2.7%来源于细菌和病毒,进一步证明间隔序列可能来源于外部的可移动遗传因子。
本研究发现,携带CRISPR的鸡源大肠杆菌广泛存在,CRISPR阳性菌株的多重耐药率显著高于CRISPR阴性菌株,预测大肠杆菌CRISPR的存在可能与多重耐药性具有相关性,而CRISPR的序列差异性与耐药性无相关性。本研究进一步对大肠杆菌CRISPR结构特征和序列功能进行探讨,发现重复序列有差异性,间隔序列多来源于外源基因,推测在抗生素选择压力下,细菌的CRISPR序列可能会发生变异,使细菌获得外源性耐药基因而存活下来,提示CRISPR重复序列的变异和间隔序列的多样性可能与大肠杆菌的耐药性有关。
[参考文献]
[1] Kunin V,Sorek R,Hugenholtz P.Evolutionary conservation of sequence and secondary structures in CRISPR repeats [J].Genome Biology,2007,8(4):R61.
[2] Lillesto P K,Redder P,Barrettr A,et al.A putative viral defence mechanism in archaeal cells [J].Archaea,2006,2(1):59-72.
[3] Mojica F J,Diez-Villasenor C,Soria E,et al.Biological significance of a family of regularly spaced repeats in the genomes of archaea,bacteria and mitochondria [J].Molecular Microbiology,2000,36(1):244-246.
[4] Barrangou R,Marraffini L A.CRISPR-Cas systems:prokaryotes upgrade to adaptive immunity [J].Mol Cell,2014,54(2):234-244.
[5] Jansen R,Van Embden J D,Gaastra W,et al.Identification of a novel family of sequence repeats among prokaryotes [J].OMICS A Journal of Integrative Biology,2002,6(1):23-33.
[6] Pourcel C,Salvignol G,Vergnaud G.CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA,and provide additional tools for evolutionary studies [J].Microbiology,2005,151(3):653-663.
[7] Cain A K,Boinett C J.A CRISPR view of genome sequences [J].Nat Rev Microbiol,2013,11(4):226.
[8] Comas I,Homolka S,Niemann S,et al.Genotyping of genetically monomorphic bacteria:DNA sequencing inMycobacteriumtuberculosishighlights the limitations of current methodologies [J].PLoS One,2009,4(11):e7815.
[9] Horvath P,Romero D A,Coute-Monvoisin A C,et al.Diversity,activity,and evolution of CRISPR loci inStreptococcusthermophilus[J].J Bacteriol,2008,190(4):1401-1412.
[10] Li T,Huang S,Zhao X,et al.Modularly assembled designer TAL effector nucleases for targeted gene knockout and gene replacement in eukaryotes [J].Nucleic Acids Res,2011,39(14):6315-6325.
[11] Joung J K,Sandrer J D.TALENs:a widely applicable technology for targeted genome editing [J].Nat Rev Mol Cell Biol,2013,14(1):49-55.
[12] Grissa I,Vergnaud G,Pourcel C.The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats [J].BMC Bioinformatics,2007,8:172.
[13] Bland C,Ramsey T L,Sabree F,et al.CRISPR recognition tool(CRT):a tool for automatic detection of clustered regularly interspaced palindromic repeats [J].BMC Bioinformatics,2007,8:209.
[14] 邓凯波,霍贵成.嗜热链球菌中CRISPR序列的检测与同源性分析 [J].食品科学,2013,34(3):153-157.
Deng K B,Huo G C.Detection and homology analysis of CRISPR inStreptococcusthermophilus[J].Food Science, 2013,34(3):153-157.
[15] Biswas A,Gagnon J N,Brouns S J,et al.CRISPRTarget:bioinformatic prediction and analysis of crRNA targets [J].RNA Biol,2013,10(5):817-827.
[16] Brouns S J,Jore M M,Lundgren M,et al.Small CRISPR RNAs guide a ntiviral defensein prokaryotes [J].Science,2008,321(5891):960-964.
[17] Burley K M,Sedgley C M.CRISPR-Cas,a prokaryotic adaptive immune system,in endodontic,oral,and multidrug-resistant hospital-acquiredEnterococcusfaecalis[J].Journal of Endodontics,2012,38(11):1511-1515.
[18] Palmer K L,Gilmore M S.Multidrug-resistant enterococci lack CRISPR-Cas [J].Mbio,2010,1(4):e00227-10.
[19] Toucho M,Charpentier S,Pognard D,et al.Antibiotic resistance plasmids spread among natural isolates ofEscherichiacoliin spite of CRISPR elements [J].Microbiology,2012,158:2997-3004.
[20] 王琳琳,王颖芳,段广才,等.志贺菌CRISPR的检测及其与耐药的关系 [J].微生物学报,2015,55(4):476-483.
Wang L L,Wang Y F,Duan G C,et al.Detection of CRISPR and its relationship to drug resistance in Shigella [J].Acta Microbiologica Sinica,2015,55(4):476-483.
[21] Haft D H,Selengut J,Mongodin E F,et al.A guild of 45 CRISPR-associated (Cas) protein families and multiple CRISPR/Cas subtypes exist in prokaryotic genomes [J].PLoS Comput Biol,2005,1(6):e60.
[22] Makarova K S,Grishin N V,Shabalina S A,et al.A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery,functional analogies with eukaryotic RNAi,and hypothetical mechanisms of action [J].Biol Direct,2006,1:7.
[23] 王 琰,喻 婵,王阶平,等.蜡状芽孢杆菌群中规律成簇间隔短回文重复序列的生物信息学分析 [J].中国生物工程杂志,2011,31(7):72-78.
Wang Y,Yu C,Wang J P,et al.Bioinformatics analysis of clustered regularly interspaced short palindromic repeats (CRISPR) in the genomes ofBacilluscereusgroup [J].China Biotechnology,2011,31(7):72-78.
[24] Kunin V,He S,Wamecke F,et a1.A bacterial metapopulation adapts locally to phage predation despite global dispersal [J].Genome Res,2008,18(2):293-297.
[25] 王琳琳.CRISPR/Cas系统与志贺菌耐药的关系 [D].郑州:郑州大学,2015.
Wang L L.The relationship between CRISPR/Cas system and drug resistance in Shigella [D].Zhengzhou:Zhengzhou University,2015.
