菊花AINTEGUMENTA克隆与功能分析
2018-05-14温立柱孙霞樊红梅郭芸珲于媛媛任红王文莉郑成淑
温立柱,孙霞,樊红梅,郭芸珲,于媛媛,任红,王文莉,郑成淑
菊花克隆与功能分析
温立柱,孙霞,樊红梅,郭芸珲,于媛媛,任红,王文莉,郑成淑
(山东农业大学园艺科学与工程学院/山东省中日韩菊花国际合作研究中心,山东泰安 271018)
【目的】从菊花中分离克隆,分析其序列特征和在菊花中的时空表达特性,通过基因沉默研究其对菊花花序发育的影响,分析可能的调控方式和潜在机理,为菊花花序大小的调控奠定理论基础。【方法】使用RACE方法从菊花中克隆全长,通过DNAMAN等软件对其序列进行分析。采用荧光定量的方式检测其在菊花不同器官和发育时期的表达。构建35S::-GFP融合蛋白载体进行亚细胞定位,构建TRV2-沉默表达载体侵染菊花。使用SPSS软件统计分析沉默系菊花表型变化,通过光学显微镜观察舌状花花瓣表皮细胞变化,使用荧光定量方式检测相关基因在沉默系中的表达。【结果】从菊花中克隆了全长,其编码的氨基酸序列长度为540,理论等电点为7.39,蛋白质的理论分子量为60.4 kD,含有两个AP2保守功能区域和VYL修饰位点。在与其他物种的ANT蛋白构建的系统进化树中,CmANT与AtANT聚在一起。荧光定量结果表明:(1)在花蕾中的表达量最高,其次是根>茎>叶。(2)在花序不同部位的比较中,舌状花中的表达量最高,其次是筒状花,在花萼中的表达量最低。(3)在舌状花中的表达随着发育时期的延续而降低。(4)在2,4-D诱导下的3—6 h内表达量持续上升。WoLF PSORT软件预测和35S::-GFP融合蛋白在洋葱表皮细胞中的定位结果显示,CmANT蛋白定位在植物细胞核中。TRV--1和TRV--2沉默系的平均花径相比对照分别减小18.93%和27.47%,舌状花的数目分别减少11.39%和14.66%,其中TRV--2与对照差异显著(<0.05),筒状花数目分别减少14.55%和36.56%。顶端花序的舌状花平均长度分别减少34.17%和54.68%,舌状花的宽度分别比对照减小24.05%和10.13%。叶片平均鲜重分别比对照组减小13.19%和21.98%,叶片鲜重与筒状花数目显著相关(<0.05)。舌状花花瓣表皮细胞显微观察发现,沉默系舌状花花瓣表皮细胞长度和宽度与对照差异不明显。在两沉默系中的表达明显上升,在小花原基分化期时分别是对照中的1.8和1.78倍,同期的表达分别下降了32.28%和38.19%,和的表达量在多个时期也明显降低。【结论】根据沉默系表型与相关基因的表达,推测的沉默可能解除了对抑制,促进了生长素的转运和过量积累,间接抑制了的活性,限制了细胞的分裂增殖,引发细胞数目变少,导致沉默系器官变小。
菊花;AINTEGUMENTA;花径;基因表达;基因沉默
0 引言
【研究意义】‘杭白菊’是中国传统的菊花品种,不仅具有较高的观赏价值,而且富含黄酮和酚酸类等营养物质,成为重要的饮用保健产品,但由于其花序较小,限制了产量提高。()对花器官的形成和大小发育具有重要作用。开展调控菊花花序生长发育的机理研究,可以为杭白菊的产量和品质提高奠定理论基础,对杭白菊产业的可持续发展具有重要意义。【前人研究进展】- like()属于()转录调控家族中的亚族[1],在模式植物拟南芥中包含、,共8个成员[2],其序列均包含两个AP2保守功能区域,但不含有miR172结合位点[3]。家族成员对于植物的生长发育调控具有重要作用,参与了胚的发育,茎的伸长,花器官发育的起始和生长等[4-5],其中对于花器官大小的影响最为明显[2]。缺失突变体的花器官变小,胚珠发育受损导致雌性不育[6-7],而其过表达导致花器官增大[8]。Krizek等[9]发现拟南芥中生长素转运相关基因可能是的直接调控目标,YAMAGUCHI等[10]研究表明,可以响应生长素的诱导,参与了()的表达调控,进而影响营养生长向开花起始的转变,该路径与()平行。在花器官生长过程中,的活性受到上游生长素响应基因()的调控,影响花器官的大小发育[11]。除花器官发育起始和生长大小以外,Krizek等[12-13]发现参与的其他生理过程也与生长素有关,如花器官身份确定和雌蕊群的发育等。CHIALVA等[14]研究表明,的表达水平与葡萄果实的大小有关。植物花器官的发育由A、B、C、E四类转录调控基因相互作用而决定[15],能够调控(,B类)和(,C类)的表达,影响花器官位置和大小的发育[4],其缺失突变导致花器官发育紊乱。MIZUKAMI等[8]的研究认为能够致使器官增大的原因是其在器官形成过程中保持分生组织较强的分生能力,拟南芥中可能通过()调控细胞分裂周期,使分生组织保持活力从而影响器官的大小。拟南芥中过表达导致叶片细胞数目变多[16]。在ant ail6双突变体和野生型中,细胞壁重塑相关基因的表达存在较大差异[9],HARADA等[17]证明/()和()参与了康乃馨花瓣的伸长过程,笔者实验室前期研究也发现菊花中和与花瓣细胞的分裂和伸长有关。【本研究切入点】关于在菊花花序生长发育中的功能有待研究。【拟解决的关键问题】使用RACE方法从菊花中克隆全长,分析其在菊花不同器官和时期以及2,4-D诱导下的表达特性,统计观察沉默的菊花花瓣大小和形态特征,从而鉴定在菊花花序生长发育中的功能。
1 材料与方法
试验于2016—2017年在山东农业大学园艺科学与工程学院实验中心进行。
1.1 试验材料与处理
选取长势均匀一致的杭白菊扦插苗生根后栽培到温室中,经30 d长日照(16 h光照,8 h黑暗)营养生长后,置于短日照(8 h光照,16 h黑暗)环境中促进其花芽分化。采取不同时期的根、茎、叶片和花器官,存储在-80℃低温冰箱中保存,备用。
1.2 菊花CmANT克隆
试验采用天根TRIzol试剂盒提取菊花花序的RNA。使用SMARTer RACE cDNA扩增试剂盒(Takara,日本)反转录所需的cDNA一链。在菊花转录组数据库(http://www.icugi.org/chrysanthemum/)中检索获得的初始中间片段。使用诺维赞高保真酶进行5′和3′端克隆,通过DNAMAN软件进行全长拼接,设计引物进行全长验证。
1.3 生物信息学分析
通过NCBI网站(http://www.ncbi.nlm.nih.gov)的BLAST程序对CmANT蛋白进行保守功能区域搜索。使用DNAMAN软件进行多氨基酸序列比对,在PBIL网站(https://prabi.ibcp.fr/htm/site/web/home)对其信号肽等特征位点进行检测。使用Mega 7.0软件以邻近结合的方式构建系统发育进化树,青蒿AaANT、向日葵HaANT、马铃薯StANT、枣树ZjANT、棉花GhANT、苦瓜McANT与拟南芥AILs家族成员序列从NCBI网站获得。
1.4 CmANT实时荧光定量分析
为分析在菊花不同器官中的表达情况,采取现蕾期(花蕾直径约0.5 cm)菊花的根、茎段、植株中间部位的功能叶片和顶端花蕾分别进行检测。采取植株顶端盛花期(花序直径约6 cm)的菊花花序,检测在花序不同部位的表达。采取初开期(舌状花长约1.5 cm)、盛花期(舌状花长约2.8 cm)和衰败期(舌状花出现萎蔫变色)的舌状花,检测在舌状花不同生长时期的表达。参照YAMAGUCHI等[18]的方法对营养生长状态下的菊花植株喷施10 mmol∙L-12,4-D,检测在菊花顶芽中的表达量。使用菊花的作为定量的内参基因,试验中所需引物见表1。使用罗氏Light Cycler 480荧光定量仪进行PCR检测,具体程序如下:94℃预变性10 min;94℃变性30 s,60℃退火30 s,40个循环。每个试验重复3次,根据2−ΔΔCt公式计算结果,通过SPSS软件计算标准误差和差异显著性。
1.5 亚细胞定位观察
根据全长设计引物扩增其编码区序列(表1)。使用T4连接酶连接到p1258-GFP载体中,转化大肠杆菌DH5α,测序验证后,提取质粒转化到农杆菌GV3101中,制备菌液侵染洋葱表皮细胞,在超高分辨率激光共聚焦显微镜(LSM880)下观察拍照。
1.6 CmANT的沉默
参照LÜ等[19]的方法对菊花进行病毒诱导沉默。根据与转录组中相似基因的序列比对,筛选出的特异序列,用于沉默扩增,所需引物见表1。将沉默所需片段连接到p-TRV2载体上,转化到GV3101农杆菌中,按需配制含pTRV1、pTRV2和pTRV2-质粒的混合侵染液。将扦插生根后的菊花幼苗分为两组进行侵染,一组由含有pTRV1和pTRV2空载的农杆菌侵染,作为对照;一组由含有pTRV1和pTRV2-的农杆菌侵染,每组处理50株菊花。经真空负压侵染后置于黑暗条件中2 d,然后转移到温室长日照(光照16 h,黑暗8 h)环境中培养30 d,后转移到短日照(光照8 h,黑暗16 h)环境中促进其花芽分化。采取小花原基分化期的花芽,初开期和盛花期的花序,通过半定量PCR的方式检测病毒诱导基因沉默效果。采集沉默系与TRV对照组菊花花序和叶片进行形态特征调查分析。
1.7 沉默系的表型性状观察
采取沉默系和TRV对照组的盛花期花序和叶片,调查花序直径、舌状花和筒状花数目、舌状花的长度和宽度,称量所有叶片鲜重。使用IBM SPSS软件进行统计分析,花器官形态使用数码相机拍照。制作舌状花花瓣中部上表皮细胞切片,在光学显微镜下观察拍照,随机选取视野中9个细胞,测量记录其长度和宽度,使用SPSS软件统计分析。
1.8 CmANT相关基因实时荧光定量分析
在菊花转录组中检索调控相关的基因序列,设计定量引物(表1)。根据文献查阅和预试验检测,采取沉默系和对照组菊花的小花原基分化期(短日照处理14 d)花芽[20],初开期和盛花期的内外轮舌状花,参照1.4的方法进行荧光定量检测。
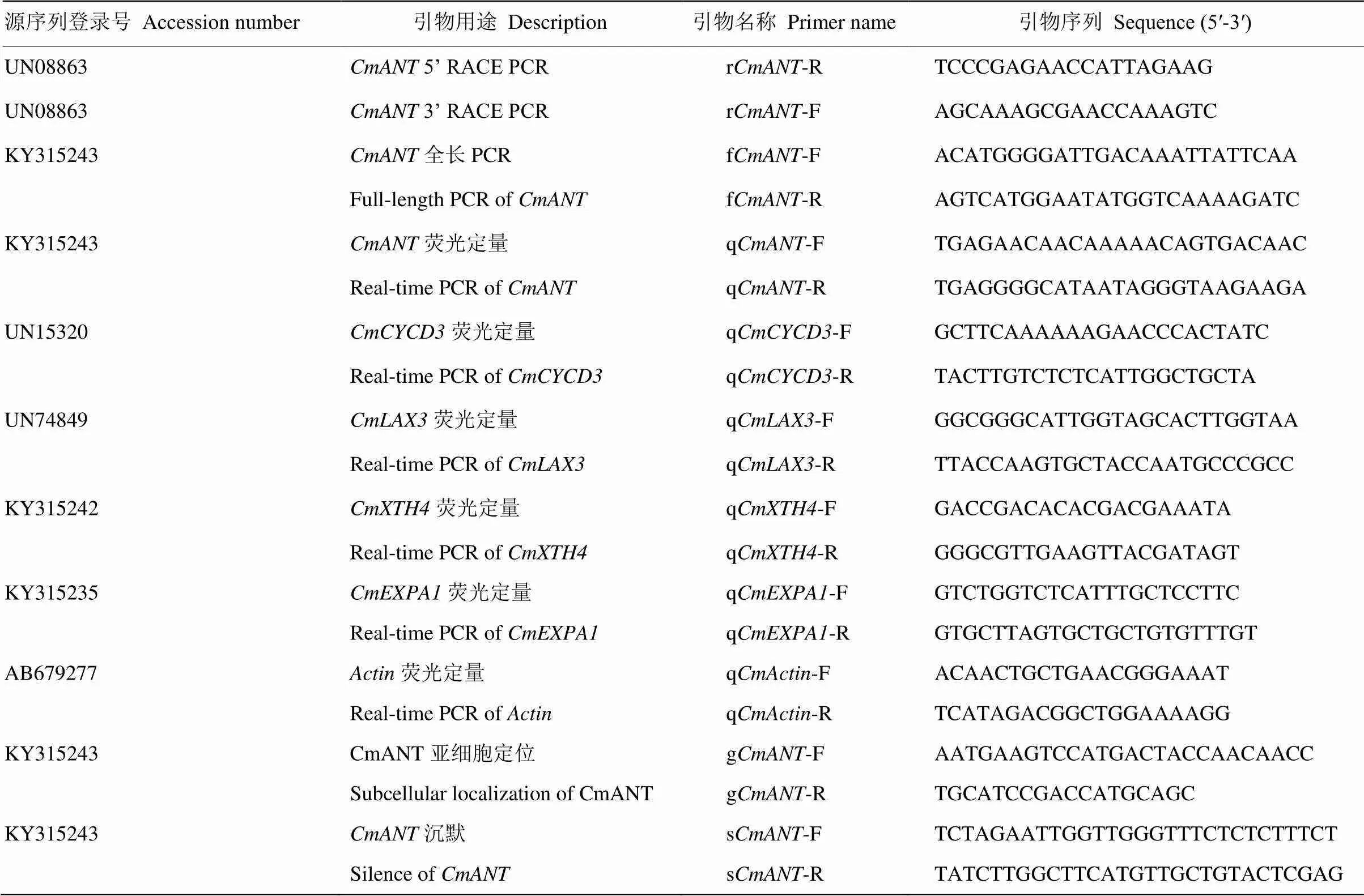
表1 相关基因试验所用引物序列
2 结果
2.1 菊花CmANT克隆与序列分析
根据拟南芥中序列在菊花转录组数据库中的blast结果,筛选出相近的基因中间片段,通过RACE克隆的方法获得全长序列,共1 995 bp,将其序列提交到NCBI基因库中,命名为,登录号为:KY315243。使用DNAMAN软件预测,发现编码540个氨基酸,其理论等电点为7.39,蛋白质的理论分子量为60.4 kD。
2.2 ANT氨基酸序列比对分析
通过NCBI网站BLAST搜索相似蛋白序列,获得了其他物种的ANT蛋白,其中CmANT与青蒿的AaANT相似度最高,为80.78%,由于拟南芥中AtANT的氨基酸序列研究较为深入,所以将菊花的CmANT与其进行详细序列比对(图1),两者的相似度为39.97%。对CmANT结构分析表明,在中间部位有两个AP2/ERF家族特有的AP2保守功能区域,是AP2-like亚族区别于ERF-like亚族的重要特征[21]。两个AP2保守区域之间有29个氨基酸组成的连接区域[22]。此外,CmANT含有与转录活性有关的VYL修饰位点[23]。

两个AP2保守结构域由下划线标示,VYL修饰位点由黑色三角标示
2.3 系统进化分析
为进一步确认CmANT的身份,将菊花的CmANT与其他物种的ANT蛋白以及拟南芥的8个AtAIL蛋白成员一起通过Mega 7.0软件以邻近结合的方式构建系统发育进化树。结果显示菊花的CmANT与青蒿的AaANT聚在一组(图2),两者的遗传距离更为接近。此外,各物种的ANT蛋白聚在一起,与拟南芥的其他AIL成员遗传距离较远。由此说明,CmANT为菊花中的ANT蛋白。
2.4 菊花CmANT在菊花中的表达
通过荧光定量的方式检测在菊花不同器官中的表达(图3-A),发现在茎和叶中的表达量较低,在花蕾中的表达量最高,是叶中的16.21倍,同时在根中也有相当较高的表达,是叶中的8.28倍。比较在菊花花序不同部位中的表达(图3-B),发现其在花萼中的表达量最低,在筒状花中次之,在舌状花中的表达量最高,是花萼中的10.46倍。本试验进一步检测了在舌状花不同生长时期的表达(图3-C),结果表明在衰老期的表达量最低,在初开期和盛花期均有较高的表达量,分别是衰老期的14.25和8.45倍。是生长素调控网络中重要的响应基因[10],参照YAMAGUCHI等[18]的方法,检测了在2,4-D诱导下的表达情况,发现其表达量在经2, 4-D处理3 h后上升了4.64倍,在6 h后仍持续上升,是对照的7.49倍(图3-D)。
2.5 菊花CmANT表达载体的构建与亚细胞定位
经WoLF PSORT软件预测,发现CmANT蛋白定位在植物细胞核中。根据全长设计引物扩增其编码区序列,连接到p1258-GFP过表达载体中,转化到土壤农杆菌中侵染洋葱表皮细胞,使用475 nm蓝光激发,发现绿色荧光聚集在细胞核区域,表明CmANT蛋白主要定位在洋葱表皮细胞的细胞核中。两种方法的结果互相吻合(图4),说明CmANT蛋白的作用区域可能更多集中在细胞核中。
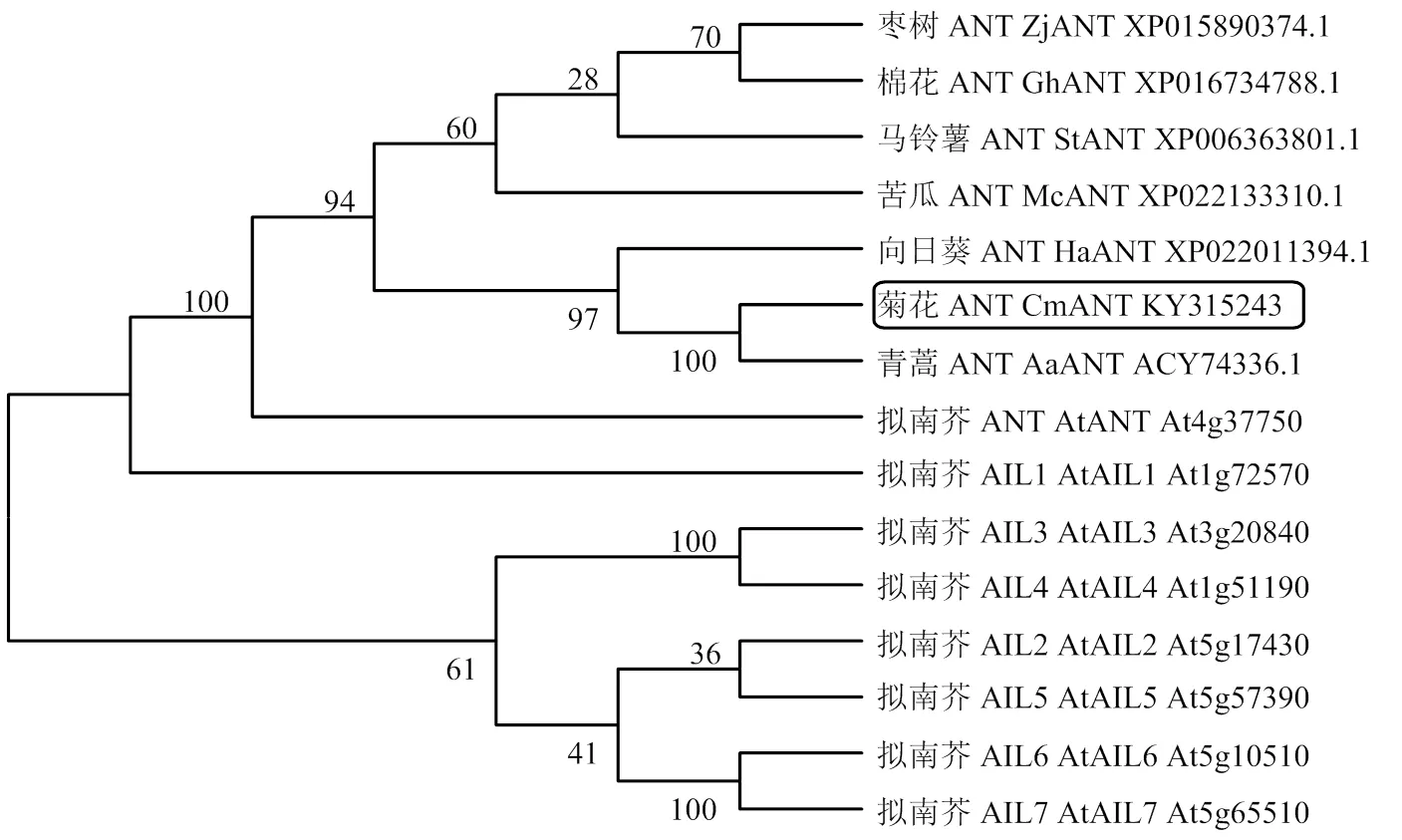
图2 AILs蛋白系统进化分析
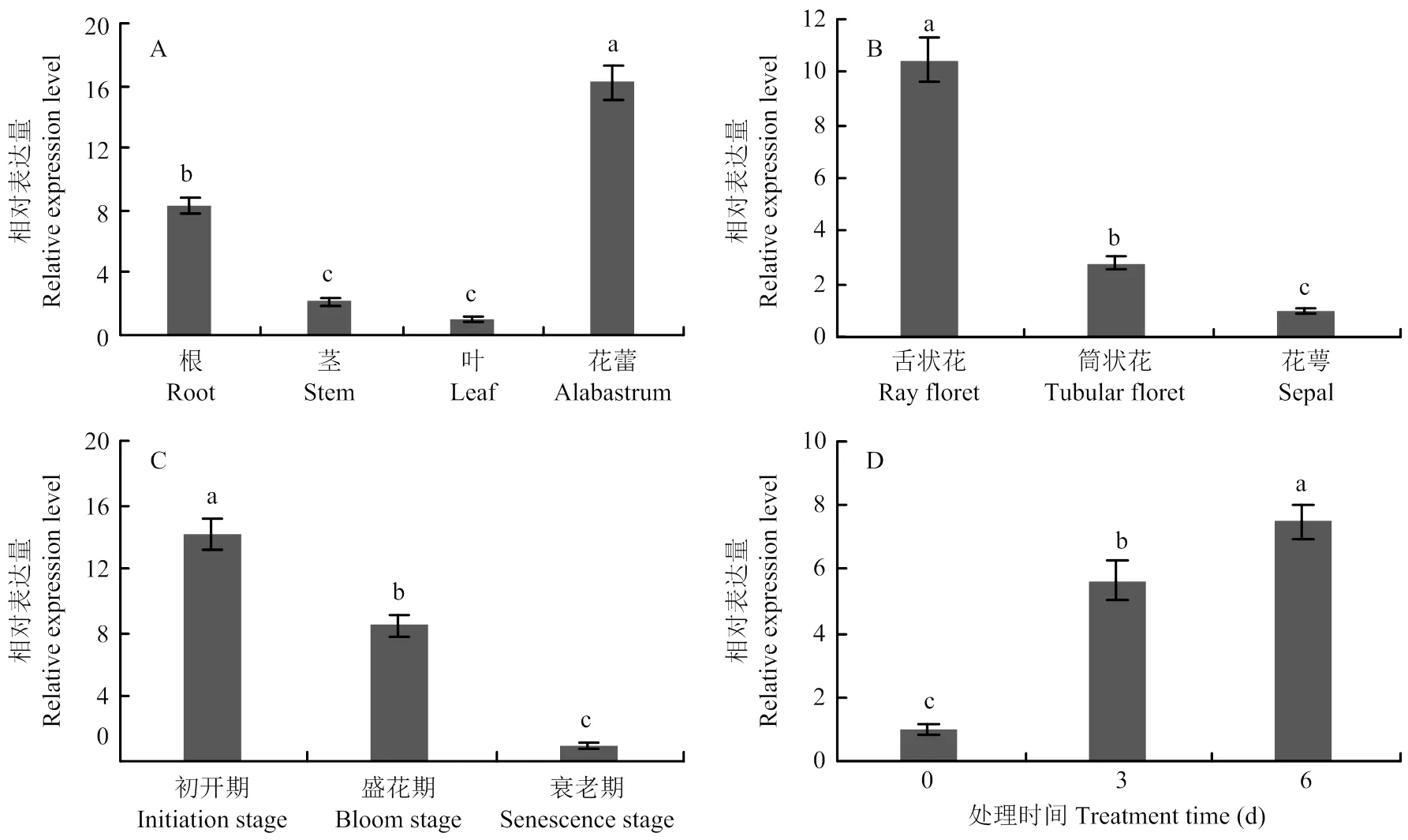
A:CmANT在菊花不同器官中的表达;B:CmANT在菊花花序不同部位的表达;C:CmANT在不同生长期的舌状花中表达;D:CmANT在2,4-D诱导下的表达。误差线代表3个生物学重复计算的标准误差。不同小写字母表示差异显著(P<0.05)。下同
2.6 菊花CmANT沉默的菊花花序和叶片
通过半定量PCR方式检测在各株系小花原基分化期花芽中的表达,鉴定出6个TRV-沉默株系,选择表型变化最为明显的TRV--1和TRV--2进行下一步研究,并在初开期和盛花期时持续检测沉默效果(图5)。

A、D为绿色荧光下的图片,B、E为明场下的图片,C、F为叠加下的图片
TRV--1和TRV--2沉默系中花序直径与TRV对照组差异明显(<0.01),分别减小了18.93%和27.47%(图6-A,表2)。舌状花的数目分别减少了11.39%和14.66%,其中TRV--2与对照差异显著(<0.05),而筒状花的数目变化更为明显,分别减少了14.55%和36.56%。舌状花的大小对菊花花序直径具有重要影响,本试验采集了各株系顶端花序的内、外轮舌状花进行观察和统计分析(图6-B,表2),发现两株沉默系的舌状花平均长度与对照相比差异显著(<0.01),分别减少了34.17%和54.68%;舌状花的宽度分别比对照减小了24.05%和10.13%。

图5 CmANT在不同沉默系和TRV对照组小花原基分化期花芽中的表达
舌状花大小的变化可能源自细胞体积和数目的改变,为进一步研究TRV-沉默株系舌状花花瓣变小的原因,本试验采集了体积差异明显的沉默系和对照组舌状花,制备花瓣中部表皮细胞玻片,在400倍光学显微镜下观察统计(图6-C,表2),发现两个TRV-沉默株系的表皮细胞长度和宽度与对照组差异不明显。说明的沉默并没有引起花瓣表皮细胞的体积变化,推测舌状花花瓣变小的原因可能是由于花瓣细胞数目的改变。菊花的营养对花序的发育具有重要影响,本试验初步检测了作为主要营养供应的叶片的鲜重(表2),发现沉默系中叶片平均鲜重分别比对照组减小了13.19%和21.98%,但差异不显著(>0.05)。进一步通过SPSS软件了解其与花序各项指标的相关性,发现叶片鲜重与筒状花数目显著相关(<0.05),而与其他指标的相关性均不显著(表2),说明沉默系中筒状花数目的改变可能与叶片鲜重存在一定关系,而其他花器官指标的变化大多与叶片改变关系可能不大。
2.7 CmANT及其相关基因在菊花沉默系中的表达
在对照组的小花原基分化期时表达量最高,初开期时有所下降,盛花期时表达量最低;在TRV--1和TRV--2沉默系中在小花原基分化期和初开期时的表达量极显著低于对照组(<0.01),在盛花期时,TRV--2沉默系与对照组也有显著差异(<0.05)。说明在两个沉默系的表达受到了明显抑制。在TRV--1和TRV--2沉默系中各时期的表达均高于对照组,在小花原基分化期时差异最为显著(<0.05),分别是对照中的1.8和1.78倍,而表达的改变可能会影响生长素的运输和积累。在两个沉默株系中的表达均比对照中低,如在小花原基分化期时分别下降了32.28%和38.19%,说明的沉默影响了的表达。在初开期时分别减少了27.76%和29.88%,在初开期时分别比对照降低了37.03%和33.78%(图7)。和的表达降低,从侧面说明沉默系中花瓣细胞的分裂活动可能受到了影响。

A:花序,B:舌状花,C:舌状花花瓣表皮细胞,I:TRV对照组,II:TRV-CmANT-1沉默系,III:TRV-CmANT-2沉默系,Bar=25 µm

表2 CmANT沉默系与对照组菊花花序形态指标
表中数值为平均值±标准误差。每一行中*代表在<0.05水平上与对照相比有显著差异,**代表在<0.01水平上有显著差异
Values are mean ± SE. In each line, * indicates statistically significant differences at<0.05, ** indicates statistically significant differences at<0.01
3 讨论
CmANT氨基酸序列中含有AILs家族中保守的两个AP2功能区域,前人研究发现两个AP2区域会同时作用于ANT和靶基因的结合[24],CmANT的第一个AP2功能域中含有与转录活性有关的VYL修饰位点,说明菊花的CmANT可能与AtANT类似,具有转录调节功能。
3.1 菊花CmANT表达特性
本试验发现在花蕾中的表达量最高,说明其可能更多的调控花器官的发育,但也参与了根、茎、叶等营养器官的生长。MIZUKAMI等[8]研究证明能够保持细胞的分生能力,实现细胞的持续增殖,调控器官不断生长。比较在菊花不同时期舌状花中的表达发现,其表达量随着舌状花发育的持续而降低,推测可能是由于初开期的舌状花处于快速生长期,伴随着大量细胞的生长和增殖,的高表达可能会维持此时细胞增殖所需的能力,而衰老期时舌状花几乎停止了生长,细胞生长和增殖的需求较小,所以的表达量较低。同理,在初开期花序的不同部位中,舌状花的数量较多,生长发育较快,需要的高表达来实现其较大的生长需求,其次是筒状花和花萼。在生长素调控花序发育的代谢网络中,生长素与受体结合促进了转录抑制蛋白AUX/IAAs的降解,解除了对转录调控因子AUXIN- RESPONSE FACTORs(ARFs)的抑制[25-26],其中ARF MONOPTEROS(MP/ARF5)能够直接激活的表达[18]。本试验中,的表达在菊花经2,4-D处理3—6 h内出现快速响应而持续上升,说明可能受到生长素含量的间接调控。通过35S::- GFP融合蛋白在洋葱表皮细胞中的亚细胞定位发现,荧光信号聚集在细胞核中,这可能与CmANT作为转录调控因子的性质有关,在细胞核中实现对下游基因的转录调控。

相邻列中*代表在<0.05水平上与TRV对照相差异显著,**代表在<0.01水平上差异显著
* indicates statistically significant differences at<0.05 compared with TRV control among adjacency lists, ** indicates statistically significant differences at<0.01
图7及其相关基因在沉默系和对照中的表达
Fig. 7 Expression ofand related genes in silenced and control chrysanthemum
3.2 CmANT沉默对菊花花序和叶片的影响
通过构建TRV-沉默表达载体侵染菊花进行功能验证,发现TRV--1和TRV--2沉默株系的花序平均直径相比TRV对照组明显减小。而在拟南芥强突变体中,不仅花器官的体积变小,同时出现了外轮花瓣和雄蕊的数量减少[7],NOLE-WILSON等[27]发现突变体中分生组织细胞的不足导致形成的花器官原基比野生型要小,进而可能使花器官数目减少。虽然本试验沉默系中未发现菊花单个小花的器官缺失,但舌状花和筒状花数目均明显减少,说明的沉默可能会影响菊花小花原基的分化,影响小花数目。筒状花数目减少的幅度要大于舌状花,可能是由于筒状花相比舌状花在小花原基分化时发育较晚的缘故,因此筒状花数目的改变更为明显。对舌状花观察统计发现,其平均长度和宽度较对照组明显变小,通过对花瓣表皮细胞的显微观察,发现细胞的长度和宽度与对照相比无明显变化,类似的是拟南芥突变体的叶片和花瓣表皮细胞体积没有变小,反而异常变大[8]。排除细胞体积的变化的因素,则引起花瓣变小的原因可能是细胞数目的减少,因此推测的沉默可能会影响花瓣细胞的增殖,进而影响花瓣的大小。本试验发现沉默系叶片平均鲜重明显小于对照组,说明的沉默也会对叶片产生类似的影响。通过相关性分析发现除筒状花数目以外,花序其他指标与叶片的相关性不明显,推测沉默系中花序的改变除部分来自叶片营养供应的差异外,可能更多的是由于的沉默。
3.3 CmANT调控花序生长发育机理的分析
是调控细胞分裂起始的重要基因,主要在花器官中表达[28],拟南芥中过表达导致细胞数目变多[16]。MIZUKAMI等[8]发现拟南芥中过表达使在成熟叶片中持续表达,在突变体中却没有表达,认为可能会直接调控的表达,从而实现细胞的持续分裂。但RANDALL等[29]的研究表明的启动子序列与AtANT并无直接互作关系,并推测与在调控器官生长过程中是独立运行的。本试验中发现,在的沉默系中的表达有所降低,推测可能会以间接的方式影响的表达。在沉默系的小花原基分化时,的表达降低可能会限制分生组织的细胞分裂,影响小花的数目,尤其是发育较晚的筒状花。是重要的生长素转运相关蛋白,影响侧生器官的生长发育[30],可能受到的直接调控[9]。在拟南芥双突变体中,发现虽然花器官的发育受阻,但有更多的生长素积累[9]。在本试验中,在两个沉默系中的表达均明显上升,所以猜测可能是负向调控的转录抑制因子,的沉默导致抑制减弱,而激活了的表达,进而可能会促进生长素的积累。Garza- AGUILAR等[31]的研究表明在IAA处理过的玉米种子萌发过程中,CYCD3;1蛋白含量降低,同时相关蛋白激酶的活性也有所改变,因此推测持续过高浓度的生长素含量对的转录表达和相关激酶的活性可能具有抑制作用。另一方面,植物体内激素调控网络存在交叉联系和相互影响[32],诱导的生长素积累,可能会对其他激素的代谢网络产生影响,如拟南芥双突变体花器官中不仅生长素大量积累,同时伴随着其他激素含量的改变[21],这些激素的代谢紊乱可能会对产生影响,抑制其转录表达或者蛋白激酶活性。因此也有可能是菊花的沉默激活了的表达,造成花序中的激素代谢紊乱,对的表达产生了间接影响,抑制了其活性,导致细胞分裂受阻。PEAUCELLE等[33]发现细胞壁中果胶成分的改变,引起细胞壁的松散,有利于侧生器官的起始发育,而引起的生长素积累,调控一系列细胞壁重塑相关基因的表达,如和。Harada等[17]证明了和参与了康乃馨花瓣的伸长生长。本试验中,和在两个沉默系中的表达均有所降低。因此推测的沉默可能间接的降低了细胞壁重塑基因的表达,限制了细胞壁的舒张,影响了花器官分生组织的发育,也有可能是的沉默导致细胞分裂活动的降低,减少了细胞壁重塑相关蛋白的需求,从而影响了和的转录表达,本研究更倾向于后者。
4 结论
本研究克隆的具有典型AP2家族序列特征,在花蕾和初开期的舌状花中具有较高的表达。的沉默导致花序直径、小花数目和舌状花的体积明显变小,推测原因是由于对抑制的解除,促进了生长素的转运而过量积累,间接抑制了的活性,限制了细胞的分裂增殖,以致细胞数目变少,伴随着细胞壁的重塑活动,从而影响器官的生长。
[1] Mizoi J, Shinozaki K, Yamaguchi-Shinozaki K. AP2/ERF family transcription factors in plant abiotic stress responses., 2012, 1819(2): 86-96.
[2] Krizek B A.genes have partly overlapping functions withbut make distinct contributions toflower development., 2015, 66(15): 4537-4549.
[3] Zumajo-Cardona C, Pabón-Mora N. Evolution of thegene lineage in seed plants., 2016, 33(7): 1818-1832.
[4] Horstman A, Willemsen V, Boutilier K, Heidstra R. AINTEGUMENTA-LIKE proteins: Hubs in a plethora of networks., 2014, 19(3): 146-157.
[5] Mudunkothge J S, Krizek B A. Threegenes act in combination to regulate shoot apical meristem function., 2012, 71(1): 108-121.
[6] Elliott R C, Betzner A S, Huttner E, Oakes M P, Tucker W, Gerentes D, Perez P, Smyth D R., an-like gene ofwith pleiotropic roles in ovule development and floral organ growth., 1996, 8(2): 155-168.
[7] Klucher K M, Chow H, Reiser L, Fischer R L. Thegene ofrequired for ovule and female gametophyte development is related to the floral homeotic gene., 1996, 8(2): 137-153.
[8] Mizukami Y, Fischer R L. Plant organ size control:regulates growth and cell numbers during organogenesis., 2000, 97(2): 942-947.
[9] Krizek B, Bequette C, Xu K, Blakley I, Fu Z, Stratmann J, Loraine A. RNA-Seq links the transcription factorsandto cell wall remodeling and plant defense pathways., 2016, 171(3): 2069-84.
[10] Yamaguchi N, Jeong C W, Nole-Wilson S, Krizek B A, Wagner D. AINTEGUMENTA and AINTEGUMENTA-LIKE6/ PLETHORA3 induceexpression in response to auxin to promote the onset of flower formation in., 2016, 170(1): 283-293.
[11] Hu Y, Xie Q, Chua N H. Theauxin-inducible genecontrols lateral organ size., 2003, 15(9): 1951-1961.
[12] Krizek B A.andregulate auxin-mediated flower development in., 2011, 4(1): 176.
[13] Krizek B A. Auxin regulation of Arabidopsis flower development involves members of the AINTEGUMENTA-LIKE/PLETHORA (AIL/PLT) family., 2011, 62(10): 3311-3319.
[14] Chialva C, Eichler E, Grissi C, Muñoz C, Gomez- Talquenca S, Martínez-Zapater J M, Lijavetzky D. Expression of grapevine-like genes is associated with variation in ovary and berry size., 2016: 1-14.
[15] Smaczniak C, Immink R G H, Muiño J M, Blanvillain R, Busscher M, Busscher-Lange J, Dinh Q D, Liu S, Westphal A H, Boeren S, Parcy F, Xu L, Carles C C, Angenent G C, Kaufmann K. Characterization of MADS- domain transcription factor complexes inflower development., 2012, 109(5): 1560-1565.
[16] Dewitte W, Riou-Khamlichi C, Scofield S, Healy J, Jacqmard A, Kilby N, Murray J. Altered cell cycle distribution, hyperplasia, and inhibited differentiation incaused by the D-type cyclin CYCD3., 2003, 15(1): 79-92.
[17] Harada T, Torii Y, Morita S, Onodera R, Hara Y, Yokoyama R, Nishitani K, Satoh S. Cloning, characterization, and expression of xyloglucan endotransglucosylase/hydrolase and expansin genes associated with petal growth and development during carnation flower opening., 2011, 62(2): 815-823.
[18] Yamaguchi N, Wu M-F, Winter C M, Berns M C, Nole-Wilson S, Yamaguchi A, Coupland G, Krizek B A, Wagner D. A molecular framework for auxin-mediated initiation of flower primordia., 2013, 24(3): 271-282.
[19] Lü P, Zhang C, Liu J, Liu X, Jiang G, Jiang X, Khan M A, Wang L, Hong B, Gao J.mediates the antagonism of gibberellins to ABA and ethylene during rose () petal senescence., 2014, 78(4): 578-590.
[20] 杨娜, 郭维明, 陈发棣, 房伟民. 光周期对秋菊品种’神马’花芽分化和开花的影响. 园艺学报, 2007, 34(4): 965-972.
YANG N, GUO W M, CHEN F D, FANG W M. Effects of photoperiod on floral bud differentiation and flowering ofRamat. ‘Jinba’., 2007, 34(4): 965-972. (in Chinese)
[21] Jofuku K D, Den Boer B, Van Montagu M, Okamuro J K. Control offlower and seed development by the homeotic gene., 1994, 6(9): 1211-1225.
[22] Krizek B A. AINTEGUMENTA utilizes a mode of DNA recognition distinct from that used by proteins containing a single AP2 domain., 2003, 31(7): 1859-1868.
[23] Masaki T, Mitsui N, Tsukagoshi H, Nishii T, Morikami A, Nakamura K. ACTIVATOR of Spomin::LUC1/WRINKLED1 ofTransactivates Sugar-inducible Promoters., 2005, 46(4): 547-556.
[24] Nole-Wilson S, Krizek B A. DNA binding properties of the Arabidopsis floral development protein AINTEGUMENTA., 2000, 28(21): 4076-4082.
[25] 盛慧, 秦智伟, 李文滨, 周秀艳, 武涛, 辛明. 黄瓜生长素反应因子(ARF)家族鉴定及表达特异性分析. 中国农业科学, 2014, 47(10): 1985-1994.
SHENG H, QIN Z W, LI W B, ZHOU X Y, WU T, XIN M. Genome-wide identification and expression analysis of auxin response factor (ARF) family in cucumber., 2014, 47(10): 1985-1994. (in Chinese)
[26] Müller B, Sheen J. Cytokinin and auxin interplay in root stem-cell specification during early embryogenesis., 2008, 453(7198): 1094-1097.
[27] Nole-Wilson S, Tranby T L, Krizek B A.() genes are expressed in young tissues and may specify meristematic or division-competent states., 2005, 57(5): 613-628.
[28] Menges M, Samland A, Planchais S, Murray J. The D-type cyclin CYCD3;1 is limiting for the G1-to-S-phase transition in., 2006, 18(4): 893-906.
[29] Randall R, Sornay E, Dewitte W, Murray J.and the D-type cyclin CYCD3;1 independently contribute to petal size control in: evidence for organ size compensation being an emergent rather than a determined property., 2015, 66(13): 3991-4000.
[30] Porco S, Larrieu A, Du Y, Gaudinier A, Goh T, Swarup K, Swarup R, Kuempers B, Bishopp A, Lavenus J, Casimiro I, Hill K, Benkova E, Fukaki H, Brady S, Scheres B, Péret B, Bennett M. Lateral root emergence inis dependent on transcription factorregulation of auxin influx carrier., 2016, 143(18): 3340-3349.
[31] Garza-Aguilar S, Lara-Núñez A, García-Ramírez E, Vázquez-Ramos J. Modulation of CycD3;1-CDK complexes by phytohormones and sucrose during maize germination., 2017, 160(1): 84-97.
[32] Vandenbussche F, Petrásek J, Zádníková P, Hoyerová K, Pesek B, Raz V, Swarup R, Bennett M, Zazímalová E, Benková E, Van Der Straeten D. The auxin influx carriersandare involved in auxin-ethylene interactions during apical hook development inseedlings., 2010, 137(4): 597-606.
[33] Peaucelle A, Braybrook S, Le Guillou L, Bron E, Kuhlemeier C, Höfte H. Pectin-induced changes in cell wall mechanics underlie organ initiation in., 2011, 21(20): 1720-1726.
(责任编辑 赵伶俐)
Cloning and Functional Verification ofGene in Chrysanthemum
WEN LiZhu, SUN Xia, Fan HongMei, GUO YunHui, YU YuanYuan, REN Hong, WANG WenLi, ZHENG ChengShu
(College of Horticulture Science and Engineering, Shandong Agricultural University/Chrysanthemum Research Center of China, Japan and Korea in Shandong Province, Tai’an 271018, Shandong)
【Objective】 To understand the role ofgene in chrysanthemum inflorescence development, we clonedgene from chrysanthemum, analyzed its sequence, and characterized its temporal and spatial expression pattern. We further analyzed the impact ofsilence on inflorescence development and its possible regulation mode. This study was carried to reveal the potential mechanism ofin governing inflorescence development, which in turn can provide a theoretical basis for chrysanthemum inflorescence diameter adjustment. 【Method】 The chrysanthemumgene was cloned by RACE method and its sequence was analyzed with DANMAN software. Its expression in different developmental stages and organs of chrysanthemum was detected by real-time fluorescent quantitation PCR. The plasmid of 35S::-GFP was constructed for the subcellular localization. The silence vector of TRV2-was constructed to infect chrysanthemums. The statistics of phenotypic changes insilenced chrysanthemums were analyzed via SPSS software. The ray florets petal epithelial cells were observed with optical microscope. The expression ofand related genes were detected by real-time fluorescent quantitation PCR. 【Result】 The full-length ofwas cloned from chrysanthemum. It encodes 540 amino acids and contains two AP2 conserved function domains and VYL modification sites. The theoretical isoelectric point of CmANT is 7.39 and its molecular weight is 60.4 kD. The phylogenetic tree composed of ANT proteins from various plants species showed that CmANT and AaANT had been grouped together. The real-time fluorescent quantitation results showed that: (1)was expressed most in floral buds, followed by roots, stems and leaves. (2) Gene expression in different parts of inflorescence indicated thatwas expressed the most in ray florets followed tubular florets and the lowest in the sepal. (3) The expression ofdeclined during the development of ray florets. (4) The expression ofunder 2,4-D treatment increasedbetween 3 h to 6 h. WoLF PSORT prediction and 35S::-GFP fusion protein localization in onion epithelial cells indicated that CmANT protein was located in cell nucleus. Compared with the control, the mean inflorescence diameters of chrysanthemums in TRV--1 and TRV--2 lines were decreased by 18.93% and 27.47% respectively and the numbers of ray florets were decreased by 11.39% and 14.66% respectively, among which the difference between TRV--2 lines and the control was statistically significant (<0.05 ). The numbers of tubular florets were decreased by 14.55% and 36.56%, the mean length of ray florets in top inflorescences was decreased by 34.17% and 54.68%, and the width was decreased by 24.05% and 10.13% respectively in TRV--1 and TRV--2 lines compared with the control, the mean fresh weight of leaves was decreased by 13.19% and 21.98% respectively in TRV--1 and TRV--2 lines and the correlation between leaf fresh weight and tubular floret number was significant (<0.05 ). The microscopic observation of epithelial cells in ray floret petals indicated that the cell length and width of petals in the silenced lines and the control had no visible differences. The expression ofwas decreased by 32.28% and 38.19% in two silenced lines and the expression ofandalso decreased at most developmental stages. 【Conclusion】Due to the phenotypic changes andexpression pattern in silenced chrysanthemums lines, we speculated that the silence ofmight relieve the repression on, facilitate the transport and accumulation of auxins, indirectly repress the activation of, limit the cell division, and cause the decrease of cell number, combined of which would eventually lead to the smaller organ sizes in silenced chrysanthemums lines.
;; inflorescence diameter; gene expression; gene silence
10.3864/j.issn.0578-1752.2018.09.014
2017-09-20;
2017-12-20
国家科技支撑计划(2011BAD10B07)、国家自然科学基金面上基金(31670663)
温立柱,E-mail:wlizhu@163.com。孙霞,E-mail:sunxia65@sina.com。温立柱与孙霞为同等贡献作者。
郑成淑,Tel:0538-8246139;E-mail:zcs@sdau.edu.cn
