利用回交和标记辅助选择快速培育高油酸花生品种及其评价
2018-05-14张照华王志慧淮东欣谭家壮陈剑洪晏立英王晓军万丽云陈傲康彦平姜慧芳雷永廖伯寿
张照华,王志慧,淮东欣,谭家壮,陈剑洪,晏立英,王晓军,万丽云,陈傲, 康彦平,姜慧芳,雷永,廖伯寿
利用回交和标记辅助选择快速培育高油酸花生品种及其评价
张照华1,王志慧1,淮东欣1,谭家壮2,陈剑洪3,晏立英1,王晓军4,万丽云1,陈傲2, 康彦平1,姜慧芳1,雷永1,廖伯寿1
(1中国农业科学院油料作物研究所/农业部油料作物生物学重点开放实验室,武汉 430062;2湛江市农业科学研究院,广东湛江 524094;3泉州市农业科学研究所,福建泉州 362212;4江苏徐淮地区徐州农业科学研究所,江苏徐州 221131)
【目的】高油酸育种是花生品质改良的重要方向,利用回交育种结合标记选择可快速实现现有推广品种的高油酸化改良,探讨利用这一技术体系进行花生高油酸遗传改良的实践和效率。【方法】以目前推广的优质高产抗病品种中花16、中花21、泉花551、徐花13为轮回亲本(母本,基因型AABB),以高油酸材料冀花13为非轮回亲本(父本,基因型aabb)配制4个杂交组合,一年种植两季并进行人工杂交或自交,夏季在武汉种植,冬季在湛江南繁基地种植,通过1次杂交、4次回交和1次自交得到BC4F2后代。利用PCR产物测序方法,鉴定杂交和回交后代的基因型:根据回交后代基因型分离规律及与序列高度同源性的特点,用引物F0.7/R3在一个PCR反应内同时高效扩增F1和回交后代(BC1F1-BC4F1)的和片段,并利用R3作为测序引物进行反向测序,读取测序峰图判别基因型,在回交后代中筛选基因型AaBb的后代作为下代回交父本。自交后代(BC4F2-BC4F3)基因型鉴定采用KASP分型,获得高油酸(基因型aabb)后代。对获得的基因型为aabb的高油酸后代与其对应轮回亲本进行重要农艺性状、品质和重要抗病性的调查和SSR标记检测。【结果】在3年时间内,4个组合分别获得10、5、6、8株BC4F2高油酸纯合隐性基因型(aabb)单株,通过一代自交获得相应的BC4F3株系,对获得的高油酸株系与轮回亲本进行植物学、农艺性状、品质和青枯病抗性的考察,最终,4个组合均获得了与轮回亲本综合性状最接近的株系,分别为ZJ019、ZJ109、ZJ160和ZJ805,其油酸含量为82.54%、79.85%、79.22%、和78.94%,可作为轮回亲本对应的高油酸新品种。另外,本研究还对中花16回交组合中获得的高油酸株系的遗传背景进行了SSR分子检测,发现ZJ019株系的回复率达94.8%,在该组合中回复率最高,这一结果与植物学、农艺性状鉴定的结果一致。【结论】利用连续回交、南繁加代和分子标记辅助选择等技术可在3年内快速实现现有推广花生品种的高油酸化改良。
花生;高油酸;回交;分子标记辅助选择;品种
0 引言
【研究意义】花生(L.)是中国重要的油料和经济作物。近几年,中国花生种植面积约4.67×106hm2,总产1.68×107t,约占世界花生产量的40%。国产花生原料的55%用于榨油,年产花生油2.6×105t,是国产植物油的第二大来源[1-2]。因此,中国是世界上最大的花生生产、消费和出口国。花生种子中含8种主要脂肪酸,而油酸(45%—80%)、亚油酸(2%—35%)和棕榈酸(6%—12%),这三种脂肪酸占比达到92%[3]。油酸含量是评价植物油品质的重要指标。油酸可以降低对人体健康有害的低密度胆固醇(low-density lipoprotein,LDL),同时不破坏对人体有利的高密度胆固醇(high-density lipoprotein,HDL),而且高油酸花生(油酸含量约80%)产品还可延长货架寿命[4-6]。但目前国内主推的花生品种绝大多数为普通油酸含量(55%以下),高油酸花生品种较少,导致国内花生原料和加工产品的油酸含量低,品质不优[7]。因此,如何快速实现中国花生主推品种的高油酸化,已成为花生品种改良亟需解决的问题。【前人研究进展】花生油酸到亚油酸的转化,主要由分别位于A09、B09染色体上的、两对同源非等位基因控制[8]。目前,在花生中广泛应用的高油酸突变材料F435[9],是由于的ORF-448 bp G/A替换和的ORF-441_442的“A”插入导致FAD2酶部分或全部失活,2个基因同时突变导致了花生高油酸性状的产生[10-11]。目前,针对这两个突变基因的特定突变位点开发的基因型鉴定方法主要有:1)CAPS标记法;2)荧光定量PCR法;3)位点特异性PCR(Allele-specific,AS-PCR)法;4)竞争位点特异性PCR(Kompetitive Allele Specific PCR,KASP)等[12-15]。上述方法均是鉴定的间接方法,由于、基因序列的高度相似性,导致鉴定结果具有一定比例的假阳性,用于回交后代的检测准确性不高,并且由于鉴定方法需要用到内切酶和荧光物质、荧光PCR仪等设备,鉴定成本和操作技术复杂,因此需要建立更准确的回交后代基因型鉴定方法。杂交育种是转移和聚合优良性状的常用技术方法[16],自从花生高油酸突变体F435被引入中国以来,国内利用该突变体或其衍生材料,培育出了多个高油酸花生新品种,但这些新品种普遍存在产量较低、抗性不高、耐寒性差等问题[17]。回交育种是改良推广品种个别不良性状的最为快速有效的方法,目前国内推广的主要花生品种均经过生产上的广泛种植,在一定生态区具有良好的适应性,通过回交育种快速地将这些优良的推广品种转育成高油酸品种,将加快中国花生高油酸品种的推广和应用进程。【本研究切入点】中国高油酸花生遗传改良起步晚,且目前培育的品种多为利用杂交和系谱法育成,杂交后代选择和品系的适应性评估周期较长,导致高油酸品种培育进展缓慢,不能满足产业发展需求。为此,需要建立简便高效的高油酸育种基因型鉴定方法和技术体系。【拟解决的关键问题】针对目前花生高油酸改良存在的问题,本研究利用回交技术提高推广花生品种的油酸含量,根据回交分离的特点,实现回交后代基因型的简便高效鉴定,结合自交后代基因型的KASP分型技术,快速地获得高油酸株系。同时,综合利用植物学性状、农艺性状、抗病抗逆性鉴定和SSR标记检测,发掘与轮回亲本(推广品种)高度相似的高油酸回交后代,以加速高油酸品种的培育和推广进程。
1 材料与方法
1.1 材料
1.1.1 轮回亲本(推广品种) 1)中花16(ZH16,基因型AABB),中国农业科学院油料作物研究所选育,2009年通过国家鉴定和湖北省审定,2013年通过江苏省鉴定,珍珠豆型中果早熟品种。含油量57.76%,油酸54.00%,亚油酸23.00%,为高产高油品种。2)中花21(ZH21,基因型AABB),中国农业科学院油料作物研究所选育,2009年通过湖北省审定,2015年通过国家鉴定,珍珠豆型中果早熟品种。含油量53.09%,油酸41.20%,亚油酸36.00%,为高产抗青枯病品种。3)泉花551(QH551,基因型AABB),福建省泉州市农科所选育,2015年通过国家鉴定和福建省审定,珍珠豆型小果花生品种。高抗青枯病,百果重210.9 g,含油量50.9%,油酸含量49.40%,亚油酸含量31.80%,为高产抗青枯病品种。4)徐花13(XH13,基因型AABB),徐州农业科学研究所选育,2008年通过国家鉴定,中早熟大粒花生品种,百果重234.2 g,含油量56.43%,油酸45.70%,亚油酸33.30%,为高产高油品种。
1.1.2 非轮回亲本(高油酸基因供体) 冀花13(JH13,基因型aabb),河北省农林科学院粮油所选育,2010年通过国家鉴定,普通型中早熟小果,百果重191.2 g。油酸含量81.4%,亚油酸2.9%,属高油酸小果花生品种。其高油酸基因来源是自然突变材料F435(基因型aabb)。
1.2 选育过程
杂交获得F1代:以轮回亲本为母本,非轮回亲本为父本配置杂交组合,通过人工授粉获得F1杂交种子,鉴定F1杂交种子的基因型,获得F1真杂种(基因型AaBb)。
回交获得BC1F1-BC4F1种子:以轮回亲本为母本,以获得的F1真杂种为父本进行人工授粉,获得BC1F1种子,鉴定BC1F1种子基因型,保留基因型为AaBb的BC1F1种子作为下一代回交的父本。重复回交直至获得基因型为AaBb的BC4F1种子。
自交获得BC4F2-BC4F3种子:将BC4F1种子(基因型AaBb)种植,自交获得BC4F2种子,利用近红外初筛单粒种子的油酸含量,保留65%以上的种子进行基因型鉴定(KASP法)。为加快回交育种进程,分别在湖北武汉种植春季杂交(4月—8月)、广东湛江(11月—次年2月)进行每年两代人工杂交(图1)。
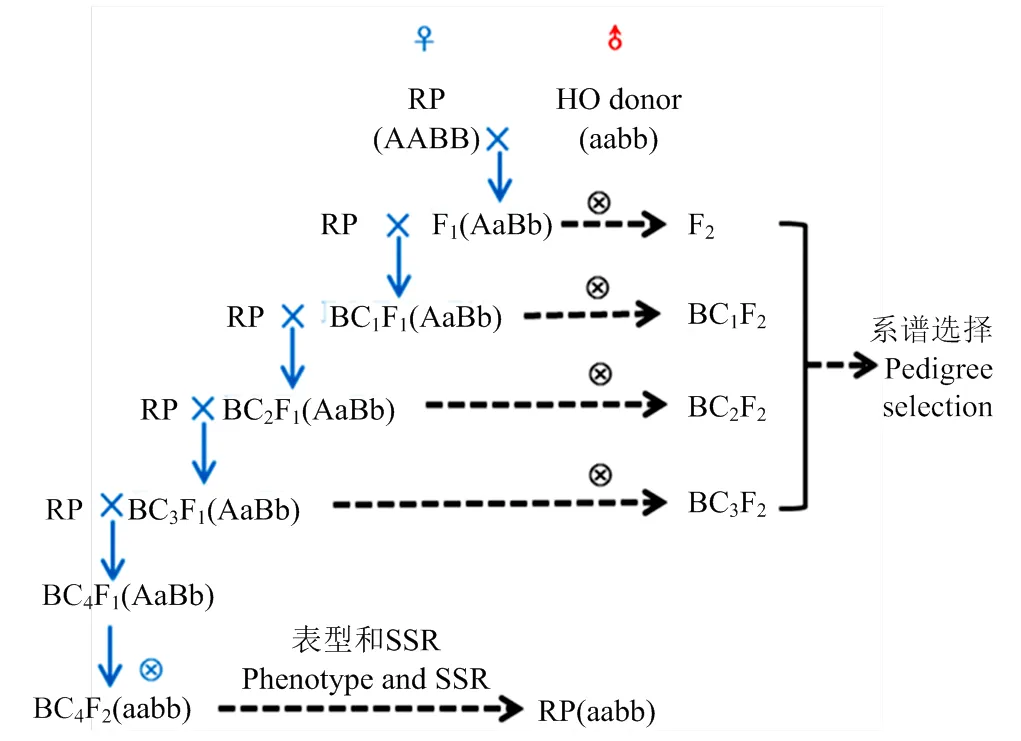
图1 高油酸花生回交育种流程
1.3 人工杂交方法
人工杂交父本一般比母本提前5天种植,以保证母本初花时有足够的父本花粉供应。母本初花后即可开始人工杂交,一般每天下午16:00—18:00对母本上花萼微裂花瓣黄色、第二天早上能正常开花的花蕾进行人工去雄,去雄后除去本节位上的多余花苞,然后用棉线绑在该节位上以示标记;次日清晨5:00—7:00父本花粉小心涂抹在去雄的母本花柱上,人工杂交一般持续15—20 d,人工杂交结束后一周用丝线标记杂交成功的果针。
1.4 DNA的提取
叶片DNA提取:分别取1片未展开的花生小叶,采用改良CTAB法提取[18]DNA。
花生子叶DNA提取:切取种子远胚端的40 mg组织(最好从一片子叶上切取),采用王传堂等[19]方法提取DNA。
1.5 ahFAD2A、ahFAD2B基因型鉴定
根据回交育种不同世代的分离特点,采用不同的基因型鉴定方法。1)亲本基因型的鉴定:用Sanger测序法鉴定亲本材料的基因型。其中引物对Fad2A-F/ Fad2A-R用于单独扩增;Fad2B-F/Fad2B-R用于单独扩增,使用2个测序引物:Internal1和Internal2进行双向测序(表1)。PCR扩增体系(50 μL)为2×SYBR® Premix Ex TaqTM(包含缓冲液,dNTPs) 25 μL、上下游引物(10 μmol·L-1)2.5 μL和模版DNA 30 ng。PCR程序为95℃ 5 min;94℃ 30 s,61℃ 30 s,72℃ 60 s,30个循环;72℃ 10 min,4℃保存。得到的PCR产物原液(未纯化),送擎科生物公司测序。2)F1、BC1F1- BC4F1基因型的鉴定:杂交和回交后代种子采用廖伯寿[20]描述的PCR产物测序法进行。PCR扩增引物:F0.7/R3。PCR扩增体系(50 μL)为2×SYBR® Premix Ex TaqTM(包含缓冲液,dNTPs)25 μL、上下游引物(10 μmol·L-1),2.5 μL和模版DNA 30 ng。PCR程序为95℃ 5 min;94℃ 30 s,56℃ 30 s,72℃ 60 s,30个循环;72℃ 10 min,4℃保存。得到的PCR产物原液(未纯化),送擎科生物公司测序,以反向扩增引物R3为测序引物。3)自交后代BC4F2- BC4F4:采用位点竞争性PCR(Kompetitive Allele Specific PCR,KASP)方法进行。取自交后代幼嫩的叶片,送去中玉金标记公司,按照ZHAO[21]描述的方法进行BC4F2等自交后代的基因型分型。随后用Sanger测序法验证KASP法鉴定获得的aabb单株基因型(具体方法同亲本基因型鉴定)。

表1 花生ahFAD2A、ahFAD2B的PCR扩增引物名称、序列和扩增目标
1.6 油酸含量的测定(参照国家标准方法,GB/ T17377-2008测定)
脂肪酸提取方法:切取10—20 mg花生种子于10 ml玻璃管中,加入2 ml石油醚,超声波震荡30 min,再加入1 ml 0.4 mol·L-1NaOH-甲醇溶液,超声波震荡30 min,静置,取上清。
气相色谱仪Agilent 7890B测定油酸,色谱条件:DB-23色谱柱(30 m×0.25 mm×0.25 μm);载气N225 ml·min-1,空气 400 ml·min-1,燃气H230 ml·min-1。采用分流的方式进样,进样量1 μL,分流比60﹕1,进样口280℃,检测器280℃;程序升温:起始温度180℃,保持2 min,以10℃/min的速度升至190℃,保持1 min,再以5℃/min的速度升至200℃,保持1 min,再以20℃/min的速度升至220℃,保持6 min。
1.7 植株、农艺性状和抗病抗逆性的鉴定
aabb基因型的BC4F2-BC4F4材料与其轮回亲本表型性状的考察参照姜慧芳等[22]方法进行。
1.8 SSR分子标记的检测
采用SSR方法检测aabb基因型的BC4F2材料与其轮回亲本分子水平近似性。PCR反应体系(10 μL)为20 ng DNA模板、Taq PCR SuperMix(由北京全式金生物公司提供,包含缓冲液、dNTPs、Taq酶)5 μL、正反引物(10 μmol·L-1)各1 μL。PCR扩增采用Touchdown程序为95℃3 min;95℃30 s,65℃30 s,(每循环1次降低1℃),72℃1 min,共10个循环;93℃30 s,55℃30 s,72℃1 min,共30个循环;72℃10 min,4℃保存。PCR产物采用变性PAGE胶检测[23]。
SSR标记带型统计和分析:与轮回母本一样的带型记为“1”,与父本一样的带型记为“2”,杂合记为“3”。回复率(recovering ratio of genetic background,RRGB)计算公式G(g)=[L+X(g)]/(2L)=1/2+(1/2)(X(g) /L),G(g)指在g代的遗传背景恢复率[24];X(g)指在回交g代表现为轮回亲本带型的分子标记数量;L指所分析的分子标记数量,并用Powermarker V3.25做聚类分析。
2 结果
2.1 亲本基因型鉴定
配置杂交组合前,采用Sanger测序法对4个轮回亲本和高油酸供体材料(各10粒种子)分别扩增(图2)和鉴定和的基因型(图3),结果表明,4个亲本材料均为纯合一致的AABB基因型,高油酸供体冀花13为aabb基因型。
2.2 基因型鉴定
杂交种子收获后切取部分子叶提取DNA,并用PCR产物测序的方法进行了基因型鉴定(图4和图5),以去除假杂种(AABB)。4个组合F1总计收获F1种子91粒,其中真杂种81粒(基因型AaBb),真杂种比例89.0%,除组合BO-04(XH13×JH13)真杂种率较低外,其他组合真杂种率均在90%以上(表2)。

A:ahFAD2A扩增产物电泳图;B:ahFAD2B扩增产物电泳图
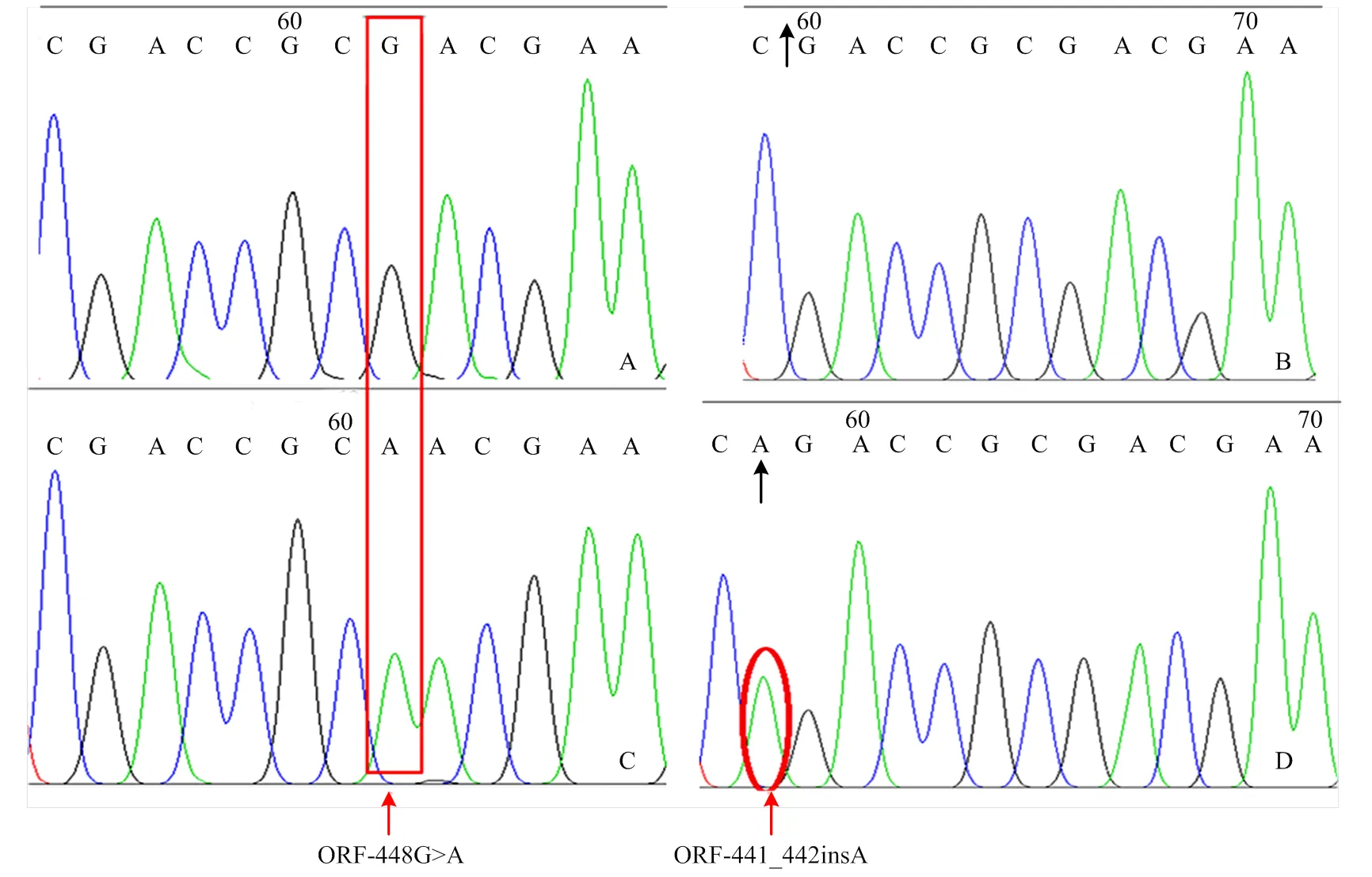
A:AA基因型;B:BB基因型;C:aa基因型;D:bb基因型 A: AA genotype; B: BB genotype; C: aa genotype; D: bb genotype

图4 F0.7/R3扩增F1种子ahFAD2A和ahFAD2B-ORF片段电泳图

A:母本(假杂种)基因型AABB;B:F1(真杂种)基因型AaBb

表2 杂交F1种子的获得与基因型鉴定结果
2.3 回交后代种子的获得及基因型鉴定
回交后代的基因型鉴定采用PCR产物测序法进行。由于和的同源性很高,设计的F0.7/R3引物可同时扩增2个同源基因499 bp的DNA片段(包含突变位点),用R3为引物进行PCR产物的反向测序,理论上测序峰图在的ORF-432应该出现序列本身固有的C/A套峰。如果所有测序峰图均出现ORF-432的C/A套峰,表明PCR扩增和测序反应均同时扩增了2个基因。如果测序峰图在ORF-448位出现A/G的叠峰,则表明基因型Aa,否则判定为AA基因型;在同一测序峰图中ORF-442位如果有A碱基插入,会导致442位点后的峰图出现一个碱基的错位,判定基因型为Bb,否则判定为BB基因型。
在4个回交组合4个回交后代中总计检测了689粒种子(表3),其中AaBb种子数170粒,约占24.7%(基本符合回交后代4种基因型1﹕1﹕ 1﹕1的分离比例)。总体来看,4种回交后代基因型中,AABB基因型个体偏多,推测可能原因为部分世代人工去雄不干净,误将母本(基因型AABB)自交的后代作为人工授粉的荚果进行标记所致,如泉花551的BC3F1世代,徐花13的BC2F1世代等。
2.4 自交后代基因型鉴定与油酸含量测定
2016年春在武汉种植从BC4F1植株上收获自交的BC4F2种子,经KASP分型鉴定(图6),获得aabb基因型植株数分别为10、5、6和8个株系,总计29株(表4)。同时也得到了基因型为AaBb、aaBb和Aabb的BC4F3代种子,后代分离可得到aabb基因型材料。再自交1繁殖代,最终4个组合分别获得基因型为aabb的株系为16、39、38和8个。

表3 回交BC1F1-BC4F1获得的种子数量与基因型鉴定结果

表4 BC4F2种子的KASP基因型分型结果

A:ahFAD2A基因分型结果:红点表示基因型为aa,蓝点表示基因型为AA,绿点表示基因型为Aa,粉点表示样品信号未检出,黑点表示未加模板的阴性对照(NTC);B:ahFAD2B基因分型结果:红点表示基因型为bb,蓝点表示此处基因型为bb,绿色表示基因型为Bb
利用Sanger测序法对KASP分型获得的基因型为aabb的种子进行了确证性检验,并利用气相色谱法进行了脂肪酸含量的测定,结果表明,KASP法获得的29粒种子种有29粒的基因型为aabb,符合率100%,其油酸含量均在75%以上。
2.5 改良的高油酸材料近似性评价
2.5.1 表型评价 对获得的BC4F4高油酸近等基因系和其轮回亲本进行表型评价,每个株系选取10个单株进行植株性状考种,含油量用FOSS近红外测定株系混合样(3次重复),青枯病抗性按照每个株系设置3次重复进行自然病圃鉴定,计算平均存活率并换算为抗病级别。百果重、百仁重取株系的混合样进行考种,脂肪酸含量用气相色谱法进行测定。利用上述数据评价获得的高油酸材料与其轮回亲本的近似度,筛选最为接近的株系作为改良的高油酸品系(表5)。
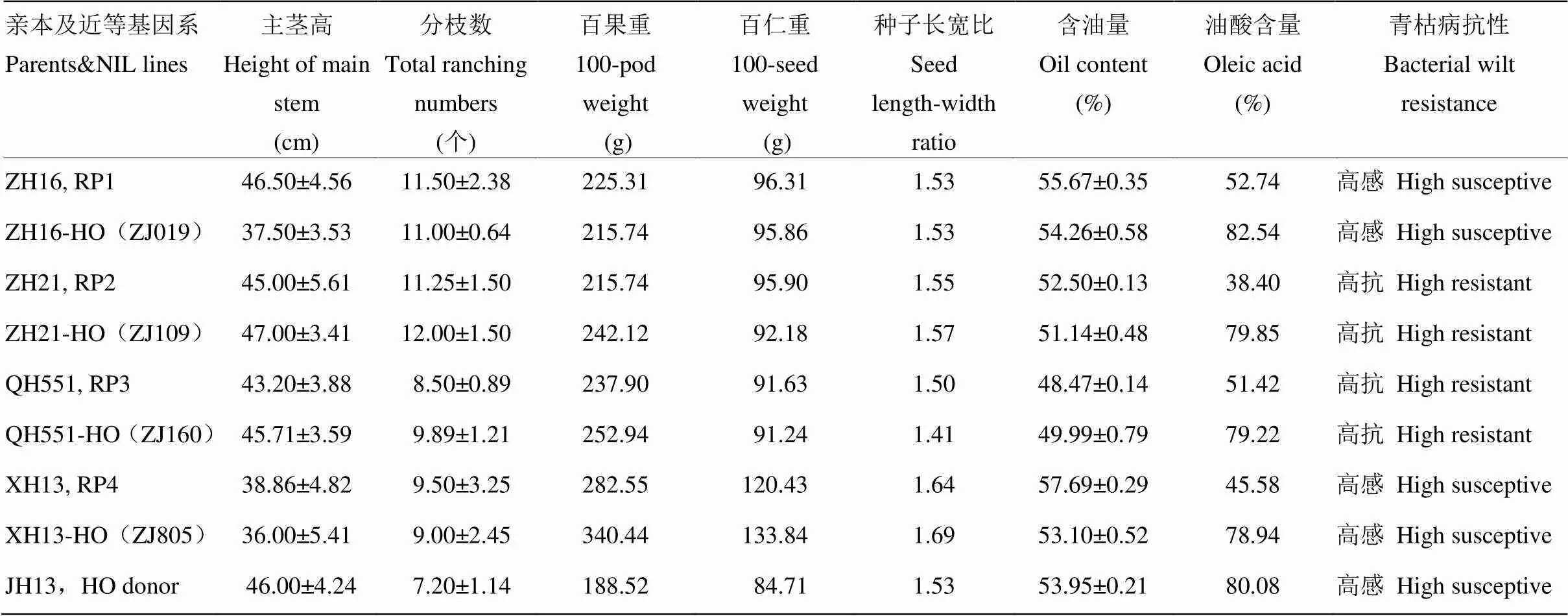
表5 决选高油酸近等基因系与其轮回亲本重要农艺性状、抗病性、品质指标的比较
经表型性状的综合评价,ZH16-HO(ZJ019)与ZH16最为相似,在分枝数、百果重、百仁重、种子长宽比、青枯病抗性上无显著差异,主茎高、含油量略有降低,油酸提高到82.54%。ZH21-HO(ZJ109)与ZH21最相似,在主茎高、分枝数、种子长宽比、青枯病抗性上无显著差异,百果重变大,百仁重、含油量略有降低,油酸提高到79.85%。QH551-HO(ZJ160)与QH551最为相似,在主茎高、分枝数、百仁重、种子长宽比、含油量、青枯病抗性上无显著差异,仅百果重略有增大,油酸提高到79.22%。XH13-HO(ZJ805)与XH13最为相似,主茎高、分枝数、种子长宽比、青枯病抗性上无显著差异,百果重、百仁重高于亲本,含油量略有下降,油酸提高到78.94%。
2.5.2 SSR标记对后代遗传背景的分析 挑选了均匀分布于花生染色体的1 263个SSR标记[25-26],在组合中花16×冀花13中,筛多态性,最终得到67个多态性标记,并用于BC4F2基因型为aabb的10个株系遗传背景的评估。
从表6可以看出,中花16×冀花13组合BC4F2代高油酸单株的遗传背景的回复率都达到83.5%以上,其中ZJ019的回复率最高94.8%,说明高油酸后代的遗传背景和轮回亲本中花16已经高度一致。
用软件PowerMarker V3.25作聚类分析得到依据遗传距离的聚类图(图7)。ZJ019与ZH16遗传背景最接近;ZJ066和ZJ077次之;而ZJ017相差最大。最终,结合表型性状的考察,选择ZJ019为中花16的最优高油酸近等基因系材料。

表6 中花16×冀花13组合高油酸单株遗传背景回复率

图7 中花16及其高油酸候选近等基因系聚类
3 讨论
高油酸育种是花生遗传改良的重要方向,通过杂交和系谱育种,可培育出高油酸花生新品种,但目前国内应用的高油酸材料主要来源于美国,多为普通型匍匐小果材料,产量潜力不高,不符合中国花生生产和种植方式,用其作为杂交亲本培育高油酸品种,后代分离周期长,并且培育的品种需经过多点试验和生产试验以评估其产量潜力、抗病抗逆性和适应性[27]。以现有推广品种作为轮回亲本,采用回交育种结合标记辅助选择技术,可在3年内快速实现推广品种的高油酸化改良,并且改良的高油酸品种在主要性状、抗病抗逆性上与原有推广品种相近,可直接在原有推广区域种植,大大加快了高油酸品种的推广进程,降低了推广风险。转育的高油酸品种还可作为优良亲本材料用于下一步的高油酸品种改良。
基于花生突变位点进行基因型鉴定已开发出多种方法,如CAPS法、荧光定量法、AS-PCR法、KASP等,但不同的方法各有优缺点。如已开发的荧光定量法、AS-PCR法、KASP法,因、序列的高度相似性,均有一定的假阳性率。而CAPS标记法需要用到限制性内切酶,成本较高且操作相对复杂。为此,本研究巧妙地利用了、序列的高度相似性,筛选出一对PCR扩增效率高的引物F0.7/R3,用于同时扩增和,通过对PCR产物进行测序并读取测序峰图,可精准地区分和鉴定回交后代中的4种基因型(AABB、AaBB、AABb和AaBb)[20]。但是由于和序列的相互干扰,也导致该方法也有一定的局限性,不能准确区分Aa/aa和Bb/bb基因型,不能用于自交后代的基因型鉴定,该方法与KASP标记法配合可完整高效地用于花生高油酸回交育种流程。
无论获得F1还是不同回交世代的人工授粉过程,本研究一直将轮回亲本作为母本,这样做的益处有3个:其一,保证获得的高油酸品系与其轮回亲本相比,其细胞质控制的遗传物质没有变化。其二,由于采用人工授粉不可避免地会有花粉去不干净导致的假杂种产生,以轮回亲本(AABB)为母本,仅仅会产生一种基因型的假杂种(AABB),母本植株上收获的基因型AaBb的后代均为人工授粉成功的真杂种。而如果将回交后代(AaBb)作为母本,一旦由于去雄不彻底而产生假杂种,假杂种(即母本自交)的基因型会非常复杂,其中AaBb基因型的假杂种可能会被误选,并作为下一代回交亲本,严重影响回交育种效果。其三,将AaBb基因型的人工杂交后代作为回交父本,不仅可在进行回交时提供花粉,父本植株上自交的种子如F2、BC1F2-BC4F2也采用系谱法进行选择,从而可最大限度地利用不同的回交后代开展高油酸育种工作。
从理论上讲,回交4代的材料与其轮回亲本相比,细胞质遗传完全相同,细胞核遗传物质有96%以上的一致性,但由于连锁累赘导致后代回复率远没理论上高[28],尤其是一些数量性状在回交后代中没能得到完全保留,如中花16和徐花13的高含油量性状在其回交后代获得的近等基因系中均未能检测到超亲的材料。然而,对于作为质量性状的青枯病抗性,在中花21和泉花551的回交后代中得到很好的保留,本研究还发现同一回交组合不同株系之间、株系与轮回亲本之间相比,在植株性状、重要农艺性状、重要抗性、品质指标上仍有部分差异,SSR分析也证实了这一结论。与表型法相比,SSR标记法是评判回交育种效率相对有效的方法,但由于SSR标记毕竟只是整个基因组的一部分,用SSR评估的回交恢复率仍然有很多不足之处。
由于提高油酸可同时提升花生的营养品质和货架寿命,高油酸已成为当前国际公认的花生品种改良重要目标。然而,种子油酸含量的大幅提高是否会给花生产量、抗性和其他品质指标带来一些不利的影响目前尚未研究清楚,仅有的研究表明高油酸花生受低温影响发芽率显著下降[29],不同研究者也发现高油酸与普通油酸花生相比,其黄曲霉产毒抗性、棕榈酸含量存在显著差异[30-32]。本项目通过回交育种获得的高油酸材料与其轮回亲本可作为遗传背景最为接近的近等基因系材料,是评价高油酸对其他重要性状影响的可靠材料。
4 结论
利用连续回交,南繁加代和分子标记辅助选择等技术可在3年内快速实现现有推广品种的高油酸化改良。对植株、农艺性状和抗病性考察并结合SSR标记检测,回交获得的高油酸后代与轮回亲本高度相似,筛选出4个最优回交后代株系ZJ019、ZJ109、ZJ160和ZJ805,作为4个优良轮回亲本中花16、中花21、泉花551和徐花13各自对应的改良高油酸酸品种,其油酸含量分别为82.54%、79.85%、79.22%、78.94%,表明利用这一技术体系实现了推广花生品种的高油酸化改良。
致谢:该研究得到“玛氏-中国花生高油酸育种计划”项目的资助,获得玛氏北京公司温若愚博士, 玛氏北美公司Victor Nwosu博士,河南省农业科学院张新友、黄冰艳研究员和美国农业部农业研究服务处作物保护和管理研究所Guo Baozhu教授在研究中所提供的帮助,在此表示感谢。
[1] http://www.fao.org/faostat/en/
[2] 禹山林. 中国花生品种及其系谱. 上海: 上海科技出版社, 2008.
Yu S L.. Shanghai: Shanghai science and technology press, 2008. (in Chinese)
[3] Janila P, Pandey M K, Shasidhar Y,VARIATH M T, SRISWATHI M, KHERA P, MANOHAR S S, NAGESH P, VISHWAKARMA M K, MISHRA G P, RADHAKRISHNAN T, MANIVANNAN N, DOBARIYA K L, VASANTHI R P, VARSHNEY R K. Molecular breeding for introgression of fatty acid desaturase mutant alleles (ahFAD2A and ahFAD2B) enhances oil quality in high and low oil containing peanut genotypes., 2016, 242: 203-213.
[4] Braddoc J C, Sims C A, O'Keefe S F. Flavor and oxidative stability of roasted high oleic acid peanuts., 1995, 60: 489-493.
[5] Mugendi J B, Sims C A, Gorget D W, O'Keefe S F. Flavor stability of high-oleic peanuts stored at low humidity., 1998, 75: 21-25.
[6] O’Bryne D J, Knauft D A, Shireman R B. Low fat- monounsaturated rich diets containing high-oleic peanuts improve serum lipoprotein profiles.1997, 32: 687-695.
[7] 赵术珍, 侯蕾, 李长生,赵传志, 任丽, 李爱芹, 邓丽, 夏晗, 王兴军. 分子标记辅助回交选育高油酸花生新种质. 中国油料作物学报, 2017, 39(1): 30-36.
ZHAO S Z, HOU L, LI C S, ZHAO C Z, REN L, LI A Q, DENG L, XIA H, WANG X J. Development of high oleic acid peanut from molecular marker assisted selection., 2017, 39(1): 30-36. (in Chinese)
[8] NAWADE B, BOSAMIA T C, THANKAPPAN R, RATHNAKUMAR A,KUMAR A, DOBARIA J R, KUNDU R, MISHRA G P. Insights into the indian peanut genotypes for ahFAD2 gene polymorphism regulating its oleic and linoleic acid fluxes., 2016, 7(1271): 173-184.
[9] Norden A J, Gorbet D W, Knauft D A, Young C T. Variability in oil quality among peanut genotypes in the Florida breeding program.1987, 14: 7-11.
[10] Okuley J, Lightner J, Feldmann K, Yadav N, Lark E, Browse J.FAD2 gene encodes the enzyme that is essential for polyunsaturated lipid synthesis., 1994, 6: 147-158.
[11] JUNG S, SWIFT D, SENGOKU E, PATEL M, TEULÉ F, POWELL G, MOORE K,ABBOTT A. The high oleate trait in the cultivated peanut [, L.] :I. Isolation and characterization of two genes encoding microsomal oleoyl-PC desaturases., 2000, 263(5):796-805.
[12] Wang C T, Hu D Q, Ding F Y, Yu H T, Tang Y Y, Wang X Z, Zhang J C, Chen D X. A new set of allele-specific PCR primers for identification of true hybrids in normal oleate × high oleate crosses in groundnut., 2011, 12(9):1-4.
[13] Chen Z, Wang M L, Barkley N A, Pittman R N.A simple allele-specific PCR assay for detecting FAD2 alleles in both A and B genomes of the cultivated peanut for high-oleate trait selection., 2010, 28: 542-548.
[14] Barkley N A, Chamberlin K D C, Wang M L, Pittman R N. Development of a real-time PCR genotyping assay to identify high oleic acid peanuts (L.)., 2010, 25: 541-548.
[15] Chu Y, Ramos L, Holbrook C C, Ozias-Akins P. Frequency of a Loss-of-Function Mutation in Oleoyl-PC Desaturase()in the Mini-Core of the U.S. Peanut Germplasm Collection.. 2007, 47(6):2372-2378.
[16] 曹永强, 孙石. 回交在转基因作物育种中的应用. 作物杂志, 2014(1): 9-14.
CAO Y Q, SUN S. Backcross breeding genetically modified crops in the application., 2014(1): 9-14. (in Chinese)
[17] 王传堂, 王秀贞, 唐月异, 吴琪, 孙全喜, 张建成, 崔凤高, 李利民, 苗昊翠.中国高油酸花生种质创制、品种选育进展与建议. 花生学报, 2015(2): 49-53.
WANG C T, WANG X Z, TANG Y Y, WU Q, SUN Q X, ZHANG J C, CUI F G, LI L M, MIAO H C. High-oleic peanut germplasm enhancement and cultivar releases in China: Main achievements and suggestions., 2015(2): 49-53. (in Chinese)
[18] 成良强. 花生遗传图谱构建及产量相关性状的QTL分析[D]. 北京: 中国农业科学院, 2014.
CHENG L Q. Construction of genetic linkage map and QTL analysis for yield related traits in peanut (L.) [D]. Beijing: Chinese Academy of Agricultural Sciences, 2014. (in Chinese)
[19] 王传堂, 王秀贞, 唐月异, 张建成, 陈殿绪, 崔凤高, 禹山林, 于树涛. 花生健康组织和病组织简便快速DNA提取方法: 中国, CN 101805730 A. 200910255786.0.
WANG C T, WANG X Z, TANG Y Y, ZHANG J C, CHEN D X, CUI F G,YU S L, YU S T. A simple and rapid DNA extraction method from healthy and disease tissues in peanut: China, CN 101805730 A, 200910255786.0. (in Chinese)
[20] 廖伯寿, 雷永, 姜慧芳, 晏立英, 王志慧, 万丽云, 淮东欣.一种花生高油酸回交育种后代基因型的快速精准鉴定方法: 中国, CN105200142 A. 201510675886.4.
Liao B S, Lei Y, Jiang H F, Yan L Y, Wang Z H, Wan L Y, Huai D X. A rapid and accurate method for identifying the genotype of backcross progenies from high oleate peanut breeding: China, CN105200142 A. 201510675886.4. (in Chinese)
[21] Zhao S Z, Li A Q, Li C S, XIA H, ZHAO C Z, ZHANG Y, LEI H, WANG X J. Development and application of KASP marker for high throughput detection of AhFAD2, mutation in peanut., 2017, 25(C): 9-12.
[22] 姜慧芳, 段乃雄. 花生种质资源描述规范和数据标准. 北京: 中国农业出版社, 2006.
Jiang H F, Duan N X.. Beijing:China agriculture press, 2006. (in Chinese)
[23] 郭建斌. 花生含油量及脂肪酸组成的QTL分析[D]. 武汉: 华中农业大学, 2016.
GUO J B. QTL Analysis for oil content and fatty acid traits in Peanut (L.)[D]. Wuhan: Huazhong Agricultural University, 2016. (in Chinese)
[24] 段红梅. 利用大豆SSR标记辅助遗传背景选择的效果分析[D]. 北京: 中国农业科学院, 2002.
DUAN H M. efficiency selection of genetic background using SSR markers in soybean [D]. Beijing: Chinese Academy of Agricultural Sciences, 2002. (in Chinese)
[25] SHIRASAWA K, BERTIOLI D J, VARSHNEY R K, MORETZSOHN M C, LEAL-BERTIOLI S C M, THUDI M, PANDEY M K, RAMI, J F, FONCÉKA, D, GOWDA M V C, QIN H D, GUO B Z, HONG Y B, LIANG X Q, HIRAKAWA H, TABATA S, ISOBE S. Integrated consensus map of cultivated peanut and wild relatives reveals structures of the A and B Genomes ofand divergence of the legume genomes., 2013, 20(2): 173.
[26] Zhou X J, Yang D, Zhao J J, HUANG L, REN X P, CHEN Y N,HUANG S M, LIAO B S, LEI Y, YAN L Y, JIANG H F. Genomic survey sequencing for development and validation of single-locus SSR markers in peanut (L.)., 2016, 17(1): 420.
[27] 李丽, 何美敬, 崔顺立, 侯名语, 陈焕英, 杨鑫雷, 王鹏超, 刘立峰, 穆国俊. 高油酸、中果型花生新材料的创制与鉴定. 中国农业科学, 2014, 47(19): 3898-3906.
LI L, HE M J, CUI S L, HOU M Y, CHEN H Y, YANG X L, WANG P C, LIU L F, MU G J. The development and identification of new peanut germplasm materials with high oleic acid and medium pod., 2014. (in Chinese)
[28] XU S S, CHU C G, HARRIS M O, WILLIAMS C E.. Comparative analysis of genetic background in eight near-isogenic wheat lines with different H genes conferring resistance to Hessian fly., 2011, 54(1): 81.
[29] Jungman B S. The effect of fatty acid profiles on peanut seed germination at low soil temperatures[D]. Lubbock: Texas Tech University, 2000.
[30] XUE H Q, ISLEIB T G, PAYNE G A, WILSON R F, NOVITZKY W P, BRIAN G O. Comparison of aflatoxin production in normal- and high-oleic backcross-derived peanut lines., 2003, 87(11): 1360-1365.
[31] HOLBROOK C C, WILSON D M, MATHERON M E, HUNTER J E, KNAUFT D A, GORBET D W.Aspergillus colonization and aflatoxin contamination in peanut genotypes with reduced linoleic acid composition., 2000, 84(2):148-150.
[32] BARKLEY N A, ISLEIB T G, WANG M L, PITTMAN R N. Genotypic effect of ahFAD2 on fatty acid profiles in six segregating peanut (L.) populations., 2013, 14(1): 62.
(责任编辑 李莉)
Fast Development of High Oleate Peanut Cultivars by Using Marker-Assisted Backcrossingand Their Evaluation
ZHANG ZhaoHua1, WANG ZhiHui1, HUAI DongXin1, TAN JiaZhuang2, CHEN JianHong3, YAN LiYing1, WANG XiaoJun4, WAN LiYun1, CHEN Ao2, Kang YanPing1, JIANG HuiFang1, LEI Yong1, LIAO BoShou1
(1Oil Crops Research Institute of the Chinese Academy of Agricultural Sciences/Key Laboratory of Biology and Genetic Improvement of Oil Crops, Ministry of Agriculture, Wuhan 430062;2Zhanjiang Institute of Agricultural Sciences, Zhanjiang 524094, Guangdong;3Quanzhou Institute of Agricultural Sciences, Quanzhou 362212, Fujian;4Xuzhou Institute of Agricultural Sciences in Jiangsu Xuhuai District, Xuzhou 221131, Jiangsu)
【Objective】The objectives of this research were (i) to establish an effective backcrossing approach combined with marker-assisted selection (MAS) of peanut high oleate breeding, (ii) to transform the elite peanut cultivars with normal oleate (NO) into high oleate (HO) ones in three years.【Method】Four cross combinations were designed named as BO-01, BO-02, BO-03 and BO-04 by selecting the elite peanut cultivars ZH16, ZH21, QH551 and XH13 with AABB genotype as recurrent parents, while JH13 with aabb genotype was selected as the high oleate donor parent. Via 1 cross, 4 backcrosses and 1 self-crossing,BC4F2progenies were obtained.According to the separation rule of backcross and high homology sequence ofand, F1and backcross progenies (BC1F1-BC4F1) were genotyped by sequencing of PCR products, in whichandwere amplified in one PCR reaction with F0.7/R3 primer pairs. AaBb genotype progenies were selected and used as male parent in next generation backcross. Self-crossing progenies (BC4F2- BC4F3) were examined by KASP (kompetitive allele specific PCR) to obtain aabb genotype progenies.Main agronomic traits, quality characters and disease resistance of aabb genotype progenies were investigated.The geneticbackgrounds of progenies with aabb genotype derived from ZH 16×JH13 were evaluated with SSR markers. 【Result】In summary, during 3 years, we obtained 10, 5, 6 and 8 high oleate progenies with aabb genotype in 4 cross combinations,respectively. By evaluating their agronomic traits, quality and bacterial wilt resistance,high oleate peanut cultivars were obtained, which were highly consistent with their corresponding recurrent female parent. ZJ019, ZJ109, ZJ160 and ZJ805 witholeic content as 82.54%, 79.85%, 79.22% and 78.94%, are regarded as high oleic cultivars of ZH16, ZH21, QH551 and XH13.【Conclusion】By using backcross, off-season multiplication for advancing generation and molecular marker selection, normal oleate peanut cultivars were successfully and quickly transited into high oleate ones within 3 years.
peanut; high oleic acid; backcross; marker-assisted selection (MAS); cultivar
10.3864/j.issn.0578-1752.2018.09.003
2017-12-01;
2018-02-25
国家自然科学基金(31671734,31071456,31371662,31461143062)、国家花生产业技术体系(CARS-13)、国家“863”计划(2013AA102602)
张照华,E-mail:zhaohuazhang9121@126.com。
雷永,E-mail:leiyong@caas.cn。 通信作者廖伯寿,E-mail:lboshou@hotmail.com
