狂蝇科昆虫线粒体COⅠ基因组研究进展
2018-05-040008乌审旗兽医局073000008
,,,, *,(. , 0008;.乌审旗兽医局, 07300;3. , 0008)
狂蝇科属节肢动物门(Arthropoda),昆虫纲(Insecta),双翅目(Diptera),短角亚目(Brachycera),从古北区到亚热带,分布于世界各地范围内,幼虫引发感染。由于狂蝇科不同的种属,寄主与寄生部位也不同,如动物呼吸道、肠道及皮下组织等能引起人类和动物的强制性蝇蛆病。蝇蛆病对动物福利和动物生产力具有严重影响,是造成畜牧业重大经济损失的一大因素。另外,有些狂蝇科幼虫引起的蝇蛆病能够在动物表皮和内部感染,导致动物健康受到危害甚至造成死亡。
狂蝇科根据现有常规分类法分为四个亚科,即狂蝇亚科、皮蝇亚科、胃蝇亚科及蛆蝇亚科,其中含有一百多个已被鉴定的种属[1]。尽管形态学分类在昆虫分类系统中一直占主导地位, 但由于受个体发育阶段的形态特征、环境条件及标本材料等因素的限制, 一些昆虫种类的地位分类和进化关系不易确定。分子信息作为形态分类的必要补充可提供具有连续性、可度量性、相对恒定性的分类和进化依据,且分子进化速率可反映物种进化变异的积累和进化距离。
细胞色素C氧化酶亚基I(COⅠ)位于线粒体基因上,是分子系统遗传标记基因,其分子量最大、基因成稳定状、基因排列及功能结构相对保守,普遍为母系遗传,极少发生重组等,因而被广泛用于进化、系统发育、种群遗传结构、基因漂流、杂交和生物地理学等方面的研究[2]。COⅠ基因包含电子传递和跨膜反应两种,其范围包括配体位点、质子通道的合成、螺旋结构及散布的亲水性环状链等不同形式和功能区域。COⅠ 基因不但保证一定程度的变异而且又很容易被通用引物扩增,其基因序列本身很少存在插入和缺失[3]。在DNA序列上比其他蛋白编码基因变异慢,包含的系统发育信号多,所以相比之下更适合用来分析亲缘关系密切的分类种群间的遗传关系。同时,COⅠ基因同其他蛋白编码基因共有某些特征,如密码子第3位碱基不受自然选择压力的影响,可以自由突变[4]。因此,在实践中绝大多数昆虫类相互区分的遗传标记选作线粒体基因组COⅠ基因5'端的区域,其长度在400到700碱基对,相邻的保守序列使得这个区域易于扩增及分析[5]。本文针对狂蝇科线粒体COⅠ基因分子信息学研究概况、研究进展等进行总结概括,为今后更多狂蝇科昆虫研究及蝇蛆病的研究提供参考。
1 狂蝇科昆虫线粒体基因组及COⅠ基因的特点
1.1 线粒体基因组
线粒体广泛存在于昆虫体内,在控制昆虫新陈代谢、生命周期、凋亡和病害等方面起着积极作用。细胞中线粒体的位置常临近于需要ATP的结构或者处于细胞进行氧化作用需要的燃料附近,如昆虫的飞行肌中和卵内。同其他双翅目昆虫一样,狂蝇科昆虫线粒体基因具有闭合双链DNA结构,大小一般在14~20 kb之间, 线粒体基因组上的多数基因在J链上编码 ( majority strand) ,少数基因在N链上编码 ( minority strand)[6]。其中稳定的包含有13个编码蛋白基因分别为细胞色素C氧化酶亚基I、II、III(即CO I、CO II、CO III),细胞色素b脱辅基酶(cytb),ATP合成酶亚基6和8(atp 6和atp 8),NADH脱氢酶亚基1-6(nad1-6)和4L(nad4l); 22 个tRNA 基因,包含18 个( trnA、trnR、trnN、trnD、trnC、trnQ、trnE、trnG、trnH、trnI、trnK、trnM、trnF、trnP、trnT、trnW、trnY 和trnV) 分别对应18 种不同的氨基酸,特殊的是丝氨酸( Ser)和亮氨酸( Leu) 的tRNA 对应2个,分别是trnL1( CUN)、trnL2( UUR)及trnS1( UCN )、trnS2( AGN);2个 rRNA 基因rrnL 和rrnS分别编码大亚基16S ( 16S rRNA) 和小亚基12S( 12S rRNA)[3]。
1.2 线粒体COⅠ基因结构与基因排列
COI氨基酸序列由五个结构二十五个区域组成,12个跨膜螺旋区(M1-M12)、6个外环区(E1-E6)、5个内环区(I1-I5)、羧基(COOH)及氨基终端(NH2)(见图1)[7]。图中方形区域具有特殊实用性作用,跨膜螺旋区M2、M6、M7和M10在COⅠ基因主要反应中提供配位金属离子,即血红素与铜原子之间互相作用反应。M8也是高度保守区域之一,与色素氧化酶质子传递途径有关联。此区域包含三个极性氨基酸残基(thr-352、thr-359和lys-362),这三个氨基酸残基在生物体内呈完全保守状态,且在传递活动中起着必不可少的作用[8]。
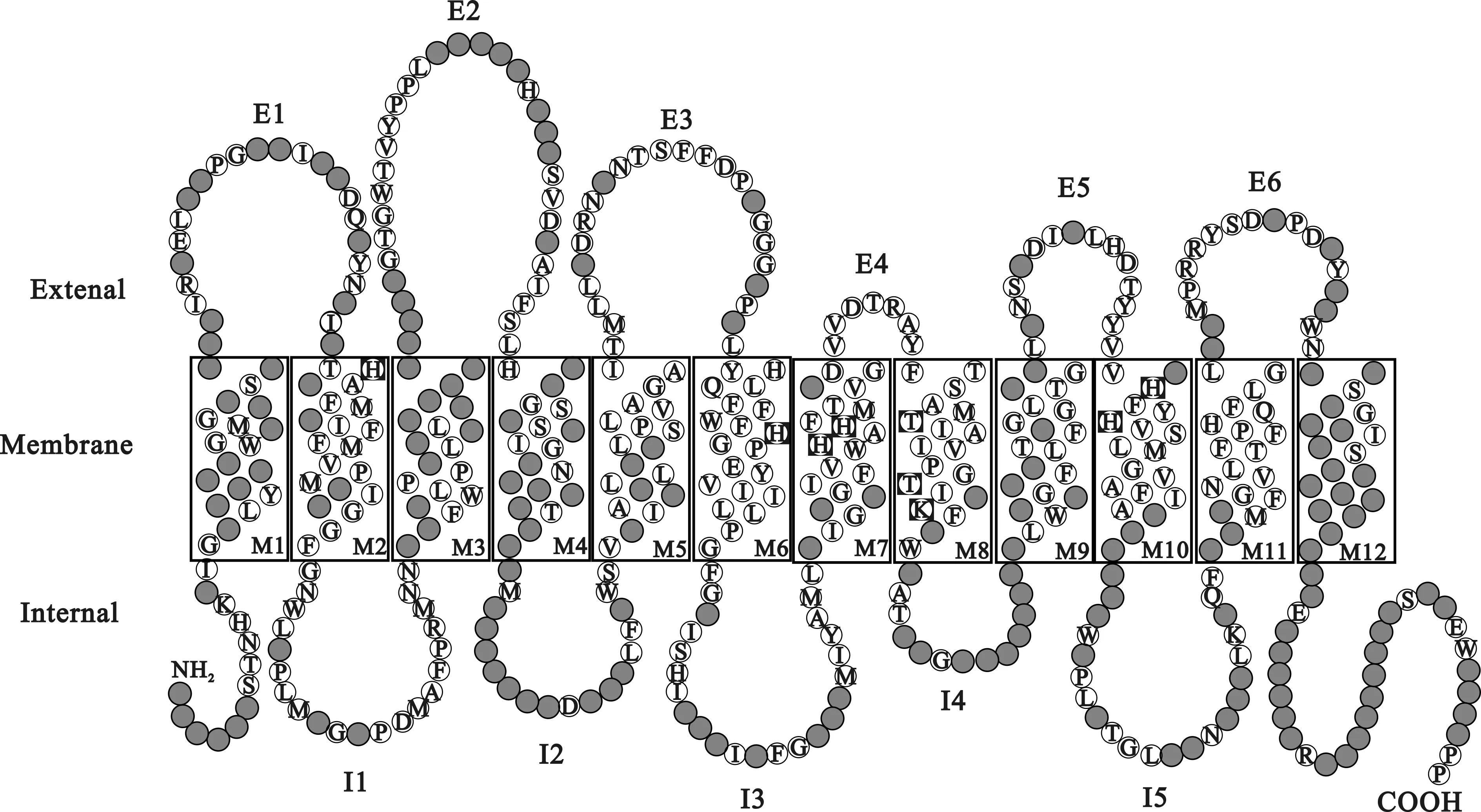
图1 昆虫COⅠ基因的二维模型图[7]填充的小圈为变异率高区 Fig.1 A two-dimensional model of the insect COⅠ gene Filled circles represent residues observed to be variable between insect species
2 狂蝇科昆虫线粒体COⅠ基因组的碱基组成及变异相关系数
特异性靶基因区域COⅠ基因作为区分狂蝇科的有力工具,不单单为种属的鉴定而提供资料,还为昆虫界的系统发育研究有所贡献。结合Clary、Crozier、Howland 等的研究显示,狂蝇科昆虫与其他昆虫线粒体基因(mtDNA)一样,由于第三密码子稳定的偏向性支持腺嘌呤和胸腺嘧啶,因而核苷酸成分显示A+T含量显著高,占60%以上[9]。Otranto等[10]早在2003对狂蝇科昆虫mtDNA基因COⅠ 序列688位点作出系统分析,其中含有385保守性位点和303可变位点,在可变位点中有48单独位点和255简约信息的变异位点。狂蝇总科内多数种属在第三位点同义替换较多,高达86%但不会影响氨基酸成分,核苷酸变异率在整个有效区域都很高(303/688)。多数第三密码子位点变异性最强;第一、二位点保守性强,且非同义替换大多数发生在第一位点达到28.4%(见表1)[11]。
3 狂蝇科昆虫线粒体COⅠ基因进化速率
综合Gennis、lunt和 Caterino的研究可推测,狂蝇科昆虫COⅠ基因羧基区域(COOH)氨基酸变异率高于跨膜螺旋区(M)、外环区(E)、内环区(I)及氨基终端(NH2)。该区域相关的几个重要的功能域M5-M8显示低水平变异率。跨膜螺旋区M3、M9、M12变异率高;外环区E3、E5尤其E4为极度保守区,内环区内I1相比另四个区显著保守(见图2)。Lunt等[7]在昆虫COⅠ基因研究中运用kruskal-wallis检验(KW test),测得结果为拒绝零假设成立,即氨基酸平均变异率、各个位点变异率及五个结构阶层之间变异率存在显著差异。当五个结构域的每个碱基位点被计算其变异率时COOH羧端变异率比其他任何区域都高,且氨基终端、跨膜螺旋区、内环区及外环区变异率无显著差异(见表2) 。
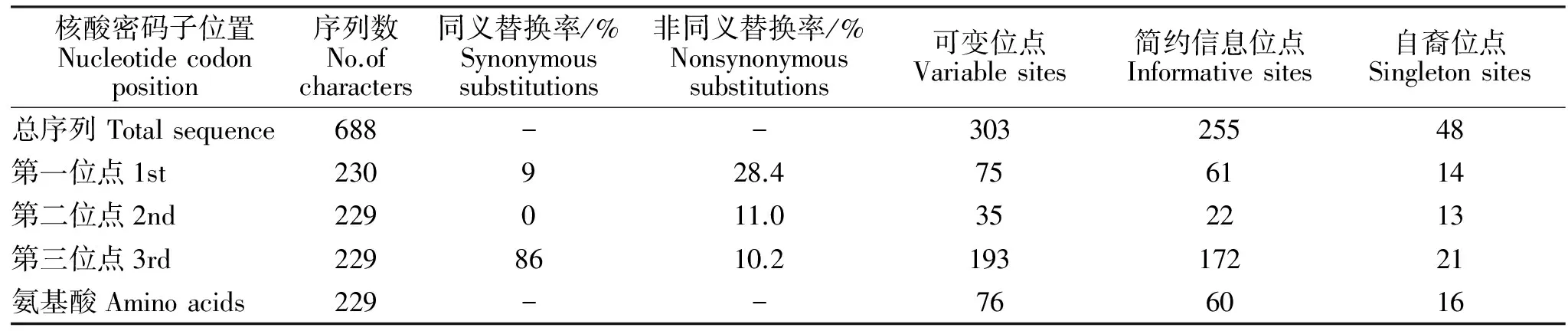
表1 狂蝇科线粒体COⅠ基因同义替换和非同义替换百分比,变异位点、简约信息位点及自裔位点的位置Table 1 Percentage of synonymous and non-synonymous substitutions and variable, informative and singleton sites occurring in total sequences at different codon positions of the COI region examined
4 mtCOI基因在狂蝇科昆虫分子系统学研究中的应用
相比狂蝇科昆虫庞大的物种数,目前已完成线粒体COⅠ基因测序的狂蝇科种类还很少,因而获得更多狂蝇科昆虫线粒体COⅠ基因序列显得极为迫切。随着测序技术的发展,研究者应针对狂蝇科的更多种属,特别是具有经济意义的种属,获得其线粒体COⅠ基因序列,通过对碱基组成、进化速率、基因重排规律及其产生的原因等方面的分析,深入了解狂蝇科昆虫线粒体COⅠ基因的进化趋势和特征,并在此基础上选择更佳的分析手段进行后续的起源进化、系统发育、种类鉴定、种群溯源以及数据库与网站建设等研究,为我国及全球狂蝇入侵防控提供客观准确的依据与技术支持,应用前景广阔。综合研究结果,显示COⅠ基因能够用于研究双翅目狂蝇科各类群昆虫的系统发育关系和进化模式,验证传统分类方法、建立物种的自然系统和进化关系,对物种的区别鉴定、发现物种隐存种、研究物种分岐分化及遗传多样性有积极作用[12]。线粒体COⅠ基因的研究是对狂蝇总科进化模式的鉴定,甚至可以在不久的将来研究宿主与环境之间的关系起着至关重要的数据依据。
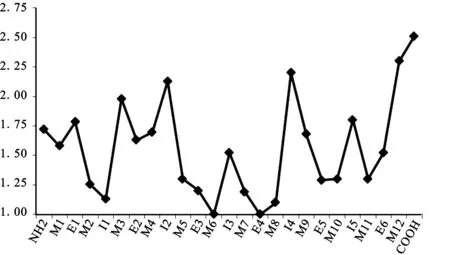

图2 昆虫COⅠ基因各个区域平均变异率Fig. 2 The mean amino acid variability for the twenty-five structural regions of the insect COⅠ gene
4.1 mtCOⅠ基因在起源和进化研究中的应用
在狂蝇科种属或在更高的水平中,COⅠ基因的研究更有意义。分子学数据构成一个基因数据库对分化时间较早的属、种及种下物种和地理种群等进行系统发育研究提供鉴定物种的有效依据。2003年,Otranto等[13]基于线粒体COⅠ基因建立了狂蝇科18个种属的的系统发育树,并估算狂蝇科昆虫线粒体COⅠ核苷酸的进化速率。表明18种狂蝇科昆虫归类于4个亚科,本结论与形态学及生物学分类法得出结论相一致[13];无脊椎动物每百万年(my)±0.5 my核苷酸置换率大约为2%[14],鉴于此资料可以推测出狂蝇科亚科间分岐时间,例如皮蝇科H.tarandi与HypodermaactaeonBrauer分歧时间为8.2±0.5 my,或者可以看出G.intestinalis和G.haemorrhoidalis、C.stimulator和C.trompe等物种分裂形成的时期。杨晓野等[15]对驯鹿狂蝇蝇蛆及其成蝇的COI 基因特异性序列进行比较分析,结果为COⅠ基因核苷酸序列在某程度上,可以反映出种属及株间进化程度。
4.2 mtCOⅠ基因种群遗传多样性及作为DNA条形码的应用
早前研究表明,Oestrus.ovis与伊伯利亚野生山羊狂蝇科昆虫在形态学及生物学上有着极大相似性[16-17],但要鉴定其种类此数据资料远远不够。在此研究的基础上,Moreno等[18]对伊伯利亚野生山羊狂蝇科昆虫COⅠ 基因进行扩增,测序结果与GenBank上的其他Oestrus.ovis对比相似度在90.3%~91.5%。基于Otranto等[10]关于COⅠ基因种属鉴定数据:COⅠ基因序列在狂蝇科种间相似值为81.9%,亚科间相似水平分别为皮蝇亚科86.8%、狂蝇亚科86.7%、胃蝇亚科90%、蛆蝇亚科 94.7%,推断此伊伯利亚野生山羊寄生昆虫与Oestrus.ovis属于同一个亚科,但绝不是同一个种类。
2004年,Otranto和Traversa[19]用分子鉴定法得出三个Przhevalskiana种的COⅠ基因序列的种间差异在0.19%~0.29%,结合前期研究得出的结论,狂蝇科种间差异高于6%且种内差异低于1%,推测Przhevalskianasilenus、Przhevalskianaaegagri和Przhevalskianacrossii属于狂蝇科内同一个物种。
Moreno等[18]对几个地方品种及野生绵羊、山羊寄生的狂蝇科昆虫DNA条形码COⅠ序列进行研究,测得狂蝇科线粒体COⅠ基因的96个新序列(登记在GenBank,KP974835-KP974930),与数据库的羊狂蝇有效CO I序列(GenBank AF497767)比较,显示相似度在98.1%~99.5%之间,由此可以鉴定属于同一个属。
Yong等[20]利用线粒体COⅠ基因对皮蝇科昆虫进行遗传多样性及群体遗传学研究,具体对CO I序列变异度、基因多样性、基因分化、种群结构及种群发生分岐时间做出了系统的结论。此研究对今后的研究给予了数据及资料参照,有利于对狂蝇科昆虫的形态学、生物地理信息及分子系统发育方面做出更完整的研究。
5 展 望
新一代测序技术为开展大规模线粒体基因组的研究提供了良好的契机,能够快速、高效地获得大量线粒体基因组序列。此技术有助于对更多狂蝇科昆虫进行线粒体COⅠ基因测序,但对测序物种种类以及规模的增加和基础原数据的积累仍是目前昆虫线粒体COⅠ基因组研究发展的首要工作。总之,对线粒体COⅠ基因组在狂蝇科昆虫系统进化研究中的价值还未进行系统评价, 这是今后需着重努力的方向。同时, 还应将线粒体基因组数据与核基因及形态学特征等数据结合起来进行综合分析, 共同推动狂蝇科昆虫的系统进化研究。
今后在对动物疫病临床检疫、诊断方面,可以利用线粒体CO I技术开展流行病学调查,对病原媒介生物等进行分型鉴定、类症鉴别,对动物蝇蛆病进行诊断、鉴定、分类等等,此研究有助于养殖工作人员增强对动物疾病的预防和控制。
参考文献:
[1] 魏书军,陈学新.昆虫比较线粒体基因组学研究进展[J].应用昆虫学报,2011,48(6):1 573-1 585.
[2] 郭仲龙,袁明龙.半翅目昆虫线粒体基因组学研究进展[J].中国科学:生命科学,2016,46(2):151-166.
[3] 王鑫,黄兵.DNA条形编码技术在动物分类中的研究进展[J].生物技术通报,2006(4):67-72.
[4] 马英,鲁亮.DNA条形码技术研究新进展[J].中国媒介生物学及控制杂志,2010,21(3):275-280.
[5] 田应峰,王东林,苗永旺.DNA条形码技术及其在畜牧兽医上的应用[J].上海畜牧兽医通讯,2016(5):47-50.
[6]SIMON C,FRATI F,BECKENBACH A,et al.Evolution,weighting and phylogenetic utility of mitochondrial gene sequences and a compilation of conserved polymerase chain reaction primers[J].Ann Entomol Soc Am,1994,87( 6) : 651-701.
[7] LUNT D H, ZHANG D-x, SZYMURA J M, et al. The insect cytochrome oxidase I gene: evolutionary patterns and conserved primers for phylogenetic studies[J]. Insect Molecular Biology, 1996, 5(3): 153-165.
[8] GENNIS R B. Site-directed mutagenesis studies on subunit I of the aa3-type cytochrome c oxidase of Rhodobacter sphaeroides: a brief review of progress to date[J]. Biochimica et Biophysica acta, 1992, 1101(2): 184-187.
[9] OTRANTO D, MILILLO P, TRAVERSA D, et al. Morphological variability and genetic identity in Rhinoestrus spp. causing horse nasal myiasis[J]. Medical & Veterinary Entomology, 2005, 19(1):96-100.
[10] OTRANTO D,MILILLO P,TRAVERSA D,et al. Morphological variability and genetic identity in Rhinoestrus spp.Causing horse nasal myiasis[J].Medical & Veterinarg Entomology,2005,19(1):96-100.
[11] HOWLAND D E, HEWITT G M. Phylogeny of the coleoptera based on mitochondrial cytochrome oxidase I sequence data[J]. Insect Molecular Biology, 1995, 4(3): 203-215.
[12] 池宇,王诗迪,张春田. 基于线粒体COⅠ基因的双翅目昆虫研究进展[J]. 昆虫分类学报,2010,32(S):71-77.
[13] OTRABTI D, TRAVERSA D, Guida B, et al. Molecular characterization of the mitochondrial cytochrome oxidase I gene of Oestridae species causing obligate myiasis[J]. Medical & Veterinary Entomology, 2003, 17(3):307-315.
[14] POWELL J R, CACCONE A, AMATO G D, et al. Rates of nucleotide substitution in Drosophila mitochondrial DNA and nuclear DNA are similar[J]. Proceedings of the National Academy of Sciences of the United States of America, 1986, 83(23):9 090-9 093.
[15] 杨晓野,李云章,包巴音仓,等. 驯鹿狂蝇(Cephenemyiatrompe)线粒体COⅠ基因序列研究[J]. 中国预防兽医学报,2006,28(5):514-517.
[16] GUITTON C, PEREZ J M, DORCHIES P. Scanning electron microscopy of larval instars and imago of Oestrus caucasicus (Grunin, 1948) (Diptera: Oestridae)[J]. Parasite-journal De La Societe Francaise De Parasitologie, 2001, 8(2):155-160.
[17] PÉREZ J M, GRANADOS J E, MORENO V, et al. In vitro rearing Oestrus caucasicus third-instar larvae and pupae (Diptera: Oestridae) from naturally-infested Iberian ibex, Capra Pyrenaica (Artiodactyla: Bovidae)[J]. Parasite-journal De La Societe Francaise De Parasitologie, 2006, 13(4):305.
[18] MORENO V, ROMEROFERNNDEZ I, MARCHAL J A, et al. Molecular characterization of bot flies, Oestrus spp. (Diptera, Oestridae), from domestic and wild Bovidae hosts[J]. Veterinary Parasitology, 2015, 212(3-4):473-477.
[19] OTRANTO D,TRAVERSA D. Molecular Przhevalskiana silensus, Przhevalskiana Przhevalskiana acrossii (Diptera, Oestridae) are one species[J]. Acta Parasitologia,2004,49:173-176.
[20] YONG F, WEI L, HONG D, et al. Genetic diversity and population genetics of the warble flies Hypoderma bovis and H. sinense in Qinghai Province, China[J]. Parasites & Vectors, 2016, 9(1):1-9.
