基于多通路CNN的多模态MRI神经胶质瘤分割
2018-05-03肖洪兵曹利红
朱 婷 王 瑜 肖洪兵 曹利红
(北京工商大学计算机与信息工程学院食品安全大数据技术北京市重点实验室 北京 100048)
0 引 言
神经胶质瘤是一种高发性脑肿瘤,它由神经胶质细胞病变引发,并且对周围组织有很强的浸润性。根据其危害程度,人们将其分为高级神经胶质瘤HGG(High Grade Gliomas)和低级神经胶质瘤LGG(Low Grade Gliomas)[1]。为保护健康组织,在任何治疗之前都要先确定脑肿瘤区域的准确位置[2]。神经胶质瘤的触角型结构以及与周围组织边界模糊,使得分割任务非常困难[3]。由于MRI具有较好的软组织对比度,能提供丰富的脑部信息[4],目前临床上大多使用MRI对神经胶质瘤作前期诊断[5]。不同模态的MRI能够提供不同类型的组织对比图像信息,常用四种标准的MRI模态,分别是FLAIR、T1、T1c、T2[6]。在临床上,通常是相关领域专家进行手动分割,这种方法耗时耗力,且不同专家存在很大意见上的差异[7]。寻找鲁棒性好的自动分割方法是近几年的一大热门课题。
目前,很多研究者已提出多种脑肿瘤分割方法,包括区域增长的方法、水平集方法、模糊聚类方法以及深度学习方法等。但是这些方法在脑肿瘤分割过程中大多需要人工干预,比如区域增长的方法需要首先在图像中手动设置种子点[8]。近期,有研究者提出自动选择种子点的方法[9],但是如果将种子点设置在正常的组织区域,分割结果将完全错误。而且,在区域增长方法的处理过程中,图像的噪声和强度不均匀性可能导致空腔或者过分割[10]。水平集方法是基于主动轮廓模型的方法,其难点在于如何选择最佳初始轮廓。例如Rana等[11]利用快限定框在肿瘤区域选择初始轮廓,使用水平集方法精确地分割出肿瘤区域的边界,但是初始轮廓选择错误将引起错误的分割结果。模糊聚类的方法通常与其他方法相结合,例如K均值或者C均值方法[12]。这些方法需要数据分布的先验知识,当数据的假定分布接近于其真实分布时,分割结果较为理想。此外,也有基于生物启发和概念证明的方法被提出,如Buendia等[13]在分组人工免疫网络GAIN(Grouping Artificial Immune Network)的基础上提出增强版GAIN+。该方法主要利用了信息理论,根据输入模式之间交互信息值的大小顺序,在训练期间更新权重,是一种全自动的分割方法,但是需要去噪、膨胀或腐蚀等繁琐的前后期处理操作。Cordier等[14]提出了基于图像块的脑肿瘤分割方法,该方法需要首先使用边界框限定肿瘤区域,然后在边界框内提取图像块,用于相似性检测,属于概念证明的方法。但是需要专家对肿瘤区域进行初步标注定位,所以属于半自动分割方法。
近期,深度学习取得了很高的脑肿瘤分割效果,是非常具有应用潜力的分割方法。深度学习方法中最典型的是CNN[15],它可以直接从数据中学习特征并进行分类[16],因此脑肿瘤分割方面的CNN研究,主要集中在模型结构设计上[17]。脑肿瘤MRI图像是三维结构,Christopher等[18]、Urban等[19]提出用三维图像块作为CNN模型的输入来训练三维CNN模型。这种方法保留了MRI图像的空间信息,但是很大程度上增加了计算量。很多人选择采用二维CNN模型,这种方法首先对三维MR图像沿轴向取切片,然后在切片上取一定尺度的图像块[20],用于训练二维CNN模型。由于CNN的局部特性,很容易丢失全局信息。此外,为了减少模型参数,很多人采用3×3像素的卷积核,并通过多层卷积代替较大的卷积核,但是这种结构容易丢失网络浅层获取的信息。
本文研究了一种新颖的结合浅层信息的多通道CNN模型,用于脑肿瘤分割。通过将图像块的局部特征信息、浅层网络提取的信息与全局特征信息相结合,获得更加丰富的特征,预测图像块中央像素的类标签,并采用3×3像素的卷积核,以减少模型参数,提高模型训练速度。该模型克服了以往CNN模型忽略全局信息与浅层信息的缺点,并通过实验证明了模型的有效性。
1 卷积神经网络结构框架
CNN是一种由数据驱动的监督学习方法[21],它能直接从原始数据中自动学习到分层的复杂特征,在人脸识别[22]、细胞有丝分裂检测[23]、语义分割[24]等方面取得了令人瞩目的效果。它通常由卷积层、激活函数层、池化层交替连接而成,最后一层为全连接层,又称分类层(或损失层),用于对模型前面卷积层、池化层部分提取的特征进行分类。
在卷积层,卷积核尺度较大时可以获得较大的感受野,但是模型参数会较多,因此选择3×3像素的卷积核。并通过增加卷积层数来获得较大感受野,卷积层输出的特征图FMs尺寸按式(1)计算。
N=M-P+1
(1)
式中:N×N是卷积层输出的FMs的大小,M×M是卷积层输入的大小,P×P是卷积核的大小。
卷积层之后是激活函数层,激活函数用于拟合非线性,常用的激活函数是线性调整单元ReLU,以及其改进形式LReLU。
激活函数层之后是池化层,池化层相当于下采样的过程,常用的池化形式是最大池化和平均池化。其中,最大池化是指选择池化窗口的最大值作为输出,平均池化是指选择池化窗口的平均值作为输出。
在全连接层FC,使用损失函数监测模型训练过程。该层通过比较网络输出与图像块中央像素点的标签值来驱动网络模型的自学习,直到损失在一定范围内不再增加,则训练结束。
2 神经胶质瘤分割方法
采用图像块训练多通路CNN分割模型一般包括三个步骤: 数据前期处理、选择CNN模型并训练、测试模型分割性能。下面详细阐述基于多通路CNN的多模态MRI神经胶质瘤分割方法。
2.1 数据前期处理
脑肿瘤的MRI图像是三维结构,其像素尺寸大小为240×240×155。3D结构计算复杂度高,一般先取切片,转化成2D结构,其过程如图1所示,再用于训练CNN模型。
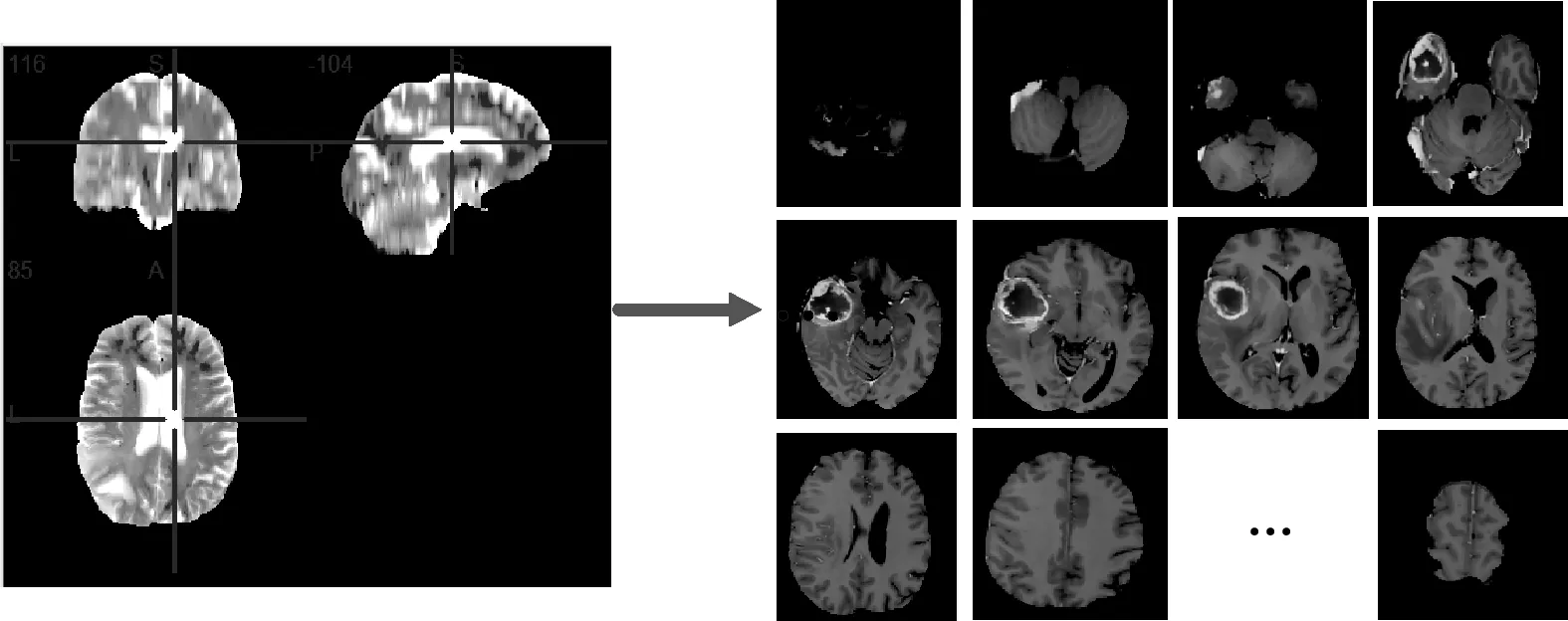
图1 三维MRI数据取切片实例
四种模态相同序列的切片需要先标准化,标准化方式如式(2)和式(3)所示,再合并在一起。

(2)
X=X/Xstd
(3)
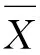
在获得的切片上取像素尺寸大小为33×33像素的图像块。具体过程是:先设定每个类标对应的图像块比例,使得每类的图像块数量近似相等。这里需要将像素分成5类,分别是0—脑部健康区域,1—脑肿瘤坏死区域,2—脑肿瘤水肿区域,3—脑肿瘤非增强区域,4—脑肿瘤增强区域。然后在四种模态相同序列的切片上随机取各类对应的图像块,如图2所示,组成训练集。
图2 240×240×4的图像结构及取切片过程
2.2 模型训练
传统CNN因其局部感知功能,只能提取到图像的局部信息,为了利用更大范围的信息,可以增加卷积核更大的通路,用于提取更大范围的信息—全局信息。然后将局部信息与全局信息相融合,获得更加丰富的图像表征方式。多通路CNN模型的结构如图3所示,包括局部信息提取通路和全局信息提取通路。
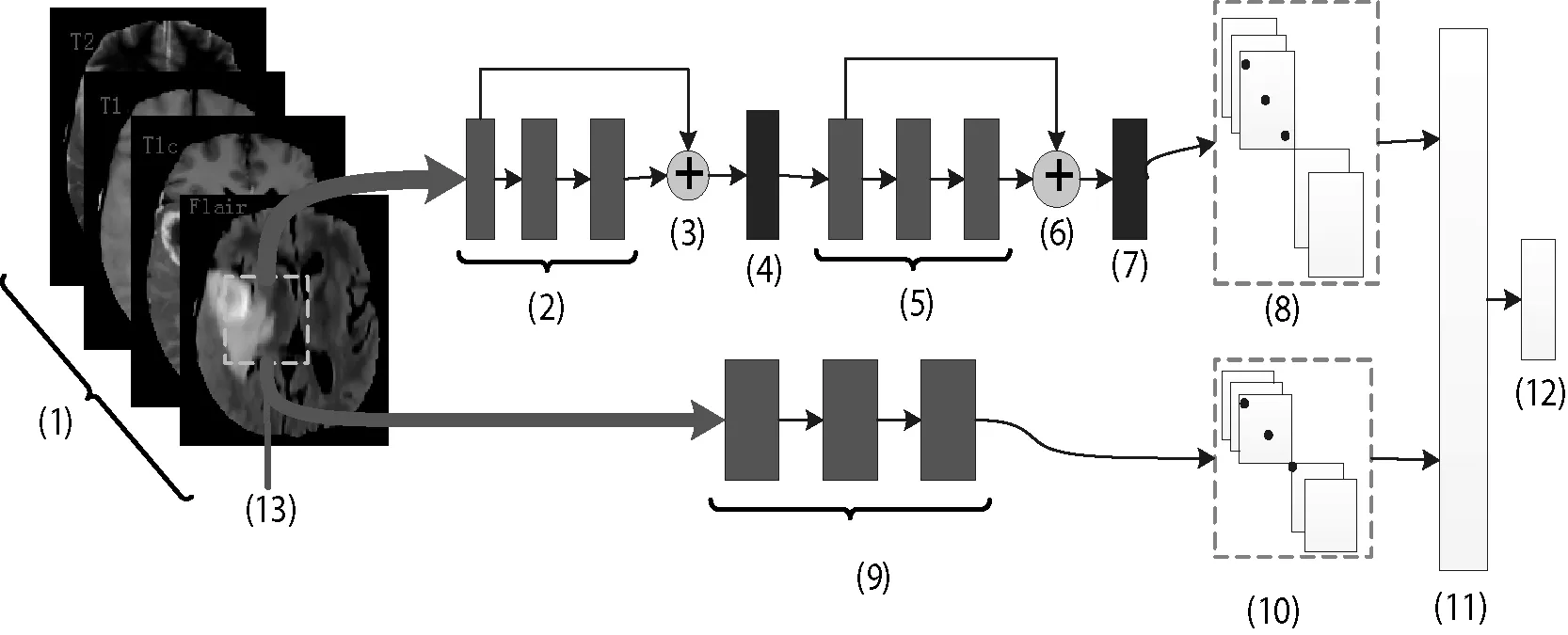
图3 多通路CNN框架结构
图3中,(2)、(5)和(9)中代表卷积块, (4)和(7)是池化层,(11)、(12)分别是特征融合层与分类层。
2.2.1 模型局部信息提取通路
CNN网络能逐层提取越来越复杂的特征,最深层的特征表征了上下文环境的解剖线索。将深层粗糙的语义信息与浅层精细的外观信息(如尺寸、位置等)相结合,组成表达能力更强的特征图谱。同时,由于局部特征提取通路采用较小的3×3像素的卷积核,其对应较小的感受野,因此所述特征图谱可称为局部特征信息。局部特征提取通路的结构形式如图3中上面支路所示。
图3中,(2)、(5)中分别包含三个卷积核为3×3像素的卷积层,(3)和(6)表示深浅层信息融合,融合方式如图4所示。
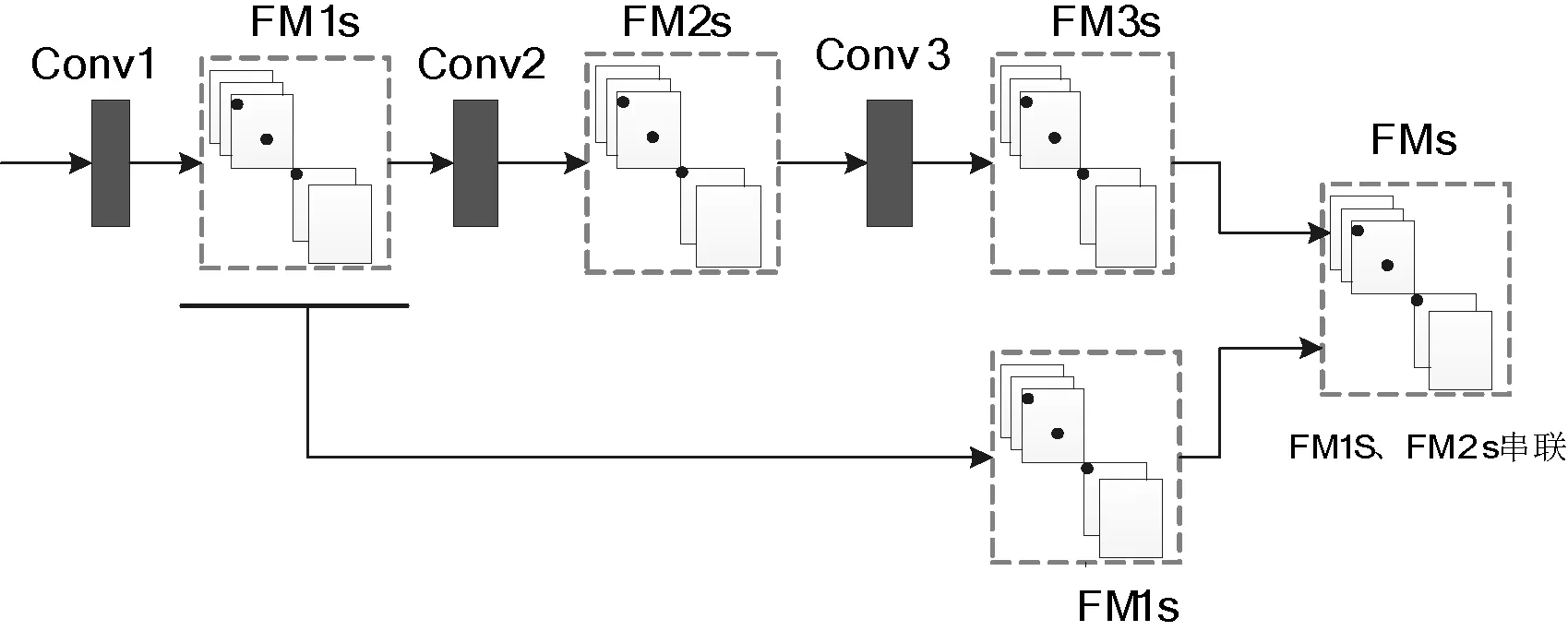
图4 浅层信息与深层信息融合示意图
图4中,Conv1、Conv2和Conv3为卷积层, FM1s、FM2s和FM3s为卷积层输出的FMs。
卷积计算过程如图5所示。
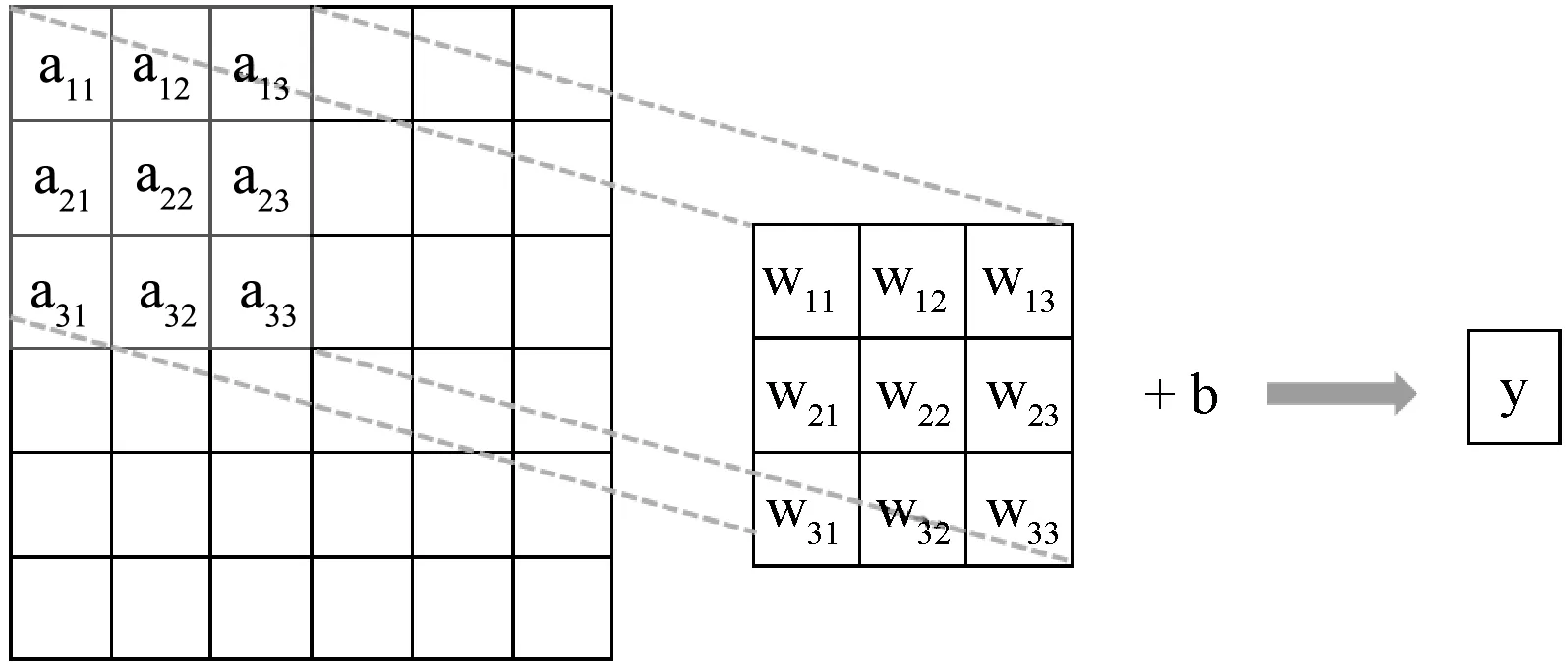
图5 卷积过程图
图5中,a11,a12,…,a33表示卷积层输入特征图的像素值,w11,w12,…,w33表示卷积核上的权值,b是偏移量,y表示卷积输出,如式(4)所示:
y=w11×a11+w12×a12+…+w33×a33+b
(4)
卷积过程后,将y输入到激活函数,获得卷积层的输出值,这里卷积层的激活函数均为LReLU。
融合浅层信息的方式,改善了传统CNN网络随层数加深,易丢失浅层精细信息的弊端,能够更加充分地利用网络提取的特征信息。
2.2.2 模型全局信息提取通路
全局信息是指感受野较大时获取的信息,即全局是个相对的概念。由于卷积核较大时,对应的感受野也较大,因此,在全局通路上,采用卷积核均为9×9像素的三个连续卷积层,由该通路获得的特征图谱被称作全局信息。全局特征提取通路的结构形式如图3中下面支路所示。
该通路提取的较大范围的信息,是对单通路CNN网络提取的信息的补充,使得网络模型提取的信息更加全面,更具有表达能力。
2.3 模型训练过程
为了更好地评估多通路CNN对MRI脑肿瘤图像的分割效果,在模型训练过程中,将训练集按4∶1分配为训练集数据和验证集数据。其中训练集数据用于训练多通路CNN 模型,验证集数据用于监督训练过程,检测模型训练程度,以及是否发生过拟合,当训练的模型精确度不再提高时,及时终止训练。
使用变化的学习率来更新权值,学习率函数如下:
(5)
式中:lr为每一轮训练的学习率,lr_base表示开始训练时的学习率,epoch为训练的轮次,epoches为设置的训练总轮数,lr_power为设置的指数参数。
为了减少过拟合,在局部通路上,为卷积层权重施加二范数正则项l2,其形式如公式所示:
(6)
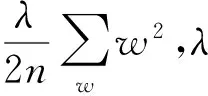
在全局通路上,在每个卷积层后使用Dropout正则化方式[25]。其中,Dropout正则化方法是指在训练模型时,随机保留全局通路的卷积层的一些节点的权重,暂时不更新。
模型最后一层采用softmax函数。softmax函数是多分类函数,输出属于各个类别的概率。这里,模型的输出有5个节点,分别表示输入图像块的中央像素点属于5个类别的概率,最终选择概率最大的类别为中央像素点的类别。
2.4 模型评价指标
本文采用Dice系数、Sensitivity系数和Specificity系数评估模型性能[3],具体计算公式如式(7)-式(9)所示。
Dice系数计算方式:
(7)
Sensitivity系数计算方式:
(8)
Specificity系数计算方式:
(9)
式中:P为预测结果,T为真实结果,P0为P的“非”,T0为T的“非”,比如,当P、T为脑肿瘤区域时,P0、T0则为非脑肿瘤区域。“∧”表示取交集,“+”表示取并集。
3 实验结果及分析
3.1 实验数据
实验数据是真实病人的脑部MRI图像,来自BRATS2013(The 2013 Brain Tumor Segmentation Challenge)。BRATS2013数据集包含20个HGG病人的数据,每个病人的数据均有四种模态,分别是FLAIR、T1、T1c和T2。如图6所示。
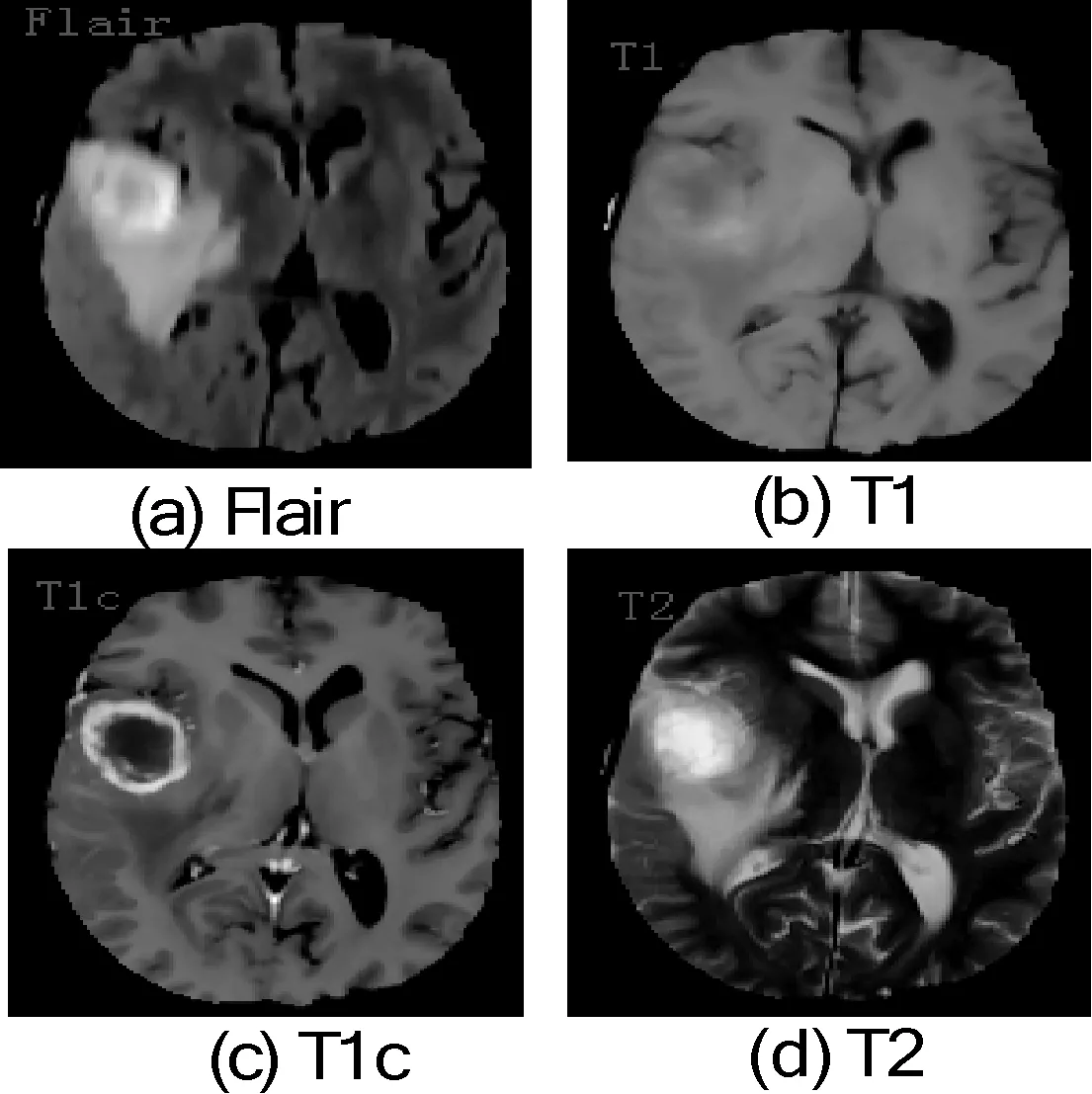
图6 四种模态的切片图像
3.2 实验结果分析
为了验证本文多通道CNN的有效性,精心设计了模型的训练过程。实验使用深度学习框架Keras搭建多通道CNN模型,在一个GPU(NVIDIA Titan X(Pascal))上训练模型。
设置最大训练迭代次数为150次,训练集与验证集的数据量之比为4∶1。验证集不参与训练,用于监督模型训练过程,当验证集的精确度不再提高时,训练过程会提前终止。在训练过程中,训练集误差与验证集误差随训练次数的变化如图7所示。
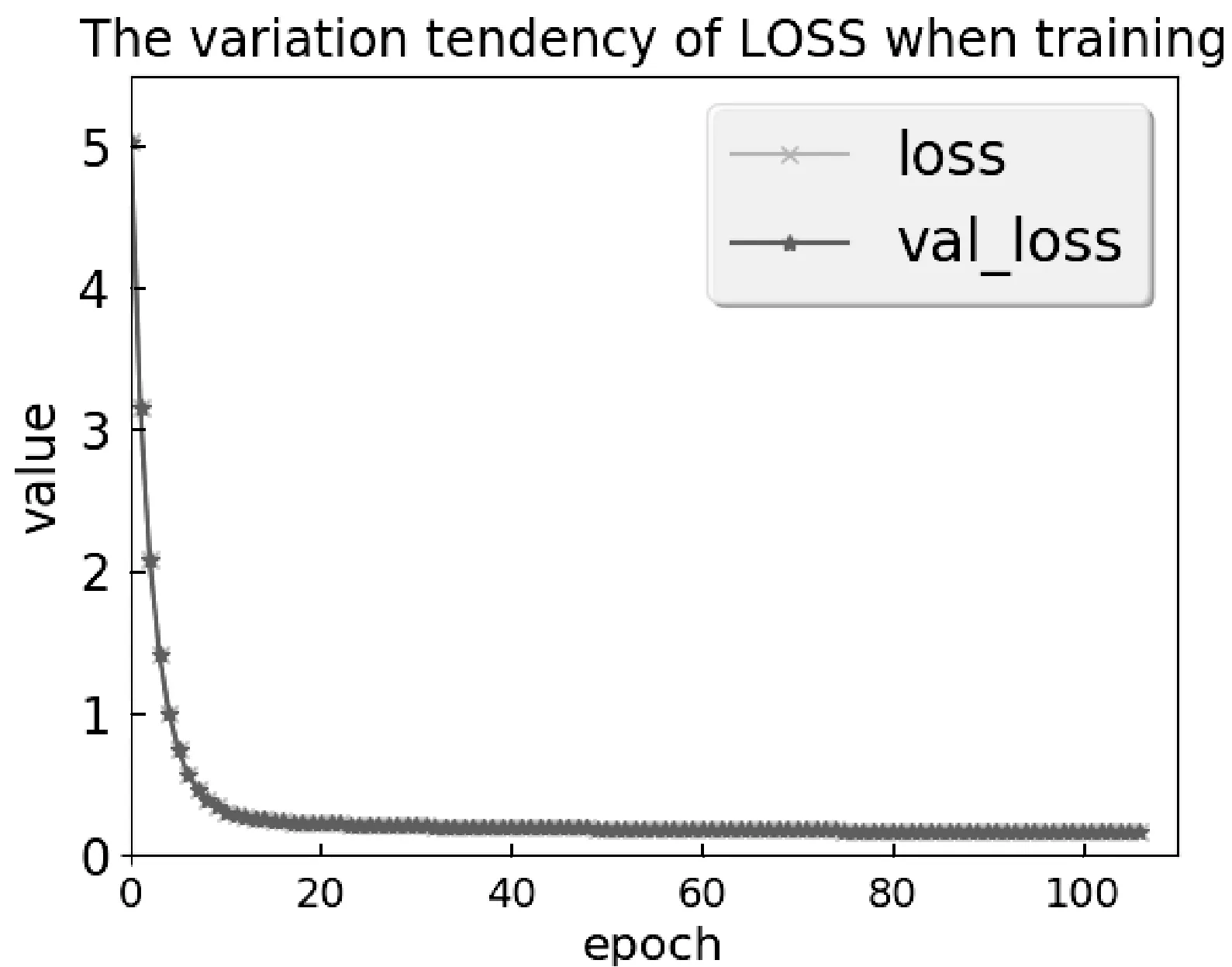
图7 训练过程中,误差变化过程
图7中,横轴表示训练次数,纵轴表示误差值。从图中可以看出,训练误差在训练初期很快下降,误差呈相对平稳的下降趋势,且训练过程中验证集误差与测试集误差相同。可以看出,模型稳定性强,且具有很好的泛化能力。
由于Buendia等[13]和Cordier等[14]给出了BRATS2013数据集的20个HGG病人数据的分割结果,而本文也是对2013年的数据集进行分析,因此采用这两种方法的结果进行比较。训练和评估模型方式如下: 首先,将20个数据分成A、B两组,每组有10个HGG数据。然后,在A组数据中所有相同序列的四种模态的切片上按比例随机取图像块,其形式为(503 784,33,33,4),表示用于训练的数据包含50多万个大小为33×33×4像素的图像块。用所得图像块训练模型,使用训练好的模型对B组数据做分割,获得B组10个HGG数据的分割结果。接着,使用同样的操作在B组数据上随机取图像块,获得40多万个大小为33×33×4像素的图像块用所得图像块训练模型,使用训练好的模型对A组数据做分割,获得A组10个HGG数据的分割结果。分割结果包括肿瘤区域(体素标签为1、2、3、4), 肿瘤核区域(体素标签为1、3、4)以及肿瘤增强区域(体素标签为4)。 脑肿瘤分割的可视化效果如图8所示。图8(a)、(c)、(e)中亮色区域以及其周边较深区域表示使用CNN模型分割某位患者3张脑肿瘤切片的结果示例;(b)、(d)、(f)中亮色区域及其周边较深区域表示专家分割相应切片的脑肿瘤结果示例。其中亮色区域包括坏死部分、非增强区域 、增强区域,水肿部分为周边较深区域。
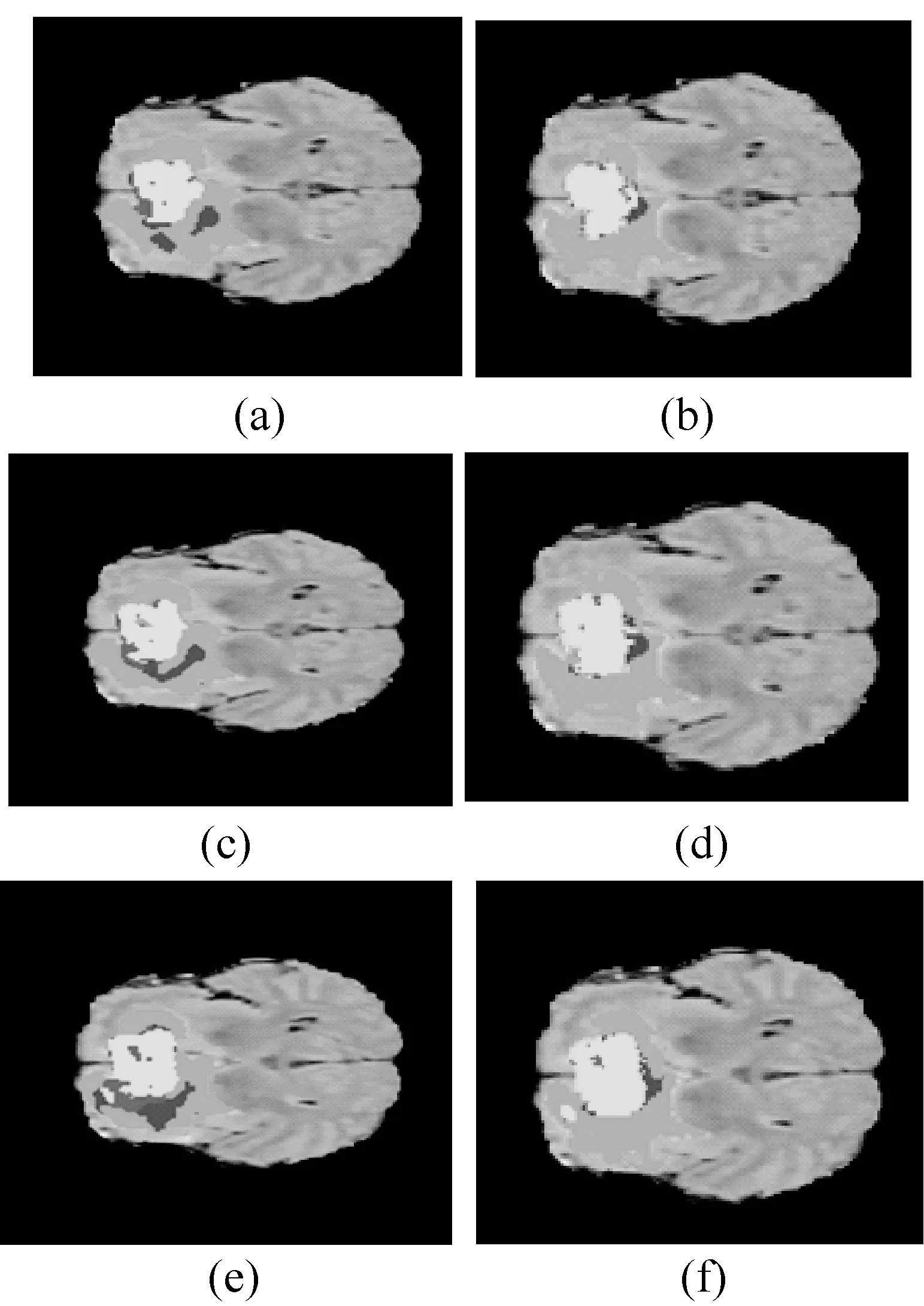
图8 脑肿瘤分割的可视化效果图
从图8中可以看出,多通路CNN模型能够将脑肿瘤的区域准确分割出来,但对脑肿瘤各区域的划分能力还有待提高。不过,其分割结果已经达到了可以接受的程度,是非常有前景的方法。
使用多通路CNN模型分割脑肿瘤的结果与其他方法的结果相对比,其对比结果如表1所示。其中,区域1表示脑肿瘤区域,区域2表示肿瘤核区域,区域3表示肿瘤增强区域。
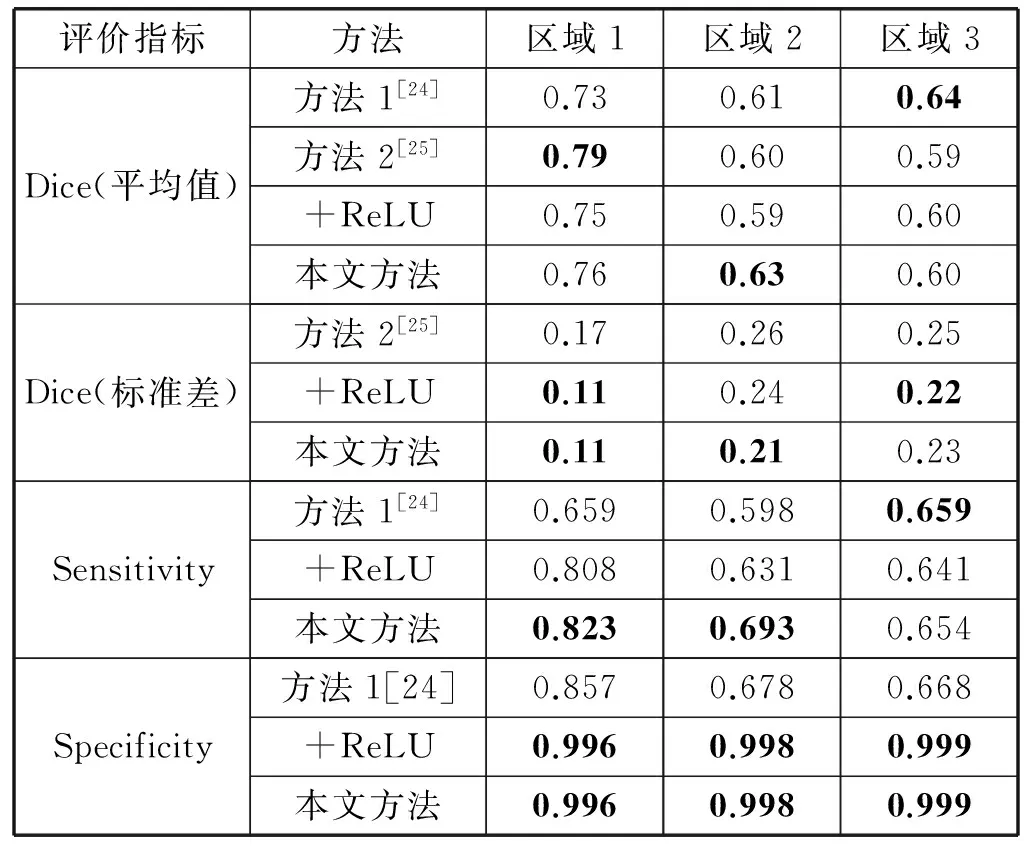
表1 不同方法的分割结果
从表1可以得到如下结论:
采用Dice系数评估方法性能时,三种方法各有优势。方法1对脑肿瘤的增强区域比较敏感,方法2能较好的分割出整个脑肿瘤区域,而本文方法更擅长分割出肿瘤核部分,且本文方法的分割性能更加稳定。
方法1对脑肿瘤增强区域分割效果优于本文方法,原因在于方法1在做训练时采用的Flair、T1c、T1、T2数据量的比例为7∶7∶1∶1,本文方法中四种模态的图像数据比例为1∶1∶1∶1。前人实验结果表明,Flair、T1c图像对分割脑肿瘤的增强区域更有效。
方法2对整个脑肿瘤分割效果优于本文方法,原因在于方法2在预处理部分首先使用边界框限定了图像中的脑肿瘤区域。然后只针对图像边界框内区域取图像块,而本文方法是针对整个图像区域取图像块,方法2的针对性更强。
采用Sensitivity系数评估方法性能时,本文方法的灵敏度只在肿瘤增强区域的分割方面薄弱一些,对另外两个区域分割的灵敏度均超过方法1,这说明本文方法能更好地预测出脑肿瘤各区域。
采用Specificity系数评估方法性能时,本文方法在脑肿瘤各区域分割方面的特异性远远超过方法1,这说明本文方法对非脑肿瘤区域非常敏感。
方法1需要去噪、去除非均匀性、强度直方图校准等前期处理以及寻找和提取连通区域进行膨胀和腐蚀等后期处理。该方法过程复杂,且针对小批量的数据,适用性不强。方法2是基于图像块的分割方法,需要校准强度、定位脑肿瘤、选择图像块、检索相似图像块等相关操作,过程比较繁琐。
+ReLU表示将本文方法的卷积层函数由LReLU换成ReLU的实验结果,可以看出使用LReLU激活函数时,模型的分割效果更好。
从表1中可以看出,本文方法在既没有对图像数据做去噪、强度校准等前期处理,也没有做任何后期处理的情况下,对脑肿瘤区域的分割效果仍然优于方法1、方法2,且本文方法操作简单,能够较准确地实现全自动分割脑肿瘤的任务。
4 结 语
目前使用深度学习方法分割脑部神经胶质瘤非常具有研究前景。传统脑肿瘤分割方法需要专家手动标注,而不同专家或者是同一专家在不同时刻的标注结果都有可能不同。这导致标注可能偏差过大,所以通常会选择多个专家进行标注,并取综合后的标注结果。此外,MRI数据量大,使用传统方法耗时耗力,已经无法满足目前大数据的需求。本文研究的多通道CNN全自动脑肿瘤分割方法解决了上述这些问题,并且不需要对数据做太多前期处理,而且提取特征与分类一步完成,操作简单且快速。实验表明,与传统方法相比,使用多通路CNN进行脑部神经胶质瘤分割,在分割精度、分割速度等方面获得了优秀的性能。
为了进一步提高本文方法的性能,并使用更多的数据集验证本文方法的有效性,未来的实验将完成一些其他工作。例如数据的前期处理等,其中最重要的是增加实验数据,为了与其他方法做比较,本文实验仅采用了BRATS2013的数据,未来的工作将增加BRATS2015的数据。由于深度学习数据驱动的特性,增加数据无疑能提高该类方法的性能,而不仅限于本文方法。
[1] Menze B H,Jakab A,Bauer S,et al.The multimodal brain tumor image segmentation benchmark (BRATS)[J].IEEE transactions on medical imaging,2015,34(10):1993-2024.
[3] Havaei M,Davy A,Warde-Farley D,et al.Brain tumor segmentation with deep neural networks[J].Medical image analysis,2017,35:18-31.
[4] Rewari R.Automatic Tumor Segmentation from MRI scans[Z].cs231n.stanford.edu.
[5] Müller S,Weickert J,Graf N.Automatic brain tumor segmentation with a fast Mumford-Shah algorithm[C]//SPIE Medical Imaging.International Society for Optics and Photonics,2016:97842S-97842S-7.
[6] Bauer S,Nolte L P,Reyes M.Fully automatic segmentation of brain tumor images using support vector machine classification in combination with hierarchical conditional random field regularization[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention.Springer Berlin Heidelberg,2011:354-361.
[7] Hasan A M,Meziane F,Aspin R,et al.Segmentation of brain tumors in MRI images using three-dimensional active contour without edge[J].Symmetry,2016,8(11):132.
[8] Levine M D,Shaheen S I.A Modular Computer Vision System for Picture Segmentation and Interpretation[J].IEEE Transactions on Pattern Analysis & Machine Intelligence,1981,PAMI-3(5):540-556.
[9] Balasubramanian C,Saravanan S,Srinivasagan K G,et al.Automatic Segmentation of Brain Tumor from MR Image Using Region Growing Technique[J].Life Science Journal,2013,10(2).
[10] Xiao Z,Huang R,Ding Y,et al.A deep learning-based segmentation method for brain tumor in MR images[C]//IEEE,International Conference on Computational Advances in Bio and Medical Sciences.IEEE,2016:1-6.
[11] Rana R,Bhdauria H S,Singh A.Brain tumour extraction from MRI images using bounding-box with level set method[C]//Contemporary Computing (IC3),2013 Sixth International Conference on.IEEE,2013:319-324.
[12] Abdel-Maksoud E,Elmogy M,Al-Awadi R.Brain tumor segmentation based on a hybrid clustering technique[J].Egyptian Informatics Journal,2015,16(1):71-81.
[13] Buendia P,Reyes M,Ryan M,et al.A Grouping Artificial Immune Network for Segmentation of Tumor Images[C]//Proceedings of NCI-MICCAI BRATS,2013:1-5.
[14] Cordier N,Menze B,Delingette H,et al.Patch-based segmentation of brain tissues[C]//MICCAI Challenge on Multimodal Brain Tumor Segmentation.IEEE,2013:6-17.
[15] Bengio Y,Courville A,Vincent P.Representation learning:A review and new perspectives[J].IEEE transactions on pattern analysis and machine intelligence,2013,35(8):1798-1828.
[16] Kamnitsas K,Chen L,Ledig C,et al.Multi-scale 3D convolutional neural networks for lesion segmentation in brain MRI[J].Ischemic Stroke Lesion Segmentation,2015,13.
[17] Pereira S,Pinto A,Alves V,et al.Brain tumor segmentation using convolutional neural networks in MRI images[J].IEEE transactions on medical imaging,2016,35(5):1240-1251.
[18] Elamri C,de Planque T.A New Algorithm for Fully Automatic Brain Tumor Segmentation with 3-D Convolutional Neural Networks[Z] .cs231n.stanford.edu.
[19] Urban G,Bendszus M,Hamprecht F A,et al.Multi-modal brain tumor segmentation using deep convolutional neural networks[C]//MICCAI BraTS (Brain Tumor Segmentation) Challenge.Proceedings,winning contribution.2014.
[20] Dvorak P,Menze B.Structured prediction with convolutional neural networks for multimodal brain tumor segmentation [C]//Proceeding of the Multimodal Brain Tumor Image Segmentation Challenge,2015:13-24.
[21] Zhao L,Jia K.Multiscale cnns for brain tumor segmentation and diagnosis[J].Computational and mathematical methods in medicine,2016,2016.
[22] Matsugu M,Mori K,Mitari Y,et al.Subject independent facial expression recognition with robust face detection using a convolutional neural network[J].Neural Networks,2003,16(5):555-559.
[24] Long J,Shelhamer E,Darrell T.Fully convolutional networks for semantic segmentation[C]//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition.2015:3431-3440.
[25] Jodoin A C,Larochelle H,Pal C,et al.Brain Tumor Segmentation with Deep Neural Networks[EB/OL].http://www.professeurs.polymtl.ca/christopher.pal/miccai2014/brats2014_lisa_cnn_paper.pdf.
