南江黄羊ARHGAP11A基因SNPs筛选及其与生长性状的关联分析
2018-05-02王嘉欣张德鹏谭永乾宋天增张红平
王嘉欣,张德鹏,谭永乾,冯 静,宋天增,陈 愉,张红平,李 利*
(1.四川农业大学动物科技学院,成都 611130;2.四川农业大学动物医学院,成都 611130;3.西藏自治区农牧科学院,拉萨 850000;4.四川南江黄羊原种场,四川南江 635600)
ARHGAP11A(Rho GTPase-activating protein 11A)基因,也被称为 KIAA0013 或 MGC70740,属于RhoGTPs(Rho GTPase-activating proteins)酶激活蛋白的一员,与细胞内信号分子Rho GTPases的调控息息相关[1]。RhoGAPs结合Rho GTPases在包括能动性、收缩性、生长、分化等细胞进程中至关重要[2-3]。
ARHGAP11A通过Rho GTPase和GPCR(G protein coupled receptor)信号通路参与激活GTPase活动,从而间接控制细胞的生物学功能,包括细胞的形态、扩散、迁移、内吞以及细胞周期进程[4]。在哺乳动物癌细胞中发现,ARHGAP11A诱导Rac1的活性从而抑制RhoA的相对增强是癌细胞迁移和侵袭的关键[5]。在有丝分裂中ARHGAP11A基因沉默则会导致有丝分裂细胞膜起泡,RhoA的激活速率增强[6]。因此,ARHGAP11A表达水平影响细胞的增殖和凋亡[1],但目前关于ARHGAP11A基因在家畜上的研究未见报道。
南江黄羊是我国培育的第一个肉用山羊品种,具有体格高大,生长发育快,四季发情,繁殖率高,抗病力强,采食性好,适宜粗放饲养等特点[7]。同时,南江黄羊骨骼肌卫星细胞(SMSCs)具有突出的增殖和融合率能力[8]。因此,本试验采用PCR产物直接测序法,检测南江黄羊ARHGAP11A基因外显子及非编码区域的单核苷酸多态(single nucleotide polymorphism,SNP)位点,并将这些位点与其生长性状进行关联分析,以期筛选出对南江黄羊生长性状有显著影响的多态位点,为南江黄羊的分子育种提供参考依据。
1 材料和方法
1.1 试验动物和血样采集
试验中南江黄羊母羊群体(n=243)均来自南江黄羊原种场。所有羊只在同一环境条件下进行相同的饲养管理。羊群饲养方式为放牧加适当补饲,饲料营养水平满足羊生长需求。
每一只试验羊颈静脉采集全血1.5mL,加入肝素钠抗凝,-20℃冰箱中保存以备基因组DNA提取。
1.2 南江黄羊生产性能测定
按照常规方法分别测定南江黄羊的初生重(birth weight,BWT)以及 2月龄、6月龄、12月龄及24月龄共 4个生长阶段的体重(weight,WT)、体长(body length,BL)、体高(body height,BH)和胸围(chest circumference,CC)。
1.3 引物设计
为了扩增山羊ARHGAP11A基因的12个外显子以及部分非编码区,根据NCBI数据库中公布的山羊基因组序列以及预测的ARHGAP11A mRNA(XM_005685462.3),采用Primer Premier 5.0软件设计了14对引物(见表1)并用Oligo 6.0和Blast验证其特异性。引物由成都擎科生物公司合成。
1.4 基因组DNA的提取及DNA的质量检测
采用常规血液基因组提取试剂盒(天根生化科技北京有限公司)提取山羊基因组DNA后,利用1.5%琼脂糖凝胶电泳并在凝胶图像分析仪BIO-RAD ChemDOC XRS中紫外成像,用Quality One 4.6.2软件分析图像以判定DNA完整性。DNA的纯度与浓度利用核酸蛋白检测仪(BIO-RAD,USA)测定。对符合要求的样品-20℃保存备用。
1.5 目标片段的PCR扩增和测序
以获得的山羊基因组DNA为模板,根据PCR扩增总体系(2×Master Mix Taq 酶 15.0 μL,DNA 模板 1.0 μL,上、下游引物(10 μmol/L)各 1.0 μL,灭菌dd H2O 12.0 μL)和PCR程序(95℃预变性 5 min,然后38个循环(95℃变性30s,各引物特异性Tm 30 s,72℃延伸30 s),72℃延伸10 min)进行扩增后,1.5%琼脂糖凝胶电泳检测PCR产物,选取目的条带亮且无杂带的PCR产物送至深圳华大基因科技服务有限公司进行双向测序。
1.6 数据分析
将测序结果进行核对、组装以确定ARHGAP11A基因的 CDS区和 UTR;利用 RegRNA 2.0(http://regrna2.mbc.nctu.edu.tw/)分析序列上的调控元件。用Sequencher 4.7软件比对各SNPs,在线进行群体中SNPs位点的基因型频率和基因频率以及Hardy-Weinberg 检验(http://www.oege.org/software/hardyweinberg.html)。通过PHASE 2.1.1进行单倍型构建,Haploview 4.2进行连锁不平衡分析。

表1 PCR扩增山羊ARHGAP11A基因序列的引物信息Table 1 Primers'information in amplifying goat ARHGAP11A
南江黄羊体重体尺性状与SNPs关联分析利用SAS(19.0)软件中的一般线性模型进行。分析模型为:Yijkl=μ+Gk+eijkl,其中 Yijkl为个体性状的观测值,μ为群体均值,Gk为基因型效应,eijkl为随机误差。结果以平均值±标准差表示。P<0.05为差异显著,P<0.01为差异极显著。
2 结果与分析
2.1 山羊ARHGAP11A基因序列及其与其他物种的比较分析
基于NCBI中预测的山羊ARHGAP11A mRNA序列(XM_005685462.3),利用14个测序片段组装获得山羊ARHGAP11A基因序列4235bp,其中CDS序列全长3 069 bp,其余为5′UTR和3′UTR的部分序列(分别 486 bp和680 bp)。将山羊ARHGAP11A基因核苷酸序列与小鼠(NM_181416.3)、绵羊(XM_004010425.3)、牛(XM_870134.6)、人(NM_014783.5)和豚鼠(XM_003475662.2)的进行分析,发现物种之间相似性达到78%以上。其中,山羊与绵羊的相似性最高(99%),其次是与牛(97%)(见图1)。利用预测出的ARHGAP11A氨基酸序列进行对比也发现物种间ARHGAP11A蛋白高度相似,尤其是绵羊、山羊和牛这3种反刍动物。这些说明ARHGAP11A基因在物种之间高度保守,暗示其在个体发育中的可能具有重要作用(见图2)。
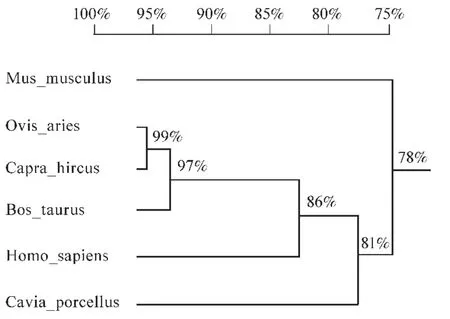
图1 物种间ARHGAP11A基因外显子序列相似性分析Figure 1 Sequence similarity of ARHGAP11A exons among species
2.2 南江黄羊群体中ARHGAP11A基因的SNP位点
在南江黄羊群体中共发现ARHGAP11A基因16个SNPs位点(见图3a)。其中1个突变(g.-340A>G,以翻译起始为+1)位于 5′UTR,在 Exon 1中也只有 1个(g.39G>C),其余 14个分别位于 Exon 12(7个 SNPs)和 3′UTR(7 个 SNPs)(见图 3b)。ARHGAP 11A基因编码区的8个 SNPs中有 3个SNPs(g.39G>C,g.2565C>T,g.3060T>A)是同义突变,其余 5个 SNPs(g.2852T>C,g.2856G>A,g.2907G>A,g.2912 G>A,g.2950G>A)为错义突变。对应的氨基酸改变如下:缬氨酸→丙氨酸(g.2852T>C)、甲硫氨酸→异亮氨酸(3371C>T)、甲硫氨酸→异亮氨酸(g.2907G>A)、精氨酸→赖氨酸(g.2912G>A)、缬氨酸→异亮氨酸(g.2950G>A)。

图2 6个物种间预测的ARHGAP11A氨基酸序列对比Figure 2 Comparison of predicted amino acid sequence of ARHGAP11A among 6 species
利用在线软件 RegRNA 2.0(http://regrna2.mbc.nctu.edu.tw/)分析发现,ARHGAP11A具有包括myogenin(cagctg)以及 MEF2A(tatttttaaa)、MEF2C(tattttt)和MEF2D(aaaatag)在内的多个转录因子调控基序(Transcriptional regulatory motif,TRM)。有趣的是,在南江黄羊群体中发现的16个SNPs中7个位于转录调控元件内(见表2),其中既涉及内含子剪切增强子(intron splicing enhancer,ISE),如 g.-340A>G和g.3155T>C;也有外显子剪切增强子(exon splicing enhancer,ESE),如 g.3680 T>C,而且 g.3680 T>C 位点还涉及多聚腺苷化(Polyadenylation sites,PAS)。另外还有 4个位点(g.39G>C、g.3060T>A、g.3483A>G和g.3525A>G)均位于调控基序(见表2)。值得注意的是,南江黄羊群体中发现的7个与转录调控有关的SNPs中就有 3个(g.3483A>G、g.3525A>G、g.3680T>C)集中在3′UTR端约200 bp的片段上。因此,后续重点分析这范围内的4个SNPs以及它们与南江黄羊生长发育的关系。
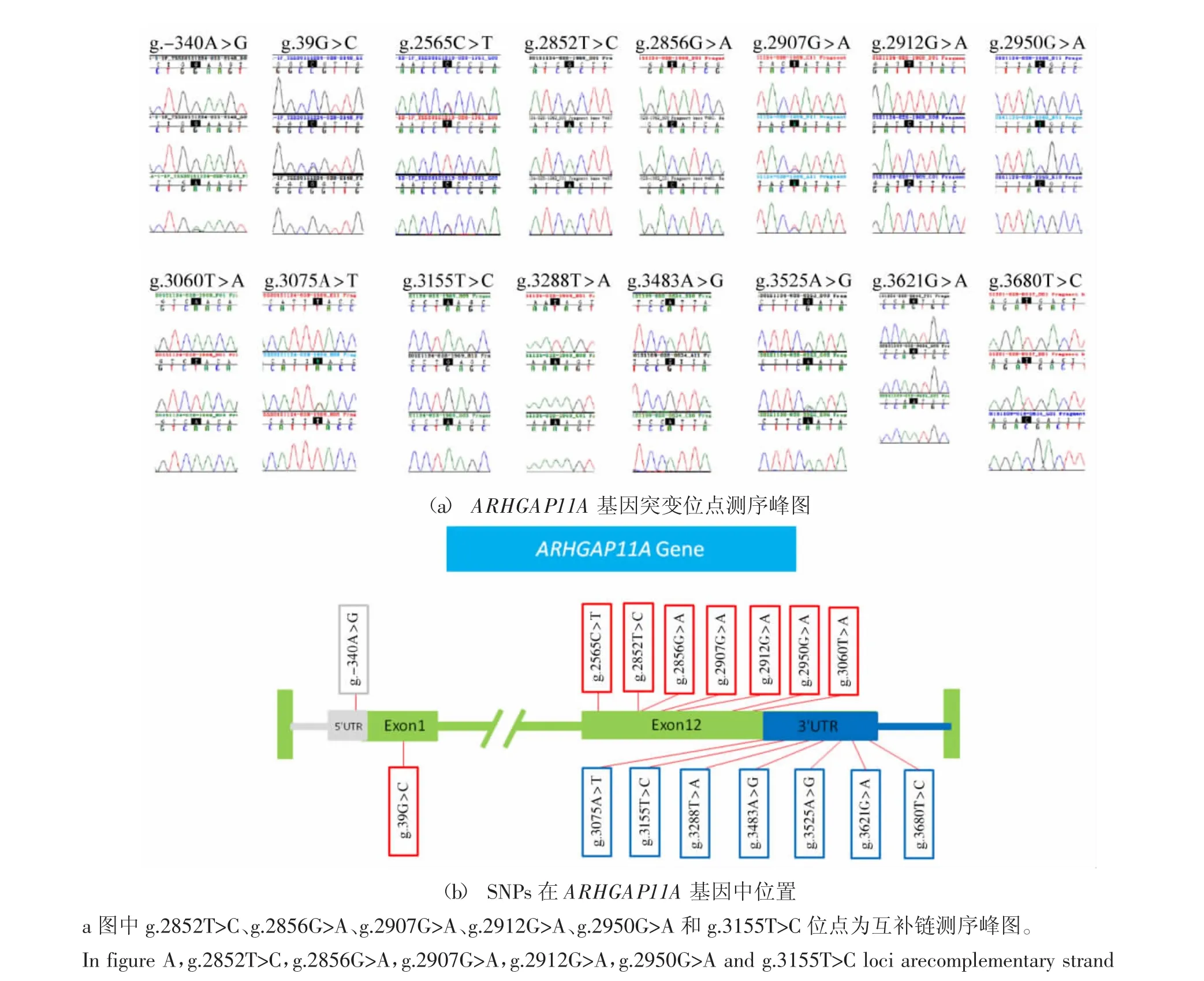
图3 南江黄羊羊ARHGAP11A基因突变位点测序峰图以及SNPs在ARHGAP11A基因中位置Figure 3 Mutation sites of ARHGAP11A gene in Nanjiang Yellow goats and their schematic locations
南江黄羊群体ARHGAP11A基因中g.3483A>G、g.3525A>G、g.3621G>A 和 g.3680T>C 这 4 个位点优势等位基因的频率范围为0.525(g.3525A>G)到0.996(g.3680T>C),各位点的观测杂合度和期望杂合度分别介于0.005-0.451和0.499-0.003之间(见表3)。除了g.3525A>G以外,其余3个位点(g.3483A>G,g.3621G>A 和 g.3680T>C) 处于 Hardy-Weinberg不平衡状态(P<0.01),暗示群体中这 3个位点可能受到选择的影响。利用这4个位点构建获得4种单倍型,其中AAGT与AGGT占绝对优势,频率分别为0.514和0.467(见图4a),并且这4个SNPs位点存在一定程度的连锁不平衡,尤其是g.3483A>G 与g.3680T>C之间(见图4b)。

表2 南江黄羊群体中ARHGAP11A基因16个SNP的基因型及相关调控元件Table 2 Genotypes of 16 mutations and related regulatory motifs in ARHGAP11A gene of Nanjiang Yellow goats
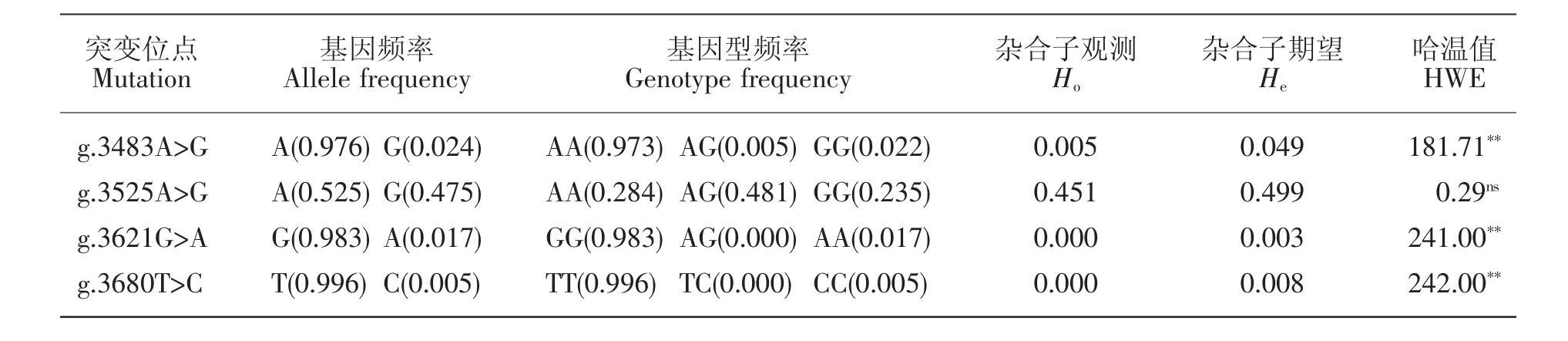
表3 南江黄羊群体中ARHGAP11A基因4个SNP的基因分型检测结果Table 3 Genotypes of 4 SNPs in ARHGAP11A gene of Nanjiang Yellow goats
2.3 南江黄羊群体ARHGAP11A基因与生长发育性状的关联分析
由于群体中只有1个个体在g.3680 T>C位点突变(CC型,其余为野生型TT),因此本研究进一步分析了3个SNPs(g.3483A>G、g.3525A>G和g.3621G>A)与南江黄羊不同生长阶段体重、体高、体长、胸围等指标之间关系(见表4)。结果发现,g.3483A>G和g.3621 G>A上纯合突变个体(分别为GG型和AA型)具有2周岁前体重高于野生型(分别为AA型和GG型)个体的趋势(P>0.05)。在g.3525 A>G位点,AG型个体不同阶段的体重、体尺指标均高于GG型和AA型个体,且BWT、WT-12和CC-12与突变型GG个体差异显著(P<0.05),但与AA个体之间差异不显著(P>0.05)。进一步分析发现 g.3483A>G、g.3525A>G、g.3621G>A 和 g.3680T>C 位点构建的 3种单倍型中(频率小于1%的单倍型GGAC未分析),具有AAGT单倍型个体的体重体尺指标值最高,GGAT型个体的最低,尤其是6月龄和12月龄平均体重两者相差多达2.5 kg以上,但两者差异不显著(P>0.05)(见表4)。总的来看,无论从单SNP位点还是联合的单倍型,所分析的ARHGAP11A基因突变均不同程度影响南江黄羊的体重体尺指标。
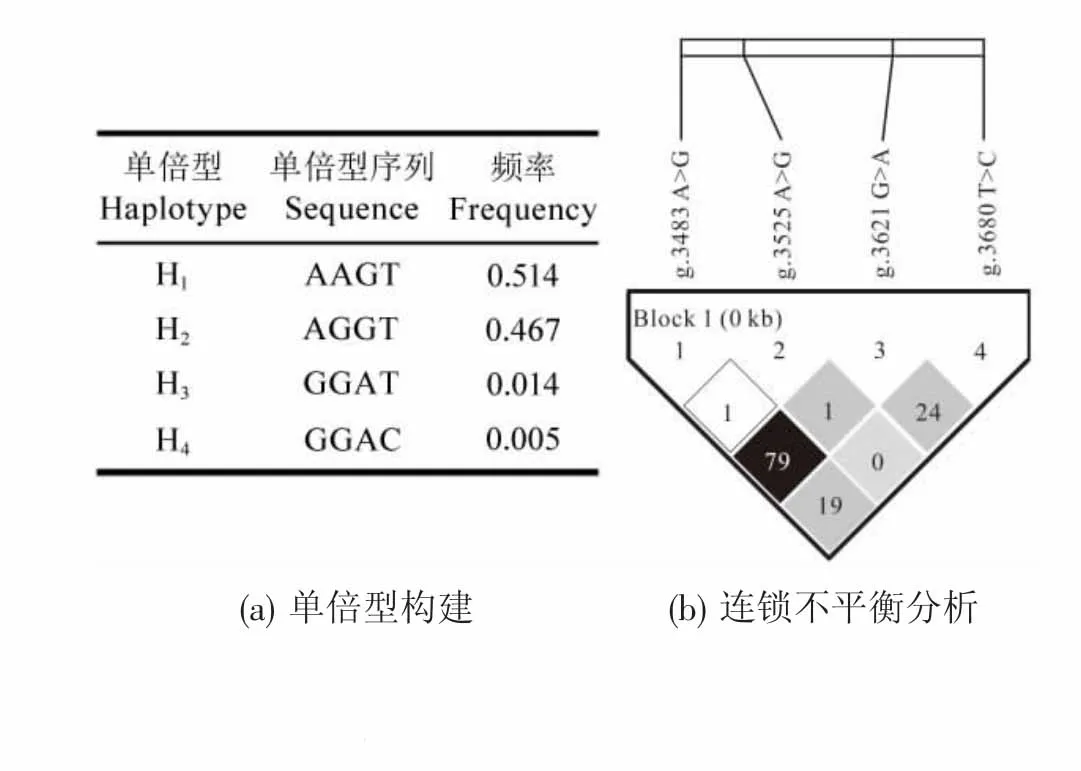
图4 南江黄羊群体中ARHGAP11A 基因4个SNP 的单倍型构建和连锁不平衡分析Figure 4 Haplotype construct and linkage disequilibrium analysis of 4 SNPs in ARHGAP11A gene in Nanjiang Yellow goats
3 讨论
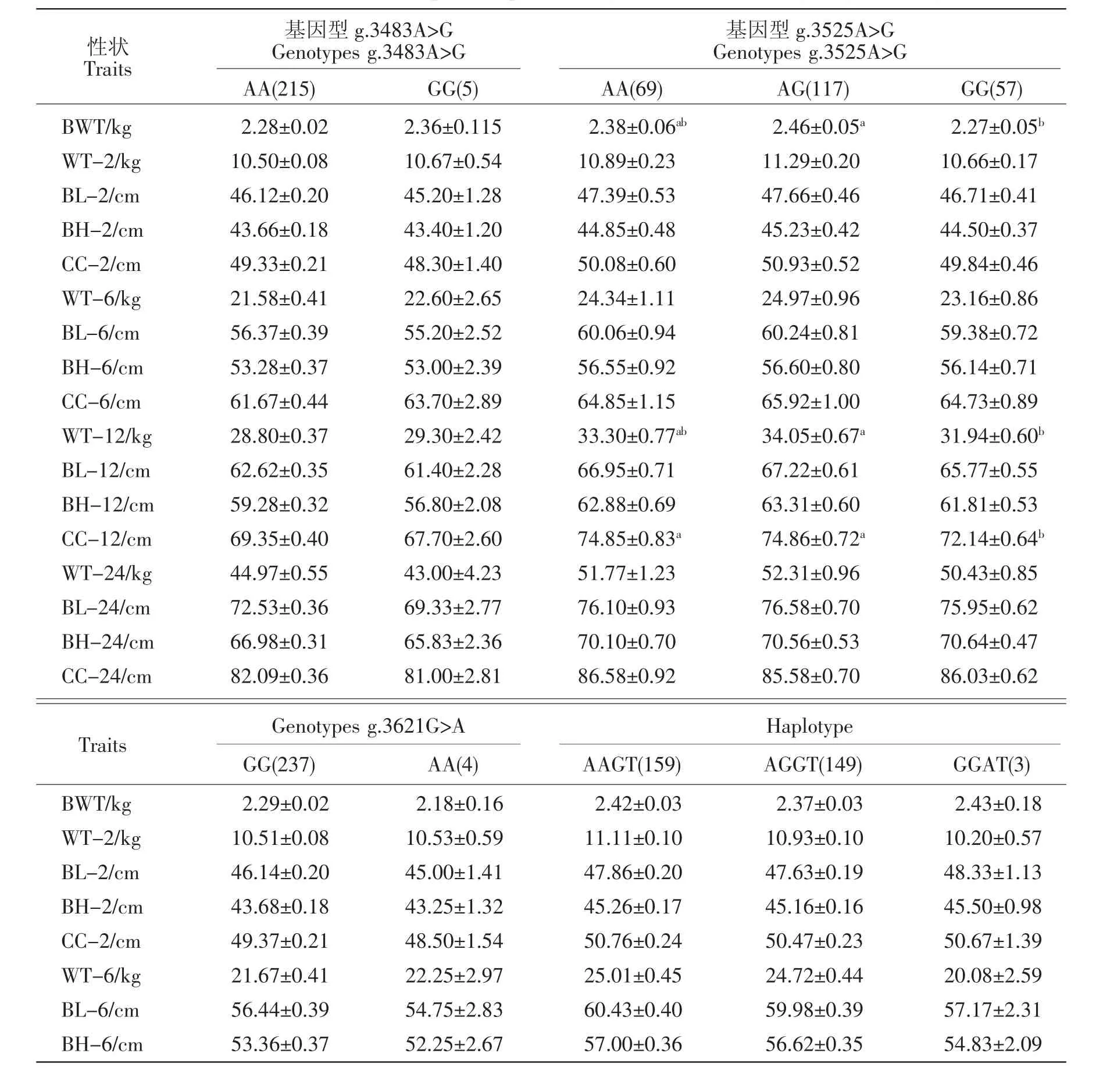
表4 南江黄羊ARHGAP11A基因多态性位点对其生长性状的影响Table 4 Effect of ARHGAP11A polymorphisms on growth traits in Nangjiang Yellow goats
本实验测序组装得到ARHGAP11A基因包含外显子和部分UTR的片段(4 235 bp),其中CDS区3 069 bp。对CDS以及预测的氨基酸序列比对均发现小鼠、绵羊、山羊、牛、人和豚鼠间ARHGAP11A基因高度保守。另外,ARHGAP11A基因还可能受到转录因子 myogenin以及MEF2家族中MEF2A、MEF2C和MEF2D的调控。作为肌肉特异性的转录因子,myogenin主要参与骨骼肌的发育和修复[9],而肌肉组织约占哺乳动物机体重量的40%~50%。MEF2家族基因则广泛参与动物组织器官的生长发育[10]。同时,ARHGAP11A通过Rho GTPases和GPCR信号通路参与调控众多信号转导途径,从而间接控制细胞的生物学功能如细胞周期进程[4]。这些结果暗示,在进化上高度保守的ARHGAP11A基因与动物生长发育的密切相关。

续表
基因突变与畜禽生长发育性状密切关联,从而常作为畜禽育种的分子标记以大幅度提高育种效率[11-13]。最为著名的是myostatin基因(也称为GDF8基因),其SNP位点显著影响夏洛莱肉羊的肌肉深度[14]以及新西兰罗姆尼羊的胴体重[15]。在南江黄羊ARHGAP11A基因4 235 bp上发现16个SNPs,其中14个集中在最后一个外显子(Exon 12;7个)和3′UTR(7个),并且位于转录调控元件内的7个SNPs中有 4个(g.3155T>C、g.3483A>G、g.3525A>G和 g.3680T>C)在 3′UTR。大量研究表明,3′UTR 与mRNA在细胞核外的转运、翻译效率、稳定性及亚细胞定位紧密相关[16]。该区域的变异会对目标基因功能的发挥产生一定程度的影响,如肉牛ANGPTL4基因的3′UTR SNPs位点与肉用性能显著相关[17]。本研究在南江黄羊群体中分析了3个3′UTR内的SNPs(g.3483A>G、g.3525A>G 和 g.3621G>A),发现它们均与南江黄羊的体重体尺性状密切相关。其中,在g.3483A>G和g.3621 G>A位点纯合突变个体体重(2、6和12月龄)高于野生型。在g.3525 A>G位点,AG型个体不同阶段的体重体尺指标均高于GG型和AA型个体。
值得注意的是,在南江黄羊群体ARHGAP11A基因上发现7个SNPs位于转录调控元件基序内,如g.3483A>G与g.3525A>G分别位于转录激活因子 IPF1(pancreatic and duodenal homeobox 1,也称为 PDX1)和 OC-2(one cut homeobox 2)的基序内。IPF1主要涉及能量代谢和肝脏发育,并且与FOXO1(forkhead box O1)具有互作[18],而 g.3060T>A 位于FOXO1基序内,OC-2则结合在RNA聚合酶II核心增强子区域参与基因激活[19]。另外,ARHGAP11A基因在CDS区中发现的5个错义突变SNPs位点可直接改变氨基酸序列。因此,在ARHGAP11A基因突变位点上,这些潜在转录因子的调控作用以及其CDS区内的SNP位点与南江黄羊生长发育的关系需进一步研究。
4 结论
实验测得山羊ARHGAP11A基因全长CDS(3 069 bp)序列和部分UTR。在南江黄羊ARHGAP11A基因外显子和UTR内检测到16个SNPs,主要分布于最后一个外显子(7个)和3′UTR区(7个)。3个3′UTR 内的突变 (g.3483A>G、g.3525A>G 和 g.3621G>A)与 3种单倍型(AAGT、AGGT 和 GGAT)均与南江黄羊的体重、体尺性状密切相关。初步研究表明ARHGAP11A基因可作为南江黄羊生长发育性状的候选基因。
参考文献:
[1]JIE X,ZHOU X,WANG J,et al.RhoGAPs attenuate cell proliferation by direct interaction with p53 tetramerization domain[J].Cell Reports,2013,3(5):1526.
[2]AMIN E,JAISWAL M,DEREWENDA U,et al.Deciphering the molecular and functional basis of RhoGAP family proteins:A systematic approach towards selective inactivation of Rho family proteins[J].Journal of Biological Chemistry,2016,291(39):M116-M736967.
[3]JACOBS T,HALL C.Rho GAPs—Regulators of Rho GTPases and More[M].Dordrecht:Springer Netherlands,2005:93-112.
[4]ROMANOV V S,POSPELOV V A,POSPELOVA T V.Cyclindependent kinase inhibitor p21(Waf1):contemporary view on its role in senescence and oncogenesis[J].Biochemistry Biokhimiia,2012,77(6):575-584.
[5]KAGAWA Y,MATSUMOTO S,KAMIOKA Y,et al.Cell cycledependent Rho GTPase activity dynamically regulates cancer cell motility and invasion in vivo[J].Plos One,2013,8(12):e83629.
[6]ZUO Y,OH W,FROST J A.Controlling the switches:Rho GTPase regulation during animal cell mitosis[J].Cellular Signalling,2014,26(12):2998-3006.
[7]张红平,王维春,熊朝瑞,等.南江黄羊的种质特性[J].中国草食动物科学,2004(s1):113-114.
[8]陈利.南江黄羊和波尔山羊骨骼肌卫星细胞增殖分化能力的比较[D].雅安:四川农业大学,2014.
[9]HASTY P,BRADLEY A,MORRIS J H,et al.Muscle deficiency and neonatal death in mice with a targeted mutation in the myogenin gene[J].Nature,1993,364(6437):501.
[10]Potthoff M J,Olson E N.MEF2:a central regu-lator of diverse developmental programs[J].Development,2007,134(23):4131-4140.
[11]CHANG M T,CHENG Y S,HUANG M C.A novel non-synonymous SNP of the COLX gene and its association with duck reproductive traits[J].Molecular&Cellular Probes,2012,26(5):204-207.
[12]FAN B,DU Z Q,DANIELLEM G,et al.Development and application of high-density SNP arrays in genomic studies of domestic animals[J].Asian Australasian Journal of Animal Sciences,2010,23(7):833-847.
[13]VIGNAL A,MILAN D,SANCRISTOBAL M,et al.A review on SNP and other types of molecular markers and their use in animal genetics[J].Genetics Selection Evolution,2002,34(3):1-31.
[14]HADJIPAVLOU G,MATIKA O,CLOP A,et al.Two single nucleotide polymorphisms in the myostatin(GDF8)gene have significant association with muscle depth of commercial Charollais sheep[J].Animal Genetics,2008,39(4):346-353.
[15]HICKFORD J G H,FORREST R H,ZHOU H,et al.Polymorphisms in the ovine myostatin gene(MSTN)and their association with growth and carcass traits in New Zealand Romney sheep[J].Animal Genetics,2010,41(1):64-72.
[16]MIGNONE F,GISSI C,LIUNI S,et al.Untranslated regions of mRNAs[J].Genome Biology,2002,3(3):1-4.
[17]MA Y,LI R R,HOU F,et al.Comparative Mapping and 3'UTR SNP Detection of ANGPTL4 Gene in Beef Cattle[J].Journal of Animal&Veterinary Advances,2011,10(13):1649-1655.
[18]ELBEIN S C,WANG X,KARIM M A,et al.Role of a proline insertion in the insulin promoter factor 1(IPF1)gene in African Americans with type 2 diabetes[J].Diabetes,2006,55(10):2909-2914.
[19]JACQUEMIN P,LANNOY V J,ROUSSEAU G G,et al.OC-2,a novel mammalian member of the ONECUT class of homeodomain transcription factors whose function in liver partially overlaps with that of hepatocyte nuclear factor-6[J].Journal of Biological Chemistry,1999,274(5):2665.
