玻璃缺鳍鲶线粒体全基因组序列测定与分析
2018-04-26孙志鹏吕伟华匡友谊郑先虎佟广香孙效文程磊何立川曹顶臣吴学农
孙志鹏,吕伟华,匡友谊,郑先虎,佟广香,孙效文,程磊,何立川,曹顶臣,吴学农
(中国水产科学研究院黑龙江水产研究所淡水鱼类育种国家地方联合工程实验室,黑龙江 哈尔滨 150070)
玻璃缺鳍鲶(玻璃猫鱼)Kryptopterus vitreolus属鲶科、缺鳍鲶属,主要分布于泰国、马来西亚和印度尼西亚,体长一般12~18cm,是一种常见的观赏鱼。玻璃缺鳍鲶全身无鳞,缺乏色素,游泳时看上去像一副鱼类骨骼在飘动,因此又称为“幽灵鱼”。鱼类线粒体基因组(Mitochondrial DNA,mtDNA)与其他脊椎动物相似,是闭合双链环状DNA,长度一般在15.7~19.5kb,由37个编码基因、轻链复制起始区(OL)、主要的控制区(CR)及散布于不同基因间的非编码序列组成。mtDNA作为细胞核外具有自主复制、转录及翻译能力的遗传因子,相比于核DNA,其结构简单、拷贝数多、母系遗传、进化速度快、不同区域进化速率不同,而且易于扩增测序。基于这些优点,线粒体基因已作为分子标记应用到鱼类的物种鉴别、分类及系统发育等研究领域中[1-6]。
缺鳍鲇属分类较为混乱,相关研究较少,直至2013年玻璃缺鳍鲶才被正式定种。路斌等[7]在西双版纳罗梭江鱼类资源调查中发现了湄南缺鳍鲇Kryptopterus moorei;杨光等[8]对8种鲶科鱼类的ITS序列与18s序列的研究中报道,双须缺鳍鲇Kryptopterus bicirrhis18s序列为 1 833bp,ITS1序列为408bp,ITS2序列为339bp。本研究测定了玻璃缺鳍鲶全线粒体基因组,对丰富缺鳍鲇属基础数据和系统进化研究具有重要意义。
1 材料与方法
1.1 材料
30尾玻璃缺鳍鲶购自哈尔滨四平观赏鱼公司,现有24尾养殖于黑龙江水产研究所斑马鱼生态实验室。
1.2 方法
1.2.1 基因组DNA的提取
取玻璃缺鳍鲶尾部肌肉,采用Sambrook等[9]的方法和酚-氯仿提取总DNA,用Nanodrop 8000及1.2%琼脂糖凝胶电泳检测DNA核酸含量和质量。
1.2.2 引物设计与PCR的扩增
利用已发表的瓦氏黄颡鱼Pelteobagrus vachelli[6]16对线粒体基因组扩增引物对玻璃缺鳍鲶线粒体基因组DNA进行PCR扩增,其中6对(P.V3、P.V5、P.V8、P.V9、P.V13和 P.V15本实验中分别编号为KV.2、KV.4、KV.6、KV.7、KV.11 和 KV.13)可以扩增得到目的片段。利用NCBI比对测序得到的序列与南方鲇Silurus meridionalis相似度最高。根据比对结果,利用Primer 3再设计9对引物(KV.1、KV.3、KV.5、KV.8、KV.9、KV.10、KV.12、KV.14 和 KV.15)。15对引物见表1。

表1 玻璃缺鳍鲶线粒体基因组扩增引物Tab.1 Mitochondrial genome amplification primers of catfish Kryptopterus vitreolus
PCR反应体系为50μL,包括10mmol/L Tris-Cl(pH8.0),50mmol/L KCl,1.5mmol/L MgCl2,200μmol/L dNTP,上下游引物(10μmol/L)各 2μL,Taq DNA 聚合酶 3U,DNA 模板(50ng/μL)6μL,补无菌水到50μL。反应程序为 94℃预变性 3min;94℃变性30s,52~62℃复性 30s,72℃延伸 30s,35 个循环;最后72℃延伸5min,4℃保存。
1.2.3 测序与序列拼接
PCR产物经琼脂糖凝胶回收试剂盒(康为世纪)纯化,纯化产物连接到pMD18-T(TaKaRa)载体(反应体系及反应时间按照说明书进行),转化至DH5α感受态细胞内进行克隆,选择阳性克隆进行测序。由DNAMAN分析测序结果,用Staden(1.7)进行序列拼接。
1.2.4 序列分析与系统进化分析

表2 玻璃缺鳍鲶线粒体基因组结构Tab.2 Organization of the mitochondrial genome of catfish K.vitreolus
拼接得到线粒体全基因组序列,DNAMAN统计序列全长、碱基百分比等信息,利用GenBank数据库在线工具 Blast(http://blast.ncbi.nlm.nih.gov/Blast.cgi)分析序列同源性,结合 tRNA Scan-SE 1.21(http://lowelab.ucsc.edu/tRNAscan-SE/)定位tRNA基因、蛋白质编码基因、rRNA基因和D-loop区,以确定玻璃缺鳍鲶线粒体基因组的基因结构。
查找GenBank中脊椎动物线粒体核苷酸序列,使用Clustal W软件对5种鱼类全序列及13个编码蛋白基因的核苷酸序列分别进行比对分析。基于5个科12种鱼与2个外群的线粒体D-loop区核苷酸序列,由Modeltes选择最适模型,使用MEGA 6.0软件构建系统进化树,Bootsrap值1 000次。
2 结果与分析
2.1 基因组结构
玻璃缺鳍鲶线粒体基因组全长16 662bp(Genbank accession number,KY710750),包括 2 个 rRNA基因、22个tRNA基因、13个编码蛋白质基因和一个非编码控制区。玻璃缺鳍鲶线粒体基因组结构和基因排列顺序与已公布的鲇形目鱼类完全一致。其中 ND6、tRNA-Pro、tRNA-Glu、tRNA-Ser、tRNA-Alu、tRNA-Asn、tRNA-Cys、tRNA-Tyr和 tRNA-Gln 在 L链外其他大多数基因在H链编码(表2)。
A、T、C和 G四种碱基组成分别为:30.54%、26.34%、27.76%和15.36%,其中A+T含量(56.88%)明显高于G+C含量(43.12%)。基因组共包含9个基因间隔区,间隔长度1~34bp,最大间隔在tRNA-Asn和tRNA-Cys基因之间;基因间共存在12处重叠,重叠碱基数1~10bp,最长的重叠区位于ATP6和COX3基因。玻璃缺鳍鲶线粒体全基因组中除COX1以GTG为起始密码子外,其余12个编码蛋白的基因都以ATG开头。7个编码蛋白基因(ND1、Cox1、ATP8、ATP6、ND4L、ND5 和 ND6) 以TAA终止,其余6个编码蛋白的基因没有完整的终止密码子,包括Cox2、ND3和ND4以T终止,ND2、Cox3和CYTB以TA终止。
2.2 控制区(D-LOOP)结构
鱼类mtDNA D-loop区一般分为三个部分:终止序列区(termination associated sequences,TAS)、中央保守区(central conserved sequence block,CSB)和保守序列区(conserved sequence block,CSB)。参考其他鱼类控制区分析结果[10]推测出,本实验得到的玻璃缺鳍鲶的线粒体控制区结构包括:潜在终止区域(ETAS)位于 15 712~15 733bp,5个保守区域分别为CSB-1(16269~16288bp)、CBS-2(16377~16394bp)、CBS-3(16 402~16 420)、CBS-D(16 023~16 050bp)和 CBS-E(15 982~16 007 bp)。保守区域以 CSB-E开始,CSB1为中央保守区域和保守区域的分界线。
2.3 核苷酸序列比对分析
使用ClustalW软件对玻璃缺鳍鲶、南方鲇、鲇Silurus asotus、黑翅反游猫鱼Synodontis schoutedeni及斑马鱼Danio rerio的线粒体基因组全序列及13个编码蛋白基因的核苷酸序列分别进行比对分析(表3)。序列分析表明,玻璃缺鳍鲶与鲇科其他种属间具有较高的同源性(序列一致性介于76.23%~86.7%之间),其中与南方鲇最高(86.7%)。5种鱼之间线粒体基因组中COX1基因相对最保守,相似度为78.98%~92.78%。
2.4 系统进化分析
基于12种鱼类与人Homo sapiens和小鼠Mus musculus(外群)线粒体基因组D-loop区核苷酸序列,使用MEGA6.0软件构建系统进化树(图1)。所选择的12种鱼类包括:玻璃缺鳍鲶、南方鲇、鲇、黑翅反游猫鱼、荷马条鳅Homatula variegatus、虎头鲨Pangasianodon hypophthalmus、鳙 Hypophthalmichthys nobilis、新疆高原鳅Triplophysa strauchii、蒙古鲌Chanodichthysmongolicus、虹鳟 Oncorhynchusmykiss、斑点叉尾Ictalurus punctatus、大西洋鲑 Salmo salar。从图1中可以看出,玻璃缺鳍鲶独自为一支,但与鲇和南方鲇亲缘关系最近。

图1 玻璃缺鳍鲶D-loop核酸序列的系统进化树Fig.1 Phylogenetic tree of catfish Kryptopterus vitreolus based on sequences of D-loop

表3 5种鱼类线粒体基因组比较Tab.3 Similarity of 13 pairs of homolog gene between 5 fish species
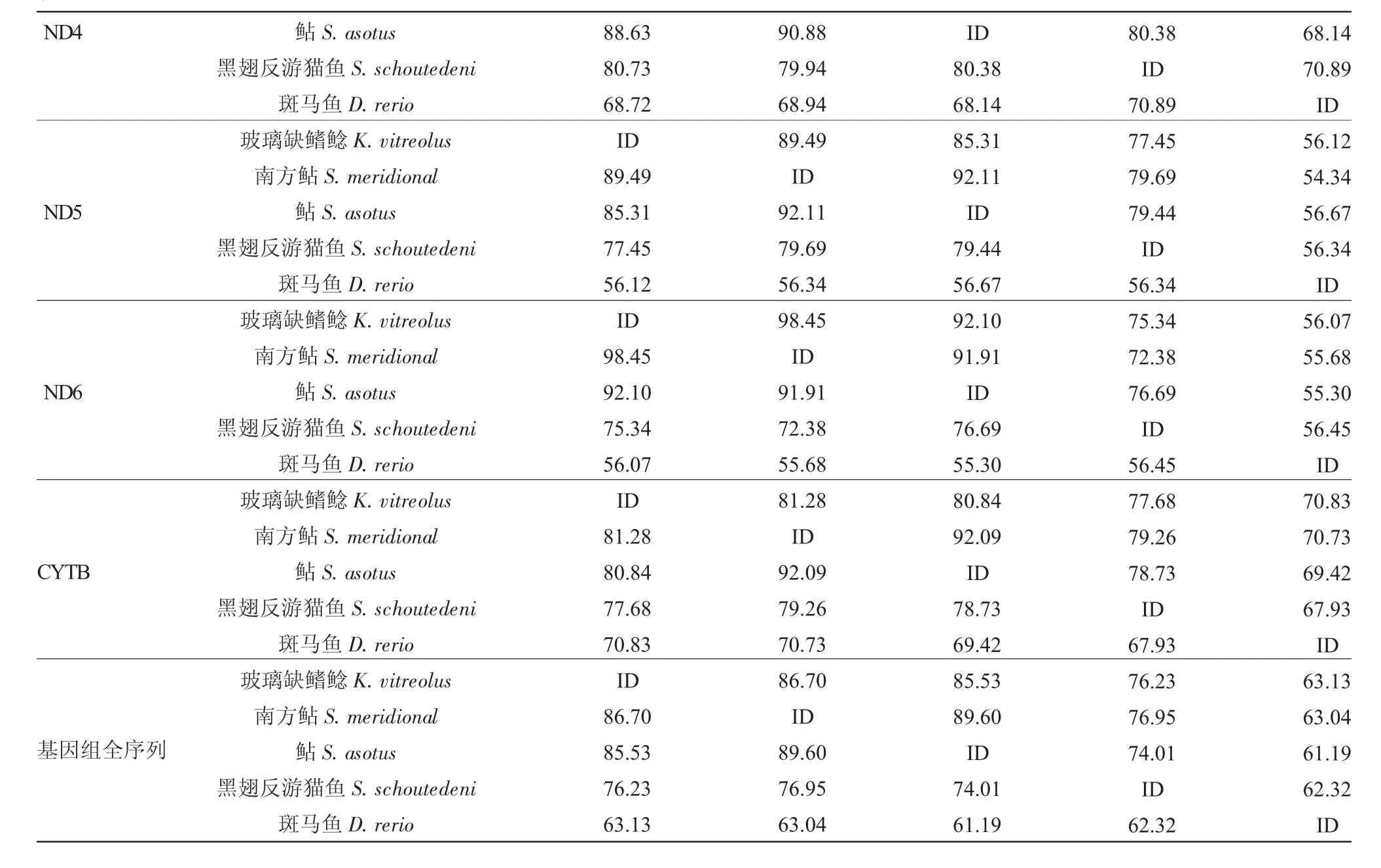
续表3
3 讨论
3.1 线粒体基因组全序列分析
玻璃缺鳍鲶线粒体基因组的基因排列顺序与已公布的鲇形目鱼类线粒体基因组完全一致,各基因长度也基本一致,证明了线粒体基因组在进化上的高度保守型。玻璃缺鳍鲶mtDNA的tRNATrp-tRNAAla-tRNAAsn-tRNACys-tRNATyr区包含一个典型的“茎-环”结构[11],tRNAAsn与tRNACys之间有一段34bp区域,这段区域可能是L链复制起点(OL)的“Stemloop”结构[12]。在其他脊椎动物中,如两栖类[13]、鱼类[14]及哺乳类[15]等也有类似结构,但在鸟类[16]和鳄类[17]中尚未发现。蒲友光等[18]认为该结构可能具有种属特异性,推测它为脊椎动物线粒体基因组的进化特征。
在13个编码蛋白的基因中有6个没有完整的终止密码子,包括Cox2、ND3和ND4以T终止,ND2、Cox3和CYTB以TA终止,这种现象普遍存在于线粒体基因组中。虽然没有终止密码子,但其DNA序列表明其转录产物3'末端为T或TA,加上线粒体mRNA 3'末端含有PloyA尾,因此转录产物在加工过程中加接上多腺苷酸后可以形成TAA终止密码子。
3.2 控制区(D-loop)结构分析
终止区域(ETAS)是D-loop区变异最大的部分,存在不同程度的串联重复序列。一般认为,每个重复序列中都含有一个保守的终止相关序列(termination associated sequences,TAS),由于 TAS 序列均存在着TACATATGTA这一个稳定的发夹结构,它可能是与H链复制终止有关的信号。本实验参照TAS发卡结构序列,在玻璃缺鳍鲶D-loop区仅识别到一个潜在终止序列E-TAS,沙鳅亚科(Botiinae)[19]、鳜属Siniperca[20]和白鱼Anabarilius grahami[21]也仅含有一个终止区域。而朱世华等[22]在大部分鲹科(Carangidae)鱼类中识别到了2个终止相关序列。对于这种多终止相关序列的现象,刘焕章[23]认为只有1个TAS行使功能,其他的都是复制的没有终止功能。但具体该区含有几个终止相关序列及其功能问题研究的较少,有待进一步研究。
中央保守区CSB是整个控制区最为保守的区域,几乎在所有的种类中都十分保守。曾青兰等[24]识别了大口胭脂鱼Ictiobus cyprinellus的CSB-D序列;Lee等[25]对众多鱼类的序列进行比较时,也仅识别了CSB-D的存在,而在大部分鱼类的研究中都已识别出CSB-F、CSB-E和CSB-D,并确定了其关键序列。其中CSB-D序列较稳定,很容易识别,CSB-E含有GTGGG-box特殊结构[19-23]。本实验对照其他鱼类的中央保守区序列,成功识别了玻璃缺鳍鲶的CSB-D和CSB-E序列,但未找到CSB-F。
鱼类中一般都存在保守序列区CSB的3个保守序列区。有学者认为,CSB1一般与将近完成H链替代合成的终止信号有关;而CSB2和CSB3可能与H链的复制起始有关[26]。其中CSB1在鱼类中的变异较大,不易识别。虽然Broughton等[27]和刘焕章[23]认为GACATA最为保守,可以帮助识别CSB1的存在,但在很多鱼类却并非如此。赫崇波等[28]在圆斑星鲽Verasper variegatus识别出的CSB1并未存在该段保守序列。张燕等[29]在科识别出的CSB1与之描述也不同,但本实验测得的玻璃缺鳍鲶CSB1序列(TCTATTGACATAACTGGTCC)的特征与大多数鱼类的CSB1一致。
3.3 系统进化分析
线粒体DNA的D-loop区长度和进化速率适中,以及富含系统发育信息等特点,被作为DNA通用标签广泛应用于种质鉴定、物种分类和系统进化研究。彭士明等[30]利用银鲳Pampus argenteus的线粒体D-loop区序列比较分析了养殖与野生银鲳Pampus argenteus群体的遗传多样性。董志国等[31]利用三疣梭子蟹Portunus trituberculatus的线粒体D-loop区序列分析了中国海三疣梭子蟹的遗传多样性。姬南京等[32]利用仿刺参Apostichopus japonicas Selenka的线粒体D-loop区序列分析了大连、朝鲜罗津和俄罗斯海参崴3个仿刺参群体的遗传结构和系统发生。本研究应用玻璃缺鳍鲶线粒体DNA D-loop区序列与其他鲇形目鱼类进行系统进化关系分析和物种鉴定分析。结果表明,以D-loop区序列为标记可有效分析鲇形目鱼类的系统进化与分类关系。目前关于缺鳍鲶属的研究有限,线粒体相关数据更是甚微,本实验通过对玻璃缺鳍鲶线粒体全基因组序列的测定,遗传控制区的分析,以及系统进化的讨论,为丰富缺鳍鲶属基础研究数据和促进鲶属系统进化研究具有重要意义。
[1]Thacker C E.PhylogenyofGobioidei and placement with in Acanthomorpha,with a new classification and investigation of diversification and character evolution[J].Copeia,2009,9(1):93-104.
[2] Keith P,Lord C,Lorion J,et al.Phylogeny and biogeographyofSicydiinae(Teleostei:Gobiidae)inferred from mitochondrial and nuclear genes[J].Mar Biol,2011,158(2):311-326.
[3]Lord C,Lorion J,Dettai A,et al.From endemism to widespread distribution:phylogeography of three amphidromous Sicyopterus species(Teleostei:Gobioidei:Sicydiinae)[J].Mar Ecol,2012,455:269-285.
[4] Jeon H B,Choi S H&Suk H Y.Exploring the utility of partial cytochrome c coxidase subunit for DNAbarcodingof gobies[J].AnimSyst Evol Diver,2012,28(4):269-278.
[5] Thacker C E.Molecular phylogeny of the gobioid fishes(Teleostei:Perciformes:Gobioidei)[J].Mol Phylogenet Evol,2003,26(3):354-368.
[6]李林,梁宏伟,李忠,等.瓦氏黄颡鱼线粒体全基因组序列分析及系统进化[J].遗传,2011,33(6):627-635.
[7]路斌,仝向荣,王德斌,等.云南西双版纳罗梭江的鱼类物种组成及现状[J].昆明学院学报,2009,31(6):60-63.
[8]杨光,俞菊华,徐跑,等.利用18S和ITS序列揭示8种鲇形目鱼类的系统发育[J].动物学杂志,2010,45(4):110-117.
[9]Sam brook J&Russell D W.Molecular Clonging:A LaboratoryManual,3rd edn[M].Cold SpringHarbor LaboratoryPress,NY,2001:463-471.
[10]邹芝英,杨弘,罗永巨,等.长鳍鲤mtDNAD-loop区序列结构和多态性分析[J].江苏农业科学,2013,41(8):27-30.
[11] Rafael Z,Edward M T,Michael V,et al.Complete nucleotide sequence of the mitochondrial genome of a salamander,Mertensiellaluschani[J].Gene,2003,317:17-27.
[12]Kumazawa Y&Nishida M.Complete mitochondrial DNA sequences of the green turtle and blue-tailed mole skink:statistical evidence for archosaurian affinity of turtles[J].Mol Biol Evol,1999,16(6):784-792.
[13]Zardoya R,Malaga-Trillo E,Veith M,et al.Complete nucleotide sequence of the mitochondrial genome of a salamander,Mertensiella luschani[J].Gene,2003,31(7):17-27.
[14] Rasmussen A S&Arnason U.Phylogenetic studies of complete mitochondrial DNA molecules place cartilaginous fishes within the tree of bony fishes[J].J Mol Evol,1999,48(1):118-123.
[15]Horai S,Hayasaka K,Kondo R,et al.Recent African origin of modern humans revealed by complete sequences of hominoid mitochondrial DNA[J].Proc Natl Acad Sci,1995,92(2):532-536.
[16]Mindell D P,Sorenson MD,Dimcheff D E,et al.Interordinal relationships of birds and other reptiles based on wholemitochondrial genomes[J].Sys tBiol,1999,48(1):138-152.
[17]吴孝兵,王义权,周开亚,等.扬子鳄的线粒体全基因组与鳄类系统发生[J].科学通报,2003,48(18):1954-1958.
[18]蒲友光,彭巧玲,王志方,等.乌龟线粒体全基因组序列和结构分析[J].动物学报,2005,51(4):691-696.
[19]唐琼英,刘焕章,杨秀平,等.沙鳅亚科鱼类线粒体DNA控制区结构分析及系统发育关系的研究[J].水生生物学报,2005,29(6):645-653.
[20] Zhao J L,Wang W W,Li S F,et al.Structure of the mitochondrial DNA control region of the sinipercine fishes and their phylogenetic relationship[J].Acta Genetica Sinica,2006,33(9):793-799.
[22]朱世华,郑文娟,邹记兴,等.科鱼类线粒体DNA控制区结构及系统发育关系[J].动物学研究,2007,28(6):606-614.
[23]刘焕章.鱼类线粒体DNA控制区的结构和进化:以鳑鮍鱼类为例[J].自然科学进展,2002,12(3):266-270.
[24]曾青兰,刘焕章.大口胭脂鱼线粒体DNA控制区序列的研究[J].湖北大学学报:自然科学版,2001,23(3):261-264.
[25]Lee W,Conroy J,Howell W H,et al.Structure and evolution ofteleost mitochondrial control regions[J].J Mol Evol,1995,41(1):54-66.
[26] SbisàE,Tanzariello F,Reyes A,et al.Mammalian mitochondrial D-loop region structural analysis:identification ofnewconservedsequencesandtheirfunctionalandevolutionaryimplication[sJ].Gene,1997,205(1-2):125-140.
[27]Broughton R E&Dowling T E.Length variation in mitochondrial DNA of the minnow Cyprinella spilloptera[J].Genetics,1994,138(1):179-190.
[28]赫崇波,曹洁,刘卫东,等.圆斑星鲽及相关种类线粒体DNA控制区结构分析[J].遗传,2007,29(7):829-836.
[29]张燕,张鹗,何舜平.中国科鱼类线粒体DNA控制区结构及其系统发育分析[J].水生生物学报,2003,27(5):463-467.
[30]彭士明,施兆鸿,侯俊利.基于线粒体D-loop区与COI基因序列比较分析养殖与野生银鲳群体遗传多样性[J].水产学报,2010,32(1):19-25.
[31]董志国,李晓英,王普力,等.基于线粒体D-loop基因的中国海三疣梭子蟹遗传多样性与遗传分化研究[J].水产学报,2013,37(9):1304-1312.
[32]姬南京,常亚青,赵冲,等.3个地理群体仿刺参D-loop序列的变异及系统发生分析[J].水产学报,2014,38(4):476-481.
