油茶钙调素基因的cDNA克隆与表达分析
2018-04-20王保明谭晓风胡孝义石明旺
王保明 ,谭晓风 ,胡孝义 ,石明旺 ,莫 华
(1.湖南应用技术学院 农林科技学院,湖南 常德 415000;2.中南林业科技大学 a.经济林培育与保护教育部重点实验室;b.经济林培育与利用湖南省高校“2011”协同创新中心,湖南 长沙 410004)
油茶Camellia oleifera属于山茶科山茶属,为常绿小乔木或灌木,是我国重要的木本食用油料树种,从其种子中提取的茶油中不饱和脂肪酸含量普遍达90%以上,其中油酸达80%以上,亚油酸8%左右,它不仅具有较高的食用价值,而且具有较好的药用价值。它主要分布在秦岭—淮河以南的14个省(区),栽培面积超过3.00×106hm2,适应性强[1-2]。钙调素(calmodulin, CaM)是高度保守的钙离子结合蛋白,位于胞浆、细胞器或者它们的膜上,它将钙离子信号传递给下游而调控细胞的代谢[3-4]。它是一类依赖于Ca2+的热稳定小分子蛋白,由19种氨基酸组成,等电点为3.9~4.3。该蛋白含有谷氨酸(G)和天冬氨酸(D),具有疏水性,包括4个EF-手臂(螺旋-线-螺旋结构),每个EF-手臂均含有一个D-x-D (第14~16位氨基酸),并分别结合一个Ca2+;每2个EF臂由可自由伸展的螺旋连结成一组。结合Ca2+后,其分子构象由哑铃状变为球状,疏水区激活与靶蛋白结合[5-9]。CaM作为Ca2+的下游靶蛋白,在Ca2+所介导的反应中充当中间媒介,通过与细胞内酶或蛋白质结合调节细胞的生理生化过程,在细胞增殖、分化、凋亡、迁移中发挥重要作用,参与冷热刺激、伤害、盐胁迫、水胁迫的信号转导[3-4,10-12]。在植物中,已经从拟南芥Arabidopsis thaliana[7-8,13-14]、水稻Oryza sativa[15]、大麦Hordeum vulgare[16]、玉米Zea mays[17]、马铃薯Solanum tuberosum[18]、苜蓿Medicago sativa[19]、菜豆Phaseolus vulgaris[20]、花 生Arachis hypogaea[21]、 柑 橘Citrus unshiuMarc[22]、苹果Malus domestica[23]、猕猴桃Actinidia deliciosa[24]中分离出钙调素基因。它们编码的蛋白存在于叶绿体、线粒体、原生质膜、核膜,在分生组织中最多,在成熟组织中表达较为一致,在花粉管、柱头、发育中的雌蕊细胞核、核仁中大量表达[25-27]。笔者在构建油茶cDNA文库的基础上,分离克隆了油茶钙调素基因并分析其结构特征,揭示了它们在叶、花芽、幼果、成熟种子中的表达模式,旨在为油茶组织发育、种子油脂代谢、胁迫应答等研究提供可利用的资料。
1 材料与方法
1.1 油茶cDNA文库的构建
以油茶优良无性系‘湘林1 号’和‘湘林4号’的近成熟种子为材料构建cDNA文库。裂解法提取总RNA,磁珠法分离mRNA后,SuperScriptⅡ合成cDNA第一链,第二链合成后未端补平、加接头、酶切、回收cDNA、重组cDNA,与载体pBluescript Ⅱ连接,转入大肠杆菌。以X-gal平板筛选重组克隆[28]。
1.2 油茶CaM基因的克隆、鉴定及序列测定
挑取单克隆活化培养,参照《分子克隆实验指南》(第3版)[29]中的SDS碱裂解少量制备质粒DNA法提取质粒DNA。通用引物T3(5′-ATTAACCCTCACTACCCGGGA-3′) 和 T7(5′-GCGTAATACGACTCACTATA-3′) 作 为 引物进行PCR扩增,反应体系为20 μL,其中,含 有 1×PCR buffer,1.5 mmol MgCl2,0.5 mmol dNTPs,0.5 μmol T3和 T7引 物,10 ng质 粒DNA,1 U Taq DNA聚合酶。PCR扩增条件为:94 ℃预变性5 min;94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸1 min,35个循环;72 ℃继续延伸5 min。取2 μL扩增产物在1.5%琼脂糖凝胶上电泳。目的克隆双向测序。
1.3 钙调素蛋白的结构、理化性质预测、同源性及遗传进化分析
利用生物软件DNAstar Protean、ANTHEPROT V4.3c 以及在线分析(www.embl-heidelberg. de/predictprotein/predictprotein.html和http://www.predictprotein.org/newwebsite/submit.php)油茶钙调素蛋白的结构特征。DNAStar Protein 预测该蛋白的可溶性,Karplus-Schultx方案[30]预测该蛋白的柔性,Jameson-Wolf方法[31]预测该蛋白的抗原性。利用NCBI Blast、Vector NTI、Gendoc和 Treeview 分析油茶钙调素基因的氨基酸序列特征和遗传进化关系。
1.4 RNA 制备、cDNA 第一链合成、标准cDNA制备、标准曲线和荧光定量表达分析
提取油茶优良无性系‘华硕’叶、茎、花芽、子房、幼果和成熟种子的总RNA,用DNA-free kit (Ambion)消除样品中的基因组DNA,凝胶电泳检测;然后测定其浓度和纯度。从每个样品的总RNA中取2.0 μg,用 RevertAid第一链cDNA试剂盒和oligo (dT)18引物进行反转录,获得cDNA第一链。
引 物(CoCaMF1,5′-CTTGGAACAGTGATG CGGTC-3′;CoCaM R1,5′-CAGCCTCACGAATCA TCTCA-3′;CoCaMF2,5′-CTTGGAACGGTG ATGCGGTC-3′;CaMR2,5′-CAGCCTCACGAATC ATCTCA-3′)分别用于扩增cDNA标准样品,扩增体系与上述PCR体系类似。PCR扩增条件为:94 ℃预变性 5 min;94 ℃变性 30 s,60 ℃退火 30 s,72 ℃延伸2 min,36个循环;72 ℃ 7 min终止反应。PCR产物纯化,然后测定其浓度。利用公式C=(D×6.02×1023)/(l×1×109×660), 计 算 cDNA标准样品的拷贝数。其中,C为标准样品的拷贝数,D指光密度(OD),l代表模板DNA长度。将cDNA标准样品分别稀释1倍、10倍、100倍、1000倍和10000倍,计算标准样品的拷贝数。利用Roter Gene Q300072孔(QIAGEN)荧光PCR仪扩增,反应体系为10 μL,包括Maxima SYBR Green qPCR Master mix (2×)5.0 μL,引 物 10 μmol 0.3 μL,2.0 μL 20 倍 稀 释 合 成 的cDNA第一链作为模板。反应条件为预变性94 ℃5 min,变性 94 ℃ 30 s,退火 60 ℃ 40 s,72 ℃延伸30 s,40个循环。每个反应中含有2个不加模板的对照,重复3次。采用Software Version Rotor-Gene 2.0.2.4 分析数据,油茶钙调素基因的表达量通过标准曲线分析获得。
2 结果与分析
2.1 油茶cDNA文库的构建
在含X-gal平板上观察到的大量白色菌落,即为重组克隆,计算后得知:cDNA文库的库容量为8.68×106,即,该文库约含有8.68×106个克隆。
2.2 油茶钙调素基因的鉴定
从油茶cDNA文库中挑取单克隆rzots0_000451. y1.scf和 rzots0_02160 y1.scf,活化培养提取质粒DNA,以通用引物T3和T7 为引物,质粒DNA为模板进行PCR扩增,得到2条长度约为1100 bp的电泳条带(见图1)。
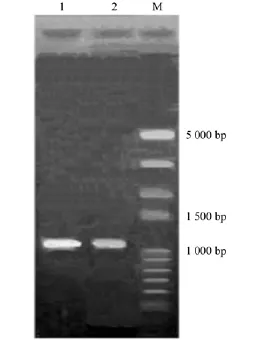
图1 油茶cDNA克隆rzots0_000451.y1.scf和rzots0_02160y1.scf的PCR检测Fig.1 The PCR result of the cDNA clones of rzots0_000451.y1.scf and rzots0_02160 y1.scf
将单克隆rzots0_000451和rzots0_002160进行T3和T7双向测序,把获得的序列在NCBI blast分析,结果显示:它们的核苷酸和氨基酸序列与Genbank中甜樱桃Prunus avium、狗枣猕猴桃Actinidia kolomikta等钙调素基因的编码区相似性超过85%,表明它们为钙调素基因,命名 为CoCaM1和CoCaM2(GenBank登 录 号:EU856536和FJ649316)。CoCaM1的核苷酸序列全长为953 bp,包含450 bp的CDS(coding regions)、77 bp 5′ 端 非 编 码 区(5′-untranslated region,5′ UTR) 和 425 bp 含 有 Poly(A) 的 3′UTR。其AT含量为59.00%。CoCaM2的cDNA全长为1024 bp,包含450 bp的CDS、84 bp的5′UTR、406 bp的3′ UTR以及84 bp的poly(A),其AT含量为57.44%。它们的起始密码子和终止密码子相同(分别为ATG和TGA);在5′UTR和3′ UTR中,AT含量较高。其中,在3′ UTR中较高的AT含量能够降低mRNA稳定性,因而有利于转录解链。这2个基因的核苷酸序列一致性为72%,编码区的核苷酸序列一致性高于拟南芥钙调素(AtCaM-2和AtCaM-3)[32]。它们编码的149个氨基酸完全相同,只是同一氨基酸的核苷酸存在C-T、T-C、C-A、A-G、G-C和C-A等碱基替换(图2涂灰部分)。研究显示:在花生钙调素基因的不同cDNA间也有碱基替换[21],这表明钙调素基因存在着碱基替换现象。“多个基因编码一种蛋白”的假设可以解释CaM在组织和器官中的特异表达[32-33]。油茶钙调蛋白的显著特点是结构高度保守,包括4个EF-手臂(图2深灰色部分)和1个半胱氨酸残基(Cys-27)(图2浅灰色部分)。
2.3 钙调素蛋白的结构、理化性质、同源性及遗传进化分析

图2 油茶CoCaM1和CoCaM2基因全长cDNA的核苷酸序列和氨基酸序列Fig. 2 The nucleotide sequences and amino acid sequence of the full-length cDNAs of CoCaM1 and CoCaM2 of C. oleifera
应用http://www.expasy.ch/tools/protparam.html和DNAStar Protean分析,结果显示:油茶钙调素蛋白含有149个氨基酸,相对分子质量为16.83 kDa,是由19种氨基酸组成的酸性蛋白。其中,酸性氨基酸残基天冬氨酸(D)和谷氨酸(E)占25.5%,碱性氨基酸残基(K, R)占12.25%,疏水性氨基酸残基(A,I,L,F,V)占28.90%,极性氨基酸残基(N,C,Q,S,T,Y)占18.89%。研究显示:一些氨基酸残基可能分别在N端、C端构成疏水穴,与中心螺旋共同负责与靶酶、拮抗剂的结合[34]。当它与靶酶结合时,由于中央螺旋具有可伸展性,因而能与不同的靶酶相互适合[34]。第66~93位氨基酸构成中央α-螺旋,其中,第77~82位氨基酸为柔性较强的非螺旋区(见图3和图4)。由于甲硫氨酸本身含有不分枝的4个重原子(Cβ、Cγ、Sδ和Cε)构成疏水侧链,而该蛋白含有9个甲硫氨酸,因此,甲硫氨酸残基在Ca2+-CaM-靶酶的疏水结合中发挥重要作用;同时,带负电的谷氨酸残基(E)可能与靶酶的正电荷存在静电作用,所以,Ca2+-CaM-靶酶的稳定性主要靠疏水力和静电引力[34]。该蛋白是稳定性亲水性蛋白,含有1个N连接糖基化位点(NGTI)、1个PKC磷酸化位点(protein kinase C phosphorylation site)(TTK)、6个酪蛋白激酶Ⅱ磷酸化位点(SLFD、TTKE、TEAE、TDSE、SAAE和TDEE)和2个N端酰基化位点(GCITTK和GQNPTE)等结构域。
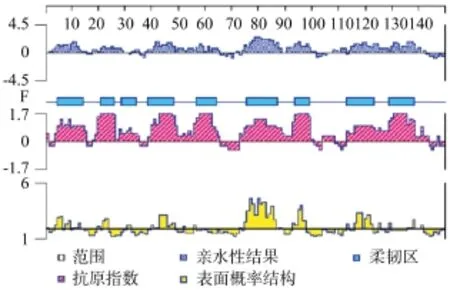
图3 基于DNAStar Protein 的油茶钙调素蛋白CoCaM亲水性、柔性及抗原性预测Fig.3 Prediction of hydropathy plot, fl exibility and antigenicity of CoCaM by DNAStar Protean

图4 油茶钙调素的氨基酸序列与其它动植物钙调素的氨基酸序列比对Fig.4 Amino acid sequence alignment of CaMs from Camellia oleifera, animals and other plants
采用自优化预测方法——SOPMA[35]预测蛋白有较好的准确性(超过70%),结果显示油茶钙调蛋白包括64.43%的α-螺旋、3.36%的β折叠、8.72%的转角和23.49%的卷曲。采用PHD[36-38]方法也得到相似的结果。该蛋白含有8个α-螺旋,其中7个α-螺旋均含有11氨基酸(见图5)。ANTHEPROT V4.3c软件分析显示,它的N端第2个α-螺旋(30~40)和C-端的第7个α-螺旋(119~129)(PHD法)分别有4个疏水性残基位于螺旋轮的同一侧,亲水性残基多位于螺旋轮的另一侧,这符合两亲性α-螺旋的特征,表明该蛋白同时具有亲水性和疏水性(见图5)[39]。当它与Ca2+与结合时,构象发生变化,暴露出强疏水性表面与靶蛋白作用,这与拟南芥等钙调素的结构特征相似[40]。
DNAStar Protean软件分析显示:油茶钙调素蛋白的抗原指数与转角、卷曲关系密切,潜在抗原多在转角和卷曲区。亲水区域分布较为均匀,对亲水性贡性献最大的氨基酸残基位于亲水中心(第78~84位氨基酸),含有较多的谷氨酸(E)和天冬氨酸(D)。该蛋白的可溶性和亲水性较好,抗原区几乎都在亲水区,并且表现高度的柔性。Karplus-Schultx[30]法分析结果显示:该蛋白在第10~14、39~48、76~87、113~123、129~138位氨基酸具有较好的柔性,容易与抗体嵌合。Jameson-Wolf法[31]分析结果显示:该蛋白具有良好的抗原性(见图3)。
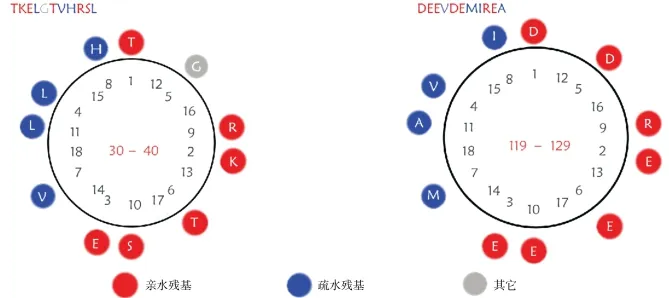
图5 油茶钙调素蛋白CoCaM的α-螺旋两亲性分析Fig.5 Amphiphatic α-helix analysis of CoCaM
在线网站预测(http://bioweb.pasteur.fr/seqanal/interfaces/toppred.html、http://www.ch.embnet.org/software/COILS_form.html、http://www.ch.embnet.org/software/TMPRED_form.html、http://www.cbs.dtu. dk/services/SignalP/)发现油茶钙调素蛋白没有跨膜结构,并且不含信号肽。http://kr.expasy.org/tools/blast/、http://swissmodel.expasy.org//SM_FIRST.html分析显示4个EF 手臂分别结合1个Ca2+(见图4)。在每个EF手臂的第14~16位氨基酸含有D-x-D模块(D-K-D、D-E-D、D-K-D、D-V-D),这符合Mohantaet al.[5]所述的钙调素EF 手臂特征。油茶钙调素与动物(非洲爪蟾和人类)和高等植物(拟南芥、玉米、油菜等)钙调素的氨基酸序列具有一致性;同时也暗示了它与人类钙调素二级结构的相似性,如α-螺旋和β折叠。通过在线预测(http://pfam.janelia.org/)、多重序列比对和隐马尔可夫模型分析发现:该蛋白还有Death effector daim、Dockenn type reapt 以及1个未知功能区。油茶钙调素的氨基酸序列与花生钙调素(PCaM-1,AAR99409)的氨基酸序列Ⅰ和Ⅱ结构域100% 相同;Ⅲ和Ⅳ结构域分别只有L-F、E-D各1个氨基酸替换,表明钙调素蛋白的Ⅰ、Ⅱ、Ⅲ、Ⅳ结构域高度保守[21]。
从图4可以看出,油茶钙调素的氨基酸序列高度保守,与其它植物钙调素的同源性高达90%以上。其中,它与拟南芥CaM(NP_180271)和小麦(AAC49582)只有2个氨基酸残基差异,与烟草钙调素(BAB61915) 具有100%的同源性,与玉米、小麦和油菜钙调素的同源性高达90%左右。但与动物(非洲爪蟾和人类)的遗传距离较远。根据Vector NTI blast和TreeView分析结果,得到油茶钙调蛋白与其它植物钙调蛋白氨基酸的遗传进化树(见图6),结果显示:该蛋白与烟草、油菜的钙调素蛋白有较近的亲缘关系,其中它与烟草钙调素的亲缘关系最近。
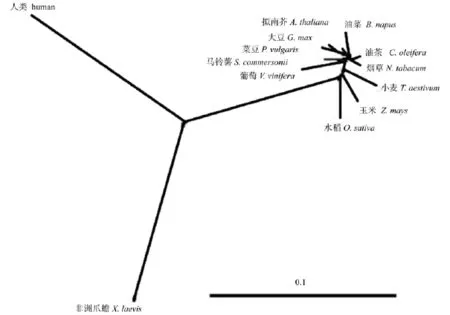
图6 油茶钙调素与其它动植物钙调素的聚类树状图Fig.6 The phylogenetic analysis of CaMs from Camellia oleifera, animals and other higher plants
2.4 油茶钙调素基因的标准cDNA制备、标准曲线和荧光定量表达分析
通过RT-PCR获得油茶CoCaM1和CoCaM2的cDNA标准样品,它们长度均为292 bp。这2个基因的cDNA标准样品梯度稀释扩增的标准曲线呈线性关系,基因拷贝数的变化范围分别为103~108和103~ 108拷贝 /μL(见图 7A-1, B-1)。CoCaM1和CoCaM2的荧光定量PCR结果显示,它们表达量存在较大的差异,其中,CoCaM1的表达量在‘华硕’组织器官中变化不大,在叶、茎、花芽、子房、幼果(4月)、幼果(7月)和成熟种子中的表达量分别为6.01×105、2.23×106、3.51×106、4.19×106、2.33×106、2.07×106和9.71×105拷贝/μL。CoCaM1在花芽到子房形成中的表达量较高;其中,在子房中最多,在花芽中次之,在4月幼果、7月幼果、成熟种子中的表达量逐渐减少(见图7A-2);而CoCaM2在油茶叶、茎、花芽、子房、果实(种子)不同发育期的表达量分别为1.20×106、2.28×107、2.36×107、2.62×107、4.73×107、3.42×107和 2.84×106拷贝 /μL,从子房到 4 月份幼果形成,它的表达量急剧增加,在4月份幼果中达到最高,然后逐渐下降,特别是从7月份到种子成熟过程中的表达量急剧下降(见图7 B-2)。
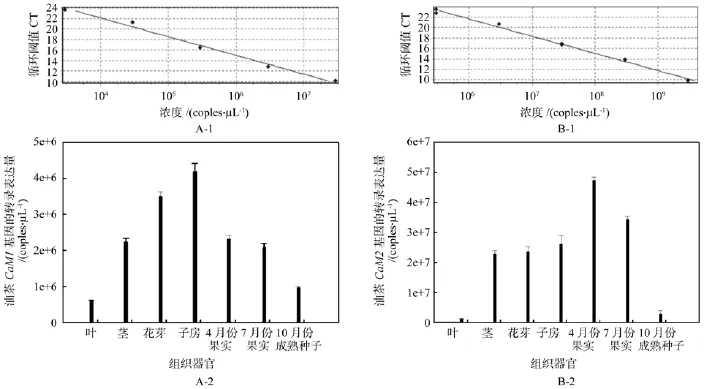
图7 油茶CoCaM1、CoCaM2的标准曲线及它们在不同组织器官中的表达Fig.7 The standard curves and expression levels in different tissues and organs of CoCaM1, CoCaM2
3 结论与讨论
在构建油茶cDNA文库基础上,分离了2个全长的钙调素基因,它们编码完全相同氨基酸序列,这种特性确认了“多个基因编码一种蛋白”假设。在编码区的核苷酸碱基替代可能是由于基因复制和剪辑产生了不同的油茶钙调素异构体。根据蚕豆[20]、拟南芥[32]、以及人类[33]钙调素的研究结果,推测油茶为了保持核苷酸序列最大的差异性,在选择压力下可能至少存在2个基因编码同一个钙调素蛋白,并且时空表达存在差异性[7]。油茶与其它植物钙调素基因序列的高度一致表明它们起源于相同的祖先[41]。这种高度保守性对于结合钙离子十分重要[42]。
EF手臂结合Ca2+后,产生构象变化以识别靶蛋白;柔韧性能使它缠绕并紧密结合靶蛋白。大量研究也提供了钙调素和钙调素样蛋白结合的证据:如,它结合抑制蛋白FLICE样蛋白调节并诱导Fas的传导,为癌症治疗提供可能靶蛋白[10];钙调素作为传导媒介,参与植物病原体免疫反应[43-46];调节结合K+、Ca2+通道开闭。这表明油茶钙调素在免疫和胁迫反应[8,43-45]、生长反应[46]、淀粉积累[46]、基因表达、激素分泌、细胞运动[47-48]、增殖[49]和凋亡[10]中具有重要作用[50]。有关研究[51]显示:双亲性α-螺旋能结合并改变浆膜或胞膜,具有稳定膜结构和亲合油脂的作用。由于油茶钙调素具有两亲性α-螺旋TKELGTVMRSL和DEEVDEMIREA,因而在油脂合成中具有重要作用。蛋白质二级结构与抗原表位的分布关系密切[52]。α-螺旋、β折叠常处于蛋白质的内部,难以与抗体结合,油茶钙调素含有较多的α-螺旋,所以它的抗原性可能较弱。该蛋白的亲水性、抗原表位、可及性、柔性具有较好一致性,可能存在多个潜在的抗原表位位点。今后有待于利用原核表达,制备油茶钙调蛋白的抗体,进一步确定它的抗原结构,鉴定叶、花、果实(种子)的CaM蛋白与它们互作蛋白的关系[53]。
从油茶钙调素的表模式可以发现:在组织器官和果实(种子)发育中,CoCaM1的表达量多低于CoCaM2,其中,CoCaM1在从花芽到子房形成中的表达量较高,在果实前期形成中作用较为明显;而CoCaM2在幼果期形成到果实膨大、油脂合成与积累高峰期的表达量较大,而在叶片和成熟种子中较少。这种差异表达表明它们在花芽发育、果实形成、油脂合成积累中发挥的作用不同。前者对花芽果实形成的作用较大,后者对果实发育及油脂形成的作用较大。这两个钙调素基因的差异表达和不同的调控作用可以用“多个基因编码一种蛋白” 假设解释[32-33]。油茶钙调素的差异表达暗示了它们在胁迫反应、生长发育、油脂合成与积累中不同的调控作用,这有待于今后结合脂肪酸和油脂形成及含量变化,进一步明确钙调素的功能。在此基础上,对油茶进行冷热、干旱等胁迫处理,从转录组和代谢的组角度进一步研究CaM的代谢网络与调控关系,确认它在胁迫条件下对油脂形成和积累的作用[54]。
总之,在构建油茶近成熟种子cDNA文库的基础上,分离鉴定了油茶钙调素基因CoCaM1和CoCaM2的cDNA序列,它们编码蛋白的氨基酸序列完全相同,证实了“多个基因编码一种蛋白”的假设。该蛋白含有EF-手臂、半胱氨酸等结构域,其亲水性/疏水性、柔性、抗原性具有较好一致性,并表现高度的柔性。它们在叶、花、果实(种子)等器官中差异表达,这暗示了它们在油茶生长发育、油脂合成与积累中具有不同的调控作用。
参考文献:
[1] 庄瑞林.中国油茶[M].第2版.北京:中国林业出版社,2008.
[2] 谭晓风,马履一,李芳东,等.我国木本粮油产业发展战略研究[J].经济林研究,2012,30(1):1-5.
[3] Tomlinson S, MacNeil S, Walker SW,et al.Calmodulin and cell function[J]. Clin. Sci.,1984,66:497-507.
[4] Veigl M L,Vanaman T C,Sedwick W D. Calcium and calmodulin in cell growth and transformation[J].Biochim Biophys Acta,1984,738:21-48.
[5] Mohanta T K, Kumar P, Bae H. Genomics and evolutionary aspect of calcium signaling event in calmodulin and calmodulinlike proteins in plants[J].BMC Plant Biol,2017,17:38.http://dx.doi.org/10.1186/s12870-017-0989-3.
[6] Nakayama S, Moncrief N D, Kretsinger R H. Evolution of EF hand calcium-modulated proteins. II. Domains of several subfamilies have diverse evolutionary histories[J].J. Mol. Evol.,1992,34:416-448.
[7] Perera I Y, Zielinski R E.Structure and expression of theArabidopsisCaM-3 calmodulin gene[J].Plant Molecular Biology,1992, 19:649-664.
[8] Zielinski R E. Characterization of three new members of theAarbidopsis thalianacalmodulin gene family:conserved and highly diverged members of the gene family functionally complement a yeast calmodulin null[J]. Panta,2002,214:446-455.
[9] Babu Y S, Sack J S, Greenhough T J,et al.Three-dimensional structure of calmodulin[J]. Nature,1985,315:37-40.
[10] Pawar P S, Micoli K J, Ding Haitao,et al. Calmodulin binding to cellular FLICE-like inhibitory protein modulates Fas-induced signaling[J]. Biochem. J., 2008,412:459-468.
[11] Means A R, Vanberkum M F, Bagchi I,et al.Regulatory functions of calmodulin[J]. Phamacol. Ther., 1991,50:255-270.
[12] Botella J R, Arteca R N. Differential expression of two calmodulin genes in response to physical and chemicals timuli[J].Plant Molecular Biology, 1994,24:757-766.
[13] Chandra A, Thungapathra M, Upadhyaya K C. Molecular cloning and characterization of a calmodulin gene fromArabidopsis thaliana[J].Plant Biochem. Biotechnol., 1994,3(1):31-35.
[14] Ling V, Perera I, Zielinski R. Primary Structures ofArabidopsisisoforms deduced from the sequence of cDNA clones[J].Plant Physical,1991,96:1196-1202.
[15] 刘芝华,吴湘钰,潘 乃,等.水稻钙调蛋白基因克隆及结构分析[J].生物工程报,1993,9(4):309-313.
[16] Ling V, Zielinski R E. Cloning of cDNA sequences enoding the calcium-binding protein,calmodulin,from barley(Hordeum vulgareL)[J].Plant Physiol., 1989,90:714-719.
[17] Griess E A, Igloi G L, Feix G. Isolation and sequence comparison of a maize calmodulin cDNA[J].Plant Physiol,1994, 104(4):1467-1468.
[18] Takezawa D, Liu Z H, An G,et al.Calmodulin gene family in potato: developmental and touch-induced expression of the mRNA encoding a novel isoform[J].Plant Mol. Biol.,1995, 27:693-703.
[19] Barnet M J, Long S R. Nucleotide sequence of analfafacalmodulin cDNA [J]. Necleotide Acids Res., 1990,18(15):3395-3340.
[20] Alberto C, Luis C, Carmen Q,et al. Expression of different calmodulin genes in bean (Phaseolus vulgarisL.): role of nod factor on calmodulin genes regulation[J].Mol. Plant-Microbe Interact, 2002, 155:428-436.
[21] 孟玉环,张君诚,单世华,等. 花生(Arachis hypogaeaL.)CaMcDNA 基因的克隆[J].中国油料作物学报,2004,26(2):27-30.
[22] 岳海林,孟海军,邓秀新,等.柑橘钙调蛋白cDNA的克隆及序列分析[J].园艺学报,2006,33(5):1075-1078.
[23] Waltillon B, kettan R, Boxus P,et al.Cloning and characterization of an apple (Malus domestica[L.] Borkh) calmodulin gene[J].Plant Sci., 1992,812(2):201-212.
[24] 任小林,金志强.猕猴桃钙调蛋白cDNA的克隆及测序[J].西北农业大学学报,1997,25(3):1-5.
[25] 夏快飞,梁承邺,叶 秀.钙调素及钙调素相关蛋白在植物细胞中的研究进展[J].广西植物,2005,25(3):269-273.
[26] Jarrett Ha W, Brown C J, Black C C,et al. Evidence that calmodulin is in the chloroplast of peas and serves a regulatory role in photosynthesis[J].The Journal of Biological Chemistry,1982, 257(22):13795-13804.
[27] Aiwu Dong, Hua Xin, YuYu,et al.The subcellular localization ofanunusual rice calmodulin isoform, OsCaM61, depends on its prenylation status [J].Plant Molecular Biology,2002,48:203-210.
[28] 胡芳名,谭晓风,石明旺,等.油茶种子cDNA文库的构建[J].中南林学院学报,2004,24(5):1-4.
[29] 萨姆布鲁克J, 拉塞尔D W.分子克隆实验指南[M].黄培堂,译.第3版.北京:科学出版社,2002:26.
[30] Karplus P A, Schultz G E. Prediction of chain flexibility in protein[J].Naturwissenschaften,1985,72(1):212-213.
[31] Jameson B A, Wolf H. The antigenic index: a novel algorithm for prediction antigenic determinants[J].CABIOS,1988,4(1): 181-186.
[32] Gawienowski M C, Szymanski D, Perera I Y,et al.Calmodulin isformase inArabidopsisencoded by multiple divergent mRNAs[J]. Plant Mol. Biol., 1993,22:215-225.
[33] Fischer R, Koller M, Flura M,et al.Multiple divergent mRNAs code for a single human calmodulin[J]. J. Biol. Chem.,1988,263:17055-17062.
[34] 马力耕,孙大业,阎隆飞.钙调素的结构生物学研究进展[J].生物化学与生物物理进展,1999,26(3):209-213.
[35] Geourjon C, Deléage G. SOPMA: Significant improvement in protein secondary structure prediction by consensus prediction from multiple alignments[J]. Cabios,1995,11:681-684.
[36] Rost B, Yachdav G, Liu J. The PredictProtein Server[J]. Nucleic Acids Research,2004,32(Web Server issue):W321-W326.
[37] Rost B, Sander C. Prediction of protein secondary structure at better than 70% accuracy[J].J. Mol. Biol.,1993,232:584-599.
[38] Rost B, Fariselli P, Casadio R. Topology prediction for helical transmembrane proteins at 86% accuracy[J]. Protein Science,1996, 5(8):1704-1718.
[39] Segrest J P, De Loof H, Dohlman J G,et al.Amphipathic helix motif:classes and properties [J].Proteins Struct Funct Genet,1990,8:103-117.
[40] 詹红利,李翠风,祁 倩,等.拟南芥调素结合蛋白cDNA克隆的分离和初步鉴定[J].南开大学学报(自然科学版),2006,39(3):6-11.
[41] Kawasaki H, Nakayama S, Kretsinger R H. Classification and evolution of EF-hand proteins[J]. BioMetals,1998,11:277-295.
[42] Criviei A, Ikura M. Molecular and structural basis of target recognition by calmodulin[J].Annu Rev. Biophys. Biomol.struct., 1995,24: 85-116.
[43] Chiasson D, Ekengren S K, Martin G B,et al.Calmodulinlike proteins from Arabidopsis and tomato are involved in host defense againstPseudomonas syringaepv.tomato[J].Plant Molecular Biology,2005,58:887-897.
[44] Sotirios K A, Argyrokastritis A, Loukas M,et al. Isolation and characterization of stress related Heat shock protein calmodulin binding gene from cultivated cotton (Gossypium hirsutumL.)[J].Euphytica, 2006,147:343-351.
[45] Ali G, ReddyV, Lindgren P,et al.Differential expression of genes encoding calmodulin binding proteins in response to bacterial pathogens and inducers of defense responses[J].Plant Mol. Biol.,2003,51:803-815.
[46] Borsics T, Webb D, Andeme-Ondzighi C,et al. The cyclic nucleotide-gated calmodulin-binding channel AtCNGC10 localizes to the plasma membrane and influences numerous growth responses and starch accumulation inArabidopsis thaliana[J].Planta, 2007,225:563-573.
[47] Dick I E, Tadross M R, Liang H,et al.A modular switch for spatial Ca2+selectivity in the calmodulin regulation of CaV channels[J]. Nature,2008,451(7180):830-834.
[48] Schumacher M A, Rivard A F, Bächinger H P,et al. Structure of the gating domain of a Ca2+-activated K+ channel complexed with Ca2+/calmodulin[J].Nature,2001,410(6832):1120-1124.
[49] Rsmussen C D, Means A R. Calmodulin is involved in regulation of cell proliferation[J].The EMBO Journal,1987,6(13): 3961-3968.
[50] Huo Longfei, Fu Guodong, Wang Xinyan,et al.Modulation of calmodulin gene expression as a novel mechanism for growth hormone feedback control by insulin-like growth factor in grass carp pituitary cells[J]. Endocrinology,2005,146(9):3821-3835.
[51] Epand R M, Shai Y, Segrest J P,et al.Mechanisms for the modulation of membrane bilayer properties by amphipathic helical peptides[J]. Biopolymers,1995,37(5):319-338.
[52] Shi D T, Booettiger D, Buck CA. Epitopes of adhesion perturbing monoclonal antibodies map with a predicted alpha helical domain of the integrin beta Ⅰsubunit[J]. Cell Sci., 1997, 110(20): 2619-2623.
[53] Singh S, Virdi A S,Jaswal R,et al.A temperature-responsive gene in sorghum encodes a glycine-rich protein that interacts with calmodulin[J].Biochimie,2017,137:115-123.
[54] 曾艳玲,谭晓风,颜亚丹,等.基于RNA-Seq的油茶种仁萜类功能组分代谢调控研究[J].经济林研究,2016,34(4):1-6.
