耐盐小麦中TaSC基因启动子的克隆及调控功能分析
2018-04-10李艳艳曹艺茹葛荣朝赵宝存
焦 博 柏 峰 李艳艳 路 佳 张 肖 曹艺茹 葛荣朝 赵宝存
河北师范大学生命科学学院, 河北石家庄 050024
全球约1/5的灌溉土地是盐渍化土地。高盐胁迫下植物细胞质中 Na+浓度升高, 体内离子失衡, 严重影响植物的生长和产量[1]。植物在长期的进化过程中形成了一套耐盐生理生化机制, 包括通过外排多余Na+或者液泡区隔化,使 Na+/K+比例稳定, 植物体内离子趋于平衡, 从而减少盐胁迫给植物带来的危害[2-3]; 通过提高抗氧化酶的活性,清除因胁迫产生的有毒的活性氧[4]; 通过增加可溶性溶质的浓度, 调节渗透压, 保护细胞膜和酶类, 缓解盐胁迫造成的危害[5-6]。这些代谢过程的完成依赖于细胞产生的有利于耐逆的植物激素和盐胁迫应答相关基因的表达[7]。可见, 植物受到胁迫时对胁迫应答相关基因的表达调控是抵抗或耐受周围环境的重要环节。
转录水平上的调控是植物体内重要的基因表达调控方式, 需要不同顺式作用元件和反式作用因子共同参与。基因的表达是受启动子(promoter)调控的, 因此, 启动子克隆及其调控功能分析是耐逆机制研究的重要内容。最早发现的盐、旱诱导型启动子是拟南芥中的rd29A, 该启动子驱动DREB1A基因的转基因植株的耐盐、耐旱性明显高于组成型表达的转基因植株[8-9]。Xiao等[10]在大麦幼苗中分析了大麦Lea基因的诱导型启动子Dhn4s, 该启动子在干旱胁迫条件下促使外源基因表达量增加, 增强了植物的耐旱性。Guo等[11]发现CBL1基因的启动子能够受低温、高盐、洪涝等多种非生物逆境信号的诱导, 并且只在维管束中特异表达, 说明CBL1的启动子是一种诱导型启动子, 同时还是一种强组织特异型启动子。可见, 逆境诱导表达的启动子与植物对非生物胁迫的耐受性关系密切。
启动子克隆和功能研究的首选方法是交错式热不对称PCR (thermal asymmetric interlaced PCR, TAIL-PCR)和5′端缺失方法。TAIL-PCR是一种基于PCR的染色体步移技术中的半随机引物 PCR, 由 Liu和 Whittier[12]于 1995年首创, 他们利用这种方法扩增出P1和YAC插入末端序列, 并对其测序。TAIL-PCR是扩增已知序列的旁侧序列的首选方法, 是克隆基因组序列未知的物种的启动子和鉴定已知序列旁侧序列的有力工具[13]。利用该技术已克隆了花生CH5B[14]、陆地棉LEA D113[15]、棉花GhRGP1[16]和GhGal1基因[17]的启动子。Xu等[18]利用该技术从小麦中成功获得了外源插入片段在转基因小麦染色体中的旁侧序列。对启动子序列的表达活性鉴定常用的是5′端缺失方法,该方法可以分析不同片段对启动子活性的贡献。Hettiarachchi等[19]利用连续缺失方法, 检测了TOP2启动子在响应盐、脱落酸等逆境信号时发挥作用的不同DNA区段。
TaSC是耐盐小麦品系RH8706-49中受盐胁迫诱导表达的基因, 该基因过表达能够提高转基因拟南芥的耐盐性[20]。为了从调控水平分析TaSC基因的耐盐性, 本研究利用TAIL-PCR结合电子克隆技术, 从RH8706-49中克隆了TaSC基因ATG上游1419 bp的启动子序列。通过检测不同长度启动子的转基因拟南芥中GUS(β-glucuronidase)基因的表达水平来鉴定其功能, 确定了启动子中不同区段的转录激活活性, 初步研究了全长启动子在自然条件和胁迫条件下的表达活性。
1 材料与方法
1.1 植物材料与菌株
小麦(Triticum aestivumL.)耐盐突变体RH8706-49为濮农3665/百农3039杂交后代F1的花药经组织培养、EMS诱变、耐盐性反复筛选得到的, 已稳定遗传18代, 其耐盐指数≥1.3[21]。哥伦比亚野生型拟南芥(Col-0)、大肠杆菌(Escherichia coli) DH5α菌株和根癌农杆菌(Agrobacterium tumefaciens) GV3101用于启动子克隆和转化。这些材料和菌株均由河北师范大学生命科学学院遗传学教研室保存。
1.2 小麦基因组DNA的提取及TAIL-PCR
参照CTAB法[22]提取小麦叶片的基因组 DNA, 参照Liu等[23]的方法, 根据TaSC基因序列设计 3条基因特异引物(其中SP2与SP3之间的距离162 bp), 分别与简并引物进行TAIL-PCR, 并将第2轮和第3轮的扩增产物同时进行电泳, 对电泳结果中第2轮和第3轮的扩增产物的大小差异接近162 bp的条带进行回收、克隆并测序。各引物序列见表1。

表1 本研究所用引物Table 1 Primers used in this study
1.3 启动子的克隆
以TAIL-PCR方法获得的片段为基础, 在http://www.gramene.org/网站进行 BLAST, 得到一条来源于中国春小麦基因组的约1419 bp的基因组逻辑序列, 参考该序列信息, 设计引物(表1), 以RH8706-49的基因组DNA为模板进行 PCR扩增。回收扩增产物并克隆和测序, 将测序正确的克隆命名为 pMD18-T-ProTaSC, 并利用 PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)对该DNA序列进行顺式作用元件分析。
1.4 启动子驱动GUS的表达载体的构建及遗传转化
为了鉴定启动子的活性, 根据启动子 ProTaSC的序列设计了 4条上游引物(表 1), 用于扩增不同长度的 5′端渐进缺失的启动子片段。以pMD18-T-ProTaSC为模板, 分别扩增不同长度的ProTaSC启动子片段, 经克隆、测序后将正确的克隆分别命名为B-1096 bp、C-681 bp、D-343 bp和E-152 bp, 全长序列标记为A-1419 bp (图1)。将这些启动子片段与含有GUS的 pCAMBIA1300载体分别进行Hind III和BamH I双酶切后进行连接, 用不同ProTaSC启动子片段分别置换载体中的CaMV35S启动子, 获得不同的ProTaSC启动子片段驱动GUS的双元报告表达载体。
利用冻融法[24]将上述重组载体转化根癌农杆菌GV3101, 再利用浸花法[25]分别转化拟南芥, 筛选阳性苗进行培养, 用于启动子活性分析。
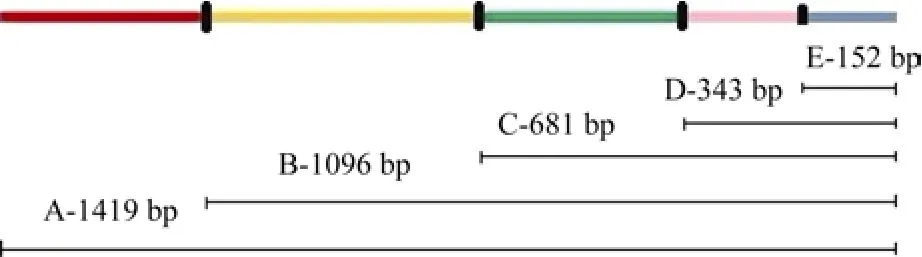
图1 用于构建GUS报告载体的TaSC启动子的不同长度片段示意图Fig. 1 Schematic diagrams of 5' deletion fragments of TaSC promoter with different lengths for constructing GUS reporter vectors
1.5 启动子的活性检测
将含不同长度 ProTaSC片段的拟南芥分别接种到1/2MS培养基上, 在普通光照培养箱(22℃, 光16 h/暗8 h)中培养。对生长7~9 d的转基因拟南芥幼苗进行GUS染色分析, 确定不同长度的启动子片段的启动活性。
将上述在1/2MS培养基上生长7~10 d后的转全长启动子的拟南芥幼苗分为两部分, 一部分转移到温室中进行蛭石培养, 28 d后取拟南芥的不同组织进行GUS染色分析; 另一部分分别转移到含 200 mmol L–1NaCl或 10 μmol L–1ABA的1/2MS培养基上胁迫处理0、24、36和72 h, 用于定量测定 GUS酶活性[26]。参考 Bradford[27]的方法定量蛋白。
2 结果与分析
2.1 TaSC启动子的克隆
以 RH8706-49基因组 DNA (图 2-A)为模板进行TAIL-PCR表明, 基因特异引物和随机引物AD4组合的第3轮扩增得到一条322 bp的片段(图2-B, 箭头所示)。该序列与第2轮扩增产物的分子量差异接近162 bp, SP3单引物扩增没有条带出现(图 2-B), 符合目的条带的特征。对该序列克隆、测序后与TaSC基因比对, 得到ATG上游313 bp的序列, 其DNA序列见图3中蓝色斜体所示部分。以这313 bp序列为基础拼接得到ATG上游1419 bp的逻辑序列, 根据逻辑序列设计引物, 扩增RH8706-49基因组DNA, 得到约1400 bp的片段(图2-C, 箭头所示), 对其克隆和测序显示, 该序列长度为 1419 bp, 与逻辑序列的同源性达90%, 命名为ProTaSC。
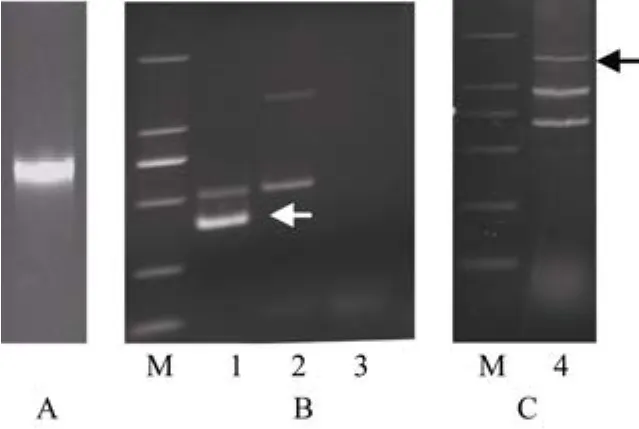
图2 TaSC基因启动子的克隆Fig. 2 Cloning of TaSC promoterA: RH8706-49的基因组DNA; B: TaSC基因启动子的TAIL-PCR扩增; C: TaSC基因的启动子扩增; M: DL2000 ladder, 条带自上而下的分子量依次外 2000、1000、750、500、200 和 100 bp; 1: SP3和AD4的三扩结果(箭头所示为目的片段); 2: SP2和AD4的二扩结果; 3: SP3引物自扩结果; 4: TaSC启动子的扩增结果, 箭头示启动子ProTaSC扩增片段。A: electrophoretic pattern of genomic DNA of RH8706-49;B: TAIL-PCR amplification of TaSC promoter; C: amplification of TaSC promoter from RH8706-49; M: DL-2000 DNA ladder in 2000,1000, 750, 500, 200, and 100 bp (from the top to the bottom);1: profile of the tertiary TAIL-PCR amplification with SP3 and AD4( target band shown by the arrow); 2: profile of the secondary TAIL-PCR amplification with SP2 and AD4; 3: the amplification profile with SP3 primer only; 4: Amplification of TaSC promoter ProTaSC (target band shown by the arrow).
2.2 启动子序列的功能预测
对ProTaSC分析发现, 序列中含有7个TATA box、13个CAAT box和多个功能顺式作用元件(图3)。不同顺式作用元件的序列、功能及其在启动子区的分布情况见表2。ProTaSC中包含非生物胁迫响应元件 ABRE和 MBS各 1个, 分别是 ABA应答元件(序列为 CACGTG, 位于-212位至-207位核苷酸区间)和MYB蛋白结合位点(序列为CAACTG, 位于-960位至-955位核苷酸区间)。
2.3 TaSC启动子的启动活性分析
对 ProTaSC启动子及其不同 5'末端缺失片段驱动GUS的转基因拟南芥植株进行GUS染色, 结果显示全长序列(1419 bp)具有启动功能, 而且在幼苗的根、茎、叶中均有表达(图4-A); 5′末端部分缺失的1096 bp 片段和681 bp片段也具有启动活性, 但启动活性较全长序列弱, 而且仅在叶片中检测到表达(图4-B, C); 转≤343 bp启动子片段的拟南芥没有GUS显色反应(图4-D, E)。上述结果表明, ProTaSC的基本启动活性中心位于起始密码子ATG上游-681至-343位核苷酸区间, 然而该区间仅具有较弱的启动功能, 表达量增强还需要-681位核苷酸上游的序列,在根中表达则需要-1096位核苷酸上游的序列。

图3 小麦耐盐突变体RH8706-49的TaSC启动子的DNA序列Fig. 3 DNA Sequences of TaSC promoter in salt-tolerant wheat mutant RH8706-49蓝色斜体字母表示TAIL-PCR的扩增结果; 下画虚线表示TATA盒; 下画实线表示CAAT盒; 阴影表示其他功能顺式作用元件, 元件名称备注在序列下方, 其中ABRE元件(CACGTG )和MBS元件(CAACTG)与高盐等逆境胁迫响应有关。The amplification fragment of TAIL-PCR is shown by blue italic letters. TATA box and CAAT box are underlined with dotted and solid lines respectively. Shadow shows other functional cis-acting elements whose name remarks under the sequence. Among them, ABRE element(CACGTG) and MBS element (CAACTG) are related to stress response.

表2 启动子ProTaSC序列中的顺式作用元件及功能Table 2 The cis-acting elements and their functions in the promoter ProTaSC sequence
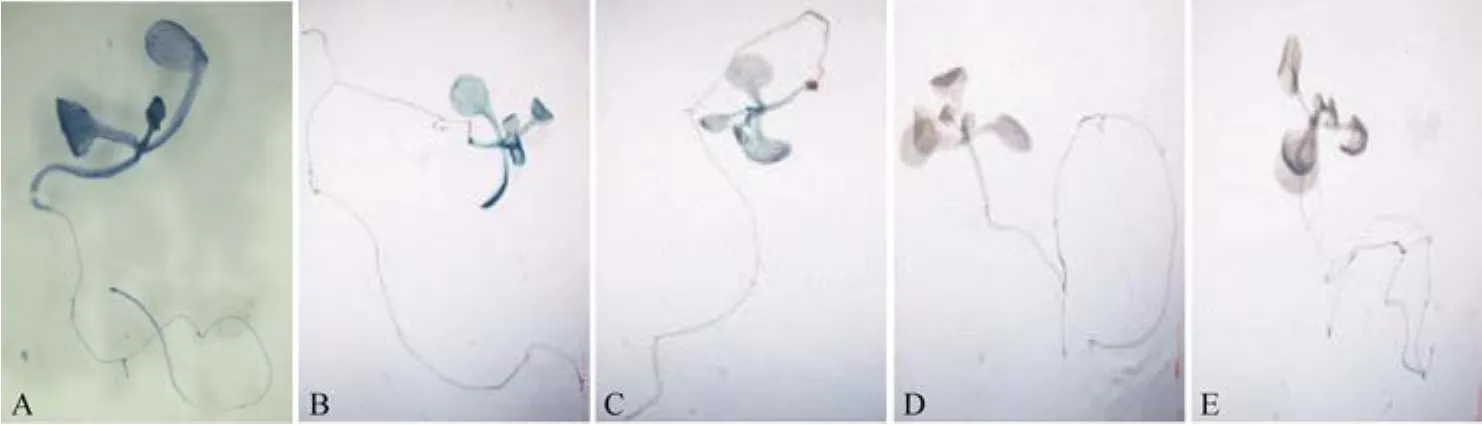
图4 转不同长度ProTaSC片段的拟南芥GUS组织化学染色结果Fig. 4 GUS assay results of transgenic Arabidopsis harboring different lengths of ProTaSC promotor fragmentsA: 转1419 bp启动子; B: 转1096 bp片段启动子; C: 转681 bp片段启动子; D: 转343 bp片段启动子; E: 转152 bp片段启动子。A: transferred with the 1419 bp promotor; B: transferred with the 1096 bp promotor fragment; C: transferred with the 681 bp promotor fragment; D: transferred with the 343 bp promotor fragment; E: transferred with the 152 bp promotor fragment.
2.4 TaSC启动子的组织表达特异性
在转全长 ProTaSC的拟南芥中, 主要在根(图 5-A)、叶片(图5-B, C)、花药和萼片(图5-D, E)及成熟果荚壳(图5-F)中检测到GUS表达, 且以根尖(图5-A)和叶脉(图5-B,C)中表达量较高, 但在主茎、花瓣、幼果和种子中没有检测到。可见, 该启动子有明显的组织表达特异性。
2.5 ProTaSC启动子受盐和ABA诱导表达
对 NaCl和 ABA分别处理不同时间的转全长ProTaSC拟南芥进行GUS酶活定量检测表明, 相对于0 h对照, NaCl处理24 h时GUS表达量上升近1倍, 表达量随后下降至初始水平, 在胁迫 72 h时表达量增长至 620 pmol 4-MU mg–1protein·min–1, 是对照的 3 倍以上(图6); ABA处理24 h时GUS表达量上调很明显, 至36 h 时表达量达到 466 pmol 4-MU mg–1protein min–1,到 72h时表达量下降至接近初始水平(图 6)。可见,ProTaSC启动子的表达活性受NaCl和ABA诱导, 但是响应时间不同。
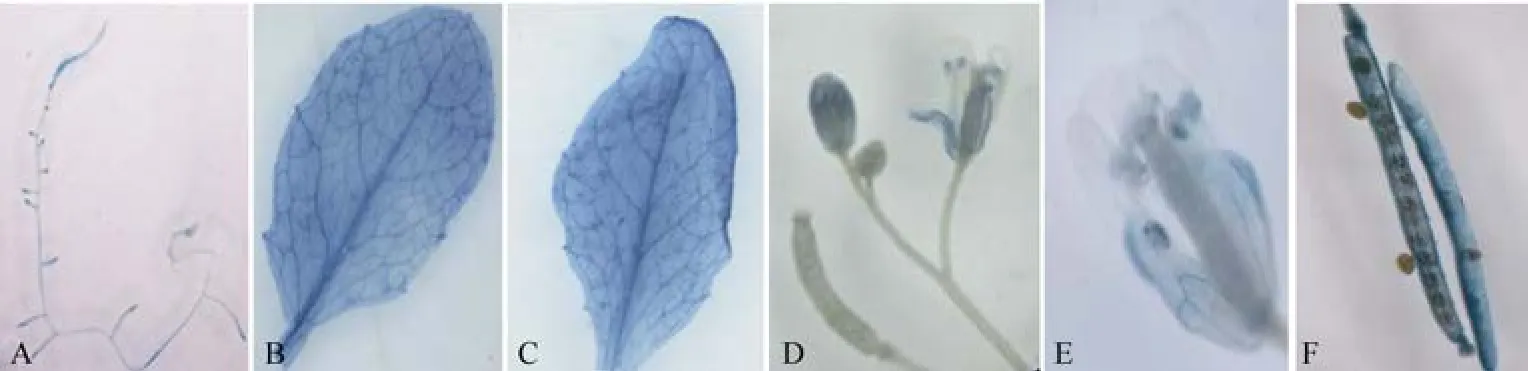
图5 转全长启动子ProTaSC拟南芥不同组织的GUS染色结果Fig. 5 GUS assay results of different tissues of Arabidopsis harboring full-length promoter ProTaSCA: 根; B: 茎上叶; C: 莲座叶; D: 花、幼嫩果荚和茎; E: 盛开的花; F: 成熟的角果。A: root; B: stem leaf; C: rosette leaf; D: floral, young pot and stem; E: blooming flower; F: mature pod.
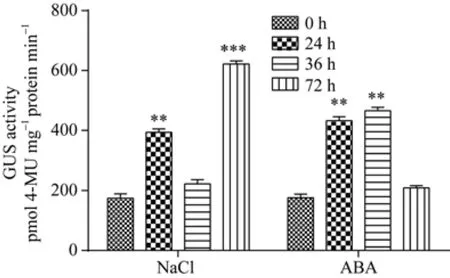
图6 不同胁迫条件下转基因拟南芥的GUS定量分析Fig. 6 Quantification of GUS activity in transgenic Arabidopsis under different stresses**和 ***分别表示胁迫处理与对照(0 h)在0.01和0.001概率水平的差异显著性(t检验)。** and *** indicate significant between the stress treatment and the control (0 h) at the 0.01 and 0.001 probability level, respectively(t-test).
3 讨论
启动子是调控基因表达的顺式作用元件, 提供特定的转录因子结合位点以调控基因的表达, 是决定基因的表达特异性的重要元件。因此, 启动子特性与基因功能密切相关。例如, 胁迫诱导型启动子可以在胁迫条件下通过提高基因的表达量来增强植物的耐逆性。拟南芥rd29A启动子受低温、干旱、ABA和高盐等诱导, 遭受胁迫时显著上调其下游基因的表达, 提高了转基因植株的耐逆性[28];rd29A驱动GmDREB3的转基因拟南芥比CaMV 35S驱动GmDREB3的转基因拟南芥有更强的耐逆性[29]。Nakashima等[30]发现, LIP9是受高盐等非生物胁迫诱导的启动子, LIP9驱动OsNAC6转基因水稻不仅耐旱性得到提高, 而且对产量没有负面影响。Jeong等[31]报道, 在根特异表达启动子RCc3的调控下, 水稻耐逆相关基因OsNAC10显著增强了转基因水稻对干旱、高盐和低温的耐受性, 改善了转基因水稻在干旱条件下的田间农艺性状, 其田间产量增加25%~42%。检测发现, RCc3:OsNAC10转基因植株的根部直径是组成型过表达植株的 1.25倍, 推测增粗的根系有利于植株吸收水分和养料, 从而提高了转基因植物的耐旱性和干旱条件下的产量。可见, 启动子类型与转基因植物的胁迫条件下的生长密切相关, 而胁迫诱导表达型启动子有利于改善植物耐逆性及提高产量。
小麦盐诱导基因TaSC具有耐盐功能, 受盐诱导上调表达[20]。本研究利用TAIL-PCR和电子克隆的方法, 从耐盐小麦RH8706-49基因组DNA中克隆到TaSC的ATG上游1419 bp的启动子序列, 命名为ProTaSC (图2-C, 图3)。鉴于中国春小麦的基因组序列已经公布, 应用 phytozome网站(https://phytozome.jgi.doe.gov/pz/portal.html)查询到Rh8706-49中的TaSCcDNA连同启动子ProTaSC序列(命名为 ProTaSC+TaSC)在中国春基因组中的同源序列(命名为 Traes_5DL_50BA3A)。对二者的比对分析表明, ProT-aSC+TaSC序列与中国春基因组中的4266 bp的连续序列能够匹配, 其中启动子区域的同源性达到 91.16%; 二者外显子部分的序列同源性达到 95.14%, 其中的编码框(coding sequence, CDS)序列的同源性达到97.65% (附图1),说明Traes_5DL_50BA3A与ProTaSC+TaSC的确是同源序列, 进一步证明了ProTaSC启动子序列与TaSC基因是连锁的。比对结果还表明, ProTaSC与中国春的序列存在一定差异, 有其序列特异性, 值得进一步研究其表达调控功能。
利用 5'末端逐渐缺失实验对 ProTaSC进行表达活性分析表明, ProTaSC的全长序列具有启动功能和组织表达特异性, 基本启动活性中心位于起始密码子 ATG上游–681位到–343位核苷酸区间内(图4和图5)。该启动子中包含2个非生物胁迫响应元件——ABRE和MBS, 分别是与ABA和MYB蛋白相关的顺式作用元件。ABA和MYB类转录因子家族参与调控植物的多种耐盐、耐旱过程[32-34],推测ProTaSC启动子具有非生物胁迫应答功能。本研究发现在盐和ABA处理的不同时间点, GUS表达量均有显著上调(图6), 表明ProTaSC是受NaCl和ABA显著诱导表达的功能序列。本研究结果为深入解析TaSC基因调控小麦耐盐性机制提供了依据。
[1] Munns R, Gillihan M. Salinity tolerance of crops—what is the cost?New Phytol, 2015, 208: 668–673
[2] Zhang X K, Zhou Q H, Cao J H, Yu B J. Differential Cl–/salt tolerance and NaCl-induced alternations of tissue and cellular ion fluxes inGlycine max,Glycine sojaand their hybrid seedlings.J Agron Crop Sci, 2011, 197: 329–339
[3] Hasegawa P M. Sodium (Na+) homeostasis and salt tolerance of plants.Environ Exp Bot, 2013, 92: 19–31
[4] Munns R, James R A, Läuchli A. Approaches to increasing the salt tolerance of wheat and other cereals.J Exp Bot,2006, 57:1025–1043
[5] Bohnert H J, Nelson D E, Jensen R G. Adaptations to environmental stresses.Plant Cell, 1995, 7: 1099–1111
[6] Holmström K, Mäntylä E, Welin B, Mandal A, Palva E. Drought tolerance in tobacco.Nature, 1996, 379: 683–684
[7] Becker D, Hoth S, Ache P, Wenkel S, Roelfsema M R G, Meyerhoff O, Hartung W, Hedrich R. Regulation of the ABA-sensitiveArabidopsispotassium channel gene GORK in response to water stress.FEBS Lett, 2003, 554: 119–126
[8] Yamaguchi-Shinozaki K, Shinozaki K. Improving plant drought,salt, and freezing tolerance by gene transfer of a single stressinducible transcription factor. In: Novartis Foundation Symposium, 2001, pp 176–186; 186–189
[9] Zhou M L, Ma J T, Zhao Y M, Wei Y H, Tang Y X, Wu Y M. Improvement of drought and salt tolerance inArabidopsisandLotus corniculatusby overexpression of a novel DREB transcription factor fromPopulus euphratica.Gene, 2012, 506: 10–17
[10] Xiao F H, Xue G P. Analysis of the promoter activity of late embryogenesis abundant protein genes in barley seedlings under conditions of water deficit.Plant Cell Rep, 2001, 20: 667–673
[11]Guo L, Yu Y H, Xia X L,Yin W L. Identification and functional characterization of the promoter of the calcium sensor geneCBL1from the xerophyteAmmopiptanthus mongolicus.BMC Plant Biol, 2010, 10: 1–16
[12] Liu Y G, Whittier F R. Thermal asymmetric interlaced PCR: automatable amplification and sequencing of insert end fragments from P1 and YAC clones for chromosome walking.Genomics,1995, 25: 674–681
[13]张晓, 张锐, 孙国清, 史计, 孟志刚, 周焘, 侯思宇, 梁成真,于源华, 郭三堆. 优化的反向PCR结合TAIL-PCR法克隆棉花线粒体atpA双拷贝基因及其侧翼序列. 生物工程学报,2012,28: 104–115 Zhang X, Zhang R, Sun G Q, Shi J, Meng Z G, Zhou T, Hou S Y,Liang C Z, Yu Y H, Guo S D. High efficiency genome walking method for flanking sequences of cotton mitochondrial double-copyatpAgene based on optimized inverse PCR and TAIL-PCR.Biotechnol Bull, 2012, 28: 104–115 (in Chinese with English abstract)
[14] Chen X L, Song R T, Yu M Y, Sui J M, Wang J S, Qiao L X.Cloning and functional analysis of the chitinase gene promoter in peanut.Genet Mol Res, 2015, 14: 12710–12722
[15] Luo K, Zhang G F, Deng W, Luo F T, Qiu K, Pei Y. Functional characterization of a cotton late embryogenesis-abundantD113gene promoter in transgenic tobacco.Plant Cell Rep, 2008, 27:707–717
[16] Wu A M, Ling C, Liu J Y. Isolation of a cotton reversibly glycosylated polypeptide (GhRGP1) promoter and its expression activity in transgenic tobacco.J Plant Physiol,2006, 163: 426–435
[17] Wu A, Liu J G. Isolation of the promoter of a cotton beta- galactosidase gene (GhGal1) and its expression in transgenic tobacco plants.Sci China C Life Sci, 2006, 49: 105–114
[18] Xu J Y, Cao J J, Cao D M, Zhao T T, Huang X, Zhang P Q, Luan F X. Flanking sequence determination and event-specific detection of genetically modified wheat B73-6-1.Acta Biochim BiophysSin, 2013, 45: 416–421
[19] Hettiarachchi G H, Yadav V, Reddy M K, Chattopadhyay S,Sopory S K. Light-mediated regulation defines a minimal promoter region of TOP2.Nucl Acids Res, 2003, 31: 5256–5265
[20] Huang X, Zhang Y, Jiao B, Chen G P, Huang S H, Guo F, Shen Y Z, Huang Z J, Zhao B C. Overexpression of the wheat salt tolerance-related geneTaSCenhances salt tolerance inArabidopsis,J Exp Bot, 2012, 63: 5463–5473
[21]沈银柱, 刘植义, 何聪芬, 黄占景, 孟庆昌, 柏峰, 马闻师, 赵松山, 陆莉, 张焕英. 诱发小麦花药愈伤组织及其再生植株抗盐性变异的研究. 遗传, 1997, 45(6): 7–11 Shen Y Z, Liu Z Y, He C F, Huang Z J, Meng Q C, Bai F, Ma W S,Zhao S S, Lu L, Zhang H Y. A Study of salt-resistant variations induced in anther calli and regenerated plants in wheat.Hereditas(Beijing), 1997, 45(6): 7–11 (in Chinese with English abstract)
[22] Allen G C, Flores-Vergara M A, Krasynanski S, Kumar S,Thompson W F. A modified protocol for rapid DNA isolation from plant tissues using cetyltrimethylammonium bromide.Nat Protoc. 2006, 1: 2320–2325
[23] Liu Y G, Chen Y L. High-efficiency thermal asymmetric interlaced PCR for amplification of unknown flanking sequences.Biotechniques. 2007, 43: 649–656
[24] An G. Binary Ti vectors for plant transformation and promoter analysis.Methods Enzymol, 1987, 153: 292–305
[25] Clough S J, Bent A F. Floral dip: a simplified method forAgrobacterium-mediated transformation ofArabidopsis thaliana.Plant J, 1998, 16: 735–743
[26] Jefferson R A, Kavanagh T A, Bevan M W. GUS fusions:β-glucuronidase as a sensitive and versatile gene fusion marker in higher plants.EMBO J, 1987, 6: 3901–3907
[27] Bradford M M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein dye binding.Anal Biochem, 1976, 72: 248–254
[28] Kasuga M, Miura S, Shinozaki K, Yamaguchi-Shinozaki K. A combination of the ArabidopsisDREB1Agene and stressinducible rd29A promoter improved drought- and low-temperature stress tolerance in tobacco by gene transfer.Plant Cell & Physiol,2004, 45: 346–350
[29] Chen M, Xu Z S, Xia L Q, Li L C, Cheng X G, Dong J H, Wang Q Y, Ma Y Z. Cold-induced modulation and functional analyses of the DRE-binding transcription factor gene,GmDREB3, in soybean (Glycine maxL.).J Exp Bot,2009, 60: 121–135
[30] Nakashima K, Tran L S, Van Nguyen D, Fujita M, Maruyama K,Todaka D, Ito Y, Hayashi N, Shinozaki K, Yamaguchi-Shinozaki K. Functional analysis of a NAC-type transcription factorOs-NAC6involved in abiotic and biotic stress-responsive gene expression in rice.Plant J, 2007, 51: 617–630
[31] Jeong J S, Kim Y S, Baek K H, Jung H, Ha S H, Choi Y D, Kim M, Reuzeau C, Kim J K. Root-specific expression ofOsNAC10improves drought tolerance and grain yield in rice under field drought conditions.Plant Physiol, 2010, 153: 185–197
[32] Klingler J P, Batelli G, Zhu J K. ABA receptors: the START of a new paradigm in phytohormone signalling.J Exp Bot, 2010, 61:3199–3210
[33] Fujii H, Verslues P E, Zhu J K.Arabidopsisdecuple mutant reveals the importance of SnRK2 kinases in osmotic stress responsesin vivo.Proc Natl Acad Sci USA, 2011, 108: 1717–1722
[34] Wang R, Jing W, Xiao L Y, Jin Y K, Shen L K, Zhang W H. The rice high-affinity potassium transporter1;1 is involved in salt tolerance and regulated by an MYB-type transcription factor 1.Plant Physiol, 2015, 168: 1076–1090

附图1 耐盐小麦RH8706-49中ProTaSC+TaSC序列与中国春(Triticum aestivum L.)基因组序列的比对Supplementary Fig. 1 Alignment of ProTaSC+TaSC sequence from salt-tolerant wheat RH8706-49 and genomic DNA sequence from Chinese spring (Triticum aestivum L.)ProTaSC+TaSC: 小麦RH8706-49中TaSC基因的cDNA序列(557 bp)及其启动子(1419 bp), 红色线条标注TaSC的起始密码子(ATG)和终止密码子(TAA); Traes_5DL_50BA3A: 中国春的4266 bp的基因组DNA序列, 其中含内含子2290 bp。ProTaSC+TaSC: the cDNA sequence (557 bp) and its promoter sequence (1419 bp) of TaSC gene in RH8706-49. Red bars show the start(ATG) and stop codon (TAA) of TaSC. Traes_5DL_50BA3A: the 4266 bp contiguous genomic DNA sequence of Chinese spring, containing 2290 bp of introns.
