转染反义微小RNA⁃155对人皮肤鳞状细胞癌细胞株A431生长的影响
2018-04-09时磊魏明师广勇刘佳龚艳杰陈河涛梁颖红涂玲
时磊 魏明 师广勇 刘佳 龚艳杰 陈河涛 梁颖红 涂玲
450052郑州大学第五附属医院药学部(时磊),检验科(魏明、师广勇、刘佳、龚艳杰、陈河涛、梁颖红、涂玲)
皮肤鳞状细胞癌(鳞癌)具有高度的侵袭能力,可能与紫外线照射损伤、免疫抑制及其他皮肤疾病有关[1]。微小RNA(microRNA,miRNA)是一类长21~25个核苷酸的非编码小分子RNA,它通过介导靶mRNA的降解或阻遏转录后翻译,在细胞生长、凋亡、分化等诸多生物学过程中起调控作用。miRNA的异常表达常与肿瘤的发生、发展密切相关,发挥癌基因或抑癌基因样作用[2⁃3]。近年研究表明,多种miRNA与皮肤肿瘤的生长、转移、侵袭等密切相关,如 miRNA⁃203[4]、miRNA⁃125b[5]、miRNA⁃128[6]等。但有关miRNA⁃155在皮肤鳞癌中的作用研究还较少。我们通过反义技术抑制miRNA⁃155在人皮肤鳞癌细胞株A431中的表达,观察miRNA⁃155对A431细胞增殖、凋亡、侵袭及迁移的影响。
材料与方法
一、材料与仪器
A431细胞株购自中国科学院典型培养物保藏委员会细胞库。脂质体LipofectamineTM2000试剂盒、RNA提取试剂Trizol(德国Invitrogen公司),噻唑蓝(MTT)、二甲基亚砜(DMSO)(美国Sigma公司);高糖改良Eagle培养基(DMEM)及胎牛血清(美国Gibco公司);脂质体试剂异硫氰酸荧光素标记的膜联蛋白Ⅴ(annexinⅤ⁃FITC)细胞凋亡检测试剂盒(美国BD公司)。反义抑制序列miRNA⁃155(5′⁃ACCCCUAUCACGAUUAGCAUUAA⁃3′)、无义对照序列miRNA⁃155(5′⁃CAGUACUUUUGUGUAGUACA A⁃3′)、Hairpin⁃itTMmiRNA荧光定量PCR试剂盒、U6核内小RNA(snRNA)荧光定量校正试剂盒(上海吉玛制药技术有限公司)。酶联免疫检测仪、FACScalibur流式细胞仪(美国BD公司),插入式细胞培养皿(Transwell)(美国康宁公司)。
二、细胞培养与转染
用含10%胎牛血清的DMEM培养基于37℃、5%CO2细胞培养箱中培养A431细胞。将对数生长期细胞以2×105个/孔接种于6孔板中,细胞铺板在2 ml含血清、不含抗生素的培养基中,24 h内细胞融合达50%~70%时,按实验要求分组:空白对照组,仅加含有Lipofectamine2000的DMEM;无义序列组,加入含Lipofectamine2000的DMEM和无义序列miRNA⁃155;反义序列组,加入含Lipofectamine2000的DMEM和反义序列miRNA⁃155。转染体系500 μl,转染终浓度100 nmol/L。孵育6 h后,换为含10%胎牛血清的RPMI 1640培养液继续培养。
三、实时定量聚合酶链反应(qPCR)检测转染后miRNA⁃155的表达
转染48 h后,使用Trizol试剂提取各组细胞总RNA,取0.5 μg总RNA,反转录合成cDNA后扩增,以U6 snRNA作为内参。miRNA⁃155正向引物序列:5′⁃GCCGTTAATGCTAATCGTGAT⁃3′,反向引物序列:5′⁃TGGGTTCATTTTCTGGGTCTT⁃3′,探针序列:FAM⁃TCTATTCCATTGGTCTACT⁃MGB;U6 snRNA正向引物序列:5′⁃CTCGCTTCGGCAGCAC⁃3′,反向引物序列:5′⁃AACGCTTCACGAATTTGCGT⁃3′,探针序列:FAM⁃AACGATACAGAGAAGATT⁃MGB。反转录特异性引物为各自的反向引物。均由上海捷瑞生物工程有限公司合成。PCR体系25 μl,反应条件:95℃预变性10 min,95℃变性10 s,60℃退火、延伸60 s,共40个循环。每个样本均测3次。采用LightCycler 480荧光定量PCR仪(美国Roche公司)检测各样本的域值循环数(Ct值),以2⁃△△Ct反映目的基因表达量,△Ct=Ct反义序列组或无义序列组-Ct内参,△△Ct=△Ct反义序列组-△Ct无义序列组。
四、MTT实验检测细胞增殖活性
将转染后培养24 h的细胞消化离心(转速3 000 r/min,离心半径13.0 cm),按每孔2 × 103个(200 μl)接种于5块96孔板,每组接种6个复孔,常规培养并隔天换液,于接种后24、48、72、96、120 h时各取1块96孔板,每孔加入20 μl MTT溶液(5 g/L),继续培养4 h,弃孔内液体,每孔加入150 μl DMSO,振荡10 min,用酶联免疫检测仪在490 nm波长处测定各孔吸光度(A值),细胞存活率(%)=(A反义序列组/A空白对照组)× 100%。
五、流式细胞仪检测细胞周期
将转染后培养48 h的细胞消化离心(转速3 000 r/min,离心半径13.0 cm),用预冷的磷酸盐缓冲液(PBS)重悬、洗涤2次,加入预冷的75%乙醇3 ml,4℃过夜,离心收集细胞,PBS洗涤后加入400 μl溴化乙锭染色液(50 mg/L)和100 μl核糖核酸酶A(RNase A)(100 mg/L),4 ℃避光孵育30 min,过300目尼龙网,于流式细胞仪中488 nm激发波长处检测处于G0/G1、S、G2/M期的细胞比例,计算细胞增殖指数(PI)。PI=(S+G2/M)/(G0/G1+S+G2/M)×100%。
六、流式细胞仪检测细胞凋亡
收集转染后培养48 h的细胞,每组设3个平行孔。按照说明书清洗、消化、离心细胞后,每管加入结合缓冲液悬浮细胞200 μl(细胞密度为1×106/ml)。避光下每管加入3 μl annexinⅤ⁃FITC,再加入溴化乙锭(其中1个对照孔两者都不加,1个对照孔只加溴化乙锭)2 μl,混匀,室温避光孵育15 min,置于冰上。流式细胞仪检测细胞凋亡,激发光波长488 nm,发射光波长530 nm。凋亡率=凋亡细胞数/(凋亡细胞数+正常细胞数)×100%。
七、Transwell法检测细胞迁移与侵袭收集转染后培养48 h的A431细胞,制备单细胞悬液,密度为1×106/ml。取3个细胞小室放入24孔板中,每个细胞小室加入对应分组稀释后的细胞悬液100 μl(即每孔细胞数为1×105个),细胞小室的下室加入500 μl含10%胎牛血清的DMEM/高糖完全培养基,置于细胞培养箱培养10 h,PBS浸洗,80%乙醇固定,伊红染色;取出小室放入含有3 ml PBS的6孔板中,于倒置荧光显微镜下随机取6个视野照相、计数(200倍),取平均值为细胞迁移数。另外将-20℃保存Matrigel胶置于4℃冰箱过夜,融化稀释后加入细胞小室上室,覆盖整个聚碳酯膜,重复以上步骤检测细胞侵袭数。
八、统计学方法
用SPSS 17.0软件进行统计学分析。计量资料用x±s表示,多组间比较采用单因素方差分析,组间多重比较采用LSD⁃t检验,以P<0.05为差异有统计学意义。

表1 A431细胞转染反义微小RNA⁃155后不同时间点细胞存活率(x±s)
结 果
一、转染后A431细胞miRNA⁃155表达水平
转染后反义序列组、无义序列组、空白对照组细胞miRNA⁃155表达水平分别为0.28±0.18、0.98±0.02、1.00± 0.01,3组间差异有统计学意义(F=634.57,P<0.001),反义序列组表达水平低于空白对照组和无义序列组,t值分别为6.371、5.863,均P<0.05;而空白对照组和无义序列组差异无统计学意义(t=0.261,P> 0.05)。
二、转染后A431细胞存活率变化
转染miRNA⁃155后孵育24~120 h,反义序列组细胞存活率呈降低趋势。见表1。转染后24、48 h时,3组细胞存活率差异均无统计学意义(P>0.05);转染后72、96、120 h时,反义序列组细胞存活率均低于空白对照组和无义序列组(均P<0.05)。
三、转染后A431细胞周期变化
转染后3组间细胞G0/G1期、S期细胞比例差异均有统计学意义(P<0.05),G2/M期细胞比例差异无统计学意义(P>0.05),细胞PI值差异有统计学意义(P<0.05)。见图1、表2。反义序列组G0/G1期细胞比例高于空白对照组、无义序列组(t值分别为3.261、3.185,P< 0.05),S期细胞比例低于空白对照组、无义序列组(t值分别为7.259、6.983,P<0.05),PI值低于空白对照组、无义序列组(t值分别为5.396、5.273,P< 0.05)。
四、转染后A431细胞凋亡率变化
反义序列组、空白对照组和无义序列组A431细胞的凋亡率分别为13.16%±0.62%、7.02%±0.80%和6.74%±1.26%,3组间差异有统计学意义(F=56.27,P<0.05),反义序列组高于空白对照组和无义序列组,t值分别为5.672、5.713,P< 0.05。见图2。
五、转染后A431细胞迁移和侵袭数变化
转染后各组细胞迁移数和侵袭数差异有统计学意义(P<0.05);反义序列组细胞迁移数和侵袭数均低于空白对照组(t值分别为7.231、7.203,均P<0.05)和无义序列组(t值分别为5.687、5.612,均P<0.05);而空白对照组和无义序列组相比,差异无统计学意义(t值分别为0.218、0.227,均P> 0.05),见表2。

图1 流式细胞仪检测转染反义微小RNA⁃155后A431细胞周期变化

表2 转染反义微小RNA⁃155后各组A431细胞周期、增殖指数及迁移数和侵袭数(x±s)
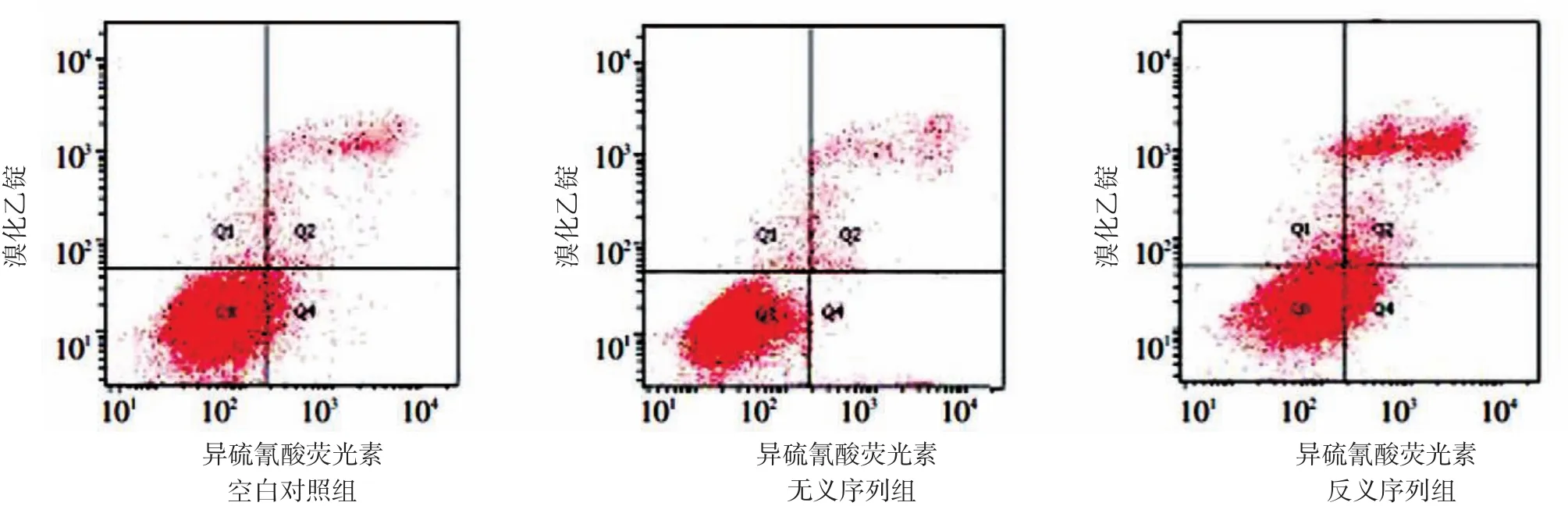
图2 流式细胞仪检测转染反义微小RNA⁃155对A431细胞凋亡的影响 反义序列组细胞凋亡比例高于空白对照组和无义序列组
讨 论
近来miRNA在恶性肿瘤发生、发展中的作用机制以及靶向miRNA基因治疗已经成为肿瘤学研究热点[7]。有研究表明,miRNA⁃155在多种肿瘤中表达上调,发挥促癌作用[8⁃12];也有研究证实,miRNA⁃155可通过减少激活诱导胞嘧啶脱氨酶产生的癌基因易位而发挥抑癌作用[13⁃14]。
我们利用反义技术抑制miRNA⁃155在A431细胞中的表达,观察细胞增殖能力及凋亡的变化。结果显示,转染反义miRNA⁃155后,A431细胞中miRNA⁃155的表达明显降低。肿瘤失控性生长的主要分子机制为细胞周期紊乱,导致肿瘤细胞增殖过多和凋亡过少。本研究中,A431细胞转染反义miRNA⁃155后,G0/G1期细胞增多,S期细胞减少,细胞的增殖能力降低,凋亡率升高,提示抑制miRNA⁃155的表达可使细胞周期阻滞,细胞增殖能力受限,促进A431细胞的凋亡。
一般认为,肿瘤细胞远处转移需要具备迁移能力及穿透周围组织进入血液或淋巴循环的能力[15⁃16]。本研究中,转染反义miRNA⁃155后穿过小室的A431细胞数明显减少,提示反义miRNA⁃155能抑制A431细胞的侵袭及迁移。
综上,转染反义序列miRNA⁃155后A431细胞miRNA⁃155表达水平降低,细胞迁移、侵袭及增殖受到抑制,提示miRNA⁃155在调控皮肤鳞癌生物学行为中发挥了重要作用。
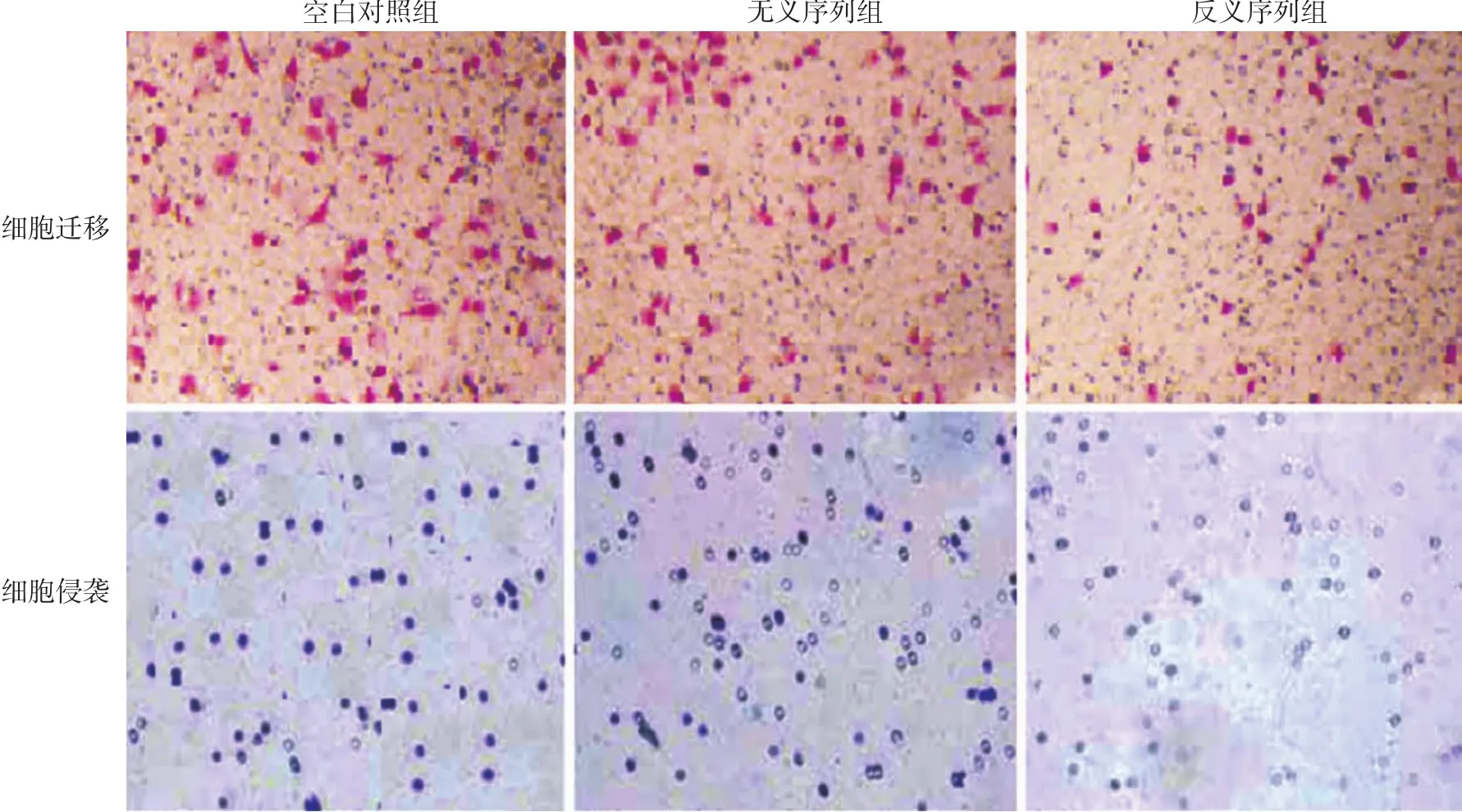
图3 显微镜下观察Transwell法检测转染反义微小RNA-155后A431细胞的迁移与侵袭(×200)反义序列组细胞迁移数、侵袭数低于空白对照组、无义序列组
[1]陈凯,常东方,段绍坤,等.miR⁃31对人皮肤鳞状细胞癌生长的影响及作用机制的研究[J].中国肿瘤临床,2014,41(9):555⁃559.doi:10.3969/j.issn.1000⁃8179.20131787.
[2]Bertero T,Bourget⁃Ponzio I,Puissant A,et al.Tumor suppressor function of miR⁃483⁃3p on squamous cell carcinomas due to its pro ⁃apoptotic properties[J].Cell Cycle,2013,12(14):2183 ⁃2193.doi:10.4161/cc.25330.
[3]Varol N,Konac E,Gurocak OS,et al.The realm of microRNAs in cancers[J].Mol Biol Rep,2011,38(2):1079⁃1089.doi:10.1007/s11033⁃010⁃0205⁃0.
[4]Sonkoly E,Lovén J,Xu N,et al.MicroRNA⁃203 functions as a tumor suppressor in basal cell carcinoma[J].Oncogenesis,2012,1:e3.doi:10.1038/oncsis.2012.3.
[5]Xu N,Zhang L,Meisgen F,et al.MicroRNA⁃125b down⁃regulates matrix metallopeptidase 13 and inhibits cutaneous squamous cell carcinoma cell proliferation,migration,and invasion[J].J Biol Chem,2012,287 (35):29899⁃29908.doi:10.1074/jbc.M112.391243.
[6]Zhou M,Liu W,Ma S,et al.A novel onco⁃miR⁃365 induces cutaneous squamous cell carcinoma[J].Carcinogenesis,2013,34(7):1653⁃1659.doi:10.1093/carcin/bgt097.
[7]Mateus C.Cutaneous squamous cell carcinoma[J].Rev Prat,2014,64(1):45⁃52.
[8]Giordano S,Columbano A.MicroRNAs:new tools for diagnosis,prognosis,andtherapyinhepatocellularcarcinoma? [J].Hepatology,2013,57(2):840⁃847.doi:10.1002/hep.26095.
[9]Nakagawa R,Leyland R,Meyer⁃Hermann M,et al.MicroRNA⁃155 controls affinity⁃based selection by protecting c⁃MYC+B cells from apoptosis[J].J Clin Invest,2016,126(1):377⁃388.doi:10.1172/JCI82914.
[10]Lerner C,Wemmert S,Bochen F,et al.Characterization of miR⁃146a and miR⁃155 in blood,tissue and cell lines of head and neck squamous cell carcinoma patients and their impact on cell proliferation and migration[J].J Cancer Res Clin Oncol,2016,142(4):757⁃766.doi:10.1007/s00432⁃015⁃2087⁃y.
[11]Hou L,Chen J,Zheng Y,et al.Critical role of miR⁃155/FoxO1/ROS axis in the regulation of non⁃small cell lung carcinomas[J].Tumour Biol,2016,37(4):5185⁃5192.doi:10.1007/s13277⁃015⁃4335⁃9.
[12]Li T,Yang J,Lv X,et al.miR⁃155 regulates the proliferation and cell cycle of colorectal carcinoma cells by targeting E2F2[J].Biotechnol Lett,2014,36(9):1743⁃1752.doi:10.1007/s10529⁃014⁃1540⁃3.
[13]Tam W,Dahlberg JE.miR⁃155/BIC as an oncogenic microRNA[J].Genes Chromosomes Cancer,2006,45(2):211 ⁃212.doi:10.1002/gcc.20282.
[14]Sun S,Sun P,Wang C,et al.Downregulation of microRNA⁃155 accelerates cell growth and invasion by targeting c⁃myc in human gastric carcinoma cells[J].Oncol Rep,2014,32(3):951⁃956.doi:10.3892/or.2014.3288.
[15]Kong XM,Zhang GH,Huo YK,et al.MicroRNA⁃140⁃3p inhibits proliferation,migration and invasion of lung cancer cells by targeting ATP6AP2[J].Int J Clin Exp Pathol,2015,8(10):12845⁃12852.
[16]Yao W,Liu Y,Zhang Z,et al.ALX1 promotes migration and invasion of lung cancer cells through increasing snail expression[J].Int J Clin Exp Pathol,2015,8(10):12129⁃12139.
