郑州地区2种菊花病毒CP基因的克隆与序列分析
2018-04-08刘瑞霞王默霏郑州师范学院生物工程研究所河南郑州450044郑州师范学院生命科学学院河南郑州450044山东省东营区实验学校山东东营57000
梁 芳,刘瑞霞,张 燕,王默霏,崔 波*,李 静(.郑州师范学院 生物工程研究所,河南 郑州 450044; .郑州师范学院 生命科学学院,河南 郑州 450044; .山东省东营区实验学校,山东 东营 57000)
菊花是我国传统名花、世界四大切花之一,具有广泛的观赏、药用及食用保健价值,在我国广泛栽培。生产上菊花的繁殖多采用无性扦插分株等方式,并且常常连作,非常利于病毒病的积累和传播,继而导致品质退化严重。由病毒病引起的菊花种质退化、品质和产量下降,严重制约了菊花产业的健康持续发展,已成为菊花生产上的一个突出问题[1-2]。迄今为止,国内外报道的菊花病毒已有20多种,其中侵染最广泛、危害最严重的为番茄不孕病毒(Tomato aspermy virus,TAV)和菊花B病毒(Chrysanthemum virus B,CVB)[3-4]。二者常常复合感染,在菊花的商业生产中,二者复合感染率为20.9%,CVB单独感染率达到97.2%[5]。
20世纪50年代,Noordam发现了CVB,该病毒属于香石竹潜隐花叶病毒属(Carlavirus),为单链正义RNA病毒,呈长形棒状结构,大小约685 nm×12 nm,含有8 990个碱基,6个开放阅读框,具有Poly A尾巴[6]。CVB与康乃馨潜隐病毒、马铃薯 Y 病毒在血清学上有近缘关系[7]。其寄主范围非常狭窄,仅局限于菊科和少部分烟草[8]。可通过汁液、插条、桃蚜等传播,发生范围广泛,全世界范围内只要有菊花生长的地方都会发生,是花卉产业的潜在威胁[9]。据报道,在我国云南地区CVB为栽培菊花的最主要病毒,田间情况下多与TAV等病毒复合侵染[10]。受CVB感染的植株常见症状有叶片斑纹、脉明,小花畸形出现褐色坏死条纹,严重时会产生褐色枯斑[1,11]。
TAV最早由Blencowe等[12]于1949年在番茄中发现。该病毒属于黄瓜花叶病毒属(Cucumovirus),为正义单链RNA病毒,病毒粒子为球形颗粒,直径约29 nm[5]。其基因组由3条正单链RNA组成,可表达产生5个蛋白质产物[13]。该病毒可感染万寿菊、旱金莲、菊花、紫菀、辣椒、莴苣、烟草、龙葵等植物,体外寿命7~11 d,失活温度65~70 ℃[14]。我国在20世纪90年代初分离鉴定了侵染菊花的TAV,根据报道,TAV变异株多,寄主范围广泛,受侵染植株表现为花朵畸形有斑点、褪色、变小,严重危害产量与品质[15-16]。史步军等[17]从安徽省药菊产区分离出TAV,这是危害药菊的病毒在我国的首次报道,其可通过桃蚜、萝卜蚜和菊小长管蚜以非持久性方式传播。
本研究以郑州地区表现明显发病症状的菊花植株为试验对象,通过RT-PCR的方法扩增TAV及CVB的外壳蛋白(CP)基因序列,并通过生物信息学分析明确其进化关系及分类地位,以期为该类病毒病的防治及菊花的抗病毒育种奠定理论基础。
1 材料和方法
1.1 试验材料
2016年6月在郑州好日子园艺有限公司菊花大棚采集叶片有花斑、卷曲、褪绿的疑似感病菊花叶片,采集后编号并立即投入液氮中速冻,置于-80 ℃超低温冰箱保存。
1.2 总RNA的提取及反转录
采用Trizol法提取植物总RNA,利用Quawell Q5000微量分光光度计测定所提RNA的OD260/280及OD260/230值,并通过琼脂糖凝胶电泳检测完整性。以总RNA为模板,用RevertAid First Strand cDNA Synthesis Kit合成cDNA第一链。
1.3 引物设计
根据NCBI上已报道的CVB和TAV的CP基因序列,利用DNAMAN和Primer Premier 5.0软件分别设计1对CP基因片段的特异性引物(表1)。

表1 引物序列与参数
1.4 病毒CP基因的PCR扩增
以反转录合成的cDNA第一链为PCR反应的模板,以合成的CVB-F/R及TAV-F/R分别为引物,扩增病毒CP基因序列。PCR反应体系为:10×PCR Buffer 2.0 μL、dNTP 1.6 μL、引物各1.0 μL、cDNA 0.8 μL、rTaqDNA聚合酶0.2 μL,以ddH2O补充至20.0 μL。PCR扩增程序为:95 ℃预变性4 min;95 ℃变性40 s,55 ℃退火40 s,72 ℃延伸40 s,35个循环;最后72 ℃延伸10 min。PCR产物用1%琼脂糖凝胶电泳检测,将正确片段切下并进行回收。
1.5 TA克隆与测序
采用琼脂糖凝胶回收试剂盒纯化目的片段,再次经电泳检测正确后与pGEM-T easy克隆载体连接,连接体系如下:缓冲液5.0 μL、pGEM-T easy1.0 μL、cDNA 3.0 μL、T4 DNA Ligase 1.0 μL,混合后置于4 ℃过夜。将连接体系转化大肠杆菌DH5α感受态细胞,涂平板后37 ℃过夜培养,经过蓝白斑筛选和2次菌落PCR鉴定均为阳性的菌液,送至上海英骏生物技术公司测序。
1.6 序列比对与进化树分析
将测得的2种病毒郑州分离物CP基因序列在NCBI网站上进行BLAST检索,用DNAMAN软件将测得的郑州菊花2种病毒分离物CP基因序列与已登录的序列进行核苷酸序列多重比对, 运用MEGA5.0软件以NJ法构建系统进化树。
2 结果与分析
2.1 菊花病毒郑州分离物CP基因的扩增
将采集的8份菊花样品用Trizol法提取总RNA,以反转录得到的cDNA第一链为模板分别进行PCR,对TAV和CVB的CP基因片段进行扩增。PCR扩增产物经琼脂糖凝胶电泳检测,结果如图1所示。在5号和8号样品中出现近500 bp的目的条带,其中8号样品目的条带很亮(图1A);在1号、4号和6号样品中出现约500 bp的目的条带,其中4号样品目的条带很亮,另外8号样品似有目的条带但不清晰(图1B)。扩增得到的目的条带均与预计大小相符,表明待测样品可能感染TAV或CVB。
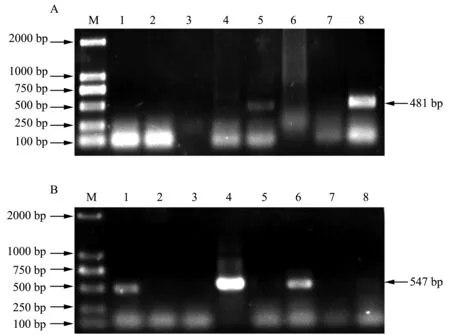
M:DL2000 DNA Marker;1—8:采集的菊花样品图1 菊花TAV(A)和CVB(B)病毒CP基因片段PCR产物
2.2 菊花病毒郑州分离物CP基因序列分析
2.2.1TAV郑州分离物通过测序,得到1条TAV的CP基因序列,长481 bp,经BLAST分析(图2),表明得到的序列即为TAV的CP基因部分片段,将此TAV郑州分离物CP基因序列登录到GenBank上,登录号为KU873003。将该基因序列与其他17种不同TAV分离物的CP基因进行核苷酸及氨基酸序列比对,结果如表2所示,核苷酸序列同源性在91%~99%,氨基酸序列同源性在86%~100%。郑州分离物KU873003核苷酸序列与印度分离物AJ580841、EF123059及EU888909,英格兰分离物D01015,伊朗分离物JQ403612,中国焦作分离物KP019200和西安分离物KP019204的同源性均为99%,而氨基酸序列则与英格兰分离物D01015、印度分离物EF123058及EU888909和伊朗分离物JQ403612完全相同。
2.2.2CVB郑州分离物测序得到3条菊花CVB病毒的CP基因序列(图3),经BLAST分析发现,其与其他已登录的CVBCP基因序列同源性最高达到99%,因此将其登录到GenBank上,登录号分别为KU873004、KU873005和KU873006。采用DNAMAN对CVB郑州分离物的CP基因序列进行比对,3条序列同源性为99.4%,有10个碱基的差异(图3)。在NCBI上搜索已经报道的CVB的CP基因序列并下载,这些分离物均来自于菊花,通过与郑州分离物KU873004比对分析表明,核苷酸序列同源性在76%~99%,氨基酸序列同源性在77%~99%(表3)。郑州分离物KU873004与其他地区分离物比较,其核苷酸序列与印度分离物AJ871367同源性最高,为93%,氨基酸序列与印度分离物AJ871367和AJ581993同源性最高,为98%。

图2 TAV郑州分离物CP基因序列比对

表2 TAV郑州分离物CP基因与其他地区分离物比对结果

图3 CVB郑州分离物CP基因序列比对
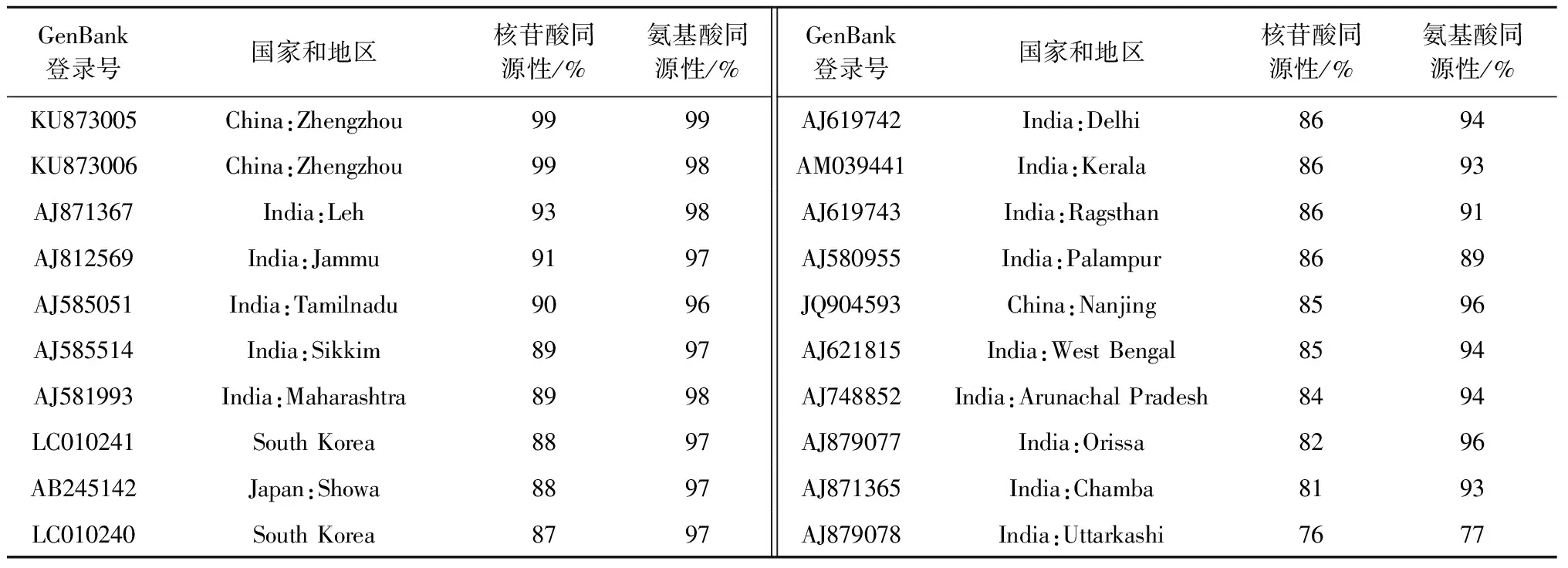
表3 CVB郑州分离物KU873004 CP基因与其他地区分离物比对结果
2.3 菊花病毒郑州分离物的系统进化分析
2.3.1TAV郑州分离物将菊花TAV郑州分离物KU873003与其他36种分离物的CP基因序列进行进化树分析,以明确TAV郑州分离物的分类地位(图4)。从图4可以看出,这些分离物可分为3个簇,本试验得到的郑州分离物KU873003与其他的31种分离物构成Ⅰ簇。在Ⅰ簇中既包括来自中国、印度和伊朗的分离物,也有来自英国、匈牙利和澳大利亚的分离物,因此该病毒的分布十分广泛。AJ550020、AJ586134、AJ965492和AM055754四种分离物构成Ⅱ簇,而AM055592单独构成Ⅲ簇,这些分离物均来自于印度。郑州分离物KU873003与北京分离物KP019201、黑龙江分离物KP019202、河南分离物KP019200和山西分离物KP019204的亲缘关系最近,与印度分离物AM055757属于同一小分支。
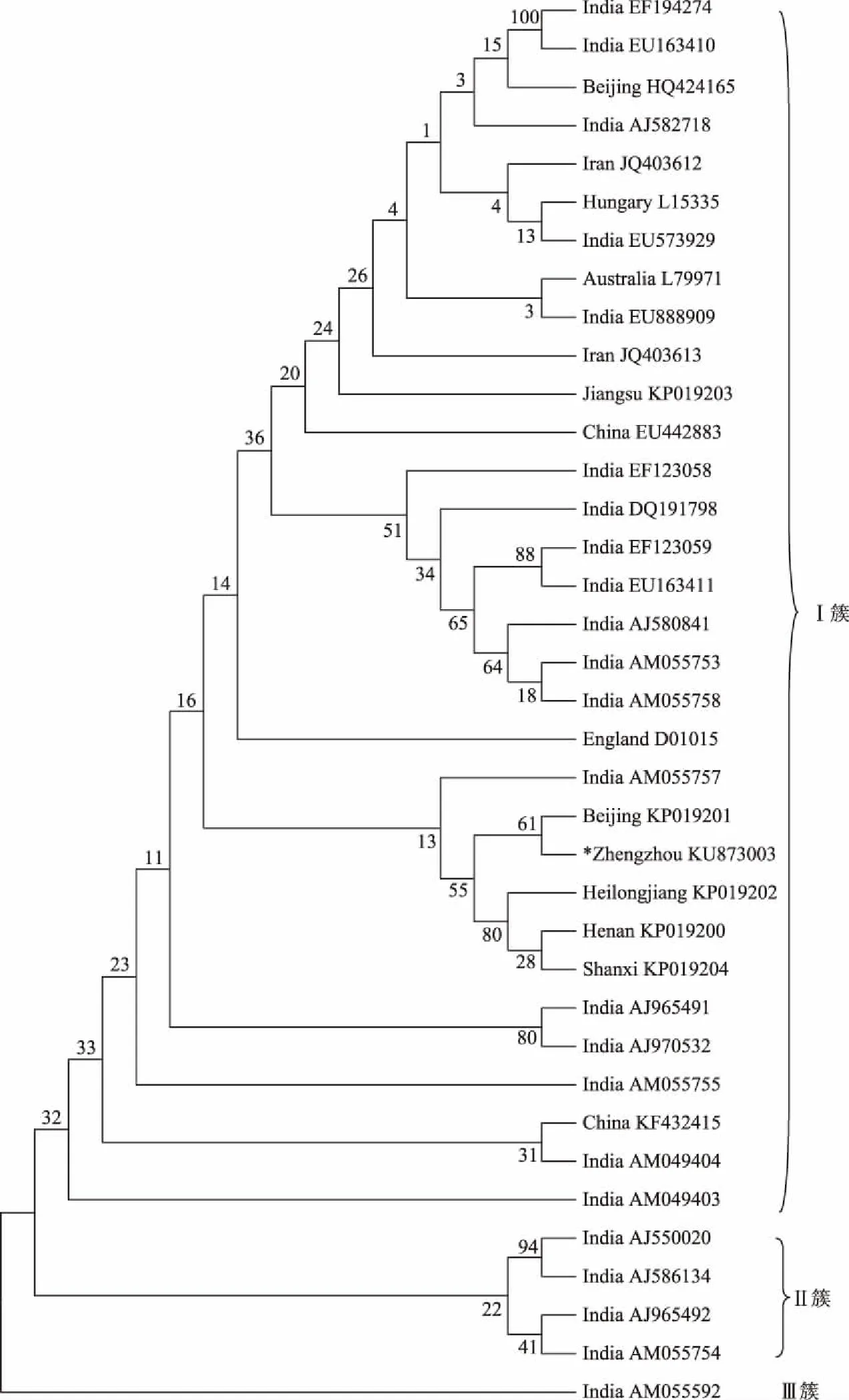
图4 不同地区TAV分离物CP基因核苷酸序列进化分析
2.3.2CVB郑州分离物将菊花CVB郑州分离物与NCBI上已登录的部分CVB的CP基因序列构建进化树,如图5所示,可分为3个簇。CVB郑州分离物与印度分离物AJ812569及AJ871367的亲缘关系最近,与韩国分离物LC010241、LC010240,日本分离物AB245142,印度分离物AJ585051、AJ581993、AJ585514和波兰分离物KJ489421属于同一个小分支,与南京分离物JQ904593和印度分离物AJ748852共同属于Ⅰ簇,其他印度分离物分别属于Ⅱ簇和Ⅲ簇。
3 结论与讨论
据相关文献报道,侵染菊花的主要病毒除了TAV和CVB,还有烟草花叶病毒(TMV)和黄瓜花叶病毒(CMV)[10,18]。本试验除了对TAV和CVB采用RT-PCR扩增其CP基因的方法进行检测外,也对TMV和CMV分别设计了2对引物,就8个菊花样品进行检测,但均未扩增出目的条带。在5号和8号样品中检测到TAV,1号、4号和6号样品中检测到CVB,8号中扩增出的目的条带很淡,可能新感染CVB,因而病毒含量很低;2号、3号和7号样品叶片出现花叶、皱褶等症状,却未扩增出目的条带,有可能并未感染病毒而是其他原因所致,也有可能是感染了其他病毒,将在后期试验中研究。
通过对菊花病毒郑州分离物进行序列分析发现,TAV郑州分离物KU873003与来自印度、英格兰、伊朗、焦作、西安的分离物核苷酸序列同源性很高,为99%,其氨基酸序列与英格兰、伊朗及印度部分分离物完全相同。从寄主方面来看,这些分离物大多来自于菊花,而印度EU573929和EU888909分离物来自于唐菖蒲(Gladiolussp.),表明该病毒无明显的寄主特异性,这与TAV的特性相符。据报道,TAV的寄主范围十分广泛,人工接种可侵染24科100多种植物,在世界种植菊花的国家均有分布[19]。从感染TAV的地区分布上看,大部分来自于印度、中国,也有部分来自英国和伊朗,因此该病毒的分布十分广泛。利用进化树来研究病毒的进化关系,已得到人们的广泛认可。本研究发现,郑州分离物KU873003与北京分离物KP019201、黑龙江分离物KP019202、河南分离物KP019200和山西分离物KP019204的亲缘关系最近,与印度分离物AM055757属于同一小分支。分析表明,TAVCP基因的进化与其地理位置的分布具有一定的关联,中国的这些分离物可能由印度分离物AM055757进化而来,然后朝着不同的方向进化。
从郑州菊花上分离出3个CVB分离物,扩增的3条序列均为547 bp,有10个碱基的差异。将NCBI上已报道的部分CVB的CP基因序列与郑州分离物KU873004进行比对分析,核苷酸序列同源性在76%~99%,氨基酸序列同源性在77%~99%。这些分离物均来自于菊花,取自不同的国家和地区。从构建的进化树可以看出,CVB郑州的3个分离物与印度分离物AJ812569及AJ871367的亲缘关系最近,与来自南京、韩国、日本、波兰的分离物同属于Ⅰ簇,郑州分离物可能由印度分离物AJ812569或AJ871367进化而来。说明菊花上CVB很有可能朝着不同的方向发展,使该病毒的防治更加棘手。
参考文献:
[1]刘辉辉,沈学根,毛碧增.菊花病毒病及其防治对策[J].药物生物技术,2015,22(1):91-94.
[2]赵喜亭,朱玉婷,田莹,等.番茄不孕病毒(TAV)脱除对怀白菊叶绿素含量及氮代谢相关指标的影响[J].南方农业学报,2016,47(7):1095-1099.
[3]Raj S K,Kumar S,Choudhari S.Identification ofTomatoaspermyvirusas the cause of yellow mosaic and flower deformation of chrysanthemums in India[J].Australasian Plant Disease Notes,2007,2(1):1-2.
[4]Ohkawa A,Yamada M,Sayama H,etal.Complete nucleotide sequence of a Japanese isolate ofChrysanthemumvirusB(genusCarlavirus)[J].Archives of Virology,2007,152(12):2253-2258.
[5]Chung B N,Choi G S,Choi Y M.Identification of tomato aspermy virus(TAV) and chrysanthemum virus B (CVB) fromDendranthemaindicumin Korea [J].The Plant Pathology Journal,1999,15(2):119-123.
[6]Noordam D.Virus diseases ofChrysanthemumindicumin the Netherlands [J].Tijdschrift Over Plantenziekten,1952,58:121-190.
[7]蒋细旺,饱满珠,薛东,等.我国菊花病害种类及危害特征[J].甘肃农业大学学报,2002,37(2):185-189.
[8]Singh L,Hallan V,Martin D P,etal.Genomic sequence analysis of four new chrysanthemum virus B isolates:Evidence of RNA recombination[J].Archives of Virology,2012,157:531-537.
[9]Singh L,Hallan V,Jabeen N,etal.Coat protein gene diversity among Chrysanthemum virus B isolates from India [J].Archives of Virology,2007,152(2):405-413.
[10]吴红芝,孔宝华,陈海如,等.昆明地区菊花病毒病的调查与鉴定[J].云南农业大学学报,2002,17(1):24-27.
[11]Mitiouchkina T Y,Firsov A P,Dolgov S V.Transgenic chrysanthemum plants transformed with the gene encoding for the virus B coat protein [J].Russian Agricultural Sciences,2013,39(5/6):431-434.
[12]Blencowe J W,Caldwell J.Aspermy—A new virus disease of the tomato[J].Annals of Applied Biololy,1949,36(3):320-326.
[13]Raj S K,Kumar S,Choudhari S,etal.Biological and molecular characterization of three isolates ofTomatoaspermyvirusinfecting chrysanthemums in India[J].Journal of Phytopathology,2009,157:117-125.
[14]Procter C H.Studies on tomato aspermy virus fromChrysanthemumindicumL.in New Zealand[J].New Zealand Journal of Agricultural Research,1975,18(4):387-390.
[15]孔宝华,李德葆.侵染菊花的番茄不孕病毒[J].浙江农业大学学报,1990,16(1):55-60.
[16]Verma N,Kumar K,Kulshrestha S,etal.Molecular studies onTomatoaspermyvirusisolates infecting chrysanthemum [J].Archives of Phytopathology and Plant Protection,2009,42(2):99-111.
[17]史步军,李彦勇,张成林,等.侵染药菊的一株病毒分离物的鉴定[J].植物病理学报,1993,23(3):231-236.
[18]赵霜.菊花病毒脱除与检测技术研究[D].北京:北京林业大学,2013:1-2.
[19]雷强,李旻,丁元明,等.菊花上2种病毒的检测及其部分外壳蛋白基因序列分析[J].华中农业大学学报,2012,31(6):704-709.
