高通量基因测序技术在自然流产胎儿绒毛染色体的核型分析
2018-03-30方有燕贺小进陈大蔚周一茹曹云霞魏兆莲
方有燕,吴 欢,贺小进,陈大蔚,周一茹,周 平,曹云霞,魏兆莲
近年,辅助生殖技术(assisted reproductive technology,ART)在个体化控制性超促排卵方案的应用、实验室技术以及仪器设备方面均取得了长足的进步,ART术后的临床妊娠率有了很大的提高,但高流产率仍是一个亟待解决的问题。常见的流产病因包括遗传因素、免疫因素、内分泌、生殖道感染、生殖道畸形及高凝血状态等,其中遗传因素尤其染色体异常被认为是最常见早期流产诱发因素[1]。绒毛细胞来源于胚胎外胚层细胞,具有与胚胎组织相同的遗传特性,因此可通过检测自然流产组织染色体核型,了解胚胎细胞的遗传性状,分析胚胎停育的可能病因。既往关于胚停绒毛染色体分析多采用传统的染色体显带和荧光原位杂交技术,且关于不同病因ART流产组织染色体分析鲜有报道。该研究采用高通量测序技术分析364例自然流产绒毛组织染色体异常的发生率、核型分布及与孕妇年龄、胎儿性别、妊娠方式、不同ART病因之间的关系,报道如下。
1 材料与方法
1.1病例资料选择2014年9月~2016年12月于安徽医科大学第一附属医院妇产科生殖中心行胎儿绒毛染色体检测的364例患者作为研究对象,年龄19~44(30.440±4.768)岁,孕5~15周,其中13~15周仅1例。根据妊娠方式,将其分为自然妊娠组及ART组。364例患者中自然妊娠246例,ART术后妊娠118例,B超提示胚胎停止发育(未见胚芽或未见心管搏动),在取得患者知情同意的前提下采集胚胎绒毛标本并行高通量测序。
1.2高通量测序法检测绒毛染色体核型① 提取基因组DNA,Wizard试剂盒进行产物纯化,Qubit法检测纯化后的产物;② 构建基因组DNA小片段文库:采用Life Tech的ION XPRESS LIBRARY KIT进行DNA片段化,并构建DNA小片段文库;③ 按照PGM测序操作指南(Ion PGM-200 Sequencing Kit V2 only)进行高通量测序及信息分析。
2 结果
2.1样本总体检测结果364例绒毛组织中检出染色体数目及结构异常178例,异常率为48.90%(178/364),其中自然妊娠组130例,异常率52.85%(130/246),显著高于ART组的40.68%(48/118)(P<0.05)。异常核型以常染色体三体型为主,占异常核型的65.17%;其次为染色体结构微缺失/微重复,占15.73%;性染色体单体占6.74%;三倍体占5.06%;嵌合体占3.93%。除1、6、11号染色体外非整倍体的发生涉及所有染色体,其中16号染色体发生率最高,占异常染色体的21.35%;22号染色体次之,占11.24%;常染色体的非整倍体多为三体型,性染色体的非整倍体则以单体型为多,仅有1例常染色体单体,核型为45,XY,-21。见表1。
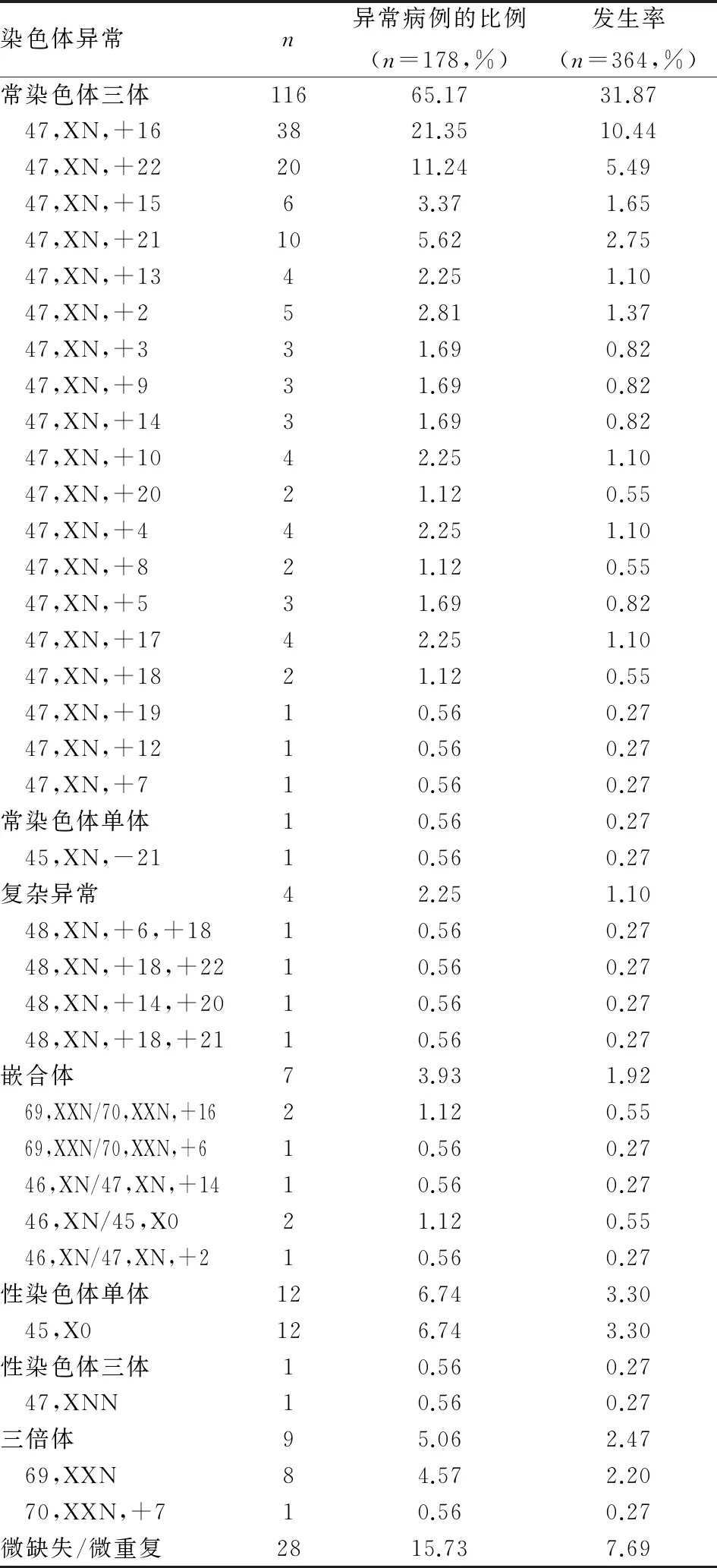
表1 早期自然流产绒毛组织染色体异常核型及分布
2.2年龄与染色体异常ART组≥35岁流产绒毛组织染色体异常发生率虽高于<35岁患者,但差异无统计学意义;自然妊娠组≥35岁流产绒毛组织染色体异常发生率显著高于<35岁患者(P<0.05)。见表2。
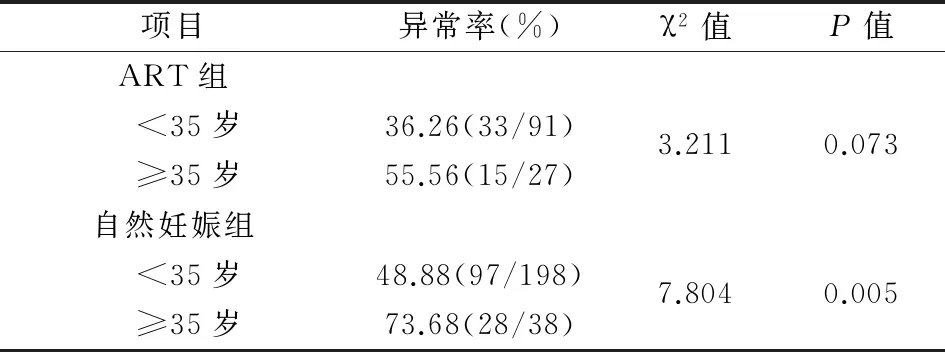
表2 年龄与染色体异常
2.3ART助孕病因与胚胎染色体异常ART组流产胎儿绒毛染色体异常率为40.68%(48/118),以复发性流产最常见,占60.00%,其次为男方因素,占48.39%。见表3。经χ2检验,差异无统计学意义,尚不能表明ART组不同病因染色体异常率不同。

表3 ART助孕妊娠组不同病因分布
2.4性别与胚胎染色体异常核型分布362例胚停组织中(其中2例因测试时要求保密,具体性别不详,予以排除,其中1例核型异常),男胚166例,女胚196例,男胚异常核型占总异常核型的53.11%(94/177),显著高于女胚的46.89%(83/177)(P=0.007)。在177例异常染色体核型中,常染色体三体型占65.64%(116/177),其中男胚63例,女胚53例。按照染色体非显带核型对三体核型进行分组,将其分为A、B、C、D、E、F、G 7组,详见表4,男、女胚均主要分布于E组(38.10%vs37.74%) 和G组(22.22%vs30.19%);男胚A组三体核型显著多于女胚(P<0.05),其余各组在性别间分布差异均无统计学意义(P>0.05),见表4。
3 讨论
绒毛滋养层细胞是受精卵有丝分裂的衍生物,因其与胎儿组织具有相同的遗传性状,故可以作为检测早期胎儿染色体异常的材料,对自然流产的遗传学病因做出诊断,为胚停夫妇再次妊娠的遗传咨询提供信息。

表4 不同性别胚胎染色体非显带核型分组分布
自然流产的发生率约占所有妊娠的15%~20%,是妊娠期常见的并发症[2]。自然流产多发生于妊娠早期,导致流产的病因众多,其中染色体异常占50%~70%[3],是导致胚胎早期流产的主要原因,因为染色体数目异常的胚胎遗传缺陷更大,导致遗传物质不平衡,致死性更高[3-4]。有研究者认为常染色体三体以16号染色体三体最常见,约占1/3,且具有高致死性[2,5]。本研究结果显示,染色体数目异常发生率为41.21%(150/364),其中16号染色体三体发生率为32.76%(38/116),与相关研究[2,5]报道一致。45,X0是人类唯一能生存的单体综合征[6],本研究中,45,X0核型有12例,占所有异常染色体核型的6.74%,该异常核型的形成除与减数分裂时性染色体不分离导致一条染色体缺失有关外,多由于父系性染色体丢失所致。通过对116例常染色体三体核型按染色体非显带核型图进行分组,显示男胚、女胚均主要分布在E、G两组,这可能与常染色体三体主要为16、22号染色体异常,而16、22号染色体分别位于E、G两组有关。本研究中男胚A组三体核型显著多于女胚,因样本量小,尚不足以得出结论,仍需扩大样本量进一步研究证实。
本研究中自然妊娠组年龄小于ART组,但绒毛染色体异常的发生率却高于ART组(52.85%vs40.68%),可能与行ART患者的配子及胚胎经体外优化、筛选后染色体异常率降低有关。有研究[5,7]显示,20~24岁、40~44岁、45岁及以上流产率分别为10%、51%、75%,≥39岁年龄组约占总人数的25%,21三体综合征的发生率占1.44%,显著高于35~38岁年龄组的0.65%。这些研究[5,7]表明,随着年龄增长,不仅流产率增加,染色体异常率也明显增高。本研究结果也证实,自然妊娠组≥35岁患者染色体异常率高达73.68%,显著高于<35岁的48.99%。ART组不同年龄间染色体异常率差异无统计学意义,可能与ART组≥35岁例数偏少有关,尚需扩大样本量进一步验证。
有研究[8]表明,不明原因性妊娠丢失与精子染色质的高频组装异常有关。本研究ART组中男方因素主要包括少、弱、畸形精子症、单/双侧输精管缺如、梗阻性无精子症等,其染色体异常发生率高达48.39%,可能与该组患者DNA碎片指数较高有关,García-Ferreyra et al[9]研究表明,DNA碎片指数较高组(37.10±17.61)胚胎染色体非整倍体率显著高于DNA碎片指数较低组(21.3±13.48,17.4±10.79)(73.9%vs61.1%vs59.1%),由于精子核染色质的去凝作用以及随后形成的雄原核对正常受精和胚胎发育都至关重要,精子DNA碎片指数高表明染色质损伤,可降低受精能力和胚胎发育潜能,最终导致妊娠丢失[8]。因此对ART后的早期流产应充分考虑到男方精子染色体异常因素,术前应常规检查男方精子染色体有无异常,以避免或减少早期妊娠丢失。
本研究中多囊卵巢综合征(polycystic ovar syndrome,PCOS)患者绒毛染色体异常发生率为30.00%,明显低于复发性流产和男方因素的患者,与Wang et al[10]报道的结果一致(胚停组织染色体非整倍体发生率为28.1%),结果表明,虽然PCOS患者早期流产率高达50%,但染色体非整倍性可能并不是其早期流产的主要原因,而母体内分泌因素(如高黄体生成素水平、高雄激素水平、孕酮产物异常、胰岛素抵抗)及其导致的子宫内膜容受性降低可能是PCOS患者早期流产的主要风险因素。子宫内膜异位症患者染色体异常发生率与PCOS患者相近(30.77%),可能与子宫内膜异位病灶的炎症反应、细胞因子的异常改变影响卵子成熟和质量以及子宫内膜容受性降低有关[11]。
早期自然流产除表现为染色体异常率增高,还存在性别特异性胚胎淘汰现象[12]。本研究362例绒毛组织中,胚停女胚占比高于男胚(196vs166),但男胚异常核型占比高于女胚(53.11%vs46.89%),与其他研究者的报道[13-14]一致,具体机制尚不清楚。
此外,本研究发现染色体结构异常占有较高的比重(7.69%),占所有异常染色体核型的15.73%,因此应重视早期流产染色体的结果异常。这可能与高通量测序技术相比于传统染色体核型分析和荧光原位杂交技术具有更高的检测深度和准确度有关。高通量测序技术可检测到0.5 Mbp以上的染色体片段缺失,能发现更多的染色体片段微缺失和微重复等结构异常。
综上所述,自然流产胚胎染色体异常核型率高于ART组;年龄与胚胎染色体异常率增高有关;染色体数目异常是导致胚胎早期流产的主要原因,16号染色体三体是最常见的胚胎染色体数目异常;胚停女胚占比高于男胚,但男胚异常核型占比高于女胚;应重视不明原因妊娠丢失的男方精子染色体异常。高通量测序技术相较于传统染色体核型分析和荧光原位杂交技术具有更高的检测深度和准确度,能发现更多的染色体结构和数目异常,对阐明早期流产的遗传学病因及再次妊娠的遗传咨询具有重要意义。
[1] Ouyang Y,Tan Y,Yi Y,et al. Correlation between chromosomal distribution and embryonic findings on ultrasound in early pregnancy loss after IVF-embryo transfer[J]. Hum Reprod,2016,31(10):2212-8.
[2] An N,Li L L,Zhang X Y,et al. Result and pedigree analysis of spontaneously abortion villus chromosome detecting by FISH[J]. Genet Mol Res,2015,14 (4):16662-6.
[3] Sugiura-Ogasawara M,Ozaki Y,Katano K,et al. Abnormal embryonic karyotype is the most frequent cause of recurrent miscarriage[J]. Hum Reprod,2012,27(8):2297-303.
[4] 郭 楠,刘雨生,周桂香,等. 129例自然妊娠与ART术后妊娠早期流产胎儿绒毛染色体核型分析[J]. 安徽医科大学学报,2013,48(2):163-6.
[5] Tur-Torres M H,Garrido-Gimenez C,Alijotas-Reig J. Genetics of recurrent miscarriage and fetal loss[J]. Best Pract Res Clin Obstet Gynaecol,2017,42:11-25.
[6] 刘长明,丛 林,袁 静,等. 荧光原位杂交技术联合染色体核型分析诊断胎儿Turner综合征[J]. 安徽医科大学学报,2011,46 (4):389-91.
[7] Zhu Y,Lu S,Bian X,et al. A multicenter study of fetal chromosomal abnormalities in Chinese women of advanced maternal age[J]. Taiwan J Obstet Gynecol,2016,55(3):379-84.
[8] Leach M,Aitken R J,Sacks G. Sperm DNA fragmentation abnormalities in men from couples with a history of recurrent miscarriage[J]. Aust N Z J Obstet Gynaecol,2015,55(4):379-83.
[9] García-Ferreyra J,Luna D,Villegas L,et al. High aneuploidy rates observed in embryos derived from donated oocytes are related to male aging and high percentages of sperm DNA fragmentation[J]. Clin Med Insights Reprod Health,2015,9:21-7.
[10] Wang Q,Luo L,Lei Q,et al. Low aneuploidy rate in early pregnancy loss abortuses from patients with polycystic ovary syndrome[J]. Reprod Biomed Online,2016,33(1):85-92.
[11] Pallacks C,Hirchenhain J,Krüssel J S,et al. Endometriosis doubles odds for miscarriage in patients undergoing IVF or ICSI[J]. Eur J Obstet Gynecol Reprod Biol,2017,213:33-8.
[12] Lebedev I. Molecular cytogenetics of recurrent missed abortions[J]. Indian J Med Res,2006,124 (1):9-10.
[13] Russo R,Sessa A M,Fumo R,et al. Chromosomal anomalies in early spontaneous abortions:interphase FISH analysis on 855 FFPE first trimester abortions[J]. Prenat Diagn,2016,36(2):186-91.
