三疣梭子蟹线粒体基因组SNP在增殖放流家系识别中的应用*
2018-03-29李志辉赖晓芳张庆起阎斌伦
赵 莲 李志辉 张 培 薛 蓓 赖晓芳张庆起 高 焕 阎斌伦①
(1. 淮海工学院海洋生命与水产学院 江苏省海洋生物技术重点实验室 连云港 222005;2. 江苏省海洋生物产业技术协同创新中心 连云港 222005; 3. 江苏省农业种质资源保护与利用平台南京 210014;4. 连云港赣榆佳信水产开发有限公司 连云港 222005)
三疣梭子蟹(Portunus trituberculatus)是我国重要的海捕经济蟹类,但随着过度捕捞,野生海捕资源迅速下降,年产量已经由1996年的5.9万t迅速下降至2001年的1.1万t (Yanget al, 2005)。为了恢复野生海捕资源,我国近年来展开了大量的增殖放流活动,2004~2006年先后在长江口、杭州湾海域共计人工增殖放流三疣梭子蟹苗种1576亿只(沈新强等, 2007);截至 2013年,山东省累计放流二期幼蟹 18.85亿只(庞景贵等, 2009; Zhanget al, 2009; 杨爽等, 2014),有效缓解了三疣梭子蟹种质资源的衰退。随着放流活动的持续开展及不断扩大,如何评估放流效果的问题也摆在了我们面前。作为甲壳类生物,三疣梭子蟹的一生需要多次蜕壳,这就大大限制了很多外部和内部物理形态标记的应用,因此,需要探索新型放流标志技术。同时,在放流过程中,放流个体对野生资源的种质有何影响,即对自然状态下三疣梭子蟹的遗传多样性和群体遗传结构有何影响,也亟待解答。而解决以上这些问题的最佳途径是开发三疣梭子蟹增殖放流效果评估的分子标志技术。
随着三疣梭子蟹线粒体全基因组的公布(Yamauchiet al, 2003),基于线粒体DNA的标记技术已广泛用于三疣梭子蟹群体的遗传多样性及遗传结构分析(蔡珊珊, 2015; 王景等, 2015; 杨爽等, 2014)。与核DNA相比,mtDNA具有分子小、结构简单、进化速度快、不同区域进化速度存在差异等特点,是一个相对独立的复制单位(李喜莲, 2007)。借助线粒体DNA具有母性遗传的特点,通过此标记很容易追踪同一母本所产生子代个体的去向,因此,非常适合用作增殖放流标志。这些优点决定了线粒体序列多态性标记技术是最适合三疣梭子蟹增殖放流效果评估的分子标志技术。
作为第三代分子标记的典型代表,单核苷酸多态性标记(Single nucleotide polymorphism, SNP)不仅具有微卫星标记易于分型的优势,而且更容易实现高通量分型,大大提高了分型效率(Beachamet al, 2009)。目前,基于核基因组的 SNP标记已被广泛应用,如遗传图谱的构建(张建勇, 2011)、种质资源遗传分析(Zhanget al, 2011; Batta-Lonaet al, 2011)以及分子标记辅助育种(Fenget al, 2014; Yanget al, 2013; Blancket al, 2013)等,而在线粒体基因组中SNP的开发及其应用研究上还相对较少。本研究通过HRM检测技术对三疣梭子蟹线粒体DNA上的SNP进行分型,以达到快速、准确鉴别不同放流家系的目的,为三疣梭子蟹种质资源的鉴定及标志放流工作的开展提供技术支持。
1 材料与方法
1.1 家系材料及DNA提取
本实验所用家系材料取自江苏连云港赣榆佳信水产开发有限公司,为2016年用于江苏省连云港海州湾增殖放流活动的4个家系,代表80–120万只放流三疣梭子蟹个体。每个家系获得母本个体各1只,子代个体分别取 28只,4个家系的亲本来源于海州湾海捕群体,为野生抱卵亲蟹,亲蟹繁育分别在4个繁育池中进行,子代个体之间无混杂。4个家系编号分别为A、B、C和D。
分别选取上述亲本和相应子代个体的肌肉组织,用液氮进行研磨,使用 Ezup柱式动物基因组 DNA抽提试剂盒[生工生物工程(上海)股份有限公司]提取三疣梭子蟹总基因组 DNA(其中包含线粒体基因组DNA),经Gene Quant Pro核酸定量仪(通用公司,美国)测定基因组DNA浓度,并均一化为20 ng/µl,–20℃保存备用。
1.2 标准品的制备
针对前期已发现的 SNPs(表1),同一对 SNP引物在不同野生个体间进行扩增,并将扩增序列送去测序比对分析,确认个体间所对应的同一片段是否发生突变与未突变情况,进而将同一 SNP扩增引物对应的未突变与突变体,在HRM中进行SNP的分型验证,为已获得的SNP标记在后续应用于4个家系群体的鉴定作标准参照。
1.3 SNP在家系间的HRM分型
利用已获得的标准品(野生/突变体) HRM分型结果,将对应SNP引物在每个家系的28只三疣梭子蟹子代个体中进行PCR反应,扩增体系为35 µl:25.3 µl ddH2O,3.5 µl 10×PCR 缓冲液,2.0 µl 25 mmol/L MgCl2,0.6 µl 10 µmol/L dNTPs,0.6 µl 5 U/µlTaqDNA 聚合酶,各 1.2 µl 10 µmol/L 上下游引物,0.6 µl 20 ng/µl DNA单个体模板。采用温度梯度PCR程序进行引物的扩增:95℃ 5 min;95℃ 30 s,Tm45 s,72℃ 45 s,循环34次;72℃ 10 min;4℃保存。
反应结束后,向35 µl PCR产物中加入高低温内标各 3.5 µl,随后按照“95℃变性 3 min,25℃复性2 min,4℃保存”的程序在PCR仪上进行变性反应。变性结束后,取扩增样10 µl加入96孔反应板中,同时加入1 µl LC Green荧光染料,并以15~20 µl矿物油进行封闭。每个扩增样品设置3个重复。在Light Scanner 96系统上进行HRM分析,反应结束后对熔解曲线进行分析并记录Tm。
2 结果
2.1 标准品的分型情况
通过HRM技术对野生个体及突变个体在22条含有24个SNP位点的同一SNP引物进行扩增,并进行分型分析。结果显示,每一对标准品(野生体/突变体)在同一SNP引物扩增序列中具有明显的差异峰,即Tm值差异明显,详见表1及图1a, b~图4a, b。
2.2 SNP位点在同一家系中的分型情况
从三疣梭子蟹线粒体基因组共得到 24个具有SNP的位点(表1)。利用HRM技术,对这些SNP位点在各个家系中的分型情况进行了研究,结果显示,9个 SNP位点在亲代(母本)及其子代的 28个个体之间具有基本一致的熔解峰,且子代个体间Tm的均一性较好,无明显差异,详见表1。
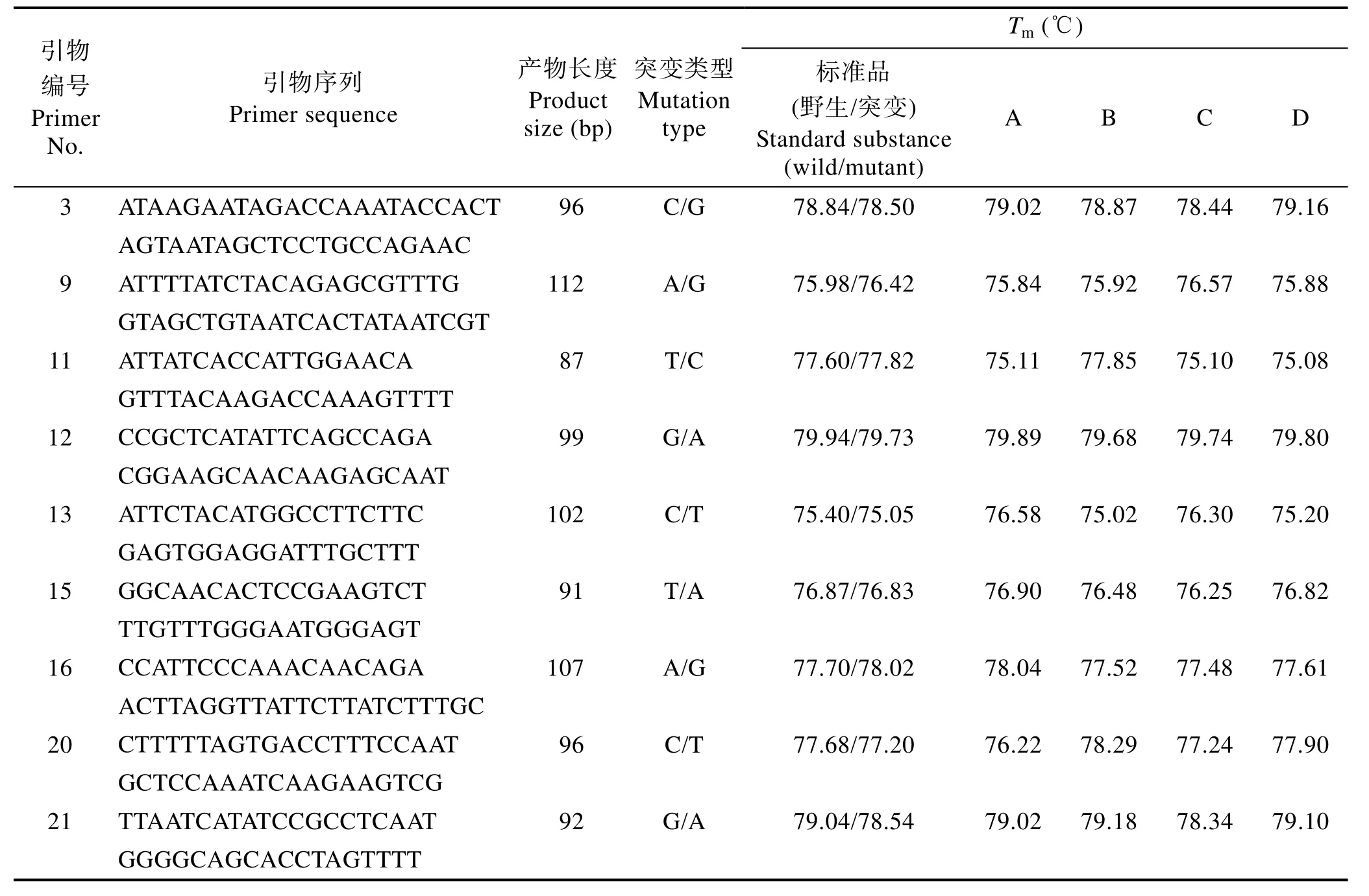
表1 SNPs引物扩增序列在HRM分析中的信息Tab.1 Ampliconic sequences of SNPs used in HRM
2.3 SNP在4个家系中的HRM分型结果
以序列已知的野生型及其突变体作为同一 SNP引物扩增片段在各家系间分析HRM的标准品,通过对含有SNP位点引物的扩增产物进行HRM分析,结果发现,在已知的线粒体基因组 24个 SNP位点中(22对引物),有9个SNPs可以用于三疣梭子蟹4个放流家系的鉴别。对相同序列的同一家系的28个个体扩增片段的熔解峰所对应Tm值取平均值,得出不同变异类型的位点相应扩增产物的Tm值(表1),与标准品中的突变体Tm相比较,各家系都有较为稳定且明显的分型效果。
通过比较各引物所对应的标准品及与家系分型后的Tm差异,均可用于三疣梭子蟹种质鉴定。其中,引物 3、9、12、20、21,这 5对引物可用于鉴别 C家系(以引物9的SNP分型结果为例,见图1);引物11和13可用于鉴别B家系(以引物13的SNP分型结果为例,见图2);引物15分别可用于鉴别D (以引物15的 SNP分型结果为例,见图3)、A家系(以引物16的SNP分型结果为例,见图4)。
图1显示,野生型与突变型的熔解曲线有明显差异(Tm>0.3℃);经过对4个家系与标准品(野生型/突变型)的HRM曲线进行对比分析(图1c和图1d),其中C家系的HRM峰型与突变型(G)的峰型重叠,而其他3个家系(A、B和 D)的熔解曲线峰与野生型(A)的峰型重叠,表明针对引物9扩增的SNP序列在C群体的HRM分型与其他3个家系的HRM分型结果有明显的不同,进而可用于C家系的鉴别。
图2显示,野生型与突变型的熔解曲线有明显差异(Tm>0.3℃);经过对4个家系与标准品(野生型/突变型)的HRM曲线进行对比分析(图1c和图1d),其中,B家系的HRM峰型与突变型(T)的峰型重叠,而其他3个家系(A、C和 D)的熔解曲线峰与野生型(C)的峰型重叠,表明针对引物13扩增的SNP序列在B群体的HRM分型与其他3个家系的HRM分型结果有着明显的不同,进而可用于B家系的鉴别。
图3显示,野生型与突变型的熔解曲线有明显差异(Tm>0.3℃);经过对4个家系与标准品(野生型/突变型)的HRM曲线进行对比分析(图1c和图1d),其中,D家系的HRM峰型与突变型(A)的峰型重叠,而其他3个家系(A、B和 C)的熔解曲线峰与野生型(T)的峰型重叠,表明针对引物15扩增的SNP序列在D群体的HRM分型与其他3个家系的HRM分型结果有着明显的不同,进而可用于D家系的鉴别。
图4显示,野生型与突变型的熔解曲线有明显差异(Tm>0.3℃);经过对4个家系与标准品(野生型/突变型)的HRM曲线进行对比分析(图1c和图1d),其中,A家系的HRM峰型与突变型(G)的峰型重叠,而其他3个家系(B、C和 D)的熔解曲线峰与野生型(A)的峰型重叠,表明针对引物16扩增的SNP序列在D群体的HRM分型与其他3个家系的HRM分型结果有着明显的不同,进而可用于A家系的鉴别。
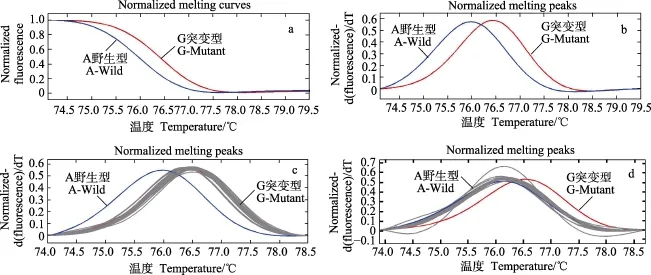
图1 引物9扩增序列在C家系(A/G突变型)中的SNP分型结果Fig.1 SNP typing in the pedigree C for the 9th primer pairs
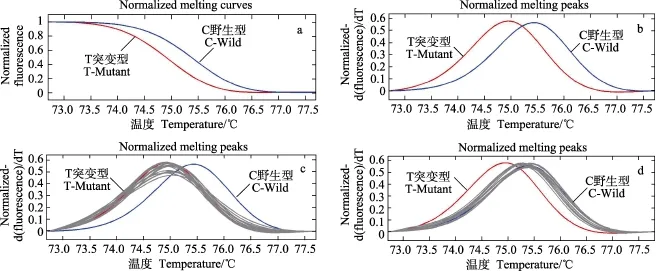
图2 引物13扩增序列在B家系(C/T突变型)中的SNP分型结果Fig.2 SNP typing in the pedigree B for the 13th primer pairs
3 讨论
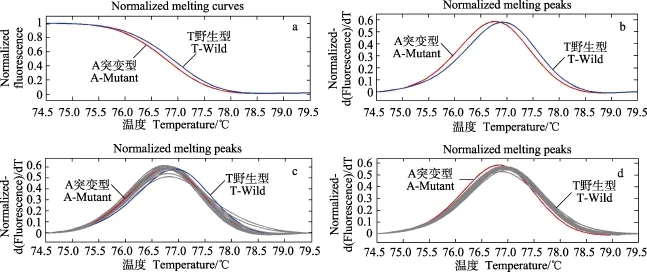
图3 引物15扩增序列在D家系(T/A突变型)中的SNP分型结果Fig.3 SNP typing in the pedigree D for the 15th primer pairs
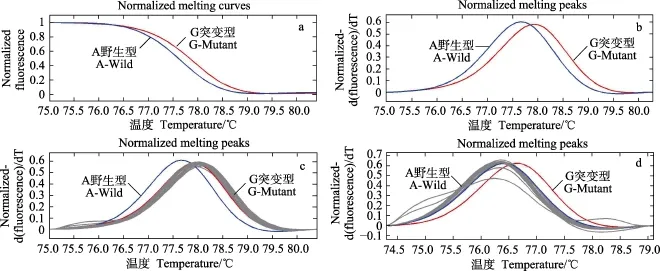
图4 引物16扩增序列在A家系(A/G突变型)中的SNP分型结果Fig.4 SNP typing in the pedigree A for the 16th primer pairs
日本学者在2004年就针对三疣梭子蟹的增殖放流标记技术进行了研究,所使用的技术为金属线码外部标记(Okamoto, 2004);后续又有学者比较分析3种外部标记(体外绑定标记、身体锚定标记及体内植入金属针绑定标记)技术在龙虾(Jasus verreauxi)的标志放流中的应用(Montgomeryet al, 2006),但这些技术应用在三疣梭子蟹这一类甲壳动物身上的效果并不理想,其原因在于:一方面三疣梭子蟹在生长发育过程中需要经历多次蜕壳,影响了外部标记的准确性;另一方面外部标记对被标记对象的身体存在一定的损伤,不仅影响其后续的生长发育,也会降低其在放流过程中的存活率。为此,我们进行了无任何损失的分子标志放流技术研究,而理论上三疣梭子蟹也是非常适合作为研究分子标志放流技术的对象:三疣梭子蟹具有产卵量大的优点,一只母蟹可产20~30万只幼蟹,因此,若知道母本的分子标志特征,则易获得子代个体的遗传特征,这就大大缩减了在大量亲蟹间或群体间寻找特异分子标记的工作量。以海州湾三疣梭子蟹增殖放流活动为例,每年在海州湾区域放流三疣梭子蟹的数量约为 600万只(董志国等, 2013),这只相当于20~30个家系(20~30个亲本)所产生的子代个体数量。本研究获得的在4个家系中存在差异的9个SNP标记中,实际只需要其中的4个就可以把4个放流家系进行准确鉴别,由此初步推断,鉴别20~30个家系所需SNP标记的数量也只需要20~30个,甚至更少。张超(2014)利用HRM技术开发出的7个SNP位点,成功用于中华鳖(Trionyx sinensis)不同品系的鉴别;Zhang等(2009)甚至利用SNPs中的2个突变位点就可将中华绒螯蟹(Eriocheir sinensis)与合浦绒螯蟹(Eriocheir hepuensis)鉴别开来。此外,由于线粒体基因组是单倍型,其上的 SNP标记对鉴别增殖放流个体要比以往利用核基因组中的微卫星和 SNP标记的研究(刘海映等, 2016; 李喜莲等, 2016)更有效率。因此,本研究利用线粒体基因组 SNP标记作为增殖放流的分子标志技术是可行的,简单、高效,如配以HRM技术,则更具有高通量分析的特点。
高分辨率熔解曲线(HRM)分型技术具有高通量、高灵敏度以及特异性强等优点。目前已被成功用于水产生物的多态性检测(吴莹莹, 2013)、种质资源的鉴定(张超, 2014)等方面。因此,根据以上HRM SNP分型技术在种质资源鉴定的基础上,为了增加其分型技术的可信度,本研究利用测序已知分型样品(野生型/突变型)作为标准品,与每个放流家系群体进行HRM比较分析,结果显示,每对 SNP引物的扩增样品所对应的标准品与家系个体间的 PCR产物熔解峰相重叠(图1~图4),且各家系内的Tm均一性也显示良好,表明此方法提高了 SNP分型的准确性,可用于三疣梭子蟹种质资源鉴定方面的研究。
从实验材料的选取上可以看出,本研究并未选取父本材料,这是因为本研究的目的之一就是希望忽略父本遗传材料的背景下,可以对放流群体进行鉴定。其主要原因在于:一方面实际放流过程中三疣梭子蟹是由各个企业自主繁育蟹苗进行放流的,亲本往往来源于野生抱卵蟹,无法准确获得父本信息;另一方面本研究采用的线粒体基因标记在解决增殖放流标记上也不需要父本的遗传信息,这是由线粒体DNA母系遗传的特点决定的。线粒体DNA属于细胞核外遗传物质,线粒体 DNA只通过母亲遗传给子代个体(Spuhle, 1988),因此,该标记非常适用于包括三疣梭子蟹在内的各种生物的增殖放流分子标志研究,只要找出不同家系母本间线粒体DNA上的特异性SNP标记,即可对不同家系群体进行鉴定,而不需要父本材料的相关信息。与核DNA相比,线粒体DNA无重组杂合的现象,不会出现因杂合产生的熔解曲线的双峰(李纪勤等, 2013),因此,较核基因SNP分型相比,线粒体SNP的HRM分型结果更直观明了。
总之,本研究所建立的不同放流家系三疣梭子蟹的线粒体特异性SNP HRM分型方法可有效鉴别用于增殖放流的不同家系群体,此方法弥补了形态、生化等传统标记方法鉴别的不准确性,并为后续的放流标志工作的展开提供技术支持。
Blanck DV, Valenti WC, Freitas PDD,et al. Isolation and characterization of SNPs within HSC70 gene in the freshwater prawnMacrobrachium amazonicum. Conservation Genetics Resources, 2013, 5(3): 631–633
Batta-Lona PG, Bucklin A, Wiebe PH,et al. Population genetic variation of the Southern Ocean krill,Euphausia superba, in the Western Antarctic Peninsula region based on mitochondrial single nucleotide polymorphisms (SNPs).Deep Sea Research PartⅡ: Topical Studies in Oceanography,2011, 58(13): 1652–1661
Beacham TD, Jonsen K, Wallace C. A comparison of stock and individual identification forChinook salmonin British Columbia provided by microsatellites and single-nucleotide polymorphisms. Canadian Journal of Fisheries & Aquatic Sciences, 2009, 67(1): 1274–1290
Cai SS. Evaluation of stock enhancement inPortunus trituberculatusandFennerropenaeus chinensisbased on molecular markers. Master´s Thesis of Ocean University of China, 2015 [蔡珊珊. 基于分子标记的三疣梭子蟹和中国对虾增殖放流效果研究. 中国海洋大学硕士研究生学位论文, 2015]
Dong ZG, Li XY, Zhang QI,et al. Genetic impact of swimming crabPortunus trituberculatusfarming on wild genetic resources in Haizhou Bay. Acta Ecologica Sinica, 2013,33(23): 7332–7339 [董志国, 李晓英, 张庆起, 等. 三疣梭子蟹增养殖过程对野生种群的遗传影响——以海州湾为例. 生态学报, 2013, 33(23): 7332–7339]
Feng N, Ma H, Ma C,et al. Characterization of 40 single nucleotide polymorphism (SNP) via Tm-shift assay in the mud crab (Scylla paramamosain). Molecular Biology Reports, 2014, 41(8): 5467–5471
Li JQ, Bao ZM, Li L,et al. The development and polymorphism analysis of EST-SNP inChlamys(Azumapecten)farreri.Periodical of Ocean University of China (Natural Science Edition), 2013, 43(1): 56–63 [李纪勤, 包振民, 李玲, 等.栉孔扇贝EST-SNP标记开发及多态性分析. 中国海洋大学学报(自然科学版), 2013, 43(1): 56–63]
Liu HY, Lü HB, Cui F,et al.Parental contribution and genetic diversity between broodstock and offsprings inPortunus tribuberculatusreleasing into natural waters. Journal of Fisheries of China, 2016, 35(6): 614–619 [刘海映, 吕海波,崔帆, 等. 放流三疣梭子蟹遗传多样性和贡献率初步研究. 水产科学, 2016, 35(6): 614–619]
Li XL. The polymorphism of mitochondrial DNA ofPortunus trituberculatus.Master´s Thesis of Northwest A&F University, 2007 [李喜莲. 三疣梭子蟹线粒体DNA多态性研究. 西北农林科技大学硕士研究生学位论文, 2007]
Li XL, Yang YJ, Li F,et al. Development of single nucleotide polymorphism markers forMacrobrachium nipponensisusing expressed sequence tags. Journal of Northeast Agricultural University, 2016, 47(2): 67–73 [李喜莲, 杨元杰, 李飞, 等. 日本沼虾 EST-SNP的筛选及多态性检测.东北农业大学学报, 2016, 47(2): 67–73]
Montgomery SS, Brett PA. Tagging eastern rock lobstersJasus verreauxi: Effectiveness of several types of tag. Fisheries Research, 2006, 27(4): 141–152
Okamoto K. Juvenile release and market size recapture of the swimming crabPortunus trituberculatus(Miers) marked with coded wire tags. In: Leber KM, Kitada S, Blankenship HL, Svasand T (eds). Stock enhancement and sea ranching:Development, pitfalls and opportunities (Second Edition).Blackwell, Oxford, 2004, 181–186
Pang JG, Guo JL. Significant enhancement effect on fishing of the Huanghai and Bohai Seas. Hebei Fisheries, 2009(1):34–35 [庞景贵, 郭金龙. 黄渤海放流增殖恢复捕捞渔业效果显著. 河北渔业, 2009(1): 34–35]
Spuhle JN. Evolution of mitochondrial DNA in monkeys, apes,and humans. American Journal of Physical Anthropology,1988, 31(Suppl): 15–48
Shen XQ, Zhou YD. Enhancement releasing and evolution of fishery resources in the Yangtze estuary and Hangzhou Bay sea area. Fishery Modernization, 2007, 34(4): 54–57 [沈新强, 周永东. 长江口、杭州湾域渔业资源增殖放流与效果评估. 渔业现代化, 2007, 34(4): 54–57]
Wang J, Zhang FY, Jang KJ,et al.Genetic diversity ofPortunus trituberculatusbased on the mitochondrial cytochrome oxidase subunit I sequence from the East China Sea. Marine Fisheries, 2015, 37(2): 114–121 [王景, 张凤英, 蒋科技,等. 基于线粒体COⅠ基因序列的三疣梭子蟹东海区群体遗传多样性分析. 海洋渔业, 2015, 37(2): 114–121]
Wu YY, Meng XH, Kong J,et al. Application of unlabeled probe by HRM in development of EST-SNPs inFenneropenaeus chinesis. Progress in Fishery Sciences, 2013, 34(1): 111–118[吴莹莹, 孟宪红, 孔杰, 等. 非标记探针HRM法在中国对虾EST-SNP筛选中的应用. 渔业科学进展, 2013, 34(1):111–118]
Yang F, Xu HT, Dai ZM,et al.Molecular characterization and expression analysis of vitellogenin in the marine crabPortunus trituberculatus. Comparative Biochemistry and Physiology, Part B: Biochemistry & Molecular Biology,2005, 142(4): 456–464
Yamauchi MM, Miya MU, Nishida M. Complete mitochondrial DNA sequence of the swimming crab,Portunus trituberculatus(Crustacea: Decapoda: Brachyura). Gene,2003, 311: 129–135
Yang S, Song N, Zhang XM,et al. Genetic diversity of swimming crab (Portunus trituberculatus) from four broodstock populations in stock enhancement inferred from mitochondrial control region. Journal of Fisheries of China,2014, 38(8): 1089–1096 [杨爽, 宋娜, 张秀梅, 等. 基于线粒体控制区序列的三疣梭子蟹增殖放流亲蟹遗传多样性研究. 水产学报, 2014, 38(8): 1089–1096]
Yang Y, Ye H, Huang H,et al. Expression of Hsp70 in the mud crab,Scylla paramamosainin response to bacterial, osmotic,and thermal stress. Cell Stress and Chaperones, 2013, 18(4):475–482
Zhang C, Zhang HQ, Xu XJ,et al.Identification of different strains ofPelodascus sinensyaby using high resolution melting(HRM) analysis of SNPs in mitochondrial DNA.Oceanologal et Limnologal Sinica, 2014, 45(2): 376–381[张超, 张海琪, 许晓军, 等. 中华鳖(Pelodiscus sinensis)不同品系线粒体SNP的分型与鉴定. 海洋与湖沼, 2014,45(2): 376–381]
Zhang D, Tang B, Ding G,et al. Molecular authentication of the fashionable daintyEriocheir japonica sinensisbased on mitochondrial DNA bar coding. European Food Research and Technology, 2009, 230(1): 173–178
Zhang JY. Development and application of SNP markers in genome ofFenneropenaeus chinensis.Doctoral Dissertation of Ocean University of China, 2011 [张建勇. 中国对虾(Fenneropenaeus chinensis)基因组 SNP标记的开发与应用. 中国海洋大学博士研究生学位论文, 2011]
