两种人牙源性iPSCs的重编程效应及生物学特性比较*
2018-03-28徐静舒戴青原谭小兵
郭 宇,徐静舒,戴青原,谭小兵△
(1.云南省第一人民医院口腔医学中心,昆明 650032;2.昆明医科大学第一附属医院心内科,昆明 650032)
最近研究发现特定转录基因可使终末分化体细胞重新获得干细胞特性,命名为诱导性多潜能干细胞(induced pluripotent stem cells,iPSCs)[1-2],iPSCs无论在形态和功能等方面均与胚胎干细胞(embryonic stem cells,ESCs)非常相似,是近年来干细胞领域的重要里程碑[3]。iPSCs技术可产生患者特异性干细胞用于疾病模型研究和细胞治疗,为个性化治疗打下了基础[4]。
iPSCs要应用于临床治疗就必须保持原有基因完整性,不能携带任何外源性病毒或基因。目前许多方法均可产生iPSCs,包括腺病毒、游离型载体、质粒,合成mRNA、miRNAs等,但都存在一定缺陷,如重编程效率低下、程序繁琐等[5]。笔者的前期研究也将人牙髓干细胞(dental pulp stem cells,DPSCs)和根尖乳头干细胞(stem cells from apical papilla,SCAP)重编程为iPSCs[6-7],但iPSCs携带有致瘤基因,且诱导效率不高,限制了其进一步应用。
为建立更高效、更安全的重编程方法,本研究采用一种新型RNA载体(仙台病毒)将人DPSCs和SCAP重编程为iPSCs,比较两种iPSCs的克隆形态、诱导效率、诱导时间等生物学特性,为iPSCs用于干细胞治疗提供更理想的细胞来源。
1 材料与方法
1.1主要试剂 α-MEM(minimum essential medium)培养基、胎牛血清(fetal bovine serum,FBS)、0.5 mmol/L乙二胺四乙酸(ethylene diamine tetraacetic acid,EDTA)购自美国Gibco公司;Ⅰ型胶原酶、中性蛋白酶购自美国Roche公司;基质胶(Matrigel,生长因子减少型)购自美国BD公司;TriZOL总RNA提取试剂盒、 SuperScript®Ⅲ Ofne-Step RT-PCR试剂盒购自美国Thermo Fisher公司;CytoTune®-iPS 2.0 Sendai Reprogramming Kit试剂盒购自美国Life公司;重编程培养基、PSC-easy iPS细胞培养基、基质胶、人胚胎干细胞(H9)购自北京赛贝生物科技公司。
1.2方法
1.2.1分离培养 收集云南省第一人民医院口腔颌面外科2017年1月拔除的年轻患者下颌第3磨牙。采用BAKKAR等[8]方法进行培养:收集牙髓、根尖乳头组织,充分剪碎,37 ℃、Ⅰ型胶原酶/中性蛋白酶(3∶4 mg/mL)消化60 min,过细胞滤器(70 μm直径)得到单细胞悬液,加入完全培养基(α-MEM+15%FBS+1%谷氨酰胺+1%青/链霉素),37 ℃、5% CO2常规培养、传代,第3~5代细胞用于后续实验。所有实验均经云南省第一人民医院伦理委员会批准(2013YL023)并取得患者书面同意。
1.2.2重编程 严格按照Sendai Reprogramming Kit试剂盒进行操作,底物为基质胶(终浓度50 μg/mL),人H9为标准对照。转染前2 d:6孔板常规培养人DPSCs和SCAP(第3代,每孔1×105),转染当天细胞70%融合。转染当天(第0天)将适量SeV溶解到70 μL完全培养基,加入6孔板,1 d后(第1天)再加入130 μL完全培养基,第2天换新鲜培养液(含SeV)继续转染。第3天收集已转染细胞,转移到基质胶覆盖6孔板,重编程培养基继续培养,3周左右可观察到小克隆出现。克隆成熟时,“十字分割法”分离克隆,转移到新培养板(基质胶覆盖),PSC-easy培养基(含10 μmol/L Y27632)培养。SeV重编程过程见图1。
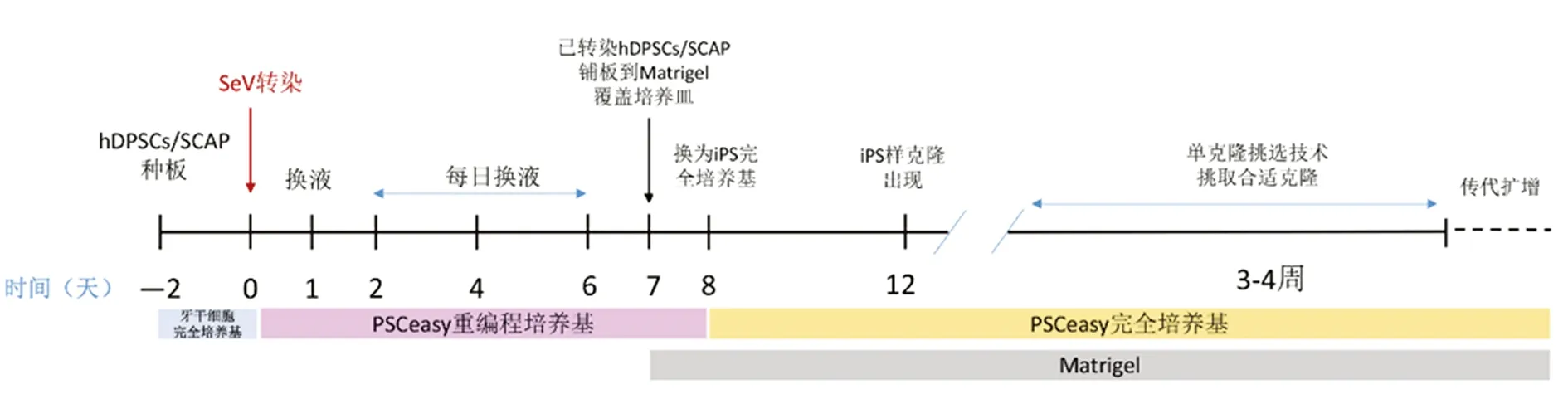
图1 仙台病毒重编程简图
1.2.3两种牙源性iPSCs克隆形态、重编程时间、重编程效率 克隆形态:观察iPSCs在不同体系底物上的生长情况(人H9为标准对照);重编程时间:计算从接种开始到ES样克隆出现的时间,即为该克隆形成时间(诱导时间),重复3次,取平均值;重编程效率:确定精确的重编程效率非常复杂,出现克隆数占重编程体细胞数量的比例是一个重要参数。计算公式:克隆数/体细胞数量×100%,重复3次,取平均值。
1.2.4逆转录-聚合酶链反应(RT-PCR) 收集两种牙源性iPSCs(第12代,5×106个),TriZOL试剂盒提取总RNA,取1 μL RNA,NanoDrop 2000分光光度机(美国Thermo公司)测量浓度,严格按照一步法RT-PCR试剂盒说明书进行反应。反应条件:反转录45 ℃ 30 min;PCR反应程序:95 ℃ 2 min,35次循环(94 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s),72 ℃ 5 min。结束后取5 μL反应产物,加入上样缓冲液电泳检测,GAPDH为管家基因。SeV:上游引物5′-GGA TCA CTA GGT GAT ATC GAG C-3′,下游引物5′-ACC AGA CAA GAG TTT AAG AGA TAT GTA TC-3′;KOS:上游引物5′-ATG CAC CGC TAC GAC GTG AGC GC-3′,下游引物5′-ACC TTG ACA ATC CTG ATG TGG-3′;Klf4:上游引物5′-TTC CTG CAT GCC AGA GGA GCC C-3′,下游引物5′-AAT GTA TCG AAG GTG CTC AA-3′;c-Myc:上游引物5′-TAA CTG ACT AGC AGG CTT GTC G-3′,下游引物5′-TCC ACA TAC AGT CCT GGA TGA TGA TG-3′;GAPDH:上游引物5′-GAA GGT GAA GGT CGG AGT-3′,下游引物5′-GAA GAT GGT GAT GGG ATT TC-3′。
2 结 果
2.1人DPSCs、SCAP的原代培养及重编程 体外培养第1天可见单细胞贴壁生长,第4天形成克隆,1周左右长满培养瓶。 DPSCs表现为多角形或长梭形,而SCAP多为成纤维细胞样或星形,很快形成致密单细胞层。SeV转染细胞接种到基质胶3~4 d可观察到纤维细胞样克隆出现,3~4周形成ES样克隆,质地均匀、排列紧密、边缘清晰,此为iPSCs第0代。挑取成熟iPS克隆继续培养,此为iPSCs第1代,以后用EDTA液消化传代,细胞维持ES样克隆特征。随机选择克隆长期培养,细胞形态稳定,边缘或内部未见分化。 RT-PCR结果表明,两种iPSCs均不表达外源性病毒或转录基因序列,具备临床应用安全性。见图2。
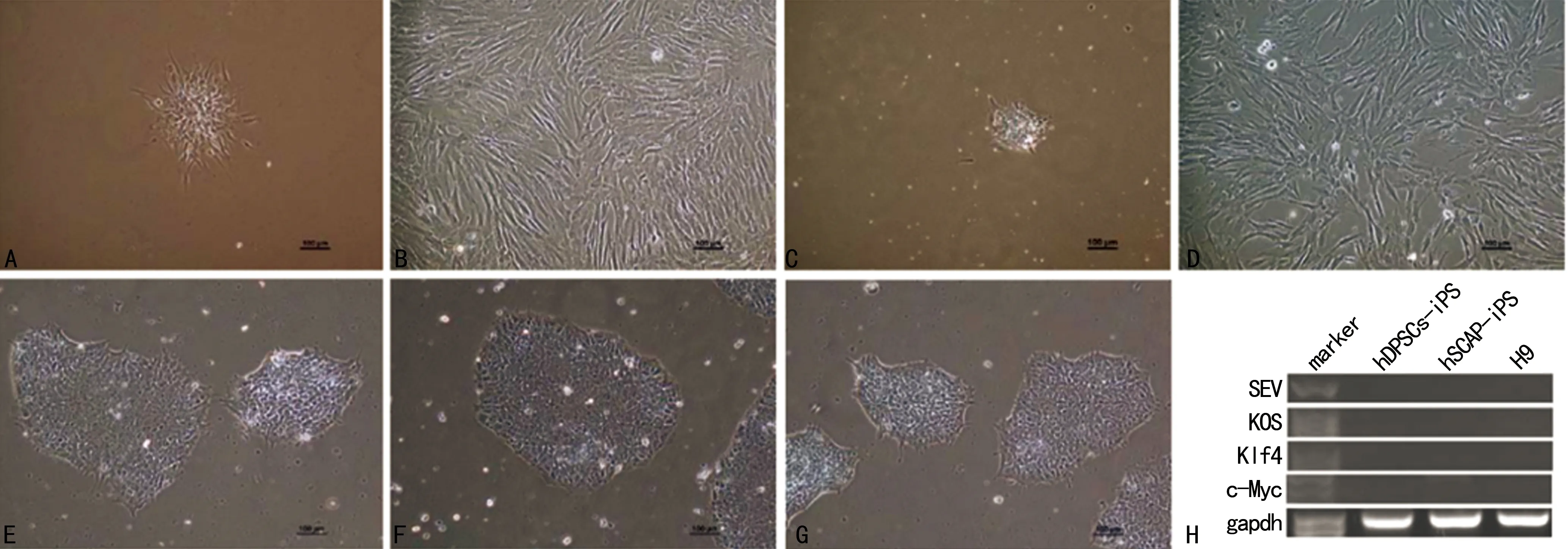
A:hDPSCs(P0,3 d,×100);B:hDPSCs(P0,8 d,×100);C:hSCAP(P0,3 d,×100);D:hSCAP(P0,8 d,×100);E:hDPSCs-iPS(P10,3 d,×100);F:hSCAP-iPS(P10,8 d,×100);G:H9(P52,3 d,×100);H:人DPSCs、SCAP(P15)RT-PCR结果
图2人DPSCs、SCAP原代培养、诱导后iPS细胞形态及RT-PCR结果
2.2人DPSCs、SCAP的重编程效率和时间 人DPSC、SCAP重编程前细胞数量大约为1×104个,重编程后分别得到68和70个左右克隆,重编程效率为(0.68±0.02)%、(0.70±0.01)%,差异无统计学意义(P>0.05)。人DPSCs与SCAP重编程时间分别为(26.0±2.1)、(27.0±1.4)d,差异无统计学意义(P>0.05),见图3。
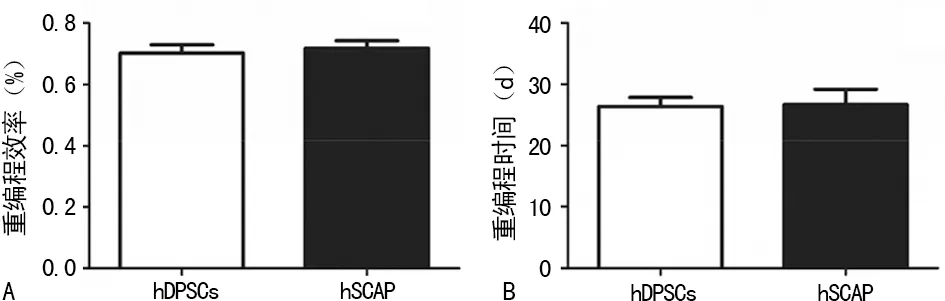
A:人DPSCs、SCAP重编程效率比较;B:人DPSCs、SCAP重编程时间比较
图3人DPSCs、SCAP的重编程时间及效率比较
3 讨 论
重编程技术的出现对干细胞研究具有重大意义,iPSCs的产生避免破坏胚胎,无免疫排斥反应,最终目的是产生患者特异性干细胞用于再生治疗[9]。早期利用病毒性载体在体细胞内过表达转录基因使其重新获得干细胞特征,后续也有研究应用非病毒性载体进行重编程,但普遍存在效率低下、方法复杂等缺点[10]。仙台病毒为15 kb单链RNA病毒,不会结合到宿主细胞基因组内,转录基因表达效率高,病毒及转录基因随着iPSCs的传代而逐渐消除,是理想的重编程转录基因的载体[11]。
iPSCs重编程技术出现以来,多种体细胞均重编程为iPSCs,包括皮肤成纤维细胞、外周血细胞、肾上皮等[12],获取方式多为有创性操作。人第3磨牙常因阻生需要拔除,临床常常丢弃处理。年轻人第3磨牙含有丰富牙髓和根尖组织,易于分离培养DPSCs和SCAP,来源简便、无创,诱导效率也较传统方法高[6-7]。有研究表明,越是处于分化早期的幼稚细胞越容易诱导为iPSCs。诱导时间与诱导效率可作为客观指标衡量诱导为 iPSCs的能力[13]。本研究利用仙台病毒转染人DPSCs和SCAP,结果发现两种细胞均有较高诱导效率(0.7%左右),可能与它们能表达内源性重编程基因Klf4、ES细胞相关基因Nanog及较高增殖效率等有关[14],与MIERE等[15]的研究结果一致。
体细胞重编程机制重点在于核心多潜能性转录基因网络,包括正常细胞发育过程中相关基因的表观遗传调控。重编程基因、miRNA和小分子调控iPSCs产生的表观遗传基因,包括DNA甲基化、组蛋白去甲基化、染色质重组等[16]。iPSCs维持多潜能性的基因主要是Oct4、Sox2、Klf4、c-Myc和Nanog。Oct4来自转录基因POU家族,主要作用为维持细胞多潜能性,能抑制分化相关基因的表达,其位点测序结果显示有广泛去甲基化,Oct4基因对形成和维持iPSCs的多能性和自我更新是和Sox2和Nanog共同完成的[17]。Sox2是性别决定区-Y框蛋白-2相关基因,在应答白血病抑制因子反应时激活,主要调控JAK-STAT信号通路,信号通路的下调导致Sox2激活[18]。Nanog在筛选维持ESCs特性的转录基因时出现,与Oct4和Sox2一起支持内细胞层,也是多潜能细胞维持自我更新的必需因素[19]。c-Myc是一种原癌基因,在重编程过程中发挥催化作用,能增加iPSCs的生成效率。c-Myc与Klf4的相互作用对重编程至关重要,c-Myc可增加增殖效率,但过多表达会增加p53水平,而Klf4能增加p21水平使增殖效率降低,同时降低p53水平以减少细胞凋亡的风险[20]。iPSCs重编程所需时间通常为16~35 d,编程时间的长短反映了重编程的难易程度,同时也体现出重编程过程中变异的风险[21]。
本研究所用仙台病毒能编码Oct4、Sox2、Klf4、c-Myc 4种基因,具备良好的重编程效率,两种细胞诱导时间为27 d左右,与LIEU等[22]的研究结果相似;得到的iPSCs经RT-PCR证实已无病毒或转录基因序列的表达,与CHICHAGOVA等[23]研究结果一致。与ZOU等[7]利用慢病毒hSTEMCCA-loxP将hSCAP重编程为TF-iPSCs相比,本研究无需后期切除外源性转录基因,程序简便,重编程效率较高。同时笔者采用无饲养层体系(基质胶)为培养底物,最大程度减少了外源性因素对iPSCs的影响。
本研究成功利用仙台病毒将人DPSCs和SCAP重编程为iPSCs,取材方便、无创,诱导效率高,得到的iPSCs具备典型干细胞生物学特征,为产生符合临床应用标准的iPSCs及干细胞治疗提供可靠的细胞来源和重编程方法。
[1]TAKAHASHI K,YAMANAKA S.Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors[J].Cell,2006,126(4):663-676.
[2]TAKAHASHI K,TANABE K,OHNUKI M,et al.Induction of pluripotent stem cells from adult human fibroblasts by defined factors[J].Cell,2007,131(5):861-872.
[3]CHRISTODOULOU C,KOTTON D N.Are embryonic stem and induced pluripotent stem cells the same or different? Implications for their potential therapeutic use[J].Cell Cycle,2012,11(1):5-6.
[4]SINGH V K,KALSAN M,KUMAR N,et al.Induced pluripotent stem cells:applications in regenerative medicine,disease modeling,and drug discovery[J].Front Cell Dev Biol,2015,3(2):1-18.
[5]ASPRER J S,LAKSHMIPATHY U.Current methods and challenges in the comprehensive characterization of human pluripotent stem cells[J].Stem Cell Rev,2015,11(2):357-372.
[6]YAN X,QIN H,QU C,et al.iPS cells reprogrammed from human mesenchymal-like stem/progenitor cells of dental tissue origin[J].Stem Cells Dev,2010,19(4):469-480.
[7]ZOU X Y,YANG H Y,YU Z,et al.Establishment of transgene-free induced pluripotent stem cells reprogrammed from human stem cells of apical papilla for neural differentiation[J].Stem Cell Res Ther,2012,3(5):43-54.
[8]BAKKAR M,LIU Y,FANG D,et al.A simplified and systematic method to isolate,culture,and characterize multiple types of human dental stem cells from a single tooth[J].Methods Mol Biol,2017,1553(2):191-207.
[9]CSOBONYEIOVA M,POLAK S,KOLLER J,et al.Induced pluripotent stem cells and their implication for regenerative medicine[J].Cell Tissue Bank,2015,16(2):171-180.
[10]HAYES M,ZAVAZAVA N.Strategies to generate induced pluripotent stem cells[J].Methods Mol Biol,2013,1029(1):77-92.
[11]GERAMI-NAINI B,SMITH A,MAIONE A G,et al.Generation of induced pluripotent stem cells from diabetic foot ulcer fibroblasts using a nonintegrative sendai virus[J].Cell Reprogram,2016,18(4):214-223.
[12]HIBAOUI Y,FEKI A.Concise review:methods and cell types used to generate down syndrome induced pluripotent stem cells[J].J Clin Med,2015,4(4):696-714.
[13]JI P,MANUPIPATPONG S,XIE N,et al.Induced pluripotent stem cells:generation strategy and epigenetic mystery behind reprogramming[J].Stem Cells Int,2016,2016:8415010.
[14]TAMAOKI N,TAKAHASHI K,TANAKA T,et al.Dental pulp cells for induced pluripotent stem cell banking[J].J Dent Res,2010,89(8):773-778.
[15]MIERE C,DEVITO L,ILIC D.Sendai Virus-Based reprogramming of mesenchymal stromal/stem cells from umbilical cord wharton′s jelly into induced pluripotent stem cells[J].Methods Mol Biol,2016,1357(1):33-44.
[16]SINGH V K,KUMAR N,KALSAN M,et al.Mechanism of induction:induced pluripotent stem cells (iPSCs)[J].J Stem Cells,2015,10(1):43-62.
[17]PARDO M,LANG B,YU L,et al.An expanded Oct4 interaction network:implications for stem cell biology,development,and disease[J].Cell Stem Cell,2010,6(4):382-395.
[18]NIWA H,OGAWA K,SHIMOSATO D,et al.A parallel circuit of LIF signalling pathways maintains pluripotency of mouse ES cells[J].Nature,2009,460(7251):118-122.
[19]WANG Z,ORON E,NELSON B,et al.Distinct lineage specification roles for NANOG,OCT4,and SOX2 in human embryonic stem cells[J].Cell Stem Cell,2012,10(4):440-454.
[20]CAI Y N,DAI X H,ZHANG Q H,et al.Gene expression of OCT4,SOX2,KLF4 and MYC (OSKM) induced pluripotent stem cells:identification for potential mechanisms[J].Diagn Pathol,2015,10(1):35-43.
[21]BROUWER M,ZHOU H,NADIF KASRI N.Choices for induction of pluripotency:recent developments in human induced pluripotent stem cell reprogramming strategies[J].Stem Cell Rev,2016,12(1):54-72.
[22]LIEU P T,FONTES A,VEMURI M C,et al.Generation of induced pluripotent stem cells with CytoTune,a non-integrating Sendai virus[J].Methods Mol Biol,2013,997(1):45-56.
[23]CHICHAGOVA V,SANCHEZ-VERA I,ARMSTRONG L,et al.Generation of human induced pluripotent stem cells using RNA-Based sendai virus system and pluripotency validation of the resulting cell population[J].Methods Mol Biol,2016,1353(30):285-307.
