辽宁地区绿豆品种SSR指纹图谱构建及品种鉴定
2018-03-20赵雅楠张东杰
赵雅楠,王 颖,2,张东杰,*
(1.黑龙江八一农垦大学食品学院,黑龙江 大庆 163319;2.国家杂粮工程技术研究中心,黑龙江 大庆 163319)
作物品种鉴定是农业生产环节必不可少的关键步骤,是保证品种优良遗传性状能够充分发挥的重要举措,同时也是名优品种保护、防止种子混杂退化的有效手段[1]。传统的品种鉴定方法主要是根据幼苗、植株的形态特征及农艺性状差异,但观察周期较长,且易受环境影响,鉴定结果准确性不高[2]。同时,由于许多品种的育种亲本更倾向于集中在少数优良品种上,导致所育品种的植物学性状比较相似,品种间差异越来越小,传统方法已无法满足鉴定需求。随着品种贸易的快速发展,在品种引进及推广过程中假冒伪劣、以次充好、种苗混杂等情况屡禁不止,给农业生产带来巨大损失[3]。DNA指纹图谱技术具有丰富的多态性和显著的个体特异性,能够在分子水平上识别品种间差异,不易受外界环境影响,可快速、准确的完成品种鉴定[4-6],主要包括限制性内切酶片段长度多态性(restriction fragment length polymorphism,RFLP)、随机扩增多态性DNA标记(random amplified polymorphic DNA,RAPD)、扩增片段长度多态性(amplified fragment length polymorphism,AFLP)、简单重复序列(simple sequence repeat,SSR)等。其中RFLP技术研究起步较早,可区分纯合型基因或杂合型基因,但操作复杂,且需要使用放射性同位素,有害身体健康;RAPD检测成本低、灵敏度高,但检测结果不稳定,重复性较差;AFLP技术检测范围广,结果重复性好,但实验成本较高且操作复杂[7-9]。而SSR分子标记技术,因其数量丰富、多态性高、稳定性强等显著特点已成为DNA指纹图谱构建及品种鉴定的重要途径[10-12]。Kumar等[13]利用39 对SSR引物对印度54 份小麦进行指纹图谱构建,为小麦品种鉴定提供重要参考;郭照东等[14]利用2 对SSR引物构建了22 个越橘品种的指纹图谱,建立了一套相对完善的越橘品种鉴定体系;姚全胜等[15]利用14 对SSR引物构建了11 个芒果品种的指纹图谱,利用其中4 对引物即可将参试品种全部区分;蒋林峰等[16]构建了我国21 个鸭茅主栽品种的DNA指纹图谱,具有唯一性,可用于鸭茅品种真伪鉴定,为其种权保护提供理论依据。
绿豆是起源于我国的优势杂粮作物,营养价值较高,富含多种维生素和矿物元素,具有清热解毒、抗癌、降血压血脂等功效,是一种广受欢迎的药食同源性作物[17]。近些年,随着人们生活水平的提高及膳食营养意识的增强,绿豆以其丰富的营养价值深受青睐,品种数量逐年上升,但因为种种因素导致种子质量管理混乱,品种真实性难以得到保证。而DNA指纹图谱可用于绿豆品种真伪鉴定,为绿豆名优品种保护、原产地溯源管理等提供科学依据[18-20]。截至目前,关于利用SSR荧光标记技术构建绿豆DNA指纹图谱进行品种鉴定的研究鲜有报道。基于此,本研究利用SSR技术构建了辽宁地区48 份绿豆品种的指纹图谱及分子身份证,将品种间DNA水平上的差异可视化,以期建立一种高效、准确的绿豆品种真伪鉴定方法,为我国绿豆品种保护及新品选育提供理论依据。
1 材料与方法
1.1 材料与试剂
选取辽宁省大连市、阜新县等21 个不同市县的48 份绿豆品种,均由中国农业科学院作物科学研究所提供,供试绿豆详情见表1。
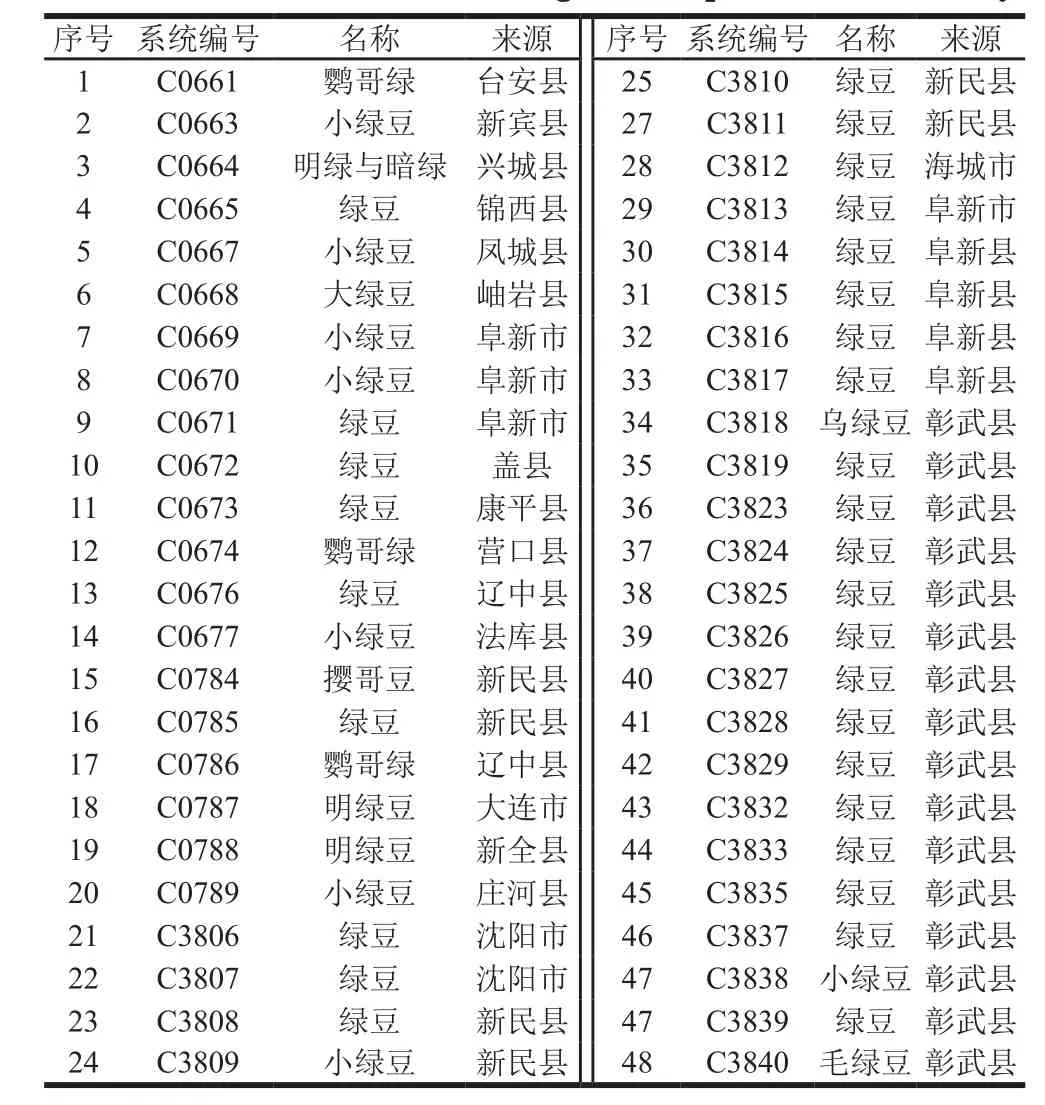
表1 辽宁地区48 份参试绿豆品种信息Table 1 Information about 48 mung bean samples tested in this study
植物基因组DNA提取试剂盒、Taq DNA聚合酶、Low MW DNA Marker-A、Mg2+、dNTPs、Buffer、琼脂糖 北京天根生物技术有限公司;15 对SSR引物由中国农业科学院作物科学研究所提供,普通引物由深圳华大基因公司合成,荧光引物由美国ABI公司合成。
1.2 仪器与设备
ABI Prism 3730XL型基因分析仪 上海创萌生物科技有限公司;Bio-rad T100 PCR仪 北京伯迈生物科技有限公司;智能核酸蛋白检测仪 上海嘉鹏科技有限公司;Biorad GelDoc XR凝胶成像系统 上海旦鼎国际贸易有限公司。
1.3 方法
1.3.1 DNA提取
采集室温条件下种植15 d左右的新鲜嫩叶,利用TIANGEN Plant Genomic DNA Kit提取绿豆基因组DNA,1%琼脂糖检测DNA提取质量,核酸检测仪测定DNA浓度,最后将母液稀释成30 ng/μL保存于4 ℃环境中备用。
1.3.2 SSR-PCR体系及毛细管电泳检测
聚合酶链式反应(polymerase chain reaction,PCR)体系为20 μL,包括30 ng/μL DNA模板2 μL,2 μmol/L引物0.6 μL,2.5 U/μL Taq酶0.2 μL,10 mmol/L dNTP 0.4 μL,20 mmol/L MgCl21 μL,Buffer 2 μL,其余用ddH2O补齐。PCR程序为94 ℃预变性5 min;94 ℃变性30 s,退火温度54 ℃,退火时间35 s,72 ℃延伸40 s,共35 个循环;最终72 ℃延伸3 min;4 ℃保存。在Bio-rad T100 PCR仪上进行。
2 结果与分析
2.1 SSR标记多态性分析
15 对SSR引物在48 份绿豆材料中共扩增出58 个等位基因,每对引物检测到2~7 个不等,平均为3.9 个;有效等位基因数介于1.135 0~5.113 4之间,平均为1.983 5;Nei’s基因多样性指数介于0.118 9~0.576 8之间,平均为0.428 1。多态信息含量(polymorphic information content,PIC)变幅介于0.114 5(GBssr-MB91)~0.776 4(X55)之间,平均为0.378 4。其中引物GBssr-MB91、P3-581、P3-627、P3-765为低度多态性位点(0<PIC<0.25),多态性较差,X55(0.776 4)、X46(0.513 2)为高多态性位点,PIC大于0.5,多态性较丰富,具有较强的检测能力,可作为辽宁地区绿豆品种遗传分析的骨干引物。其余9 对引物均为中度多态位点(0.25<PIC<0.5)。具体信息见表2。
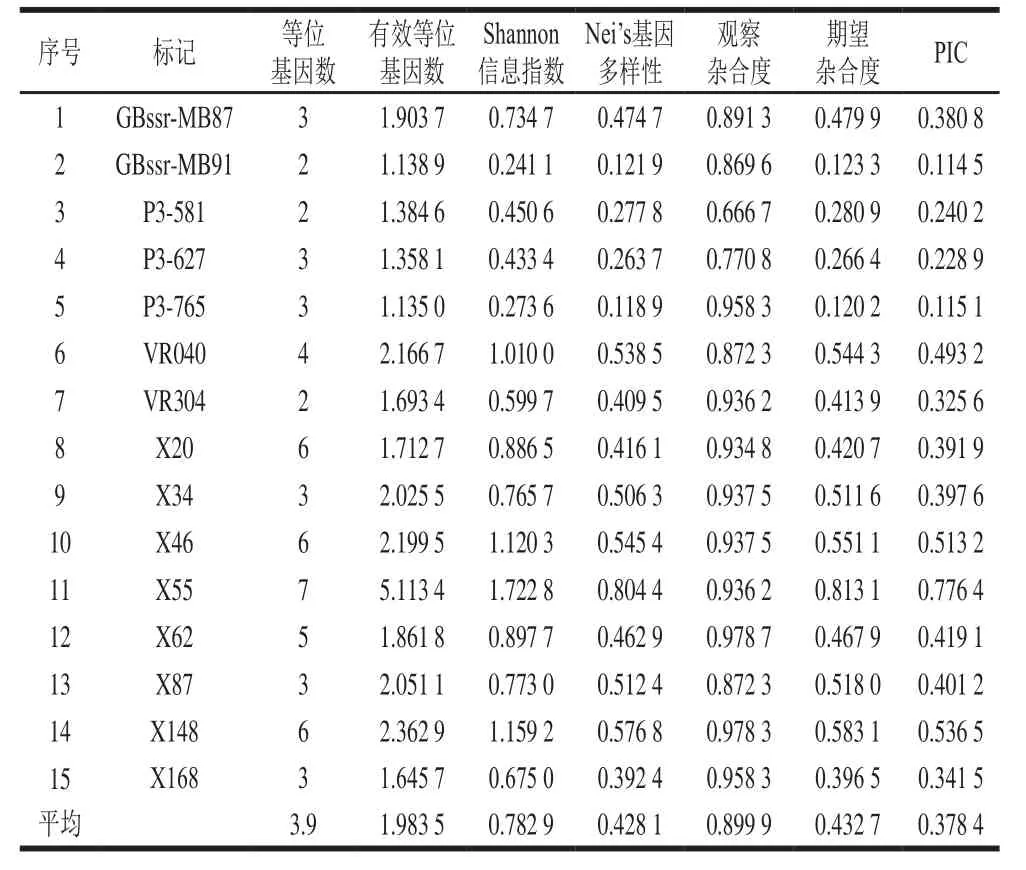
表2 15 对SSR引物多态性信息Table 2 Genetic diversity of mungbean germplasm based on 15 SSRs
2.2 指纹图谱构建及品种鉴定

图1 辽宁地区48 份绿豆品种的标准化SSR扩增指纹图谱Fig. 1 Standardized fingerprint of SSR amplification for mung bean varieties
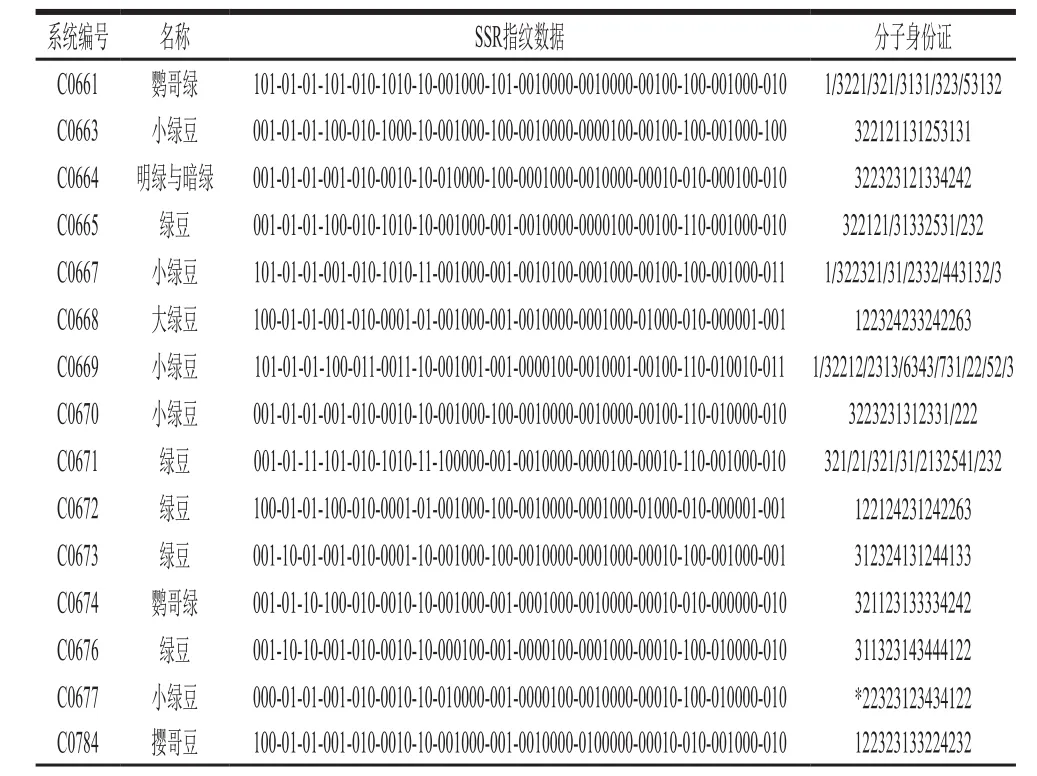
表3 48 个品种在15 个微卫星标记上的指纹数据及分子身份证Table 3 Fingerprint database and molecular identity of mung bean varieties at 15 SSR loci

续表3
依据15 对引物对48 份绿豆品种的扩增结果构建SSR指纹图谱,如图1所示。图1中横向序号表示参试的48 个绿豆品种,纵向序号表示按表2顺序的15 对引物的扩增条带分子质量。其中黑色条带处代表在该位点检测到等位基因,每个品种的指纹图谱均与表3中的指纹数据及分子身份证相对应。标准模式图甄别能力较强,表现直观,根据图谱呈现出的差异性,可快速在基因水平上实现绿豆品种的区分鉴定。如绿豆(C3823)和绿豆(C3832),在引物GBssr-MB87的第3个等位基因位点(276 bp)处存在唯一差异,可作为区分这2 个品种的标志性位点;大绿豆(C0668)和绿豆(C0672)在引物P3-627的第1个等位基因位点(155 bp)、P3-627的第3个等位基因位点(164 bp)和X34的第1个等位基因位点(141 bp)上的区分较为明显,可用于品种鉴别。纵向比较发现,15 对引物检测到的等位基因可作为参试样品的特异性位点。如标记X20是明绿与暗绿(C0664)和小绿豆(C0669)的特异性引物,分别在195 bp和211 bp处检测到二者的特异性位点,可与其他品种相区分;X46分别在107、113、115、121、123、125 bp处检测到等位基因,其中107 bp和123 bp 2 个位点仅分别在小绿豆(C3838)和小绿豆(C3809)中检测到,为其特异性位点,具有品种标志性。整体分析可知除撄哥豆(C0784)、绿豆(C3807)、绿豆(C3823)和绿豆(C3826)、绿豆(C3837)外,15 对引物可将所有参试品种完全区分,区分率为89.68%,但没有任何一对引物可将参试品种全部区分,故采用引物组合鉴定法。根据每对引物的区分效率(表4),选取鉴别能力较强的标记X20、X46、X55、X62及X148进行组合。标记X46、X55、X148组合时,品种聚类区分率为43.75%,增加标记X20,区分率增加至56.25%,5 个标记组合到一起时区分率达75%。可见,SSR指纹图谱应具备可扩充性,当品种数量增多或鉴别能力不足时可增加引物数量以保证品种间的区分效率,避免身份识别错误的情况。

表4 各标记扩增条带信息Table 4 Information about amplified bands
2.3 SSR指纹数据及分子身份证构建
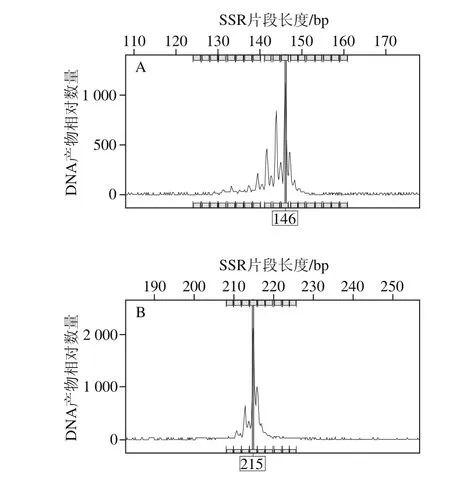
图2 明绿豆(C0787)在位点X55(A)和小绿豆(C0789)在位点X87(B)座位上的毛细管电泳峰图Fig. 2 SSR fingerprint patterns of the cultivars Minglüdou (C0787) (A)and Xiaolüdou (C0789) (B) using primer X87
利用15 对SSR引物对48 份绿豆材料进行扩增,部分毛细管电泳结果如图2所示。在同一峰值处有带记为1,无带记为0,建立起0/1形式的辽宁地区绿豆品种SSR指纹图谱。按表2引物顺序,将每对引物在参试绿豆品种中检测的变异位点按片段大小升序排列、依次编号,将编号依次串联,即得到由15 位数字(有杂合带时多于15 位)构成的分子身份证。
2.4 亲缘关系分析

表5 部分绿豆品种间的遗传相似系数Table 5 Genetic similarity coefficients among selected mung bean varieties
48 份绿豆种质资源的遗传相似系数变幅介于0.517 2~1.000 0之间,平均为0.76。其中小绿豆(C0669)和绿豆(C3839)间遗传相似系数最小,为0.517 2,说明二者亲缘关系较远;绿豆(C3832)和绿豆(C3807)间遗传相似系数较大,为0.982 8,说明二者间遗传距离较小,亲缘关系较近。部分品种间遗传相似系数如表5所示。通过用非加权组平均法利用遗传相似系数对48 份辽宁地区绿豆种质资源材料进行聚类分析,如图3所示。在遗传相似系数为0.73的阈值处,可将48 份绿豆分为4 个类群,第1类群由鹦哥绿(C0661)、小绿豆(C0663)、绿豆(C0665)等41 份品种组成,第2类群由大绿豆(C0668)、绿豆(C0672)、毛绿豆(C3840)、绿豆(C3812)、绿豆(C0673)组成,而小绿豆(C0669)和绿豆(C3839)单独聚为一类,分别组成第3、4类群。可见小绿豆(C0669)和绿豆(C3839)与该地区其他品种间的亲缘关系较远。在遗传相似系数为1.0水平上,撄哥豆(C0784)、绿豆(C3806)、绿豆(C3823)和绿豆(C3826)、小绿豆(C3838)聚为一类,并没有被完全区分,考虑可能是因为2 组品种间遗传背景相似,遗传相似性较高,15 个标记并没有检测到品种间的差异性位点,但也不排除“同名异种”的情况。
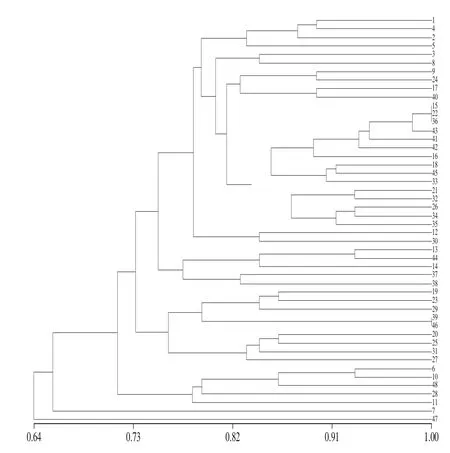
图3 基于SSR标记的48 份绿豆聚类分析Fig. 3 Dendrogram for 48 mung bean samples generated by SSR markers
3 讨 论
SSR分子标记技术操作简便、稳定性强,已逐渐发展为各作物指纹图谱构建和品种区分鉴别的有效手段,如山茶树[21]、黄麻[22]、油菜[23]、水稻[24]等遗传多样性分析和品种鉴定等,SSR技术具有较好的应用价值。目前,SSR-PCR大多采用聚丙烯酰胺凝胶电泳检测,但该法效率较低,在数据收集和整合的过程中容易产生误差。SSR荧光标记毛细管检测技术,以DNA测序仪为平台,可准确读出检测片段大小,自动化程度高,能够识别1~2 个碱基片段间差异[25]。王瑞[26]、宋伟林[27]、徐雷锋[28]等在高粱、油菜、百合研究中发现荧光标记技术检测效率高,结果精确,更加适用于高通量材料的SSR指纹图谱构建。本研究采用SSR荧光标记毛细管检测技术,获得的指纹数据更加精确,可识别出差异较小品种间的特异性位点,鉴别能力较强。
SSR引物筛选是构建高效SSR指纹图谱的重要步骤[29],选用多态性丰富、特异性强和重复性好的引物进行指纹图谱构建,可简单高效地进行品种鉴定,而遵循原则之一就是要利用少数的标记鉴定尽量多的品种,即要求引物的多态性要高。Lestari等[30]利用30 对引物对83 份绿豆品种进行分析,平均等位基因数为2.77,平均PIC为0.33;王丽侠等[31]利用26 对小豆SSR引物检测了国内外60 个绿豆品种,获得的等位基因数从2~7 个不等,平均为2.9 个,PIC从0.02~0.69不等,平均为0.36。本研究筛选得到15 对SSR引物用于指纹图谱构建及品种鉴定,共扩增出58 个等位基因,每对引物检测到2~7 个不等,平均为3.9;PIC介于0.114 5~0.776 4之间,平均为0.378 4,获得的PIC均高于前人研究,但与Sangiri等[32]获得平均等位基因数为7.3的结果相比略低,标记的等位基因数与PIC变化趋势不一致,这一方面可能与所选用的SSR标记数量有关,另一方面可能与参试种质的遗传基础有关[33-35]。因此,在筛选构建SSR指纹图谱的核心引物时,尽可能选取多态性高且具有物种特异性的引物。
SSR指纹图谱的终极目标是具备特异性和唯一性,然而由于受到研究对象基因结构、遗传基础及所选引物数量和多态性水平等因素影响,1 对引物往往不能实现完全区分,因此通常采用引物组合法进行指纹图谱构建及品种鉴定。本研究筛选得到15 对SSR引物,共扩增出58 个条带,多态性条带比率为100%,其中包含特异条带13 条,占总数的22.4%。同时,各引物的品种聚类区分效率各不相同,选取5 对鉴别能力较强的引物进行组合,区分率可达75%。但15 对引物并不能将所有参试品种全部区分,区分率仅为89.68%,其中撄哥豆(C0784)、绿豆(C3807)、绿豆(C3823)和绿豆(C3826)、绿豆(C3837)的指纹图谱并不具有唯一性,不能完全区分鉴别,这可能是因为2 组品种间的遗传背景相似,DNA水平上的差异较小,15 个标记没有检测到品种间的差异性位点,但不排除“ 同种异名”的情况。因此,当参试品种数量增加或鉴别能力不足时应选择不同的引物组合或筛选新的核心引物进行区分鉴别,以扩充和拓展SSR指纹图谱,保证其鉴别准确性。
随着DNA分子标记技术的快速发展,SSR指纹图谱身份证广泛应用,已发展成为一种种质鉴定的重要手段,其在SSR指纹图谱基础上将品种的基因型特征数字化,使每个品种具有唯一的数字代码,更加简单明了地进行品种区分鉴定。陆徐忠等[36]利用12 对SSR引物构建了安徽省127 份水稻品种的分子指纹图谱,并与各品种的基本商品信息相结合,形成了具有品种特异性的分子身份证和条码标志,为水稻品种的产权保护提供支持;陈亮等[37]利用7 对SSR引物构建了吉林省大豆品种的SSR分子身份证,具有唯一性,完善了大豆品种指纹图谱身份证技术体系。本研究利用15 对SSR引物在48 个绿豆品种中检测到的等位基因构建了0/1形式的指纹数据库,并将其数字化,形成分子身份证,将品种间DNA水平上的差异具体化、数字化,具有高度的遗传稳定性及个体特异性,对绿豆品种的科学追溯、快速鉴定和规范管理具有重要意义。此外,指纹数据库及分子身份证的构建还可为品种亲缘关系分析提供参考。如撄哥豆(C0784)和绿豆(C3832)的分子身份证分别为122323133224232和132323133224332,相似度较高,而二者间的遗传相似系数也较大,为0.982 8(文中未体现),表明其遗传背景相似,亲缘关系较近。
4 结 论
本研究以辽宁地区48 份绿豆品种为试材,利用筛选得到的15 对SSR引物对其进行分析,以扩增出的变异位点为依据进行编码,构建了SSR指纹图谱及分子身份证,数据精确、检测效率高、鉴别能力强、结果直观清晰,为绿豆品种真伪鉴定、种权保护供了有力保障。目前,分子标记所扩增的序列具体与农作物的哪个性状相关联尚不清楚[37],且分子标记的数字化具有局限性,不能反映一个品种的全部信息[38],因此在今后的研究中应将指纹图谱与田间观察的表现型相结合,同时增加品种的其他重要商品属性信息,以不断完善绿豆品种鉴定技术体系,使品种的身份信息更加详细、准确,为绿豆优良品种保护、溯源管理等提供理论依据。
[1] LUAN M B, YANG Z M, ZHU J J, et al. Identification, evaluation,and application of the genomic-SSR loci in ramie[J]. Acta Societatis Botanicorum Poloniae, 2016, 85(3): 3510-3522. DOI:10.5586/asbp.3510.
[2] 吴帅, 雷声, 杨锡洪, 等. 指纹分析技术及其在食品中的应用[J]. 食品与机械, 2015, 31(1): 249-252. DOI:10.13652/j.issn.1003-5788.2015.01.057.
[3] 左示敏, 周娜娜, 陈宗祥, 等. DNA指纹在水稻品种鉴定中的应用与展望[J]. 江苏农业科学, 2014, 42(12): 4-8. DOI:10.15889/j.issn.100-130.014.1.00.
[4] ROY S, RAY B P, SARKER A, et al. DNA fingerprinting and genetic diversity in Lentil Germplasm using SSR markers[J]. Asian Journal of Conservation Biology, 2015, 4(2): 109-115.
[5] BROWN S, HIGHAM T, SLON V, et al. Identification of a new hominin bone from Denisova Cave, Siberia using collagen fingerprinting and mitochondrial DNA analysis[J]. Scientific Reports,2016, 6: 23559. DOI:10.1038/srep23559.
[6] SALIMIZAND H, MENBARI S, RAMAZANZADEH R, et al. DNA fingerprinting and antimicrobial susceptibility pattern of clinical and environmental Acinetobacter baumannii isolates: a multicentre study[J]. Journal of Chemotherapy, 2016, 28(4): 277-283. DOI:10.108 0/1120009X.2016.1175706.
[7] 吴鑫. 柑桔DNA指纹图谱库的构建与分析[D]. 重庆: 西南大学,2008.
[8] SARAO N K, VIKAL Y, SINGH K, et al. SSR marker-based DNA fingerprinting and cultivar identification of rice (Oryza sativa L.) in Punjab state of India[J]. Plant Genetic Resources, 2010, 8(1): 42-44.
[9] SAKER M M, YOUSSEF S S, ABDALLAH N A, et al. Genetic analysis of some Egyptian rice genotypes using RAPD, SSR and AFLP[J]. African Journal of Biotechnology, 2011, 4(9): 882-890.
[10] MIN S K, CHOI B, PARK J H, et al. Assessment of genetic diversity and population structure of the sub core set in sesame(Sesamum indicum) using SSR markers[J]. Gene, 2016, 28(1): 73-83.DOI:10.12719/KSIA.2016.28.1.73.
[11] DENCIC S, DEPAUW R, MOMCILOVIC V, et al. Comparison of similarity coeきcients used for cluster analysis based on SSR markers in sister line wheat cultivars[J]. Genetika, 2016, 48(1): 219-232.DOI:10.2298/GENSR1601219D.
[12] AN M, DENG M, ZHENG S S, et al. De novo transcriptome assembly and development of SSR markers of oaks Quercus austrocochinchinensis, and Q. kerrii, (Fagaceae)[J]. Tree Genetics &Genomes, 2016, 12(6): 103-112. DOI:10.1007/s11295-016-1060-5.
[13] KUMAR S, KUMAR V, KUMARI P, et al. DNA fingerprinting and genetic diversity studies in wheat genotypes using SSR markers[J].Journal of Environmental Biology, 2016, 37(2): 319-326.
[14] 郭照东, 夏秀英, 安利佳, 等. 基于SSR标记的越橘亲缘关系分析及品种鉴定[J]. 植物遗传资源学报, 2015, 16(5): 1020-1026.DOI:10.13430 /j.cnki.jpgr.2015.05.013.
[15] 姚全胜, 詹儒林, 黄丽芳, 等. 11 个芒果品种SSR指纹图谱的构建与品种鉴别[J]. 热带作物学报, 2009, 30(11): 1572-1576.DOI:10.3969/j.issn.1000-2561.2009.11.005.
[16] 蒋林峰, 张新全, 黄琳凯, 等. 中国主栽鸭茅品种(系)DNA指纹图谱构建[J]. 植物遗传资源学报, 2014, 15(3): 604-614. DOI:10.13430/j.cnki.jpgr.2014.03.022.
[17] 刘岩, 程须珍, 王丽侠, 等. 基于SSR标记的中国绿豆种质资源遗传多样性研究[J]. 中国农业科学, 2013, 46(20): 4197-4209.DOI:10.3864/j.issn.0578-1752.2013.20.003.
[18] DEEF L E M. DNA fingerprinting of three species of family Alcedinidae (Aves: Coraciiformes) in Egypt using molecular markers[J].Environmental Science & Technology, 2016, 5(11): 393-396.
[19] PERERA D R C, GUNATHILAKA P A D H N, RODRIGO W W P,et al. DNA fingerprinting of Thunnus obesus and Thunnus albacares fish species for proper identification in large scale fish processing industry[C]// Uva Wellassa University of Sri Lanka: 6th Research Symposium Conference, 2016.
[20] PATIL V, HEMANT K. DNA fingerprinting of Indian potato cultivars by inter simple sequence repeats (ISSRS) markers[J]. Potato Journal, 2016,43(1): 70-77.
[21] CHEN Y, DAI X, HOU J, et al. DNA fingerprinting of oil camellia cultivars with SSR markers[J]. Tree Genetics & Genomes, 2016, 12(1): 1-8.
[22] ZHANG L, CAI R, YUAN M, et al. Genetic diversity and DNA fingerprinting in jute (Corchorus spp.) based on SSR markers[J]. The Crop Journal, 2015, 3(5): 416-422. DOI:10.1016/j.cj.2015.05.005.
[23] 汤天泽, 肖小余, 税红霞. SSR标记与形态学方法鉴定杂交油菜纯度的比较研究[J]. 分子植物育种, 2008, 6(2): 377-380.DOI:10.3969/j.issn.1672-416X.2008.02.029.
[24] 曾晓珊, 彭丹, 石媛媛, 等. 利用SSR标记构建水稻核心亲本指纹图谱[J]. 作物研究, 2016, 30(5): 481-486. DOI:10.16848/j.cnki.issn.1001-5280.2016.05.01.
[25] 易红梅, 王凤格, 赵久然, 等. 玉米品种SSR标记毛细管电泳荧光检测法与变性PAGE银染检测法的比较研究[J]. 华北农学报, 2006,21(5): 64-67. DOI:10.3321/j.issn:1000-7091.2006.05.016.
[26] 王瑞, 张福耀, 程庆军, 等. 利用SSR荧光标记构建20 个高粱品种指纹图谱[J]. 作物学报, 2015, 41(4): 658-665. DOI:10.3724/SP.J.1006.2015.00658.
[27] 宋伟林. 基于SSR荧光标记毛细管电泳的油菜品种DNA指纹鉴定技术平台的建立与应用[D]. 北京: 中国农业科学院, 2013.
[28] 徐雷锋, 葛亮, 袁素霞, 等. 利用荧光标记SSR构建百合种质资源分子身份证[J]. 园艺学报, 2014, 41(10): 2055-2064. DOI:10.16420/j.issn.0513-353x.2014.10.012.
[29] 潘兆娥, 孙君灵, 王西文, 等. 基于棉花参比种质的SSR多态性核心引物筛选[J]. 生物多样性, 2008, 16(6): 555-561. DOI:10.3724/SP.J.1003.2008.08132.
[30] LESTARI P, KIM S K, REFLINUR, et al. Genetic diversity of mungbean (Vigna radiata L.) germplasm in Indonesia[J]. Plant Genetic Resources, 2014, 12(Suppl 1): S91-S94. DOI:10.1017/S1479262114000343.
[31] 王丽侠, 程须珍, 王素华, 等. 小豆SSR引物在绿豆基因组中的通用性分析[J]. 作物学报, 2009, 35(5): 816-820. DOI:10.3724/SP.J.1006.2009.00816.
[32] SANGIRI C, KAGA A, TOMOOKA N, et al. Genetic diversity of the mungbean (Vigna radiata, Leguminosae) genepool on the basis of microsatellite analysis[J]. Australian Journal of Botany, 2007, 55(8):837-847. DOI:10.1071/BT07105.
[33] FUENTES F F, MARTINEZ E A, HINRICHSEN P V, et al.Assessment of genetic diversity patterns in Chilean quinoa(Chenopodium quinoa, Willd.) germplasm using multiplex fluorescent microsatellite markers[J]. Conservation Genetics, 2009, 10(2): 369-377. DOI:10.1007/s10592-008-9604-3.
[34] ALI M L R J F, BAENZIGER P S, GILL K S, et al. Assessment of genetic diversity and relationship among a collection of US sweet sorghum germplasm by SSR markers[J]. Molecular Breeding, 2008,21(4): 497-509.
[35] 陆敏佳, 蒋玉蓉, 陆国权, 等. 利用SSR标记分析藜麦品种的遗传多样性[J]. 核农学报, 2015, 29(2): 260-269.
[36] 陆徐忠, 倪金龙, 李莉, 等. 利用SSR分子指纹和商品信息构建水稻品种身份证[J]. 作物学报, 2014, 40(5): 823-829. DOI:10.3724/SP.J.1006.2014.00823.
[37] 陈亮, 郑宇宏, 范旭红, 等. 吉林省新育成大豆品种SSR指纹图谱身份证的构建[J]. 大豆科学, 2016, 35(6): 896-901. DOI:10.11861/j.issn.1000-9841.2016.06.0896.
[38] 陈亮, 郑宇宏, 范旭红, 等. 大豆SSR指纹图谱身份证的研究进展与展望[J]. 大豆科技, 2015, 34(2): 38-43. DOI:10.3969/j.issn.1674-3547.2015.02.008.
