基于BLUP和GGE双标图的林木多地点试验分析
2018-03-20张心菲张鑫鑫张卫华林元震1华南农业大学林学与风景园林学院广东广州510642广东省森林植物种质创新与利用重点实验室广东广州510642广东省林业科学研究院广东广州510640
程 玲,张心菲,张鑫鑫,张卫华,林元震 (1华南农业大学 林学与风景园林学院,广东 广州 510642:2 广东省森林植物种质创新与利用重点实验室,广东 广州510642: 广东省林业科学研究院,广东 广州 510640)
与农作物相似,林木选育的良种也存在地域适用性,因此林木遗传试验应进行多地点试验(Multi-environment trial,MET)。多地点试验分为3个阶段:初试阶段、中试阶段和大规模试验阶段,其中参试材料数一般随着试验规模的增大而减少,但试验地点数恰好相反[1]。在多地点试验中,一个突出的问题是林木基因型与环境之间往往存在显著的交互作用(Genotype by environment interaction, GEI),因此如何准确评估这种交互作用对于后续林木良种的选育和推广至关重要。目前,农业上大多使用联合回归法[2]、主效可加互作可乘模型(Additive main effects multiplicative interaction,AMMI)[3]和基因型主效加基因型-环境互作效应(Genotype main effect plus genotype-by-environment interaction,GGE)双标图[4]分析多点试验结果,并据此来进行品种评价、试验点评价和品种生态区划分,其中GGE双标图越来越受到科研人员的关注[5]。迄今,GGE双标图在林木良种选育上的应用仍然很少,仅在少数树种如辐射松[6]、杨树[7]和乐昌含笑[8]上有报道。
虽然AMMI和GGE双标图在农作物上应用广泛,但这些模型均直接通过表型数据进行分析,而且存在一些限制[5]:1)上述方法均只限定于固定效应模型;2)假定各地点误差同质;3)要求数据是平衡的。但对于林木试验地而言,首先试验地点误差同质几乎不可能,现有研究证实林地存在各种空间变异[1];其次,试验结果往往会存在缺株或缺区数据等现象;再者,作为重要遗传参数之一的育种值,需要随机效应模型才可估算。因此,这些问题制约着AMMI和GGE双标图在林木研究中的应用。针对这些问题,本研究提出采用空间变异结合因子分析法获取林木基因型在各试验地的无偏预测值(Best linear unbiased prediction,BLUP),然后再利用GGE双标图进行林木基因型评估、试验地评估和生态区划分,以避免固定效应模型、试验地点同质和缺失数据的约束,为GGE双标图在林木良种选育上的应用提供借鉴。
1 方 法
1.1 BLUP的统计模型
多地点试验的混合模型为:
yijk=μ+Si+SGi(j)+eijk。
(1)
式中:yijk为个体性状表型值;μ为总体均值;Si为第i个地点的固定效应;SGi(j)为第i个地点与第j个基因型的随机交互效应;eijk为剩余残差。
使用因子分析法[9]拟合SGi(j)效应时,其方差协方差矩阵可写为:
G=(Γ×Γ′+Ψ)⊗I。
(2)
式中:G为SGi(j)效应的方差矩阵;Γ为因子载荷矩阵;Γ′为因子载荷矩阵的转置矩阵;Ψ为特殊方差矩阵;I为单位矩阵;⊗为矩阵的Kronecker乘法。

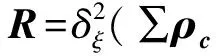
(3)

∑ρ自相关矩阵为:
(4)
上述BLUP过程采用ASReml软件[11]进行分析,具体方法参照文献[1]。
1.2 GGE双标图的统计模型
参照文献[6],GGE双标图的统计模型为:
yij-μ-βj=λ1γi1δj1+λ2γi2δj2+εij。
(5)
式中:yij为第j个地点第i个基因型的均值;μ为总体均值,为试验地点的主效应;βj为第j个地点所有基因型的均值;λ1、λ2为第1个和第2个主成分的特征值;γi1、γi2为第i个基因型在第1个和第2个主成分的特征向量;δj1、δj2为第j个地点在第1个和第2个主成分的特征向量;εij为剩余残差。
GGE双标图的分析采用R软件程序包GGEBiplotGUI[12]实现。参数设置时Scaled选择0(非标准化),Centerd选择G+GE,SVP选择Symmetrical。
1.3 测试数据
测试数据来自文献[1]。火炬松的基因型有36种(1~36),试验地点6个(S1~S6),试验设计为拉丁方设计,每个试验地设3次重复,每个重复设6个区组,测量性状为种子产量。其中,除了第3个地点为9行12列外,其余均为6行18列。
1.4 原始数据和BLUP数据的比较
原始数据和BLUP数据的热图采用R软件程序包AAfun[13]生成,方差分析采用R软件aov函数[1]完成。
2 结果与分析
2.1 原始数据的BLUP分析结果
为克服试验地误差异质对GGE双标图分析的影响,本研究采用空间分析模型拟合每个试验地的R残差,目的是:(1)求证每个试验地误差是否异质;(2)获取每个试验地测量性状的BLUP值,以用于后续的GGE双标图分析。空间变异拟合R残差的参数BLUP结果如表1所示。

表1 火炬松36个基因型的种子产量原始数据的BLUP分析结果Table 1 BLUP result of parameters for original data of seed yield of 36 Pinus teada genotypes
注:行、列自相关值的上标值是t检验统计量,为相关估计值与其标准误的比值。
Note:The superscript iststatistic autocorrelation,the ratio of estimated values to standard error.
由表1可知,各地点的环境误差差异比较大,对于随机误差,地点S1最大,而地点S2和S5的估计值为0;对于空间误差,所有地点均存在,其中地点S2和S4的比较大。此外,除地点S5以外,其余地点都存在显著的行或列自相关性(t>1.5时,代表相关值在α=0.05水平上显著),说明试验地存在显著的空间变异模式。上述结果表明,各试验地点环境误差不同,空间变异的拟合结果有效,获取的测量性状BLUP值可靠。
2.2 原始数据与BLUP数据的比较
原始数据和BLUP数据的热图结果如图1所示。

S1~S6.表示6个试验地;R1~R3.分别为重复1~3;图中的小方框代表区组S1-S6.Six trail sites;R1-R3.repeats 1-3.Small squares of the pictures represent blocks图1 火炬松种子产量原始数据(左)和BLUP数据(右)的热图Fig.1 Heatmap of original data (left) and BLUP data (right) of Pinus teada seed yield
图1显示,试验地间的种子产量差异明显,而且原始数据和BLUP数据之间的差异也比较明显。经过空间变异的校正后,BLUP数据中数据点之间的变异幅度缩小,而且BLUP数据的空间变异模式更为清晰,例如地点S2,颜色接近的几乎连成片(存在空间变异的一种典型特征),但原始数据并未呈现出这种趋势。上述结果进一步表明,各地点的确存在空间变异,与表1的分析结果一致。
原始数据和BLUP数据的方差分析结果(表2)显示,对于原始数据,基因型和地点的效应均达极显著水平(P<0.001),基因型与地点的互作效应达显著水平(P<0.05),地点与重复的互作效应达极显著水平(P<0.001),说明上述因子对种子产量影响显著;对于BLUP数据,基因型、地点及其互作效应的F值均大于原始数据的F值,而且均达极显著水平(P<0.001),但地点与重复的互作效应不显著(P>0.05),这是由于空间变异的拟合削弱了重复因子的效应,此外,误差的均方显著降低。基因型、地点及其互作效应对平方和的解释百分比之和,原始数据为67.81%,BLUP数据为88.52%,说明基因型、地点及其互作效应引起的变异是种子产量变异的主要来源,即可以使用GGE双标图进行后续分析。上述结果表明,对于产量变异的解释能力,BLUP数据比原始数据更高,表明BLUP数据用于GGE双标图的分析更为可靠。

表2 火炬松种子产量原始数据和BLUP数据的方差分析Table 2 Analysis of variance for original data and BLUP data of Pinus teada seed yield
注:*.效应达显著水平(P<0.05),**.效应达极显著水平(P<0.001)。
Note: *. significant (P<0.05), **. very significant (P<0.001).
2.3 原始数据和BLUP数据与GGE双标图结合分析结果的比较
2.3.1 试验地分组结果 GGE双标图可以展示试验地点间的关系,试验地点向量之间的夹角余弦值为试验点的遗传相关系数,夹角越小,则相关值越大,当夹角小于90°时为正相关,而大于90°时为负相关。地点间关系的分析结果(图2)显示,对于原始数据,地点S1、S2和S5之间高度相关,其中地点S1、S2的相关系数接近1,地点S4和S6间也高度相关;对于BLUP数据,地点S1、S2和S5之间仍高度相关,但地点S1和S2间的相关系数明显小于1,地点S4和S6间的相关程度也比原始数据弱。由此可见,BLUP数据可以校正试验地点间的相关关系。此外,两个主成分的方差解释百分比和,原始数据为70.51%,BLUP数据为87.50%,与表2结果一致,进一步说明BLUP数据GGE双标图的分析结果更为可靠。
GGE双标图的Which Won Where/What功能将最外围的基因型连成一个多边形,通过原点对多边形每条边做垂线,据此将试验地点分组,并得到各分组内的优秀基因型。分组结果(图3)显示,不论是原始数据还是BLUP数据,6个试验地点均分为2组,地点S4和S6为第1组,其余4个地点为第2组,分组结果与因子分析的多点结果[1]一致,说明GGE双标图的试验地分组结果是可靠的。优秀基因型筛选结果显示,2种数据在第1,2组试验地的种子最高产基因型分别是10和28。上述结果表明,虽然2种数据的试验地点分组一致,但分组内的优秀基因型存在差异。
2.3.2 试验地的区分力和代表性 试验地的选择与良种选育的可靠性直接相关,一个理想的试验地应具备较强的区分力和代表性。GGE双标图的Discrimitiveness VS.representativness功能可以展示试验地点的区分力和代表性。试验地评估结果见图4,图中各试验点与原点的虚线向量长度代表试验点的区分力,试验点向量与平均环境轴的夹度反映的是试验点的代表性,夹角越小表示试验点的代表性越强,如果夹角为钝角,则该试验地不适合作为试验点。由图4可知,对于原始数据,区分力最好的是地点S4和S3,代表性最好的是地点S5、S1和S2;对于BLUP数据,区分力最好的是地点S3、S4和S1,代表性最好的是地点S5、S1和S2。综合起来,原始数据最好的试验点是地点S5,BLUP数据最好的是地点S1。

S1~S6.表示6个试验地;第一主成分、第二主成分得分括号内的数据分别表示第一主成分和第二主成分解释的方差百分比。下图同S1-S6.Six trail sites;Data in brackets indicate the proportions explained by the first and the second principal components.The same below.图2 基于火炬松种子产量原始数据(左)和BLUP数据(右)的试验地点间关系Fig.2 Site relationship of original data (left) and BLUP data (right) of Pinus teada seed yield

1~36.表示36个基因型。下图同1-36. 36 genotypes. The same below图3 基于火炬松种子产量原始数据(左)和BLUP数据(右)的试验地分组Fig.3 Site grouping result of original data (left) and BLUP data (right) of Pinus teada seed yield
2.3.3 供试基因型的高产性和稳产性 GGE双标图的Mean VS.Stability功能可以展示供试基因型的高产性和稳产性。不同基因型火炬松的高产性和稳产性结果如图5所示,图中各基因型到平均环境轴的垂直虚线段代表各基因型在所有试验地的平均产量和稳产性,虚线段越长代表产量越不稳定;与平均环境轴垂直的实线为产量总体均值,在其左侧的基因型产量低于总体均值,距其越远产量越低,在其右侧的基因型产量高于总体均值,距其越远产量越高。由图5可知,对于原始数据,基因型28种子产量最高,其次是20,33,21和25;基因型9种子产量最低,其次是基因型32,2和31;基因型26和12种子产量接近总体均值;种子产量最不稳定的是基因型10,3,11和28,而基因型21,30和16产量比较稳定,其中16属于稳定但低产基因型。对于BLUP数据,基因型28种子产量最高,其次是33,20,21和19;基因型9种子产量最低,其次是32,2和31;基因型23种子产量接近总体均值;种子产量最不稳定的是基因型10,其次是3和28,而基因型21,30,25,22和16产量比较稳定,其中22和16属于稳定但低产基因型。综合来看,原始数据中最理想的基因型是21,BLUP数据的理想基因型也是21,该基因型种子产量既高又稳定。

图4 基于火炬松种子产量原始数据(左)和BLUP数据(右)的试验地的区分力和代表性Fig.4 Discrimination and representativeness of trial sites for original data (left) and BLUP data (right) of Pinus teada seed yield

图5 基于火炬松种子产量原始数据(左)和BLUP数据(右)的基因型高产性和稳定性Fig.5 Genotypic mean and stability of original data (left) and BLUP data (right) of Pinus teada seed yield
3 讨 论
GGE双标图的开发者严威凯[5]指出,目前AMMI和GGE双标图在多点试验分析中的主要限制有:仅限固定效应模型、各地点环境误差同质和平衡数据,这3个因素可能正是GGE双标图在林木选育和推广中应用较少的关键原因。但GGE双标图法在农业品种评价、试验点评价和品种生态区划分上已是先锋方法,林木良种的选育和推广也需要解决这3个问题,因此其在林业上的应用潜力很大。考虑到林木试验地环境差异往往比较大,GGE双标图要用于林木的遗传分析,首先要解决试验地环境的异质性问题。空间分析已成为解决环境异质性的主要方法[10],因此本研究采用空间分析结合因子分析法获取各基因型在每个地点中的BLUP数据,并对原始数据和BLUP数据的GGE双标图分析结果进行比较,结果表明空间分析可有效拟合试验地的空间变异,BLUP数据明显比原始数据有更强的空间变异趋势,方差分析的结果进一步确认BLUP数据对产量变异的解释能力要高于原始数据,这些结果说明BLUP数据已去除空间变异对目标性状的影响,即BLUP数据基本解决了环境异质性问题。原始数据和BLUP数据的GGE双标图分析结果表明,2种数据对试验地的分组结果一致,也与因子分析法的分组结果[1]一致,意味着GGE双标图对于试验地的分组结果是可靠的,但是BLUP数据中地点间的遗传相关被弱化了;同时,BLUP数据中两个主成分的方差解释百分比之和(87.50%)高于原始数据(70.51%),由此可知,BLUP数据的GGE双标图结果更为可靠。对试验地的评估结果表明,原始数据最好的试验点是地点S2,而BLUP数据是地点S1,虽然地点S1和地点S2之间的相关性比较大,但在BLUP数据中,地点S2的区分力明显偏弱,因此地点S1应为最理想的试验地。试验基因型的评估结果表明,虽然原始数据和BLUP数据中最理想的基因型均为21,但2种数据种子最高产的前5个基因型的一致性为80%,而产量稳定性强的前5个基因型的一致性仅为40%,说明2种数据的基因型评估差异较大。综上认为,GGE双标图可以用于林木多点试验分析,但需要通过空间分析解决地点环境异质性,因此采用BLUP和GGE双标图相结合的模型,无论是在试验地划分、试验地评估还是林木基因型评估上,均比原始数据的GGE双标图更为可靠。
[1] 林元震.R与ASReml-R统计学 [M].北京:中国林业出版社,2016:526-534.
Lin Y Z.R and ASReml-R statistics [M].Beijing:China Forestry Publishing House,2016:526-534.
[2] Finlay K W,Wilkinson G N.The analysis of adaptation in a plantbreeding programme [J].Aust J Agric Res,1963,14(6):742-754.
[3] Gauch H G,Zobel R W.Identifying mega-environments and targeting genotypes [J].Crop Sci,1997,37(2):311-326.
[4] Yan W.GGEbiplot:a Windows application for graphical analysis of multi-environment trial data and other types of two-way data [J].Agron J,2001,93(5):1111-1118.
[5] 严威凯.双标图分析在农作物品种多点试验中的应用 [J].作物学报,2010,36(11):1805-1819.
Yan W K.Optimal use of biplots in analysis of multi-location variety test data [J].ActaAgron Sin,2010,36(11):1805-1819.
[6] Ding M,Tier B,Yan W,et al.Application of GGE biplot analysis to evaluate genotype (G), environment (E) and G×E interaction onPinusradiata:a case of study [J].New Zealand J For Sci,2008,38(1):132-142.
[7] Sixto H,Salvia J,Barrio M,et al.Genetic variation and genotype-environment interactions in short rotationPopulus,plantations in southern Europe [J].New Forests,2011,42(2):163-177.
[8] Wang R H,Hu D H,Zheng H Q,et al.Genotype×environmental interaction by AMMI and GGE biplot analysis for the provenances ofMicheliachapensisin South China [J].J For Res,2016,27(3):659-664.
[9] Smith A,Cullis B,Thompson R.Analyzing variety by environment data using multiplicative mixed models and adjustments for spatial field trend [J].Biometrics,2001,57(4):1138-1147.
[10] Dutkowski G W,Costae S J,Gilmour A R,et al.Spatial analysis methods for forest genetic trials [J].Can J For Res,2002,32(12):2201-2214.
[11] Gilmour A R,Gogel B J,Cullis B R,et al.ASReml user guide release 4.0 [R].London:Vsn International Ltd Hemel,2016.
[12] Frutos E,Galindo M P,Leiva V.An interactive biplot implementation in R for modeling genotype-by-environment interaction [J].Stoch Environ Res Risk Assess,2014,28(7):1629-1641.
[13] Lin Y Z.AAfun:ASReml-R Added Functions.R package version 2.6.1[CP/OL].(2016-06-18)[2016-12-20].https://github.com/ yzhlinscau/AAfun.
