硫酸粘菌素对土壤反硝化细菌nirS、nosZ基因多样性的影响
2018-03-16范葶莉孙永学彭金菊牛金利钟晓霞王勉之马驿
范葶莉,孙永学,彭金菊,牛金利,钟晓霞,王勉之,马驿,*
1. 广东海洋大学农学院动物医学系,湛江 524088 2. 华南农业大学广东省兽药研制与安全评价重点实验室,广州 510642 3. 沧州职业技术学院畜牧兽医系,沧州 061001
环境介质中兽药的种类和含量随着畜禽和水产养殖业的快速发展逐年增加,兽药已成为土壤及水环境中一类重要的新型污染物。进入环境的兽药包括抗生素、消毒药、驱虫药、重金属及性激素等,其中抗生素在环境中残留对生态系统和人类健康的影响已逐步得到认识[1-4]。抗生素进入土壤、水和沉积物等环境介质,经吸附-解吸、迁移和降解等过程重新分配,已证实可影响环境介质中微生物的生物量、活性、功能和群落结构[5-8]。
硫酸粘菌素是人和动物广泛使用的一种抗生素,在临床中可用于防治家畜细菌性感染疾病,又可促进动物生长,内服难以吸收,大部分随粪便排出体外进入环境中。同时,研究表明随粪便排入环境中的硫酸粘菌素可导致土壤微生物群落结构多样性的改变,增加了微生物耐药性产生的几率[9]。土壤中氮素循环在生态环境中是一个不可或缺的过程,而反硝化作用又是氮素循环中的重要环节之一,研究反硝化细菌相应的编码基因就显得尤为重要。国内外已有报道利用DGGE、RFLP、Biolog等技术探讨反硝化细菌的群落结构多样性[10-13]。但是,目前国内关于硫酸粘菌素残留对生态环境影响的报道较少,本研究通过建立室内的土壤硫酸粘菌素暴露胁迫模型,对反硝化基因nirS、nosZ基因进行扩增、酶切、毛细管电泳扫描,以及末端限制性片段长度多态性分析(terminal restriction fragment length polymorphism, T-RFLP)分析,探讨硫酸粘菌素残留对反硝化细菌多样性的影响。
1 材料与方法(Materials and methods)
1.1 材料
土壤:采于广东海洋大学实验菜地,表层10~20 cm土壤。硫酸粘菌素(含量98%)购自山东鲁抗医药股份有限公司(批号150092214)。
1.2 方法
1.2.1 土壤处理与样品采集
新鲜土壤采集过4 mm筛后,测定土壤理化性质如下:pH 6.98;有机质24.65 g·kg-1;全氮2.41 g·kg-1;全磷6.82 g·kg-1;全钾6.2 g·kg-1;速效磷11.58 mg·kg-1;速效钾134 mg·kg-1。阴干后平均分成5组,每组3 kg。
加入硫酸粘菌素使土壤含药浓度分别为0 mg·kg-1、0.5 mg·kg-1、5 mg·kg-1和50 mg·kg-1,每组3个重复,调节土壤含水量至田间最大持水量的50%,置人工气候箱中培养,参数设为温度(28±1) ℃,湿度75%±7%,光照1 333 lx,间歇光照12 h∶12 h。分别于处理后第7、21、35、49天时采集土壤样品,各加药组均以同一采样时间的0 mg·kg-1组做对照。
1.2.2 土壤微生物总DNA提取
使用OMEGA试剂盒(美国)对不同周期土壤样品进行土壤微生物总DNA提取。
1.2.3 反硝化细菌nirS、nosZ基因扩增
采用PCR对目的基因片段进行扩增。引物序列为:NirS-F:5’-GTSAACGTSAAGGARACSGG(5’-FAM)和NirS-R:5’-GASTTCGGRTGSGTCTTGA;NosZ-1211:5’-CG(C/T)TGTTC(A/C)TCGACAGCCA(5’-FAM)和NosZ-1917:5’-CATGTGCAG(A/C/GT)GC(A/G)TGGCAGAA。50 μL扩增体系:25 μL MIX rTaq(TaKaRa,日本);2 μL Primer-F;2 μL Primer-R;4 μL总DNA;17 μL ddH2O。
nirS基因的PCR条件:94 ℃、2 min,94 ℃、3 min,56 ℃、1 min,72 ℃、1 min,36个循环,72 ℃、10 min,14 ℃保存。nosZ: 94 ℃、5 min,94 ℃、3 min,58 ℃、1 min,72 ℃、1 min,41个循环,72 ℃、5 min,14 ℃保存。
1.2.4 PCR产物限制性酶切及毛细管电泳扫描
对nirS、nosZ基因PCR产物限制性酶切,体系为:30 μL PCR产物,2 μL HaeIII(上海生工生物),2 μL 10×Buffer,6 μL ddH2O。上述酶切产物送上海生工技术有限公司进行毛细管电泳扫描。
1.2.5 数据统计与分析
使用Genemarker软件分析毛细管电泳结果,除去荧光信号小于100 RFU的峰,并且选取在3个重复中都出现的峰纳入统计[14]。
酶切图谱进行多样性指数分析使用Biodap软件,多样性指数进行方差分析采用SPSS6.12软件。
2 结果(Results)
2.1 nirS、nosZ基因PCR扩增
反硝化细菌nirS基因PCR扩增产物的目的片段大小为480 bp。nosZ基因PCR扩增产物的目的片段大小为700 bp左右。
nirS基因酶切产物扫描图谱(部分)如图1所示,各处理设置的3组重复的酶切图谱检出覆盖率均在90%以上。
2.2 反硝化细菌功能基因电泳图谱分析
2.2.1 nirS基因电泳图谱OTU个数与片段丰度
在酶切图谱中,每一个限制性片段(T-RFs)可判定为1种分类操作单元(Operational Taxonomic Units, OTU),对应的峰面积则反映出该种类的相对数量[15]。计算OTU的相对丰度值,公式:相对丰度值=单个峰面积/所有总峰面积。计算所得结果舍去小于50 bp和相对丰度小于1%的片段,并定义片段丰度值>4%为优势菌群,<1%为偶见菌群,其余则为非优势菌群[16]。不同处理组各采样点的总OTU个数与优势菌群个数见表1。每个处理组OTU总数均比对照组低,且各组随着浓度的升高逐渐降低,呈现剂量依赖效应。采样后期第35、49天时的总OTU个数均比采样前期低。而优势细菌种类无明显变化,在5~8种之间,但在49 d时优势数量降低。

图1 反硝化细菌nirS基因末端限制性片段长度多态性分析(T-RFLP)分析图谱(部分)Fig. 1 Terminal restriction fragment length polymorphism (T-RFLP) profiles of nirS gene (part)
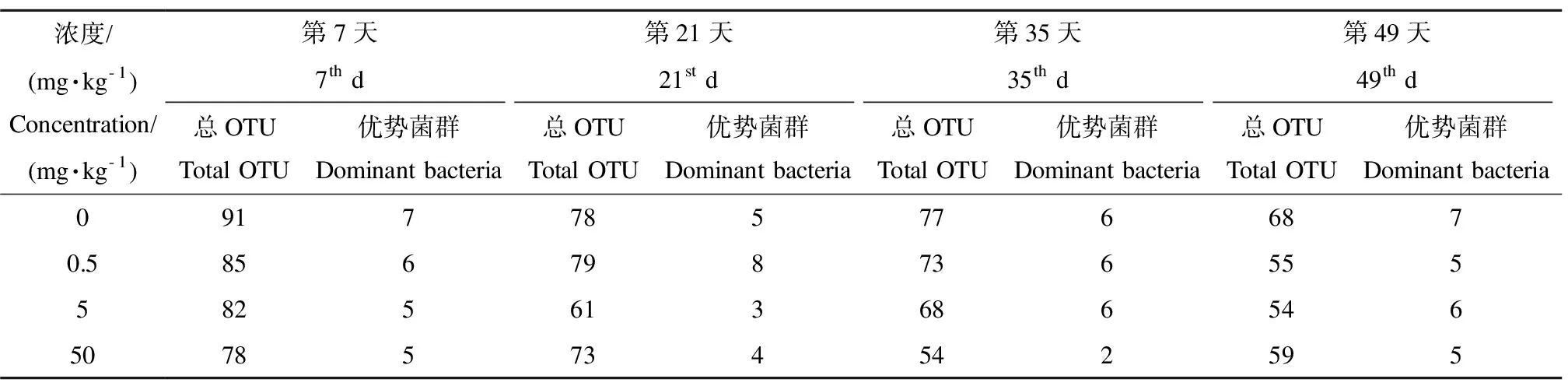
表1 反硝化细菌nirS基因总分类操作单元(OTU)个数与优势菌群个数Table 1 Total Operational Taxonomic Units (OTU) and dominant bacteria of nirS gene

图2 nirS基因优势菌群限制性片段(T-RFs)相对丰度注:图中组别中0、5表示硫酸粘菌素浓度,单位为mg·kg-1。下同。Fig. 2 Histograms of Terminal Restriction Fragments (T-RFs) relative abundances in dominant populations of nirS geneNote: the scale values on the axis of “Groups” mean 0, 5 mg·kg-1 colistin sulphate. The same below.
由图2可见,各组优势细菌主要集中在59 bp、73 bp、115 bp、116 bp、127 bp、132 bp、225 bp、259 bp、269 bp、405 bp片段中。81 bp、111 bp、364 bp片段的细菌只在采样前期的丰度较高,后期丰度检测不到或者从优势菌群变成偶见菌群;64 bp、80 bp、150 bp、197 bp、204 bp、224 bp、418 bp片段只在对照组丰度较高,而各处理组丰度较低。
2.2.2 nosZ基因电泳图谱OTU个数与片段丰度
由表2可见,各处理组OTU总数均比对照组低,但并未呈现剂量依赖效应。第35天、第49天的总OTU个数均比采样前期低,优势细菌种类无明显变化,在14~18种之间。
图3可见,各组在整个采样周期中都占有很高比例的片段有52 bp、96 bp、111 bp、112 bp、114 bp、133 bp、134 bp、209 bp;细菌只在采样前期的丰度较高的有79 bp、107 bp、212 bp,后期丰度检测不到或者从优势菌群变成偶见菌群;142 bp、151 bp片段只在对照组丰度较高,各处理组丰度较低。

表2 反硝化细菌nosZ基因总OTU个数与优势菌群个数Table 2 Total OTUs and dominant bacteria of nosZ gene

图3 nosZ基因优势菌群T-RFs相对丰度Fig. 3 Histograms of T-RFs relative abundances in dominant populations of nosZ gene
2.3 反硝化细菌功能基因多样性指数
2.3.1 nirS基因多样性指数
由表3可知,Shannon指数在第7天时,高浓度组较对照组差异显著(P<0.05);在第21、35天时,中、高浓度组较对照组差异显著(P<0.01);第49天时,各处理组较对照组差异均为显著(P<0.01)。Simpson指数在各处理组较对照组差异均显著(P<0.05)。Pielou指数在第7、49天时,高浓度处理组与对照组差异显著(P<0.05);第21天时,中、高浓度组与对照组差异显著(P<0.05);第35天时,中浓度组与对照组差异较明显(P<0.05)。

表3 nirS基因多样性指数Table 3 Diversity index of nirS gene
注:同列数据中,小写字母肩标不同,差异显著(P<0.05);大写字母不同,差异显著(P<0.01);相同字母或无字母,差异不显著(P>0.05)。下表同。
Note: In the same column, different small letter superscripts indicate significant difference (P<0.05); different capital letters indicate significant difference (P<0.01); the same or no letter superscripts indicate no significant difference (P>0.05). The same as below.
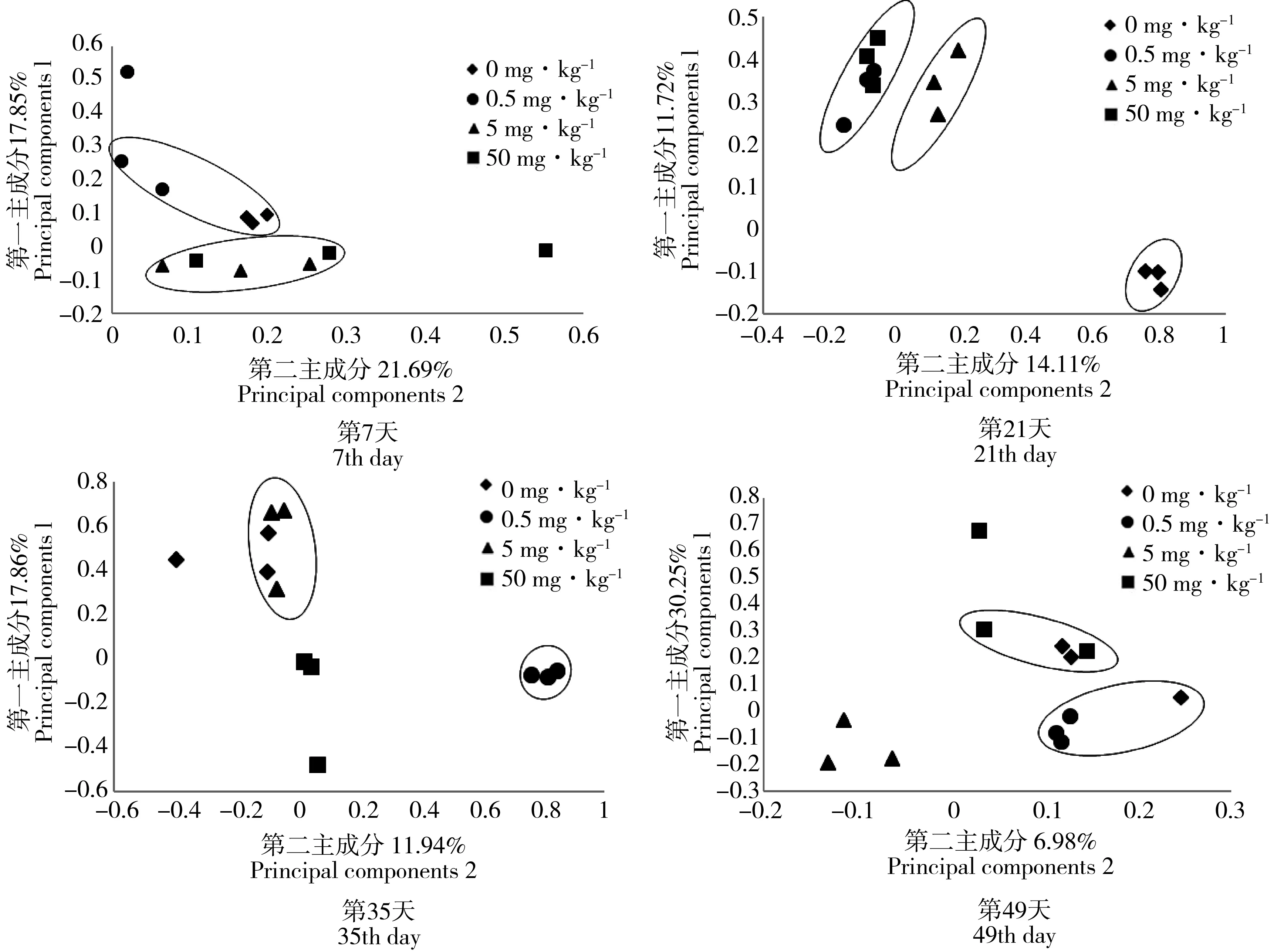
图4 nirS基因主成分分析Fig. 4 Principal components analysis of nirS gene
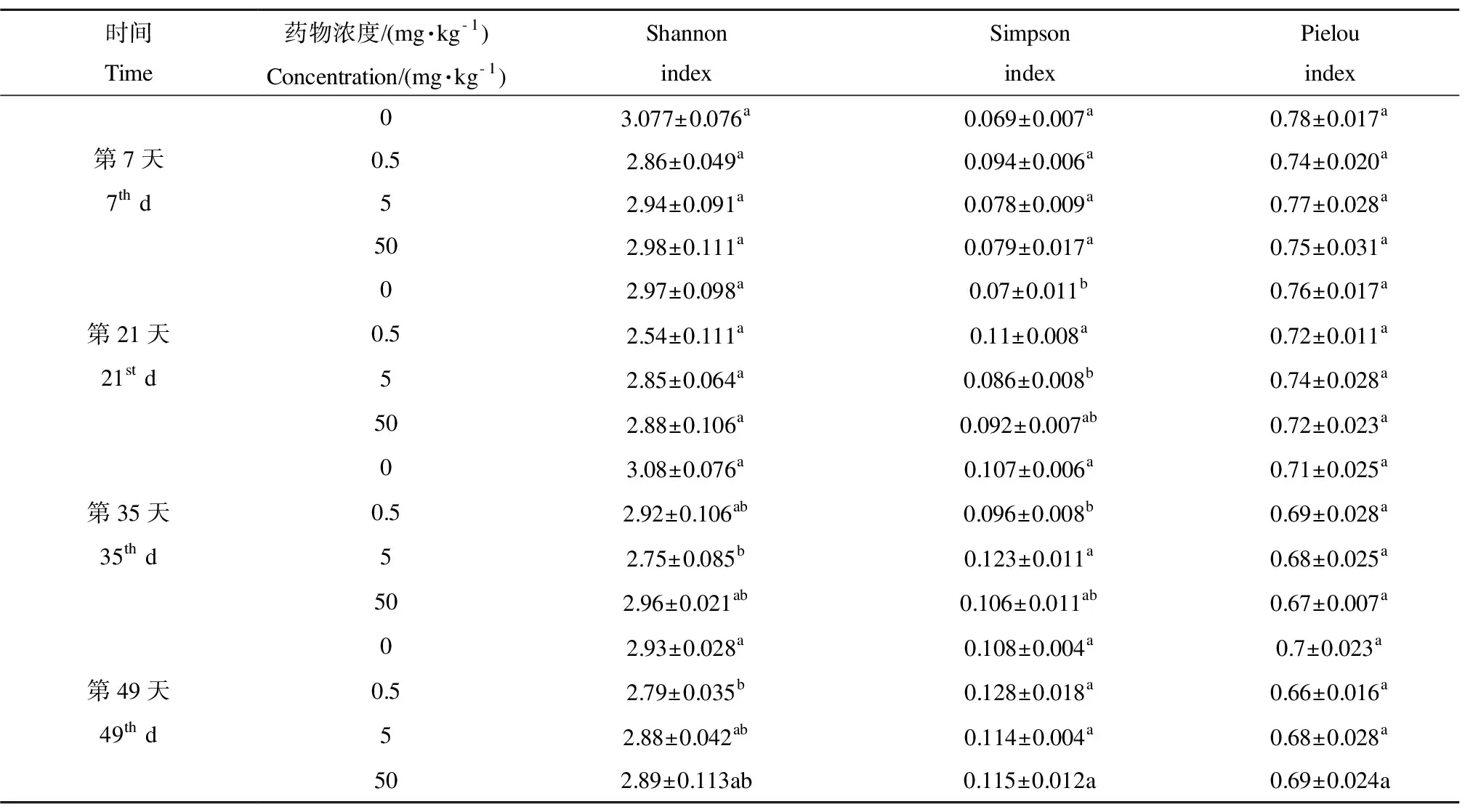
表4 nosZ基因多样性指数Table 4 Diversity index of nosZ gene
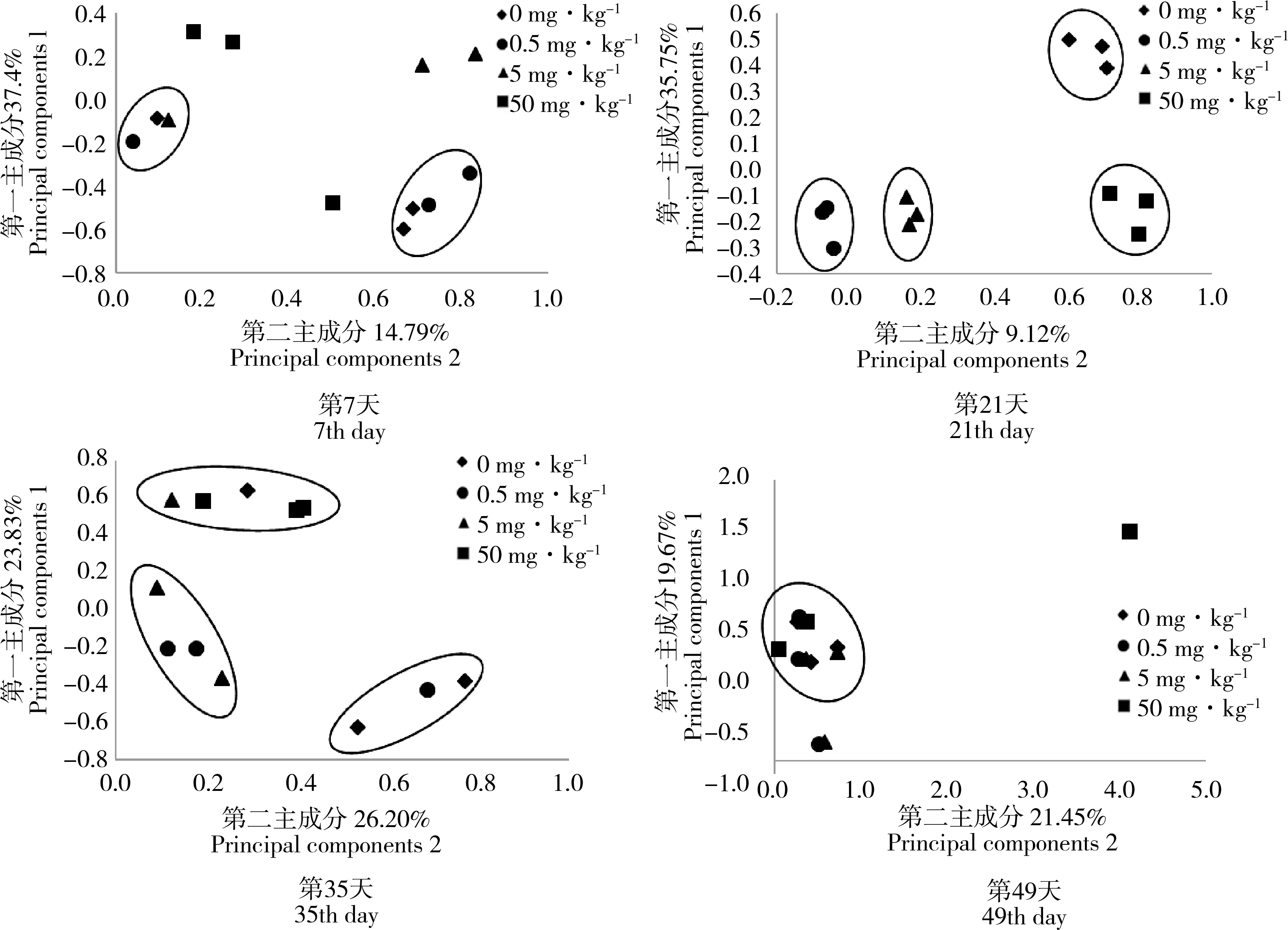
图5 反硝化细菌norZ基因主成分分析Fig. 5 Principal components analysis of norZ gene
2.3.2 nosZ基因多样性指数
nosZ基因多样性指数见表4。nosZ基因多样性在整个周期中呈现降低的趋势,但在第7、21天时,无显著差异(P>0.05);第35天时,中浓度组与对照组差异显著(P<0.05);第49天时,低浓度组与对照组差异显著(P<0.05)。Simpson指数在第7、49天时各组差异变化不显著(P>0.05),低浓度组升高较明显;第21、35天时,低浓度组与对照组差异显著(P<0.05);与Shannon指数结果相对应,说明硫酸粘菌素作用于土壤细菌对其优势度产生了影响。Pielou指数在各处理组之间差异均不显著(P>0.05),但各处理组较对照组均降低,说明硫酸粘菌素改变了土壤细菌的均匀度。
2.4 反硝化细菌功能基因主成分分析
2.4.1 nirS基因主成分分析
硫酸粘菌素对nirS基因的主成分分析(principal components analysis, PCA)结果见图4。4个图中的贡献和分别为39.54%、25.83%、29.80%、37.23%。在4个采样日期样品中,土壤微生物的群落结构特征均不相同,可能是硫酸粘菌素的作用改变了土壤微生物群落内部种群之间的关系。
2.4.2 nosZ基因主成分分析
硫酸粘菌素对norZ基因PCA分析见图5,4个图中的贡献和分别为52.19%、44.87%、50.03%和41.12%,基本可以代表整个系统状况。在前21天中,各处理组的差异均显著;第49天时,各处理组的主成分1和主成分2差异不显著(P>0.05),说明在第49天时,可能因硫酸粘菌素的迁移或降解导致浓度降低使细菌的生长活性出现了缓解。
3 讨论(Discussion)
土壤微生物是土壤环境生态系统的重要组成部分,在有机质分解、土壤养分循环等过程中具有重要作用[17],施肥、植物不同连作茬次、外来物种、污染物等对土壤微生物的多样性均会造成显著影响[18-19]。反硝化细菌群落丰度同样会受多种环境因素的影响,如pH、O2含量、含水量、养分状况、有机质含量、含氮量及其含氮素的种类等,具有不同功能基因型的反硝化细菌群落对环境因素的响应程度存在较大差异。本研究表明nirS、nosZ基因均受硫酸粘菌素影响。处理组nirS、nosZ基因的OTU总数均比对照组低,且nirS基因呈现剂量依赖效应。可能是硫酸粘菌素抑制了土壤反硝化细菌的生长,硫酸粘菌素为杀菌药,对革兰氏阴性菌的抗菌活性强,土壤中硫酸粘菌素为50 mg·kg-1时,被抑制或杀灭的细菌较多,多样性就越小。同时,分析结果显示nirS基因比nosZ基因丰度变化大。白玲[20]研究表明在洛克沙胂残留的土壤中,反硝化细菌nosZ基因丰度变化更大,本次试验结果与其相比有差异,可能是反硝化细菌对不同的药物敏感性不同,洛克沙胂是促生长添加剂,而硫酸粘菌素是抗菌型添加剂。
多样性指数结果表明,群落整体多样性随着剂量升高而降低,且nirS基因呈现了剂量依赖效应。当硫酸粘菌素≥5 mg·kg-1时,各组指数与对照组比较差异显著(P<0.05),高浓度组差异显著(P<0.01)。马驿和陈杖榴[21]研究了恩诺沙星对土壤反硝化细菌基因多样性的影响;张草[22]研究了洛克沙胂对池塘底泥微生物群落结构多样性的影响,结果显示药物作用致nirS基因多样性指数在采样前期均明显下降,而中后期群落结构出现一定程度的恢复现象。本次试验结果与上述研究结果相同,可能由于前期抗菌药导致土壤微生物数量减少,群落多样性降低,而随时间延长药物在土壤中降解,对土壤微生物的影响减弱,土壤微生物生态平衡逐渐恢复,nirS基因多样性的变化与土壤微生物数量的变化一致。PCA分析结果表明,硫酸粘菌素对nirS基因的群落结构影响更大。
T-RFLP图谱分析表明,在nirS基因图谱中,至少有3种细菌受硫酸粘菌素的影响呈现时间效应;硫酸粘菌素0.5 mg·kg-1组至少有7种细菌在第7天即被明显抑制。在nosZ基因图谱中,虽有3种细菌后期未检测到,但硫酸粘菌素抑制2种基因型细菌的种类并不相同。硫酸粘菌素浓度仅为0.5 mg·kg-1时,根瘤菌属细菌nirS、nosZ基因丰度明显降低。土壤中的反硝化细菌主要有假单胞菌属(Pseudomonadaceae)、产碱杆菌属(Chalmers),还有科奈瑟菌科(Neisseriaceae)、硝化细菌科(Nitrobacteriaceae)、红螺菌科(Rhodospirillaceae)、芽孢杆菌科(Bacillus)、螺菌科(Spirillaceae)、根瘤菌科(Rhizobiaceae)等,硫酸粘菌素对何种细菌影响最大,有待进一步对不同片段基因做克隆测序研究。
目前,研究抗生素的生态毒性多在实验室模拟的生态环境中进行,通常使用单因子变量来进行研究,条件可控。但在自然环境中存在多种影响因素,条件往往不可控,室内模拟实验与自然条件的结果有一定的差异。因此,为进一步探究抗生素对土壤微生物多样性的影响,需采集畜禽养殖实地环境中土壤垂直分布样本,同步进行抗生素残留浓度与氮素循环中硝化反硝化路径主要功能基因响应之间的量效相关性分析,为养殖源性抗菌药物残留的生态风险评估提供依据。
