高通量测序分析不同断奶时间对南方黄牛犊牛瘤胃细菌菌群的影响
2018-03-07盛笑昱羽中茅慧玲
杜 琪 ,盛笑昱 ,杨 斌 ,王 羽中*,茅慧玲 *
(1.浙江农林大学动物科技学院,浙江临安 311300;2.浙江大学奶业科学研究所,浙江杭州 310012)
断奶是反刍动物饲养过程中必须经历的一个阶段,而早期断奶是提高生产效率的技术手段之一。目前,犊牛断奶的标准主要有3种:根据体重断奶(占26.4%);根据日龄断奶(占43%);根据干物质采食量断奶(占26.9%) ,此法较为推荐。大型品种犊牛当连续3 d的干物质采食量达到700~1 000 g时即可断奶[1]。成功断奶很大程度上依赖于断奶前瘤胃的充分发育,因此,瘤胃发育状况可作为合理的断奶时间选择标准之一。反刍动物刚出生时主要依靠皱胃消化乳类为主的日粮[2],随之向反刍转变的过程需要瘤胃的不断发育和相关微生物的定植,从而利用植物性饲料[3]。幼龄反刍动物通过接触植物性饲料,刺激胃肠道尤其是瘤胃的发育,促进瘤胃微生物区系的建立[4]。Rey等[5]研究认为,瘤胃微生物的定植过程迅速,且为相继发生。然而,早期断奶幼龄反刍动物的瘤胃微生物定植规律尚未可知。因此,本研究以南方黄牛犊牛为试验对象,采用根据干物质采食量断奶的方法,利用高通量测序技术探讨不同断奶时间犊牛瘤胃细菌群落结构以及多样性的差异,进一步研究早期断奶犊牛瘤胃微生物的定植规律,为幼龄反刍动物实施早期断奶技术提供理论依据。
1 材料与方法
1.1 试验设计 选取刚出生的健康无疾病的南方黄牛犊牛18头,根据体重、出生日期相同或相近的原则,随机分为4组,对照组3头不早期断奶(Normal Weaned,NW),早期断奶组(Early weaned,EW)分为EW500组、EW750和EW1000组,每组5头牛。NW组犊牛随母牛一起生活,不饲喂开食料及代乳粉,并于120日龄断奶。3个早期断奶组逐渐增加开食料、青饲料的饲喂量,当犊牛连续 3 d每日进食固体饲料干物质分别达500 g(EW500)、750 g(EW750)和1 000 g(EW1 000)时,停喂代乳粉。
EW组犊牛出生后4 h内喝初乳,之后随母自由进食到6日龄。自7日龄起,每日饲喂乳液3 L,其中代乳粉1.5 L;14日龄起全部饲喂代乳粉,每日3 L直至断奶。自10日龄起,开食料和草料放在饲草中供犊牛自由采食。
各组犊牛断奶后,于晨饲前利用口腔取样装置[6]通过犊牛口腔采取瘤胃内容物。-80℃保存,用于提取DNA,测定瘤胃细菌的多样性。
1.2 试验日粮 试验日粮由代乳粉、开食料组成(北京精准牧业有限公司)。代乳粉营养成分见表1,开食料组成及营养成分见表2。

表1 代乳粉的营养成分
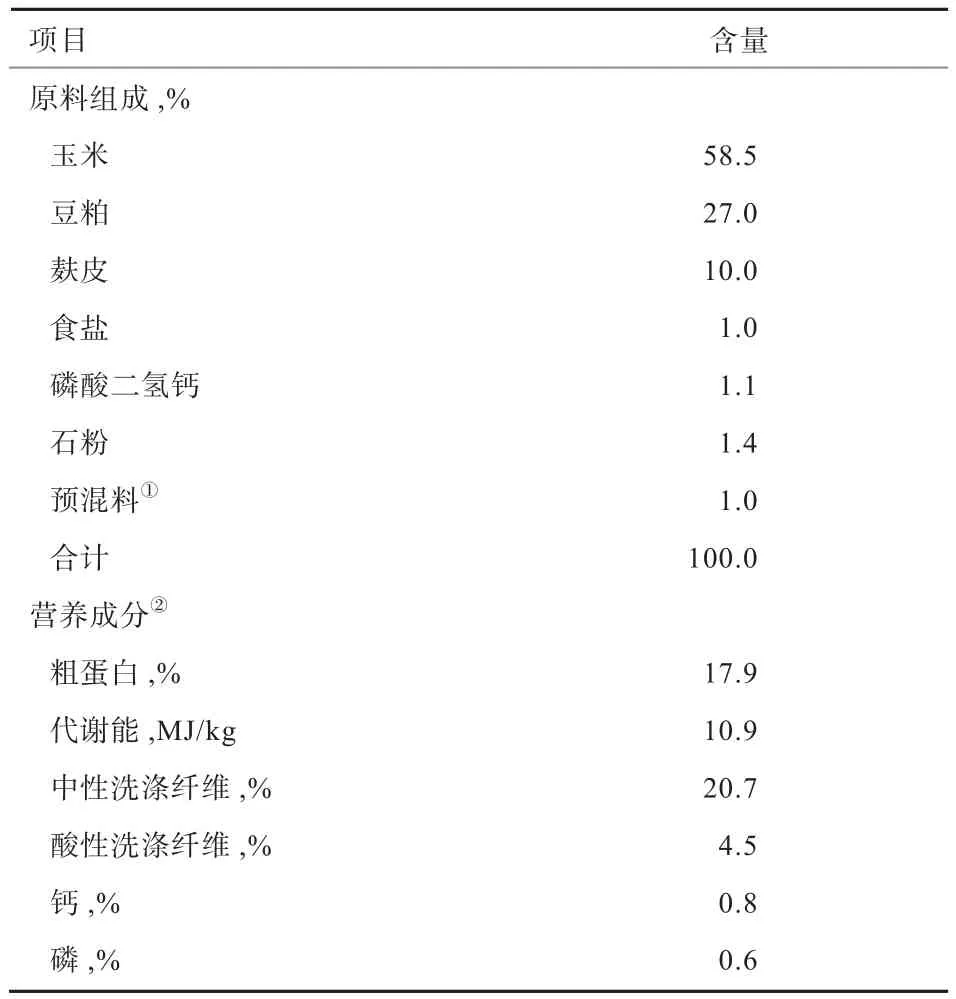
表2 0~3月犊牛开食料组成及营养成分(风干物质)
1.3 测定项目与方法
1.3.1 DNA提取 参照Gagen等[7]的珠磨法提取DNA。所有DNA样品用Qubit2.0(Invitrogen)测定浓度。
1.3.2 细菌16S rRNA基因序列扩增 16S rDNA基因序列扩增采用519F/907R通用引物[8]:上游为5'-GCCTC CCTCGCGCCATCAG-3';下游为5'-GCCTTGCCAGC CCGCTCAG-3'。为区分每个不同的样本,在通用引物的上游序列中随机添加8个核苷酸碱基的序列标签,即Barcoded-tag,形成Barcoded融合引物。PCR反应条件:95℃预变性5 min;95℃变性40 s,55℃退火40 s,72℃延伸1 min,共30循环;72℃延伸7 min[8]。
1.3.3 IlluminaMiSeq测序及下机数据分析 IlluminaMiSeq测序委托北京诺禾致源生物信息科技有限公司完成。
利用MOTHUR 软件[9]中的trim.seqs语句将Barcode错配数≥1、引物错配数≥2、序列最短长度<200 bp、最大模糊碱基数≥1的下机数据去除,并用该语句去除Barcode和primer,除杂后的文件以FASTA序列文件保存。去杂后的数据与Silva 数据库中的小核糖体RNA全长序列进行比对。比对后的序列利用MOTHUR软件中的screen.seqs语句进行二次去杂。利用MOTHUR软件中的chimera.uchime语句去除嵌合序列。
利用 MOTHUR 软件中的filter.seqs和cluster语句,以16S rRNA基因序列distance对去杂及去嵌合体后的序列进行OTU聚类。利用RDP(Ribosomal Database Project)分类器赋予每条序列物种单元分类,物种分类单元分为6层,依次为domain、phylum、class、order、family、genus。RDP分类对genus的域值为0.8。
测序深度评估:利用MOTHUR软件计算每个样品在OTU=0.03水平下的稀疏曲线(Rarefaction Curve),用于评估不同样本之间的丰度。
Alpha多样性评估:利用MOTHUR软件计算Chao、Shannon和Simpson指数。
Beta多样性评估:利用在线软件http://bioinforma tics.psb.ugent.be/webtools/Venn/绘 制 韦 恩(Venn)图,用于显示不同处理之间共有和特有的OTU情况;用jest和thetayc算法计算每个样品在OTU=0.03水平下的群落结构的相似度;通过计算所有处理间的生态距离,绘制主坐标分析PCoA(Principal Coordinate Analysis)图。
1.4 统计分析 数据采用Excel2010进行整理,结果采用SAS 8.0统计软件的Duncan´s方差分析对各组进行多重比较,以P<0.05作为差异显著性判断标准。
微生物各门属和OUT需在每个处理组的至少3头犊牛中出现(如果有组别少于或等于3头,则需该组犊牛中都出现)。
2 结果与分析
2.1 多样性分析 4个处理组瘤胃内容物中细菌的稀疏曲线见图1,虽然没有得到完全的平缓,但斜率在不断减小,说明趋向于饱和。

图1 瘤胃内容物细菌16S rRNA基因序列的稀疏曲线
4个处理组的OTU覆盖率均在95%以上(表3),能够很好地反映样品菌群情况。其中EW组的覆盖率显著高于NW组(P<0.05),说明EW组的饱和性好于NW组。NW组犊牛瘤胃内容物的OTU数、Chao指数和shannon指数显著高于EW组(P<0.05),即NW组瘤胃内细菌菌群丰度和多样性高于EW组。
基于UniFrac的加权主坐标分析其第1主成分和第2主成分的贡献率分别为20.35%和10.98%(图2)。EW组的犊牛个体差异较为显著,分布较为涣散,然而EW组和NW组分离较远,说明早期断奶会给瘤胃细菌分布造成差异。
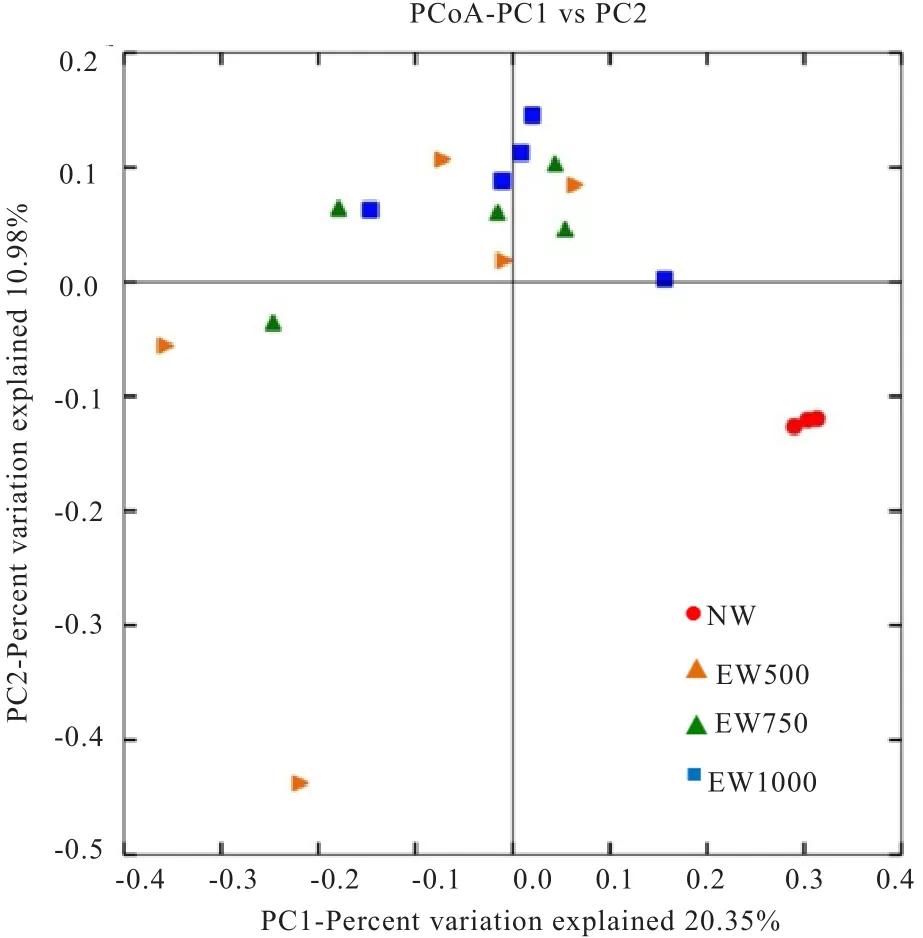
图2 瘤胃内细菌结构Unifrac加权主坐标分析图

图3 不同断奶时间犊牛瘤胃液中细菌群落共享和独有的OTU的Venn分析
不同处理之间共有和特有的OTU 情况见图3,4组共有OTU数为204个,NW组特有240个OTU,而EW组特有的OTU数较少,EW500、EW750和EW1000组特有的OTU数分别为59、72和83。
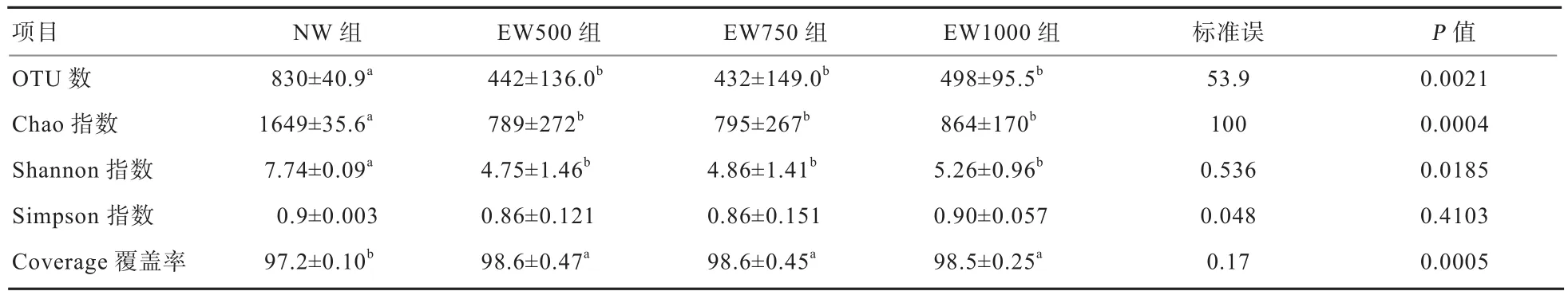
表3 97%相似性水平下物种丰富度和多样性指数
2.2 物种分类 由表4可知,瘤胃内细菌主要归于厚壁菌门、拟杆菌门和变型菌门三大类。本研究中相对含量大于0.1%的门共有7个。统计结果表明,NW组瘤胃内细菌最多的是厚壁菌门(53.9%),显著高于EW750组和EW1000组(P<0.05);与NW组相比,EW组拟杆菌门相对较高,且在EW750组和EW1000组达显著水平(P<0.05)。
由表5可知,在相对含量大于0.1%的菌属中,各组中均以普雷沃氏菌属、瘤胃球菌属和丁酸弧菌属为优势菌属,且普雷沃氏菌占比例最多。EW组的普雷沃氏菌属较NW组有所增加(P>0.05)。NW组丁酸弧菌属显著低于EW500组(P<0.05)。NW组分支杆菌属和颤螺菌属显著高于EW组(P<0.05)。EW组琥珀酸菌属显著高于NW组(P<0.05)。纤维杆菌属以EW750组最多(0.46%;P<0.05)。各组间瘤胃球菌属、月形单胞菌属、梭菌属和粪球菌属相对含量无显著差异(P>0.05)。
3 讨 论
前期研究结果发现,早期断奶有利于提高犊牛的生长性能,促进瘤胃发酵,增加挥发性脂肪酸含量,降低氨态氮浓度,提高瘤胃内源性酶活,改变瘤胃纤维降解菌的组成,且不同断奶时间存在一定差异[10]。然而,瘤胃微生物大多为未培养微生物,依靠常用的荧光定量PCR技术并不能充分反映瘤胃细菌菌群的变化,只局限于一定丰度细菌或特定微生物,对未知微生物均未涉及。本试验应用Illumina-MiSeq高通量测序技术,突破传统的培养方法和分子生物学方法,可以获得大量测序数据,通过比对或聚类分析,研究瘤胃微生物群落物种组成的变化,同时探寻一些低丰度或未知的细菌,能更加全面了解早期断奶对幼龄反刍动物瘤胃细菌菌群的影响以及不同断奶时间造成的差异。
对于成年牛而言,瘤胃是一个非常重要的发酵场所,其中含有复杂的厌氧微生物,包括细菌、古菌、真菌、原虫和病毒。这些微生物可以利用饲料生产出挥发性脂肪酸、氨态氮和微生物蛋白等;还可与宿主产生互作,影响宿主的健康、生产性能等[5]。然而,犊牛刚出生时瘤胃尚未发育,瘤胃内处于无菌且无胚芽状态,随着日粮的摄入及外界环境中的微生物进入瘤胃,逐渐形成复杂多样的微生物区系[11]。大多数瘤胃细菌如蛋白分解菌、纤维素分解菌和其他种类细菌在14日龄犊牛的瘤胃微生物区系中即可发现[12]。Jami等[13]认为,瘤胃菌群同时受日粮和年龄的影响。本研究各组优势菌门为厚壁菌门和拟杆菌门,与前人研究结果一致[14]。Thoetkiattikul等[15]研究发现,饲喂高纤维(88%)和低淀粉(2%)饲粮的奶牛瘤胃内拟杆菌门和厚壁菌门含量分别高达66.53%和24.98%。Rey等[5]对奶牛从出生至断奶时期瘤胃菌群的定植进行研究,发现犊牛2日龄时瘤胃细菌主要包含变形菌门(70%)和拟杆菌门(14%),之后拟杆菌门随日龄增长逐渐取代变形菌门。厚壁菌门中多数为梭菌目,具有纤维消化作用。本研究结果显示,EW组的厚壁菌门较NW组少,可能原因是与NW组未采食开食料相比,EW组10日龄即开始采食开食料,饲粮的摄入改变了菌群组成。
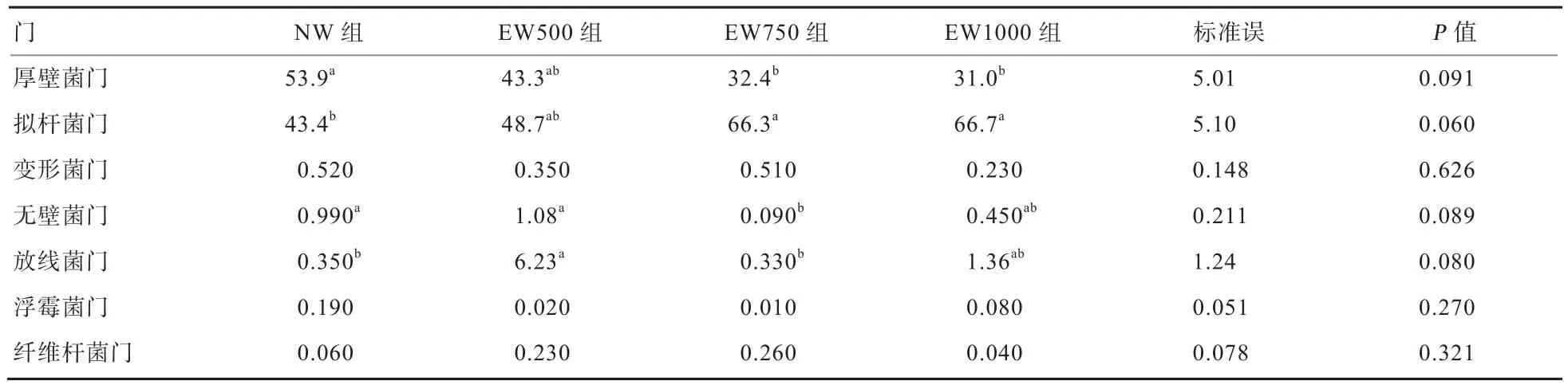
表4 相对含量大于0.1%(序列占测序总量比例)的菌门
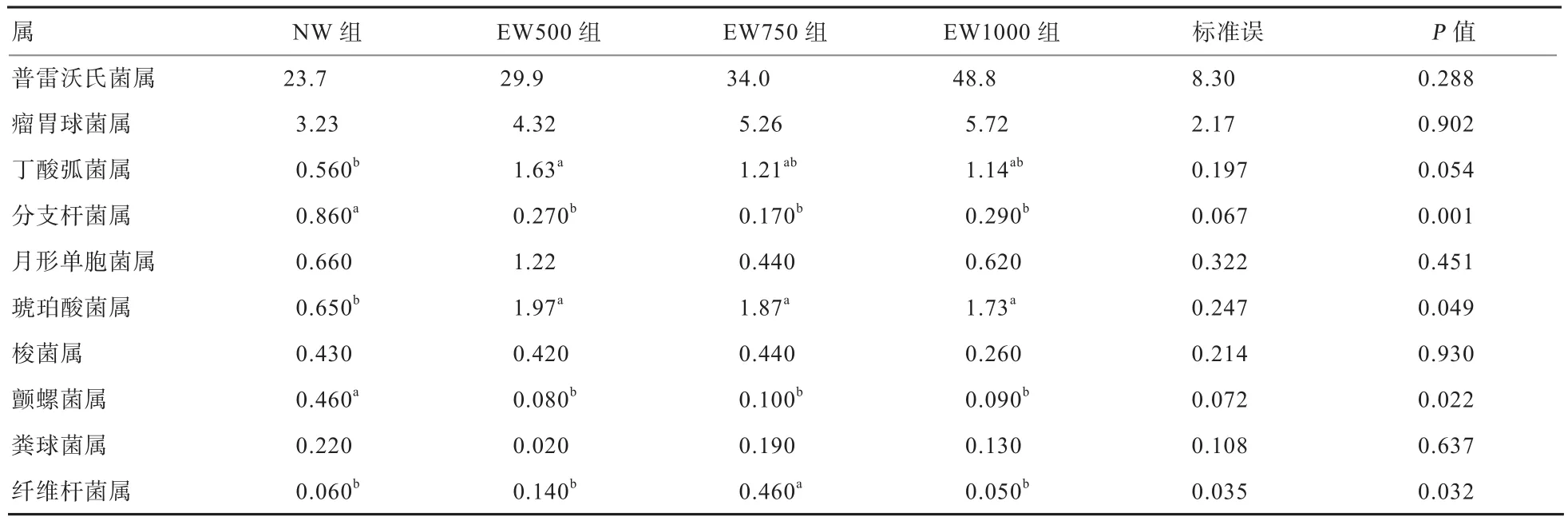
表5 相对含量大于0.1%(序列占测序总量比例)的菌属
奶牛瘤胃中普雷沃氏菌属、丁酸菌属、纤维杆菌属和密螺旋体属等为优势菌属[16],与本研究的结果有所差异,这可能是试验动物不同所造成。Wood等[17]利用纯培养的方法分离出普雷沃氏菌属,分析能够占到瘤胃细菌的60%,它拥有高活性的半纤维分解菌[18],可以降解植物非纤维多糖和蛋白质[19]。Rey等[5]研究认为,在15~83日龄,随着固体饲料的摄入,普雷沃氏菌属占有主导地位。利用454焦磷酸测序及分类学分析鹿[20]和成年奶牛[13]瘤胃菌群,结果也显示普雷沃氏菌属是优势菌属。本研究结果与这些报道较为一致,与NW组相比,EW组犊牛瘤胃中普雷沃氏菌属增多。这些结果说明犊牛越早采食固体饲料,其瘤胃菌群越接近于成年牛。丁酸弧菌属是瘤胃中主要的产丁酸菌,同样也是主要的半纤维素降解菌[21],琥珀酸菌属虽然不能发酵碳水化合物和氨基酸,但能将琥珀酸盐转化为丙酸[22]。本研究发现,EW500组的丁酸弧菌属和琥珀酸菌属较NW组均显著增加,该两菌属的变化与瘤胃内挥发性脂肪酸丁酸和丙酸含量的结果一致[10]。挥发性脂肪酸是经由瘤胃上皮代谢的,它对瘤胃容积和瘤胃乳头的发育有刺激作用,能够促进瘤胃的发育[23]。由此可见,与传统饲喂相比,尽早接触固体饲料可以使犊牛瘤胃在断奶前就得到较好的发育,为断奶后的健康生长奠定基础。Li等[24]首次在瘤胃上皮发现分支杆菌属的定植,并认为该菌属没有发酵饲料的功能。此外,有研究报道在动物饲喂高精料日粮时分支杆菌属对瘤胃上皮的健康有负面影响[25]。本试验中分支杆菌属在EW组显著降低,其原因有待进一步探究。
然而,本研究发现早期断奶3个组间瘤胃微生物变化并不明显,可能原因是这3个组同时采食固体饲料,且饲料组成相同。
4 结 论
早期断奶可以改变犊牛的瘤胃细菌多样性和物种丰度,丁酸弧菌属、琥珀酸菌属、纤维杆菌属增加,分支杆菌属降低,而3个早期断奶组间差异并不显著。从瘤胃微生物角度考虑,推荐在犊牛连续3 d每日进食固体饲料干物质达500 g时断奶。
[1] Morrill J L. The calf: birth to12 weeks[M]//Large dairy herd management. Sweden: Hawkesbury Agricultural College, 1992:401.
[2] Davis C L, Drackley J K.The development, nutrition, and management of the young calf[M]. Ames IA: Iowa State University Press,1998.
[3] Heinrichs J. Rumen development in the dairy calf[J]. Adv Dairy Technol, 2005, 17:179-187.
[4] 马志远, 李飞, 李发弟, 等. 早期断奶对湖羊羔羊生长性能及胃肠道发育的影响[J]. 动物营养学报, 2015, 27(5):1385-1393.
[5] Rey M, Enjalbert F, Combes S,et al. Establishment of ruminal bacterial community in dairy calves from birth to weaning is sequential[J]. J Appl Microbiol, 2014, 116(2):245-257.
[6] Shen J S, Chai Z, Song L J,et al. Insertion depth of oral stomach tubes may affect the fermentation parameters of ruminal fluid collected in dairy cows[J]. J Dairy Sci, 2012, 95(10):5978-5984.
[7] Gagen E J, Denman S E, Padmanabha J,et al. Functional gene analysis suggests different acetogen population in the bovine rumen and tammar wallaby forestomach[J]. Appl Environ Microbiol, 2010, 76:7785-7795.
[8] Wang Y, Qian P Y. Conservative fragments in bacterial 16S rRNA genes and primer design for 16S ribosomal DNA amplicons in metagenomicstudies[J]. PLoS One, 2009,4:e7401.
[9] Schloss P D, Westcott S L, Ryabin T,et al. Introducing mothur: open-source, platform-independent, communitysupported software for describing and comparing microbial communities[J]. Appl Environ Microbiol, 2009, 75(23):7537-7541.
[10] Mao H L, Xia Y F, Tu Y,et al. Effects of various weaning times on growth performance, rumen fermentation and microbial population[J]. Asian-Aust J Anim Sci, 2017, 30:11-12.
[11] Ziolecki A, Briggs C A.The microflora of the rumen of young calf: II. Source, nature and development[J]. J Appl Bacteriol,1961, 24(2):148-163.
[12] Li M, Zhou M, Adamowicz E,et al. Characterization of bovine ruminal epithelial bacterial communities using 16S rRNA sequencing PCR-DGGE and qRT-PCR analysis[J]. Veter Microbiol, 2012, 155:72-80.
[13] Jami E, Israel A, Kotser A,et al. Exploring the bovine rumen bacterial community from birth to adulthood[J]. ISME J, 2013,7(6):1069-1079.
[14] Wu S T, Baldwin R L, Li W Z,et al. The Bacterial community composition of the bovine rumen detected using pyrosequencing of 16S rRNAgenes[J]. Metageno, 2012, 1:1-11.
[15] Thoetkiattikul H, Mhuantong W, Laothanachareon T,et al.Comparative analysis of microbial profiles in cow rumen fed with different dietary fiber by tagged 16S rRNA gene pyrosequencing[J]. Curr Microbiol, 2013, 67(2):130-137.
[16] 徐俊, 胡丽芳, 侯玉洁, 等. 不同牧草来源饲粮对奶牛瘤胃液中细菌群落结构多样性的影响[J].动物营养学报, 2015,27(11): 3549-3557.
[17] Wood J, Scott K P, Avgustin G,et al. Estimation of the relative abundance of different Bacteroides and Prevotellaribotypes in gut samples by restriction enzyme profiling of PCR-amplified 16SrRNA gene sequences[J]. Appl Environ Microbiol, 1998,64:3683-3689.
[18] Matsui H, Ogata K, Tajima K,et al. Phenotypic characterization of polysaccharidases produced by four Prevotella type strains[J]. Curr Microbiol, 2000, 41(1):45-49.
[19] Kamrad N. Rumen microbial ecosystem[J]. Curr Sci, 2005,89(1):124-135.
[20] Pitta D W,Pinchak W E, Down S E,et al. Rumen bacterial diversity dynamics associated with changing from bermudagrass hay to grazed winter wheat diets[J]. Microbial Ecol, 2010, 59(3):511-522.
[21] Diez-Gonzalez F, Bond D R, Jennings E,et al. Alternative schemes of butyrate production in Butyrivibrio fibrisolvens and their relationship to acetate utilization, lactate production, and phylogeny[J]. Arch Microbiol, 1999, 171(5): 324-330.
[22] Van Gylswyk N O. Succiniclasticumruminis gen nov, spnov, a ruminal bacterium converting succinate to propionate as the sole energy-yielding mechanism[J]. Inter JSystemBacteriol,1995, 45(2):297.
[23] Paez Lama S, Grilli D, Egea V,et al. Rumen development and blood metabolites of Criollo kids under two different rearing systerms[J]. Livest Sci, 2014, 167:171-177.
[24] Li M, Zhou M, Adamowicz E,et al. Characterization of bovine ruminal epithelial bacterial communities using 16s rrna sequencing, pcr-dgge, and qrt-pcr analysis[J]. Veter Microbiol,2012,155(1):72-80.
[25] Liu J H, Bian G R, Zhu W Y,et al. High-grain feeding causes strong shifts in ruminal epithelial bacterial community and expression of toll-like receptor genes in goats[J]. Front Microbiol, 2015, 6: 167.
