人源CBX7全长及部分片段的表达纯化及单体结构研究
2018-03-01吕缜一陆昌瑞
吕缜一,陆昌瑞
(东华大学 化学化工与生物工程学院,上海 201600)
CBX7是存在于多梳蛋白家族(Polycomb group,PcG)中多梳抑制复合物1(Polycomb repressive complex 1,PRC1)上,对细胞的增殖及衰老起到调控作用的转录抑制因子[1]。人源CBX7蛋白位于人类染色体22q13.1上,由251个氨基酸组成,全长约28.3 KDa[2]。其能直接参与PcG复合物对其他CBX蛋白家族编码基因的转录抑制[3],且和人类的多种癌症和肿瘤的发生都有着一定联系[4-5]。到目前为止,对人源CBX7蛋白结功能的研究报道并不多,对其结构的研究仅有两篇报道[6-7]。
本实验通过对人源CBX7蛋白的二级结构预测,设计出相应的片段进行ppSUMO-CBX7/domain融合蛋白的构建[8-10],通过一系列纯化方法得到蛋白单体,为CBX7蛋白结构及功能的研究及PcG家族蛋白成员的研究奠定了基础[11]。
1 材料与方法
1.1 材料
载体:pp-SUMO表达载体(含6个His标签);菌种:DH5α、BL21感受态细胞,CBX7 Template;试剂:K+、IPTG、各种酶及各种分析纯等。
1.2 方法与步骤
1.2.1 引物的设计
根据预测得到人源CBX7的二级结构和该蛋白内在无序分布图,设计出了相应的引物及需要表达的片段[8-10],见表1。

表1 引物的设计
1.2.2 ppSUMO-CBX7/domain表达载体的构建
将PCR扩增后的CBX7/domain和SUMO质粒一同用BamH1、Xhol1酶切;再将CBX7/domain与SUMO用T4连接酶连接后转入DH5α感受态中培养,挑取单克隆再次转入BL21感受态保菌备用。
1.2.3 ppSUMO-CBX7/domain融合蛋白的表达及Ni柱亲和纯化
在37℃、225 r/min的条件下培养构建好的工程菌,其OD600到0.6时,向培养基中加入适量IPTG诱导。取菌液经4℃,3 500 r/min离心收集细胞,用 Ni-NTA Binding Buffer(500 mmol/L NaCl,pH值根据片段PI确定)重悬。用高压细胞破碎仪破碎细胞后将悬液于4℃,12 000 r/min离心1 h。使上清与Ni结合;用Wash Buffer(设置Imidazole浓度梯度,500 mmol/L NaCl,pH值根据片段PI确定)洗脱;接着用Elution Buffer(250 mmol/L Imidazole,500 mmol/L NaCl)洗脱目标蛋白;最后 用 Strip Buffer(1mol/L Imidazole,500 mmol/L NaCl)清洗 Ni柱。
1.2.4 ppSUMO-CBX7/domain离子交换纯化
经过Ni柱亲和纯化后,若存在和目标蛋白分子量大小相近的杂蛋白,需要通过离子交换纯化进行再次纯化。根据蛋白质的PI选取Q_SP_FF Prepacked预装柱,通过 Buffer A(50 mmol/L Tris-HCl,1 mmol/L DTT,1 mmol/L EDTA)柱平衡、进样、柱冲洗、Buffer A+Buffer B(50 mmol/L Tris-HCl,1 mmol/L DTT,1 mmol/L EDTA,1 mol/L NaCl)梯度洗脱的程序设定纯化蛋白。
1.2.5 ULP1酶切及酶切后Ni柱亲和纯化
将纯化好的目标蛋白加入10mL Buffer A+1 mmol/L DTT+5 μL ULP1酶的体系进行酶切,切除SUMO载体和His标签。将酶切后的样品再次用Ni柱纯化,得到不含有残基的CBX7/domain目标蛋白[11]。
1.2.6 分子筛纯化
使用GE公司Superdex 75 10/300 GL预装柱,按照 Buffer A(50 mmol/L Tris-HCl,1 mmol/L DTT,1 mmol/L EDTA,500 mmol/L NaCl)柱平衡、上样、洗脱的步骤进行纯化。
2 结果与讨论
本次实验使用PCR、双酶切、链接、转化等方法,构建了ppSUMO-CBX7(13-65aa)、ppSUMO-CBX7(51-151aa)和ppSUMO-CBX7(Full-Length)三个蛋白质片段。
2.1 ppSUMO-CBX7/domain蛋白的表达及Ni柱亲和纯化条件
根据每个蛋白的自身性质不同,改变诱导温度及IPTG浓度对3个蛋白进行诱导条件的探索;根据每个蛋白的PI不同,对Ni柱亲和纯化时Buffer的PH值进行探索[12-13]。确定ppSUMO-CBX7(13-65aa)的最适诱导温度为37℃,IPTG浓度为1 mmol/L,Buffer的 pH值为7.5;ppSUMO-CBX7(51-151aa)的最适诱导温度为18℃,IPTG浓度为0.5 mmol/L,Buffer的pH值无论是多少目标蛋白都不可溶;ppSUMO-CBX7(Full-Length)的最适诱导温度为18℃,IPTG浓度为1 mmol/L,Buffer的pH值为8.5。
随后,根据融合蛋白对Imidazole的敏感度不同进行了蛋白Ni柱亲和纯化Wash的Imidazole浓度探索,Imidazole浓度在20~80 mmol/L的范围内设置7个梯度。根据条件探索后发现:ppSUMO-CBX7(13-65aa)的Wash最适Imidazole为 20 mmol/L,ppSUMO-CBX7(Full-Length)的Wash最适Imidazole浓度为60 mmol/L。
选取每段融合蛋白的最适诱导条件及纯化条件进行亲和纯化,将纯化结果用SDS-PAGE胶检,得到图1,该结果为Western Bolt所验证。

图1 ppSUNO-CBX7全长及片段的Ni柱亲和纯化SDS-PAGE胶检
2.2 ppSUMO-CBX7/domain融合蛋白的离子交换纯化
因为ppSUMO-CBX7(13-65aa)的亲和纯化效果较好,可直接酶切,所以离子交换纯化只针对ppSUMO-CBX7(Full-Length),经过离子交换纯化的SDS-PAGE胶检如图2所示。
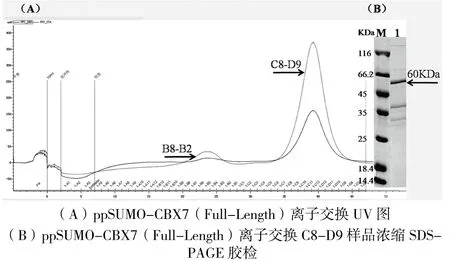
图2 ppSUNO-CBX7全长的离子交换纯化
2.3 ppSUMO-CBX7/domain融合蛋白的UPL1酶切及Ni柱亲和纯化
对 ppSUMO-CBX7(13-65aa)、ppSUMOCBX7(Full-Length)进行ULP1酶切,经过SDSPAGE和SDS-Tricine PAGE胶检得到单一目标蛋白,如图3所示。
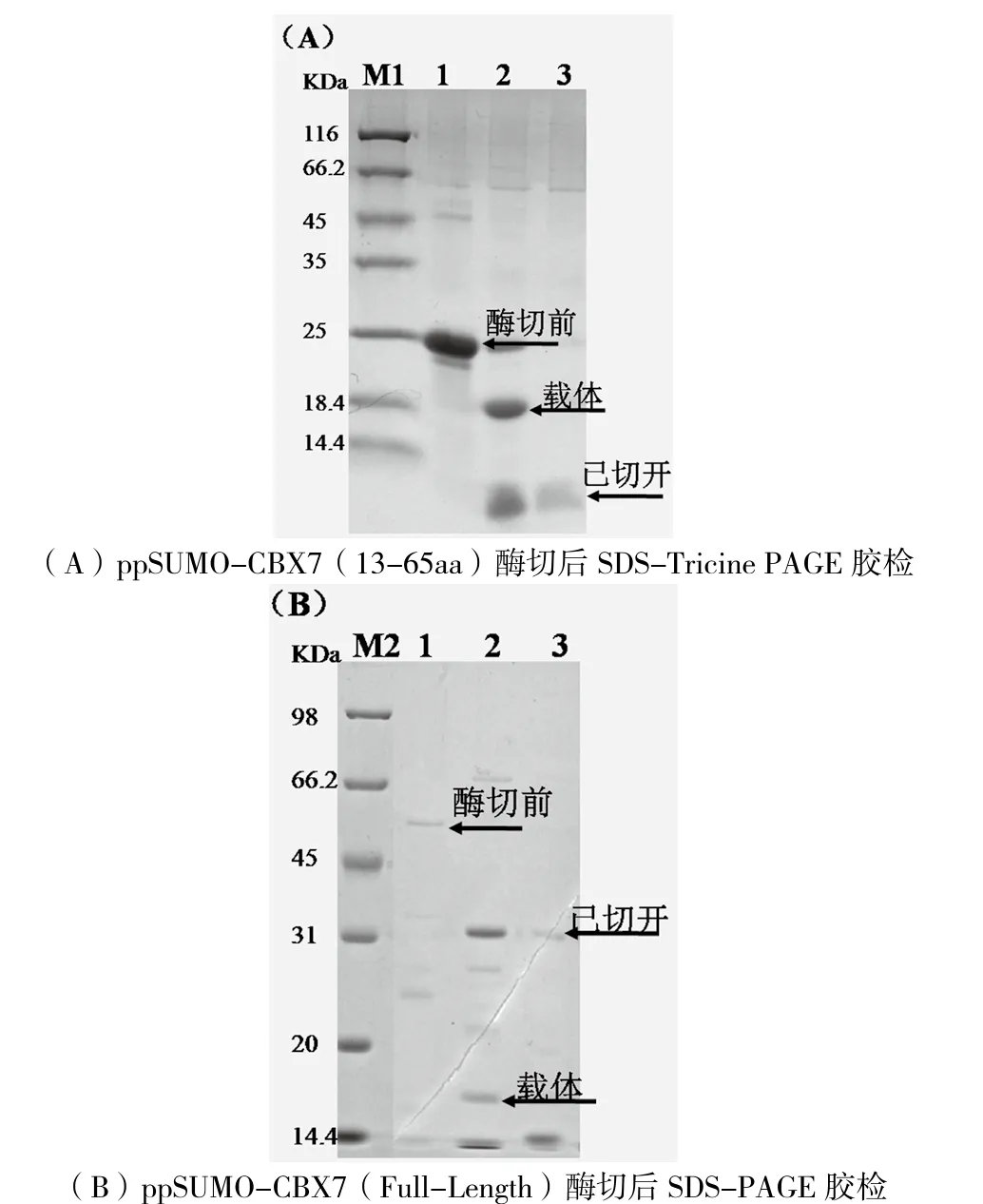
图3 ppSUMO-CBX7 ULP1酶切检测
2.4 CBX7/domain 蛋白的分子筛纯化
酶切后的样品浓度达到2 mg/mL才适合做分子筛,而CBX7(Full-Length)浓度在1 mg/mL以下,所以只针对CBX7(13-65aa)做分子筛纯化及SDS-Tricine PAGE胶检,结果如图4所示。
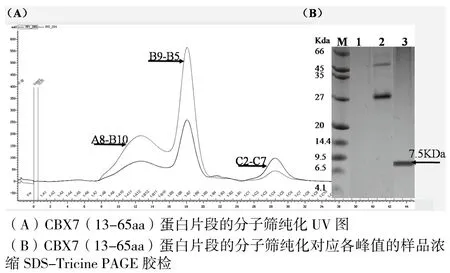
图4 CBX7(13-65aa)蛋白的分子筛纯化检测
3 结论
CBX7蛋白在人类的多种肿瘤和癌症中都有着重要的作用,但就目前而言对CBX7的研究无论是结构还是功能的报道都很少。通过Psipred网站对人源CBX7蛋白进行二级结构及功能区进行预测,根据相关文献的报道及预测结果设计了引物。通过多种纯化手段得到了较纯的两个单体蛋白以及得到已经达到结晶浓度和纯度的CBX7(13-65aa)单体蛋白。在纯化不同的蛋白质片段的过程中,发现即使所构建的蛋白片段来自于人源CBX7蛋白,但每一段蛋白的性质却大不相同。诱导温度时间的不同、Imidazole浓度的选择、Buffer的pH值等对不同片段的纯化有着很大影响。这些条件的探索结果为后续人源CBX7蛋白各片段及全长的纯化提供了重要的参考价值。
与此同时,通过实验发现人源CBX7蛋白质的中间区段存在大量不可溶性的蛋白质,而这段区域的存在对CBX7蛋白的稳定性有一定影响,凡是带有这段区域的蛋白质片段纯化时都易发生降解或部分断裂掉落等现象;并且如果单独将这段区域构建出来纯化无法得到可溶性蛋白。由此,猜想人源CBX7蛋白质可能是以C端和N端亲水区域包裹着中间疏水区域的状态存在于人体中的。目前人源CBX7蛋白的全长还没有纯化到可以结晶的条件,但是对该蛋白纯化的条件探索已基本完成,以上的猜想后续可通过结晶及X-ray晶体衍射来继续验证。
[1]Ma R,Zhang Y,Sun T,et al.Epigenetic Regulation by Polycomb Group Complexes: Focus on Roles of CBX Proteins[J].Journal of Zhejiang Universityence B,2014,15(5):412-428.
[2]Qiang Li,Xiuhong Wang,Zheming Lu,et al.Polycomb CBX7 Directly Controls Trimethylation of Histone E3 at Lysine 9 at p16 Locus[J].Plos ONE,2010,5(10):e13732.
[3]Gil J,Bernard D,Martinez D.Polycomb CBX7 has a Unifying Role in Cellular Lifespan[J].Nat Cell Biol,2004,6(1):67-72.
[4]Steffen PA,Ringrose L.What are Memories Made of? How Polycomb and Trithorax Proteins Mediate Epigenetic Memory[J].Nature reviews Molecular cell biology,2014,15(5):340-356.
[5]Mozzetta C,Pontis J,Fritsch L,et al.The Histone H3 Lysine 9 Methyltransferases G9a and GLP Regulate Polycomb Repressive Complex 2-mediated gene silencing[J].Molecular cell,2014,53(2):277-289.
[6]Wang R,Taylor AB.Polycomb Group Targeting through Different Binding Partners of RING1B C-terminal Domain[J].Structure,2010:966-975.
[7]Simhadri C,Daze KD.Chromodomain Antagonists that Target the Polycomb-group Methyllysine Reader Protein Chromobox Homolog 7(CBX7)[J].Journal of Medicinal Chemistry,2014,57(7):2874-2883.
[8]Rost B,Yachdav G,Liu J.The PredictProtein Server[J].Nucleic Acid Res,2004,31(13):3300.
[9]Rost B,Fariselli P,Casadio R.Topology Prediction for Helical Transmembrane Proteins at 86% Accuracy[J].Protein Sci,1996,5(8):1704.
[10]Ceroni A,Passerini A,Vullo A,et al.DISULFIND: a Disulfide Bonding State and Cysteine Connectivity Prediction Server [J].Nucleic Acids Res.2006,34:177-181.
[11]Mossessova E,Lima CD.Ulp1-SUMO Crystal Structure and Genetic Analysis Reveal Conserved Interactions and a Regulatory Element Essential for Cell Growth in Yeast[J].Mol Cell,2000,5(5):865-876.
[12]Muller J,Verrijzer P.Biochemical Mechanisms of Gene Regulation by Polycomb Group Protein Complexes[J].Curr Opin Genet Dev,2009,19(2):150-158.
[13]Wotton D,Merrill J.C.Pc2 and SUMOylation[J].Biochem Soc Trans,2007,35(Pt6):1401-1404.
