基于竞争性等位基因特异性PCR技术对啤酒花进行纯度检测
2018-02-28蒋培基王德良江伟宋涛李涛蒲彪栾春光
蒋培基,王德良,江伟,宋涛,李涛,蒲彪,栾春光*
1(四川农业大学 食品学院,四川 雅安,625014) 2(中国食品发酵工业研究院,北京,100015)
啤酒花,桑科草属多年生攀援草本植物,雌雄异株,是除大麦之外啤酒酿造的主要原料之一,被称之为“啤酒之魂”、“绿色黄金”[1]。其主要功能成分是酒花树脂、酒花油和多酚物质。这些物质赋予啤酒以特有的苦味与香味[2]。此外,酒花树脂还具有防腐的能力,可以延长啤酒的保质期,而多酚物质中的单宁则具有澄清麦汁的作用[3]。
2014年,全球的酒花产量在96 000 t左右[4],其中国产啤酒花产量达到了12 500 t,占到了13%[5]。我国的啤酒花主要分布在甘肃、新疆两省,甘肃种植面积最大,且甘肃省河西走廊的暖温带干旱气候有助于优良啤酒花的培育。啤酒花的品种以及添加工艺的不同会使啤酒形成不同的苦味与风味特征[6],例如Chinook和Cascade酒花能够赋予啤酒树脂和柑橘类水果风味[7]。此外,啤酒花品种对啤酒的质量也会产生影响,聂聪[8]等人的研究表明,香型酒花的品种是决定啤酒香味质量的关键,在酿造过程中应多用苦香兼优型、香型和免煮沸酒花制品。
尽管人们已经认识到酒花品种对啤酒质量起着重要作用,但一直以来,酒花的品种鉴定是一个难题。这主要是因为啤酒花品种鉴别通常使用传统方法,即从形态上加以区别,包括啤酒花的颜色、形态及主要成分[9]。但由于市场需求和工艺的改进,供应商提供的酒花都是经过粉碎后制成的颗粒酒花制品,无法使用传统的方法加以鉴别。为此分子水平的鉴定方法,例如种子贮藏蛋白或同工酶电泳等经典的物种鉴定方法被开发出来。但以上方法无法满足物种鉴定所需的效率和精确度[10]。而基于第一、二代DNA分子标记技术建立起来的鉴定方法,比如限制性片段长度多态性(restrictionfragment length polymorphism,RFLP)、随机扩增片段长度多态性(amplified fragment length polymorphism,AFLP)[11-13]和微卫星标记(simple sequence repeats,SSRS)[11,14-15]虽然相对于蛋白水平的鉴定具有一定优势,但这些方法都是基于核酸片段的多少和片段大小来完成物种的鉴别,因此对于亲缘关系相近的两个品种存在判断上的误差,且无法完成混杂或人为掺杂的样品进行检测。
作为第3代DNA分子标记技术的SNP(单核苷酸多态性)的出现为酒花品种的定性和定量鉴定提供了更为可靠的手段。有研究表明,SNP将会是一种高效、准确的品种鉴定工具。PITRA[16]对178个酒花品种进行基因测序,分析后共获得了17 128个SNP位点。HENNING[17]等对121个酒花品种进行测序后得到了1 006个SNP位点,最终确定了7个可以区分121个酒花品种的SNP位点。YAMAUCHI[18]通过对酒花品种转录组进行分析,最终得到能分开16个酒花品种的SNP组合。以上研究虽然在寻找可用于酒花品种鉴定的SNP组合的工作上取得了一些进步,但并没有建立一套系统的、可靠的酒花品种鉴定方法。而且,以上研究中存在SNP真实性验证的可靠性低、样品代表性差以及无法对混合样品定性定量测试等缺点。针对以上问题,本研究提出了一种利用最新的SNP分子标记技术与KASP基因分型检测技术相结合[19],对混合的商业啤酒花颗粒进行纯度检测的技术。该技术可以满足啤酒企业对原料的质量监测的要求,同时可为啤酒企业对原料质量的把控提供有效的方法。
1 材料与方法
1.1 材料与试剂
马可波罗、Saaz、Tradition、Magnum、Aurora、Galenq、Centinnia啤酒花样品由中国食品发酵工业研究院收集。预混样品是由Tradition啤酒花与Magnum啤酒花按照不同的质量百分比进行混合,混合比例为0%、2%、5%、10%、20%、40%、50%、100%,并标记为T1~T8。某啤酒厂送检的2份不同批次的Saaz商业啤酒花样品,标记为样品A与样品B。
Taq酶、dNTP、10×loading buffer购自大连宝生物技术有限公司;PCR引物合成由上海生工生物工程股份有限公司完成;KASP引物,上海艾吉析科技有限公司;Qiagen DNeasy Plant Mini Kit(50)植物基因组高效提取试剂盒购自德国凯杰生物公司。
1.2 仪器与设备
Veriti 96 Well Thermal Cycler、Qubit 3.0,北京Thermo Fisher Scientific有限公司;ABI7500实时荧光定量PCR仪,美国ABI公司。
1.3 方法
1.3.1 啤酒花基因组的提取
称取啤酒花样品20 mg,加液氮研磨。研磨后的样品转移至2 mL effendorf管中,按QIAGEN DNeasy Plant Mini Kit(50)DNA提取试剂盒步骤提取啤酒花基因组DNA。利用0.8%琼脂糖TAE凝胶电泳和Nanodrop核酸检测仪对提取到的酒花样品基因组进行检测。
1.3.2 SNP位点的筛选
从DDBJ数据库(http://www.ddbj.nig.ac.jp/)上查找并下载啤酒花基因组信息(登录号:PRJDB3233)。同时,结合本研究测序的7个酒花样品基因组信息(本实验室前期研究结果,未发表)完成SNP位点的筛选。然后,使用premier 5.0软件在SNP位点前后200 bp处设计引物,用来扩增对应带有SNP位点的DNA片段,引物由北京生工生物公司合成。PCR反应体系为25 μL:包括10×loading buffer 2.5 μL;dNTP 2 μL;上游引物 1 μL;下游引物 1 μL;基因组DNA 1 μL;Taq酶 0.2 μL;ddH2O 17.3 μL。 PCR反应条件:95 ℃ 5 min ;95 ℃变性30 s, 52~60 ℃退火30 s, 72 ℃延伸30 s, 35个循环;72 ℃,7 min。扩增产物使用0.8%琼脂糖TAE凝胶电泳检测。PCR扩增产物的测序由北京生工生物完成,测序结果使用MegAlign软件进行比对。筛选并统计多态性良好的SNP位点信息,以及7个啤酒花品种在位点上的基因型,用来建立区分图。
1.3.3 预混样品的检测
根据筛选出来的SNP位点信息合成KASP引物。同时,提取T1~T8预混样品的基因组DNA作为反应模板,使用筛选得到的8个SNP位点的KASP引物在ABI7500实时定量PCR仪器上进行检测。反应体系为:DNA 1 μL ,2×master mix 5 μL, 双蒸水 4 μL, primer mix 0.14 μL。KASP反应条件按照使用说明进行设置,反应条件的设置以及结果的读取均在7500 Software v2.3.软件上进行。
1.3.4 实际样品的真实性检测
分别称取5 g的样品A与样品B,加液氮在研钵中充分研磨,分别将100 mg样品转移至2mL EP管中。基因组的提取参照1.3.1提取方法进行并检测。此过程重复3次。样品分别标记为A1、A2、A3与B1、B2、B3。根据路线区分图选择合适的SNP位点。利用相应的KASP引物在ABI7500Real-time PCR仪器上进行检测。检测的条件以及结果的读取均参照1.3.3进行。
1.3.5 实际样品的纯度检测
本研究采用固定初始质量比的方法对预混样进行定量分析。根据真实性检测结果,选取具有多态性检测结果的SNP位点,以该位点2种不同基因的啤酒花标品DNA按照100%、95%、90%、80%、60%、40%、0%比例混合作为固定比例标准曲线。以提取的实际样品基因组DNA为模板,用具有多态性的SNP位点对应的KASP引物在ABI7500 Real-time PCR仪器上进行检测,检测的条件以及结果的读取均参照1.3.3进行,结果重复读取3次。统计结果中2种不同基因型的荧光信号值,通过SPSS软件建立多元回归方程,计算样品纯度。
2 结果与分析
2.1 啤酒花DNA的提取
研究中提取的啤酒花基因组经0.8%琼脂糖TAE凝胶电泳检测后,条带完整,没有降解。且条带大小处于所需目的片段范围之内。经Qubit3.0定量检测后,DNA质量浓度均大于120 ng/μL,完全符合实验所需的DNA浓度要求。
2.2 SNP位点的筛选
根据啤酒花基因组信息和生物信息学分析结果,共筛选出8个多态性较好的SNP位点。SNP位点所在片段的扩增使用的普通PCR引物见表1。筛选出来的多态性良好的SNP位点信息如表2所示(其中H07与H08位点位于同一扩增片段)。根据统计的不同啤酒花品种在SNP位点上的基因型,建立了7个啤酒花品种的SNP数据表(表3),同时建立区分路线图(图1)。

表1 本研究使用的普通PCR引物

表2 本研究所用的SNP标记
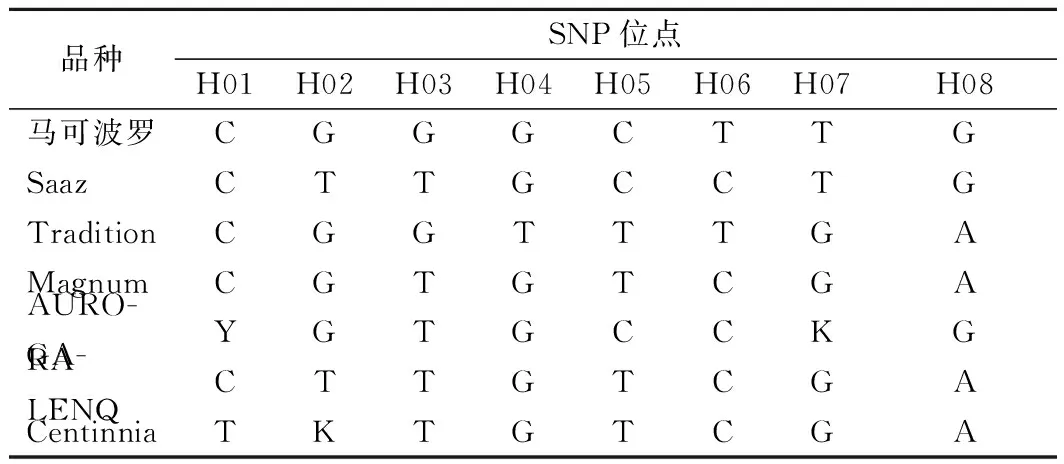
表3 研究验证并使用的啤酒花品种的SNP
2.3 预混样品的检测
通过以上8个SNP位点的检测和筛选(结果见图2),8个位点检测结果都符合预混样在位点的基因型,其中H03、H04、H06位点在T1~T8号预混样品检测过程中表现出良好的分型。从图2可以看出,2%的预混样在H03与H04号位点都被系统判定为杂合,在H06位点被系统判定为纯合。因此,H06位点无法对2%以下的混合样品进行正确的判断。进一步的检测结果表明,5%的预混样品在3个SNP位点都被判定为杂合状态,说明该方法可以满足对混杂比例>5%的啤酒花样品进行检测的要求。且图中各比例混杂样品的检测结果是呈线性比例关系分布。
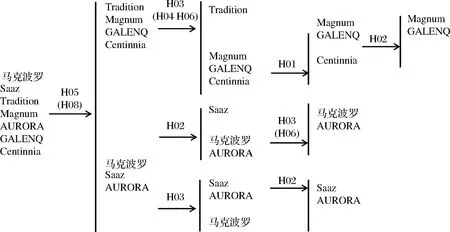
图1 七个品种基于SNP区分路线图Fig.1 Seven species differentiate route map based on SNP

图2 T1~T8样品在8个 SNP位点检测结果Fig.2 Results of T1~T8 samples at eight SNP maker
2.4 实际样品的真实性检测
根据酒花品种区分路线图可知, H05与H02位点可以用来对样品A与样品B进行区分鉴定。样品A的检测结果如图3、图4所示。结果表明,样品A基因型检测结果与Sazz完全符合,表明样品A为纯度很高的Saaz啤酒花,纯度大于95%。样品B的检测结果如图5、图6所示,其中H05的基因型检测结果与Sazz标准品一致,而H02位点出现了杂合状态,表明样品B是含有一定比例的混杂样品。

图3 H05位点样品A基因型检测结果Fig.3 The qualitative detection result of Sample A at H05
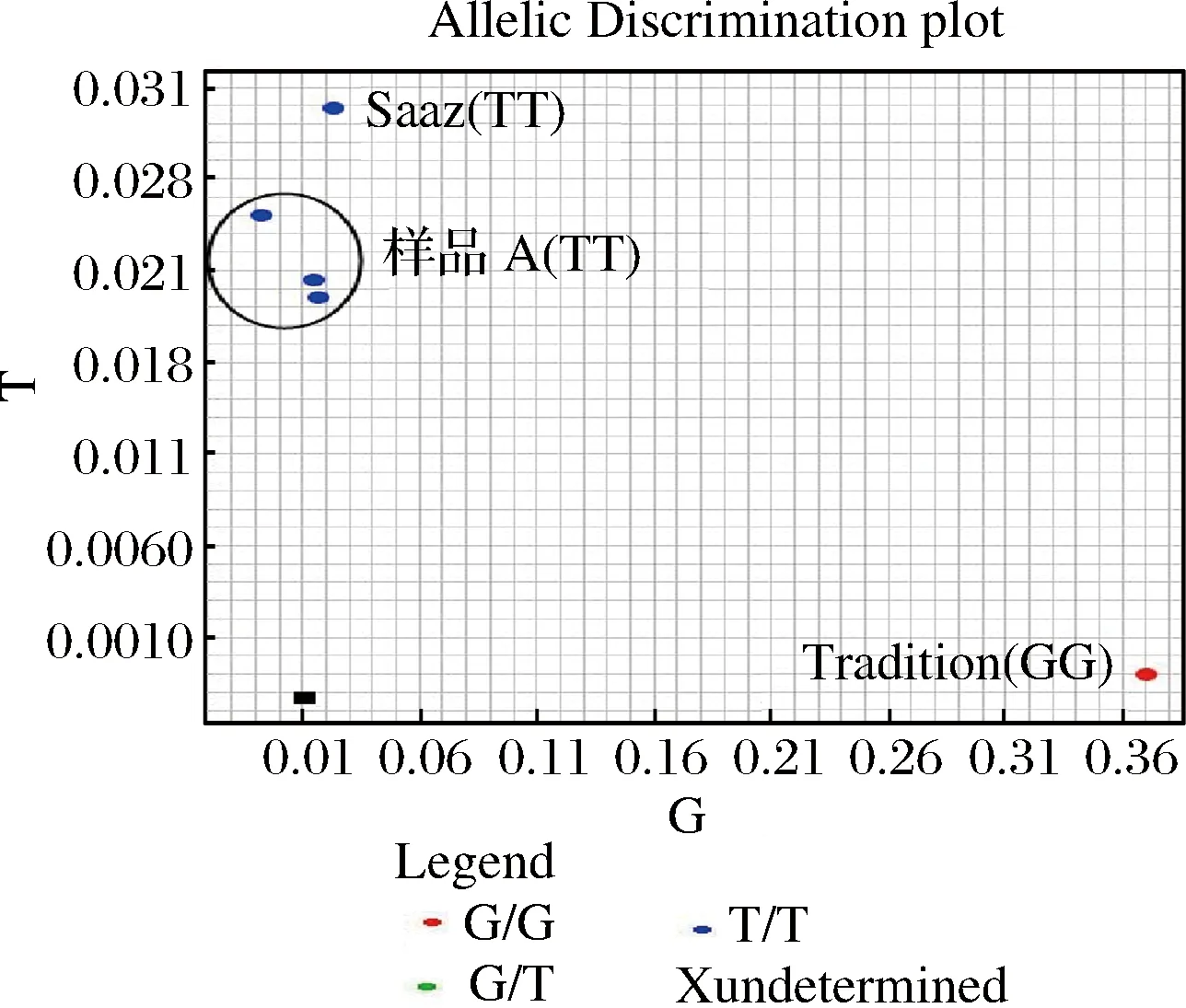
图4 H02位点样品A基因型检测结果Fig.4 The qualitative detection result of Sample A at H02

图5 H05位点样品B基因型检测结果Fig.5 The qualitative detection result of Sample B at H05
图6 H02位点样品B基因型检测结果Fig.6 The qualitative detection result of Sample B at H02
2.5 实际样品的纯度检测
根据真实性检测的结果确认H02位点可以被用于混杂样品B的纯度检测。同时使用Sazz与Tradition啤酒花标准品按照100%、95%、90%、80%、60%、40%、0%的比例混合作标准曲线,结果如图7所示。图7中黑色圈中的样品为样品B的检测结果,可见检测结果重复性很好。标准曲线的荧光信号值见表4。样品B的3次重复检测荧光值见表5。通过表4的荧光值数据建立多元回归方程,得到一次多项式:Saaz酒花所占比例Y=-0.736+0.464×A+0.669×B(A代表T基因型荧光值,B代表G基因型荧光值),R值为0.939,模型拟合度高。说明该模型能很好的对检测样品的纯度进行分析,过原点(阴性对照)做3个重复检测结果平均值做连线并与固定标准曲线相交,计算出交点的两种基因型荧光值,带入Y=-0.736+0.464×A+0.669×B中,得到最终测定结果为42.28%±2.5%。

图7 H02位点样品B纯度检测结果Fig.7 The quantitative detection of Hop using H02 SNP marker
表4 H02位点固定曲线两种基因型荧光信号值
Table 4 Two genotypes fluorescence signal values of fixed proportion curve at H02
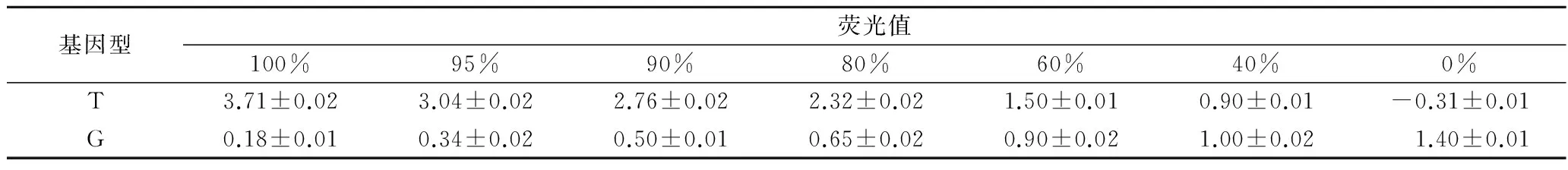
基因型荧光值100%95%90%80%60%40%0%T3.71±0.023.04±0.022.76±0.022.32±0.021.50±0.010.90±0.01-0.31±0.01G0.18±0.010.34±0.020.50±0.010.65±0.020.90±0.021.00±0.021.40±0.01

表5 H02位点样品B检测结果两种基因型荧光信号值
3 结论
本研究开发了一套基于SNP分型技术对商业啤酒花颗粒进行定性定量检测的方法,并筛选出8个可以区分7个啤酒花品种的SNP位点。并利用筛选的8个SNP标记可以准确地对7种商业啤酒花颗粒进行定性检测。同时,确定了以固定质量比例的方式对啤酒花颗粒进行定量检测的方法,并对企业送检样品进行了检测。结果表明,Sazz酒花样品A纯度大于95%,样品B纯度为42.28%±2.5%。本方法的建立为商业啤酒花颗粒品种的定性和定量检测提供了全新的检测方法。目前,关于啤酒花真实性检测的报道还很少,且大多数的研究仅限于理论上利用SNP方法分析酒花品种上,还没有建立一套实际可行的检测方法。本研究开发了的检测方法,既满足了企业实际生产检测的需要,同时也完善了啤酒花品种的SNP数据库,为酿酒原料的品种鉴定提供了一条全新的思路。
[1] 冯正睿. 5PZX-350型啤酒花采摘清洗机的研制[J].农机推广与安全, 2006(9):34.
[2] 林智平,王会军.等梯度高效液相色谱法测定酒花组份[J].啤酒科技, 2001(8):9-10,56.
[3] 高小波, 刘冬梅. 不同食用单宁对啤酒稳定效果的对比分析[J]. 啤酒科技, 2010(7):44-45.
[4] BLACKMAN J D,REES L,GLENDINNING P J.The effects of alternatives to soil residual herbicides on weed control, yield and quality of hops[J]. Journal of Horticultural Science, 2015, 71(4):629-638.
[5] 杨静.啤酒花的生产现状及机械化收获技术研究[J].农业机械, 2016(11):115-117,119.
[6] 王辉.酒花品种、新鲜度、配方和工艺对啤酒风味的影响[D].无锡:江南大学, 2012.
[7] DANIEL S,KATALIN N,MARIANM C,et al.Investigation of the aroma components of hop and beer[J]. Elelmiszervizsgalati Kozlemenyek,2010,56(2):102-107.
[8] 聂聪,付建波.香型酒花对啤酒酿造质量的影响[J].酿酒科技, 2007(4):60-64.
[9] MOMMA T,TAKAHASHI T.Developmental morphology of hop stunt viroid-infected hop plants and analysis of their cone yield[J].Journal of Phytopathology, 2010,110(1):1-14.
[10] ROSS H A.Testing the reliability of genetic methods of species identification via simulation[J]. Systematic Biology,2008, 57(2):216-230.
[11] JAKSE J,KINDLHOFER K, JAVORNIK B.Assessment of genetic variation and differentiation of hop genotypes by microsatellite and AFLP markers[J]. Genome, 2001,44(44):773-782.
[12] PATZAK J.Assessment of somaclonal variability in hop (HumuluslupulusL.)invitromeristem cultures and clones by molecular methods[J]. Euphytica,2003,131(3):343-350.
[13] PATZAK J, NESVADBA V, HENYCHOVA A.et al. Assessment of the genetic diversity of wild hops (HumuluslupulusL.) in Europe using chemical and molecular analyses[J]. Biochem Syst Ecol,2010,38(2):136-145.
[14] JAKSE J, SATOVIC Z, JAVORNIK B. Microsatellite variability among wild and cultivated hops (HumuluslupulusL.)[J]. Genome,2004,47(5):889-899.
[15] KANAI D, KIRITA M, SAKAMOTO K. Method for judging kind of hop using microsatellite DNA:200634(42IP).2006-09-02.
[16] PITRA N, MATTHEWS P D, Coles M C. Next generation sequencing for a plant of great tradition: application of NGS to SNP detection and validation in hops (HumuluslupulusL.) -Hop Special[J]. Brewing Science, 2013, 66(Nov/Dec):185-191.
[17] HENNING J A, COGGINS J, PETERSON M. Simple SNP-based minimal marker genotyping forHumuluslupulusL. identification and variety validation[J]. BMC Research Notes, 2015, 8(1):1-12.
[18] YAMCUCHI H,MUKOUZAKA Y,TANIGUCHI T,et al.Newly developed SNP-based identification method of hop varieties[J]. Journal of the American Society of Brewing Chemists, 2014,72(4):239-245.
[19] 王瑞恒,刘利民,赵金玲,等.基于等位基因特异性PCR原理建立的SNP分型新方法[J].法医学杂志, 2008, 24(3):189-193.
