9种硬骨鱼类转座组注释
2018-02-28高波沈丹陈才王赛赛杨昆仑陈伟王伟张丽宋成义
高波,沈丹,陈才,王赛赛,杨昆仑,陈伟,王伟,张丽,宋成义
扬州大学 动物科学与技术学院,江苏 扬州 225009
转座子是一类具有迁移特性的可移动遗传因子,又称转座因子 (Transposable element,TE),属于散在重复序列的一种,也是在染色体上可移位和自主复制的基本单位,其本质是具有不同结构和换位机制的移动基因序列[1]。转座子几乎存在于所有生物基因组中,依据转座机制的不同,可以分为两大类:RNA转座子 (Ⅰ类转座子,也称为逆转录转座子) 和 DNA 转座子 (Ⅱ类转座子)。RNA转座子通过 RNA聚合酶Ⅱ先转录为mRNA,然后在反转录酶的作用下反转录为cDNA,之后再在整合酶的作用下插入到基因组的新位置。这类转座子完成转座后,在其供体位点保留有原始模板。而DNA转座子则是通过“剪切和粘贴”等机制来转座,不通过RNA媒介,具体过程为:在DNA转座酶的作用下,识别DNA转座子侧翼区反向重复序列,直接将转座子供体位点切割,形成突触复合物 (Synaptic complex formation,SCF),然后再在转座酶介导下整合到基因组的新位点。转座子根据能否独立行使转座功能,分为自主转座子和非自主转座子。自主转座子一般含有完整的转座酶编码框和必要的转座元件,而非自主转座子一般没有转座酶编码框或者转座酶编码框不完整,非自主转座子可以通过自主转座子的转座酶介导进行转座[1-3]。随着测序技术不断发展,越来越多物种基因组测序完成,对这些基因组注释表明转座子是基因组进化的重要因素,影响基因组大小和结构变异等[4]。转座子的注释已成为后基因组时代重要的研究热点,现在也称为转座组 (Mobilome)[5]。研究表明转座子在不同物种中分布存在较大差异,转座子占人基因组的45%[6],鼠基因组的 40%[7],鸟类基因组的 7%–9%[8-9],在部分植物中转座子所占比例更高,如小麦基因组中达到的80%[10],玉米基因组中达到85%[11]。且研究发现转座组大小与基因组的大小具有强正相关性[12-13],相近物种间的基因组差异常常是由转座子的扩增差异导致[14-15]。
鱼类是脊椎动物所有纲中最原始的物种,在实验和进化研究中有重要的参考价值,其中硬骨鱼类遗传多样性最高,在非常相近的种系间,其基因组差异很大[16-17],但目前对硬骨鱼类基因组大小差异原因了解较少。本研究通过生物信息学手段对在Ensemble数据库能够获得基因组序列的9种硬骨鱼类 (斑马鱼、青鳉、三刺鱼、金娃娃、矛尾鱼、红鳍东方鲀、花斑剑尾鱼、罗非鱼和大西洋鳕) 转座组进行注释,以期揭示转座组在硬骨鱼类中的分布特性,及其对基因组变异和进化的影响。
1 材料与方法
1.1 基因组和已知重复序列库数据来源
9种硬骨鱼类包括大西洋鳕鱼 (Cod,Gadusmorhua,gadMor1)、矛尾鱼 (Coelacanth,Latimeria chalumnae,LatCha1)、红鳍东方鲀 (Fugu,Takifugu rubripes,FUGU 4.0)、青鳉 (Medaka,Oryzias latipes,HdrR)、花斑剑尾鱼 (Platyfish,Xiphophorus maculatus,Xipmac4.4.2)、三刺鱼(Stickleback,Gasterosteus aculeatus,BROAD S1)、金娃娃 (Tetraodon,Tetraodon nigroviridis,TETRAODON 8.0)、罗非鱼 (Tilapia,Oreochromis niloticus,Orenil1.0)、斑马鱼 (Zebrafish,Danio rerio,GRCz10) 基因组序列来自Ensembl数据库(http://www.ensembl.org/)。9种硬骨鱼类中已知重复序列库来自 GIRI的 Repbase数据库 (http://girinst.org/server/RepBase/index.php),9种硬骨鱼类进化树由在线程序phyloT (http://phylot.biobyte.de/index.html) 产生。
1.2 基因组未知重复序列库一致序列的获得
利用RepeatModeler (http://repeatmasker.org/RepeatModeler.html) 对 9种硬骨鱼类的未知重复序列进行从头注释,获得新一致序列,RepeatModeler主要由 RepeatScout和 RECON两个核心程序组成,可以对基因组上转座子的进行从头预测,以获得未知的重复序列。
1.3 重复序列参考库构建
将各物种 Repbase数据库的已知重复序列库和利用 RepeatModeler获得的未知重复序列进行合并,去除冗余,构建重复序列参考库。去除冗余根据80-80-80规则进行[18],即如果两个转座子的序列满足以下3个条件,则两个重复序列可以被划为同一个家族:1) 重复序列有 80%的相似度;2) 重复序列长度有 80%的重叠;3) 重复序列之间重叠部分至少80 bp长度。
1.4 基因组转座组注释
以构建的重复序列参考库为基础,利用本地RepeatMasker程序[19](http://repeatmasker.org/) 对各物种的转座组进行注释,其运行参数为:-cutoff 225 -w -s -gccalc -no_is。
2 结果与分析
2.1 9种硬骨鱼转座组大小和构成比较
通过RepeatMasker程序注释表明9种硬骨鱼类转座组大小和构成差异显著 (表 1)。9种硬骨鱼类转座组含量从高到低分别为斑马鱼(54.96%)、矛尾鱼 (41.69%)、青鳉鱼 (26.68%)、罗非鱼 (25.25%)、花斑剑尾鱼 (18.20%)、大西洋鳕鱼 (14.08%)、三刺鱼 (12.82%)、金娃娃 (5.89%)和红鳍东方鲀 (2.94%)。斑马鱼和矛尾鱼基因组中转座组含量较高,约占基因组大小的一半,与哺乳动物基因组转座组含量相近。矛尾鱼中的转座组含量虽然较高,但其中多数仍为未知转座子序列,占到整个基因组的26.23%。金娃娃和红鳍东方鲀,转座组含量较低 (<10%),其他几种鱼转座组含量在10%–30% (表1)。通过将基因组大小和转座子含量进行相关性分析发现,在硬骨鱼类中转座子含量和基因组大小具有较强的线性相关(图1),相关系数r=0.743P=0.022,达显著水平。在进化上,亲缘关系、基因组大小和转座组大小大体呈现一致性,基因组和转座组较小的几个物种其亲缘关系也较近,如金娃娃、三刺鱼和红鳍东方鲀,而与其亲缘关系较远的斑马鱼、矛尾鱼基因组和转座组含量均较大 (图1和图2)。其中Ⅱ类 (DNA转座子) 转座子含量差异是9种硬骨鱼类转座组含量差异的主要决定因素。斑马鱼DNA转座子含量最高 (38.37%),罗非鱼、青鳉和花斑剑尾鱼分别占到11.22%、8.9%和8.38%,其他6种硬骨鱼中则不到5%。Ⅰ类转座子 (RNA转座子) 在9种鱼中分布也有明显差异 (12.73%–0.99%),在矛尾鱼和斑马鱼基因组中比例较大,而在基因组较小的金娃娃、罗非鱼及红鳍东方鲀中占比例较小。在硬骨鱼类基因组中 SINE转座子扩增最弱,其含量在 7种硬骨鱼类基因组 (金娃娃、红鳍东方鲀、大西洋鳕、青鳉、花斑剑尾鱼、三刺鱼、罗非鱼) 中低于1%,仅在斑马鱼和矛尾鱼中较高,分别为3.28%和5.64%。LINE转座子占硬骨鱼类基因组的 0.53%–5.75%,显著低于哺乳动物基因组。LTR转座子除了在斑马鱼和三刺鱼中含量达到 5.58%和 2.51%,在大多硬骨鱼类基因组中的含量也比较低,其基因组含量不超过2%。矛尾鱼的RNA转座子显著多于DNA转座子,而红鳍东方鲀、三刺鱼和金娃娃的RNA转座子略高于DNA转座子;斑马鱼、青鳉鱼、大西洋鳕鱼、罗非鱼和花斑剑尾鱼的 DNA转座子显著高于RNA转座子。
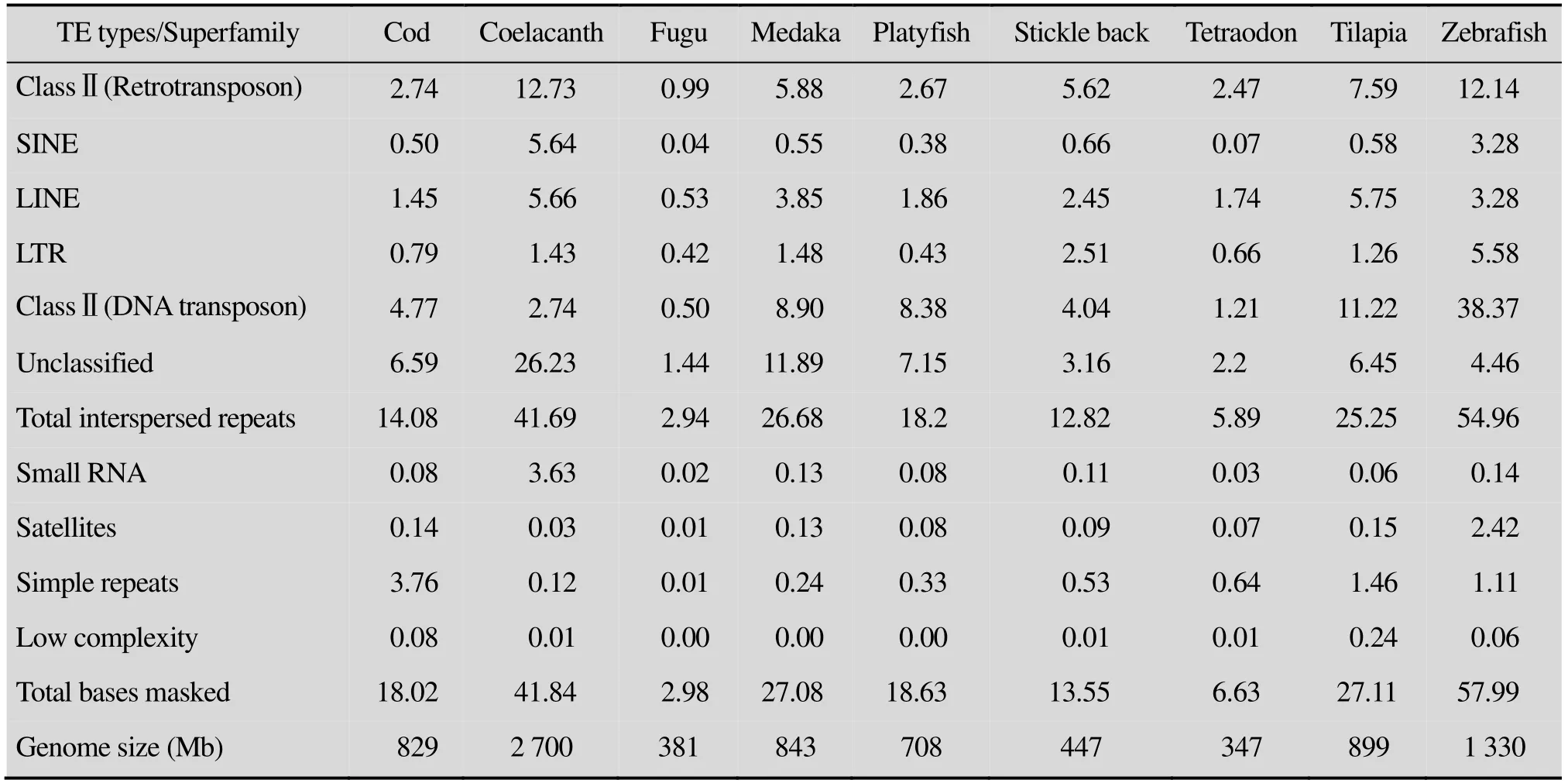
表1 9种硬骨鱼类转座组注释 (占基因组比重, %)Table 1 The annotation of mobilome in nine teleost (genome coverage, %)
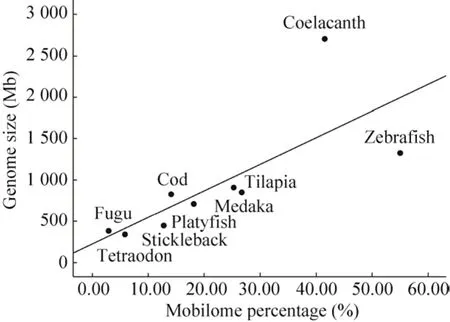
图1 转座组比重与基因组大小关联Fig. 1 The correlation between the percentage of mobilome and genome size.
2.2 DNA转座子在硬骨鱼类中的分布
转座组注释表明 DNA转座子在9种硬骨鱼类呈现很高的多样性,共有22个超家族分布,其中大西洋鳕鱼、斑马鱼、罗非鱼、青鳉、三刺鱼和花斑剑尾鱼基因组中DNA转座子超家族较多,分别含17、16、16、14、14和13个超家族,而红鳍东方鲀、矛尾鱼和金娃娃略少,分别发现5、9和8个超家族 (表2)。hAT和Tc/Mariner超家族是硬骨鱼类主要的 DNA转座子。hAT超家族是DNA转座子中含量最丰富且最具多样性的,其在斑马鱼、罗非鱼、三刺鱼、花斑剑尾鱼、青鳉、矛尾鱼和大西洋鳕鱼基因组中分别占11.14%、3.10%、1.88%、2.61%、3.64%、1.28%和1.33%,而在红鳍东方鲀 (0.23%) 和金娃娃 (0.62%) 中含量较低 (表2)。Tc1/Mariner超家族是硬骨鱼基因组中第二丰富的 DNA转座子,在斑马鱼、罗非鱼、花斑剑尾鱼和青鳉中分别占测序基因组的5.6%、4.48%、4.25%及2.3%,在红鳍东方鲀 (0.20%)、三刺鱼 (0.74%)、金娃娃 (0.43%)、矛尾鱼 (0.28%)和大西洋鳕鱼 (0.18%) 中含量较低。在大西洋鳕鱼、罗非鱼、矛尾鱼基因组中发现 Novosib超家族转座子,且在大西洋鳕鱼 (1.14%) 中占据较高比例,而在其他硬骨鱼类中没有发现。另外,CMC-EnSpm、PIF、MULE-MuDR、Kolobok和PiggyBac等转座子也出现在硬骨鱼DNA转座子中,且有些 (CMC-EnSpm和PIF-Harbinger) 在斑马鱼基因组中含量比较高 (表2)。
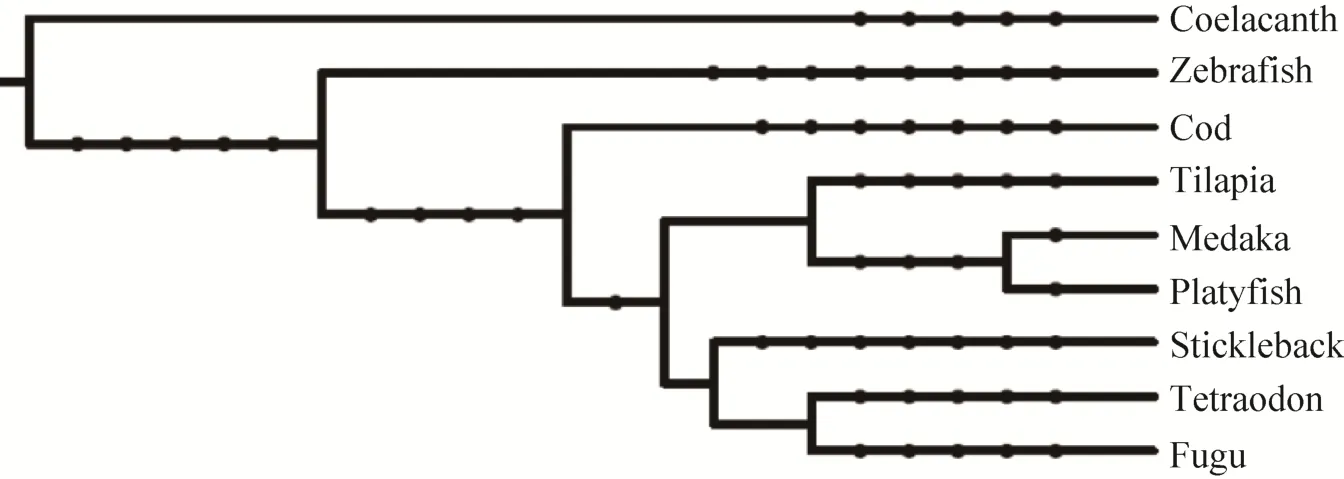
图2 9种硬骨鱼类进化树Fig. 2 The polygenetic tree of 9 teleost.
2.3 RNA转座子在硬骨鱼类中的分布
基因组注释结果表明 RNA转座子 (LINE、LTR和 SINE) 在 9种硬骨鱼类呈现不同分布特点。其中LINE和LTR转座子在9种硬骨鱼类中也呈现出很高的多样性。LINE类转座子中共有14个超家族分布 (表3)。这些LINE超家族在9种硬骨鱼类基因组中呈现不同程度的扩增,其中L1、L2、RTE和Rex-Babar转座子扩增最为明显,尤其是 L2超家族,在罗非鱼 (1.96%)、矛尾鱼(1.73%)、青鳉 (1.52%)、三刺鱼 (1.27%) 和斑马鱼基因组 (1.72%) 组成中都超过 1%;RTE组在青鳉基因组中扩增最为丰富,达到0.86%;而Rex组在三刺鱼、金娃娃和罗非鱼基因组中扩增明显,分别达到 0.66%、0.67%和 0.86。Proto2、CR1、Dong-R4、Jockey、R1、DRE和 TAD1等仅在个别鱼类中被检测出来,且基因组含量也较小(0.01%–0.08%),但CR1在矛尾鱼扩增达到2.16%。R1和DRE只分布在青鳉中,而TAD1只分布在花斑剑尾鱼中。LTR类转座子共有6类超家族分布 (Copia、DIRS、ERV、Gypsy、Ngaro和 Pao),其中扩增最为明显的是Gypsy,在青鳉、罗非鱼、大西洋鳕鱼、三刺鱼和斑马鱼中分别达到0.70%、0.0.47%、0.47%、1.57%和 1.80%。DIRS在矛尾鱼 (1.00%) 和斑马鱼 (1.30%) 中有一定程度的扩增,而在花斑剑尾鱼 (0.01%)、三刺鱼 (0.02%)、金娃娃 (0.05%) 和红鳍东方鲀 (0.00%)中扩增很弱。ERV在大多硬骨鱼类基因组中丰度较低(<0.50%),其中三刺鱼 (0.52%) 和斑马鱼 (0.47%)基因组中 ERV转座子含量高于大西洋鳕鱼(0.10%)、矛尾鱼 (0.07%) 及青鳉 (0.07%),而在花斑剑尾鱼中未检测到ERV分布。Ngaro、Copia和Pao超家族转座子在硬骨鱼类基因组中含量较少,Copia超家族在各鱼类基因组中都不超过0.1%。SINE中tRNA、5S和MIR三个超家族在部分硬骨鱼类中有一定程度扩增。其中 tRNA超家族在矛尾鱼 (4.09%) 和斑马鱼 (2.57%) 基因组中有明显扩增,5S超家族在矛尾鱼 (1.48%) 中有明显扩增,其他超家族成员占基因组的含量均很低 (<0.50%)。

表2 DNA转座子在9种硬骨鱼类基因组中分布(拷贝数/(占基因组比重,%))Table2 The istributiong of DNA transposons in nine teleost(copy number/(genome coverage,%))
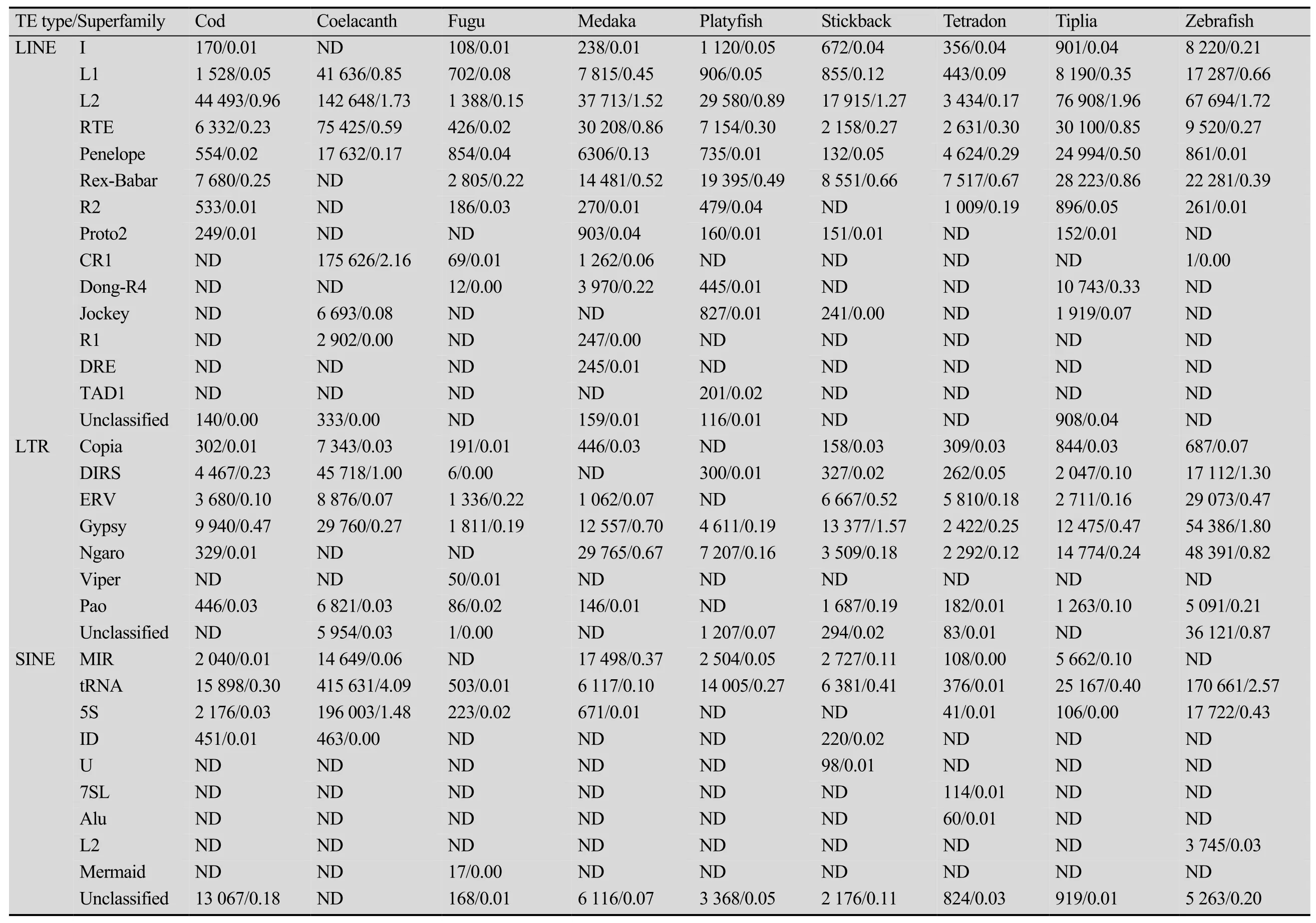
表3 RNA转座子在9种硬骨鱼类基因组中分布(拷贝数/(占基因组比重,%))Table3 The distribution of RNA transposons in nine teleost(copy number/(genome coverage,%))
3 讨论
通过RepeatMasker注释,本研究获得了9种硬骨鱼类的转座组数据。不同于其他脊椎动物,硬骨鱼基因组中转座子的扩张呈现很大的差异性(2.94%–54.96%),远远超过脊椎动物中的哺乳动物 (36%–52%)[6-7,20-22]、蝾螈 (25%–48%)[23]或鸟类等物种间的差异 (7%–9%)[8-9]。本研究中斑马鱼的转座组占基因组的比例 (54.96%) 与先前的报道 (51.83%) 相似[24],与哺乳动物转座子比例相近,高于目前大多数报道的脊椎动物,如鲤鱼(31.3%)[25]、蜥蜴 (34.4%)[26]、青蛙 (34.5%)[27]和鸟类 (7%–9%)[9,28]。本研究所注释红鳍东方鲀转座组 (2.94%) 与先前的报道相近 (2.7%)[29]。大西洋鳕鱼转座组注释结果 (14.08%) 也与前期注释 (12.31%) 相近[30],而本研究其他大部分转座组注释结果均高于前期注释,如矛尾鱼 (41.69% vs 25%)[31]、花斑剑尾鱼(18.5% vs 5%)[32]、罗非鱼(25.25% vs 14%)[33]、青鳉(26.68% vs 16.2%)[34]、金娃娃(5.89% vs <1%)[35]。造成这种差异的主要原因是之前组装的基因组数据与重复序列库数据不完整,特别是早期重复序列库数据缺乏,导致转座组比重被严重低估。本研究中三刺鱼所注释转座组的覆盖率 (12.82%) 远远低于先前的估量(25.2%)[36],由于文献中没有提供注释的方法,所以差异的原因并不清楚。
研究表明遗传关系相近的物种之间基因组大小差异可归因于转座子积累程度不同[14-15],本研究对硬骨鱼的转座子注释进一步证实了这一理论。在9个硬骨鱼中预测出的转座子比重和基因组大小呈正相关,且与进化上的亲缘关系相一致,如最小基因组红鳍东方鲀转座子比例为 5.89%,而最大基因组斑马鱼的转座组则高达54.96%,转座组差异主要是由 DNA转座子的扩增量不同导致。以 DNA转座子为主的转座子的不同扩增量是促成硬骨鱼基因组大小差异的主要分子机制。这与两栖类相似[26-27],但与多数哺乳动物和爬行动物的相反。哺乳动物和爬行动物的基因组扩张主要取决于LTR或非LTR反转录转座子[6-7,20-22]。本文为从转座子角度揭示物种进化提供参考。
相比于哺乳动物和鸟类,硬骨鱼基因组含有多种Ⅰ类和Ⅱ转座子,并且拥有大量进化分支、超家族和家族[12],本研究显示9个硬骨鱼类基因组中转座组分布差异明显。虽然 DNA转座子是引起转座子扩增差异的主要力量,但基因组中转座子的差异不仅仅局限于扩增量的不同。转座子的多样性在不同硬骨鱼类基因组中也存在较大差异,在较大的转座组物种中如斑马鱼、大西洋鳕鱼、青鳉,其转座子多样性也较高,这也是造成硬骨鱼基因组的差异原因之一。在硬骨鱼中,转座子在超家族水平上呈现出较大的差异,和较大的基因组相比,较小的基因组含有较低多样性,这与在蜥蜴中的结果相一致[23]。目前关于脊椎动物基因组内转座子多样性和活性差异原因的了解仍然非常有限,有学者认为这可能是由于抑制转座子活性的宿主防御机制差异导致的[37]。
4 结论
本研究对9种硬骨鱼类转座组进行了全面系统的注释,分析结果显示,硬骨鱼基因组中转座子多样性丰富,转座子在基因组中的分布比例差异很大,尤其是 DNA转座子,转座组差异与硬骨鱼基因组大小有很强的相关,转座组是决定硬骨鱼基因组大小的重要因素。
[1]Finnegan DJ. Eukaryotic transposable elements and genome evolution. Trends Genet, 1989, 5: 103–107.
[2]Kapitonov VV, Jurka J. A universal classification of eukaryotic transposable elements implemented in Repbase. Nat Rev Genet, 2008, 9(5): 411–422.
[3]Pray L. Transposons: the jumping genes. Nature Education, 2008, 1(1): 204.
[4]Jurka J, Kapitonov VV, Kohany O, et al. Repetitive sequences in complex genomes: structure and evolution. Annu Rev Genomics Hum Genet, 2007, 8:241–259.
[5]Babatz TD, Burns KH. Functional impact of the human mobilome. Curr Opin Genet Dev, 2013, 23(3):264–270.
[6]Lander ES, Linton LM, Birren B, et al. Initial sequencing and analysis of the human genome.Nature, 2001, 409(6822): 860–921.
[7]Chinwalla AT, Cook LL, Delehaunty KD, et al.Initial sequencing and comparative analysis of the mouse genome. Nature, 2002, 420(6915): 520–562.
[8]Wicker T, Robertson JS, Schulze SR, et al. The repetitive landscape of the chicken genome. Genome Res, 2004, 15: 126–136.
[9]Warren WC, Clayton DF, Ellegren H, et al. The genome of a songbird. Nature, 2010, 464(7289):757–762.
[10]Charles M, Belcram H, Just J, et al. Dynamics and differential proliferation of transposable elements during the evolution of the B and A genomes of wheat. Genetics, 2008, 180(2): 1071–1086.
[11]Schnable PS, Ware D, Fulton RS, et al. The B73 maize genome: complexity, diversity, and dynamics.Science, 2009, 326(5956): 1112–1115.
[12]Chalopin D, Naville M, Plard F, et al. Comparative analysis of transposable elements highlights mobilome diversity and evolution in vertebrates.Genome Biol Evol, 2015, 7(2): 567–580.
[13]Hawkins JS, Kim H, Nason JD, et al. Differential lineage-specific amplification of transposable elements is responsible for genome size variation inGossypium. Genome Res, 2006, 16(10): 1252–1261.
[14]Lee SI, Kim NS. Transposable elements and genome size variations in plants. Genomics Inform, 2014,12(3): 87–97.
[15]Zuccolo AI, Sebastian A, Talag J, et al. Transposable element distribution, abundance and role in genome size variation in the genusOryza. BMC Evol Biol,2007, 7: 152.
[16]Volff JN. Genome evolution and biodiversity in teleost fish. Heredity, 2005, 94(3): 280–294.
[17]Venkatesh B. Evolution and diversity of fish genomes. Curr Opin Genet Dev, 2003, 13(6):588–592.
[18]Wicker T, Sabot F, Hua-Van A, et al. A unified classification system for eukaryotic transposable elements. Nat Rev Genet, 2007, 8(12): 973–982.
[19]Tarailo-Graovac M, Chen N. UNIT 4.10 Using RepeatMasker to identify repetitive elements in genomic sequences//Current Protocols Bioinformatics. New York: John Wiley and Sons,2009.
[20]Li RQ, Fan W, Tian G, et al. The sequence andde novoassembly of the giant panda genome. Nature,2010, 463(7279): 311–317.
[21]Kirkness EF, Bafna V, Halpern AL, et al. The dog genome: survey sequencing and comparative analysis. Science, 2003, 301(5641): 1898–1903.
[22]Murchison EP, Adams DJ. Sequencing skippy: the genome sequence of an Australian kangaroo,Macropuseugenii. Genome Biol, 2011, 12: 123.
[23]Sun C, Shepard DB, Chong RA, et al. LTR retrotransposons contribute to genomic gigantism in plethodontid salamanders. Genome Biol Evol, 2012,4(2): 168–183.
[24]Howe K, Clark MD, Torroja CF, et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature, 2013, 496(7446): 498–503.
[25]Xu P, Zhang XF, Wang XM, et al. Genome sequence and genetic diversity of the common carp,Cyprinus carpio. Nat Genet, 2014, 46(11): 1212–1219.
[26]Alföldi J, Di Palma F, Grabherr M, et al. The genome of the green anole lizard and a comparative analysis with birds and mammals. Nature, 2011, 477(7366):587–591.
[27]Hellsten U, Harland RM, Gilchrist MJ, et al. The genome of the Western clawed frogXenopus tropicalis. Science, 2010, 328(5978): 633–636.
[28]Hillier LW, Miller W, Birney E, et al. Sequence and comparative analysis of the chicken genome provide unique perspectives on vertebrate evolution. Nature,2004, 432(7018): 695–716.
[29]Aparicio S, Chapman J, Stupka E, et al.Whole-genome shotgun assembly and analysis of the genome ofFugurubripes. Science, 2002, 297(5585):1301–1310.
[30]Star B, Nederbragt AJ, Jentoft S, et al. The genome sequence of Atlantic cod reveals a unique immune system. Nature, 2013, 477(7363): 207–210.
[31]Amemiya CT, Alföldi J, Lee AP, et al. The African coelacanth genome provides insights into tetrapod evolution. Nature, 2013, 496(7445): 311–316.
[32]Schartl M, Walter RB, Shen YJ, et al. The genome of the platyfish,Xiphophorusmaculatus, provides insights into evolutionary adaptation and several complex traits. Nat Genet, 2013, 45(5): 567–572.
[33]Shirak A, Grabherr M, Di Palma F, et al.Identification of repetitive elements in the genome ofOreochromisniloticus: tilapia repeat masker. Mar Biotechnol (NY), 2010, 12(2): 121–125.
[34]Kasahara M, Naruse K, Sasaki S, et al. The medaka draft genome and insights into vertebrate genome evolution. Nature, 2007, 447(7145): 714–719.
[35]Crollius HR, Jaillon O, Dasilva C, et al.Characterization and repeat analysis of the compact genome of the freshwater pufferfishTetraodon nigroviridis. Genome Res, 2000, 10(7): 939–949.
[36]Peichel CL, Nereng KS, Ohgi KA, et al. The genetic architecture of divergence between threespine stickleback species. Nature, 2001, 414(6866): 901–905.
[37]Abrusán G, Krambeck HJ. Competition may determine the diversity of transposable elements.Theor Popul Biol, 2006, 70(3): 364–375.
