口蹄疫病毒基因组3' 末端序列扩增及分析
2018-02-25李金娜苗书魁马文戈魏玉荣陆桂丽米晓云沙依兰卡依扎
李金娜,苗书魁,马文戈,魏玉荣,汪 萍,夏 俊,陆桂丽,米晓云,沙依兰·卡依扎,王 延,魏 婕,黄 炯
(1.新疆农业大学动物医学学院,乌鲁木齐 830052;2.新疆畜牧科学院兽医研究所(新疆畜牧科学院动物临床医学研究中心),乌鲁木齐 830011)
0 引 言
【研究意义】口蹄疫(Foot-and-mouth disease,FMD)是由FMDV引起的偶蹄动物的一种急性、热性、高度接触感染的传染病[1]。口蹄疫病毒型多易变,宿主谱广,传播途径多,自然康复动物长期排毒,同群动物发病即扑杀处置,导致经济损失巨大、政治影响严重。因此,OIE将其规定为法定报告的传染病,我国将其列为一类动物传染病[2]。FMDV属于小RNA病毒科口蹄疫病毒属,基因组为单股正链RNA,RNA分子位于衣壳内,约有8 500个核苷酸(nucleotide,nt)。基因组5'端共价连接着一个病毒编码的小蛋白质(VPg),紧接着依次由5'非翻译区(Untranslated region,UTR)、开放阅读框(Open reading fragment,ORF)、3'UTR和Poly(A)组成。试验用普通PCR法和3'RACE法扩增3'端序列,对FMDV反向遗传学研究有实际意义。【前人研究进展】FMDV全基因组序列的扩增已有很多报道,其中选用最多的扩增方法为普通PCR方法[3-10],这些学者先提取FMDV的RNA,反转录后获得cDNA,再以cDNA为模板,利用设计的多对引物分别进行普通PCR,将扩增产物测序,最后拼接得到病毒全基因组序列。Arzt J等[11]将FMDV全基因组设计为5'UTR、ORF和3'UTR三个相互重叠的片段,提取病毒RNA后,采用RT-PCR获得了FMDV全基因组序列。【本研究切入点】有关FMDV同一毒株不同亚克隆之间Poly(A)长度的研究尚未见报道。研究口蹄疫病毒基因组3'末端序列扩增。【拟解决的关键问题】试验采用普通PCR法和3'RACE法对FMDV基因组3'末端进行扩增,对3'UTR序列进行分析,比较Poly(A)长度的不同,为FMDV 3'端序列的扩增、FMDV结构与功能的研究、基因组结构、感染性克隆构建、致病机理和毒力变异机制等提供参考。
1 材料与方法
1.1 材 料
1.1.1 病毒基因组RNA和细胞
口蹄疫病毒O/Akesu/58 CE39A是1958年采集自新疆阿克苏地区的牦牛口蹄疫O型病毒,经过鸡胚适应传代39代次驯化致弱,再筛选获得的克隆毒株A。基因组RNA自1990年代由新疆畜牧科学院兽医研究所保存于液氮中。BHK-21细胞由新疆畜牧科学院兽医研究所保存。感受态大肠埃希菌DH5α购自天根生化科技(北京)有限公司。
1.1.2 主要试剂
反转录试剂Transcriptor First Strand cDNA Synthesis Kit (Cat.Not.04896866001)购自Roche公司,PrimeSTAR DNA聚合酶购自TaKaRa公司。
1.1.3 引物设计与合成
根据GenBank中O型口蹄疫参考序列(GenBank登录号:EF552696),在病毒全基因组3'端设计1对引物及3'RACE引物,由新疆昆泰锐生物技术有限公司合成。表1
表1 3'端序列扩增引物
Table 1 Primers for amplification of the 3'-end sequences
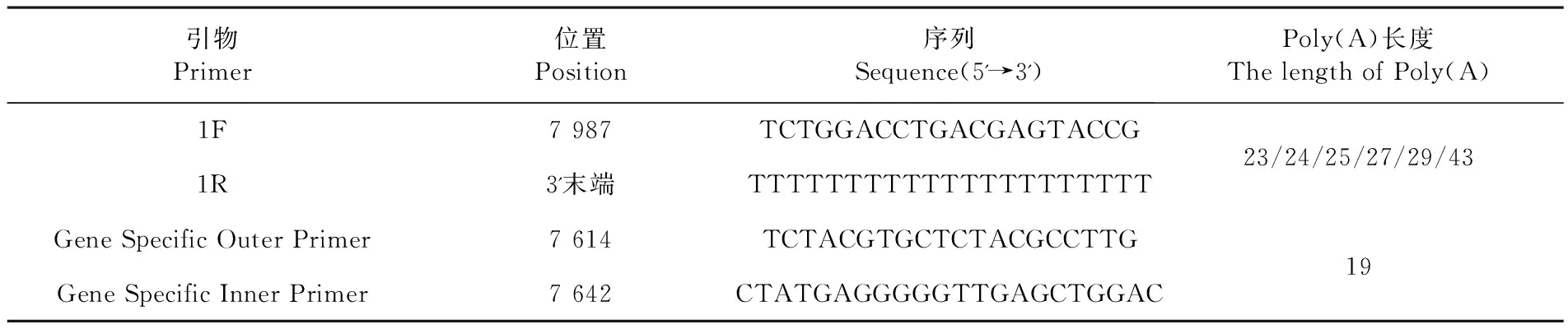
引物Primer位置Position序列Sequence(5'→3')Poly(A)长度The length of Poly(A)1F7 987TCTGGACCTGACGAGTACCG1R3'末端TTTTTTTTTTTTTTTTTTTTT23/24/25/27/29/43Gene Specific Outer Primer7 614TCTACGTGCTCTACGCCTTGGene Specific Inner Primer7 642CTATGAGGGGGTTGAGCTGGAC19
1.2 方 法
1.2.1 RNA纯度测定
取液氮中保存的FMDV O/Akesu/58 CE39A RNA 10 μL,加990 μL ddH2O,混匀,于紫外分光光度计测定其核酸纯度。
1.2.2 反转录
将保存的口蹄疫病毒O/Akesu/58 CE39A RNA反转录为cDNA,操作过程按照Transcriptor First Strand cDNA Synthesis Kit产品说明书操作,反转录引物选用Oligo (dT)18。
1.2.3 3'末端序列扩增
以反转录产物cDNA为模板,以普通PCR(引物1F/1R)和3'RACE法分别对O/Akesu/58 CE39A毒株全基因组3'端序列进行扩增。所用引物见表1。
普通PCR反应条件:94℃预变性2 min;94℃ 30 s,55℃ 20 s,72℃ 20 s进行25个循环;最后72℃延伸10 min。
3'RACE法Outer PCR反应条件:94℃预变性3 min;再以94℃ 30 s,55℃ 30 s,72℃ 40 s进行25个循环;最后72℃延伸10 min。
3'RACE法Inner PCR反应条件:94℃预变性3 min;再以94℃ 30 s,55℃ 30 s,72℃ 40 s进行25个循环;最后72℃延伸10 min。
各取5 μL PCR产物,以1%琼脂糖凝胶进行电泳。
1.2.4 目的片段的克隆与鉴定
将普通PCR产物克隆至pMD19-T载体(标记为:pMD19-T-1),将3'RACE法Inner PCR产物克隆至pMD19-T载体(标记为:pMD19-T-3),分别转化DH5α,挑单菌落培养,提取质粒。将质粒pMD19-T-1和pMD19-T-3均用Xba I和Sal I双酶切鉴定,各选7个阳性质粒送新疆昆泰锐生物技术有限公司测序。
1.2.5 3'UTR序列分析
用DNAStar、DNAMAN等软件对病毒全基因组3'UTR序列进行分析,并与参考毒株的序列进行比对。
2 结果与分析
2.1 RNA纯度测定
以分光光度计测得O/Akesu/58 CE39A株基因组RNA A260/A280为1.871,此RNA中可能有部分蛋白质或其它有机物的污染,或是完整RNA降解导致纯度降低,但在允许范围1.8~2.0之内。
2.2 3'端序列扩增
普通PCR法扩增出了目的条带,3'RACE法也扩增出了目的条带。图1

注:M:GeneRuler 1Kb Plus DNA Ladder;1,普通PCR法扩增的目的条带(237 bp);3,3'RACE法扩增的目的条带(597 bp)
Note: M:GeneRuler 1Kb Plus DNA Ladder;1,The target bands amplified by general PCR(237 bp);3,The target bands amplified by 3'RACE(597 bp)
图1 3' 端序列扩增
Fig.1 Amplification of 3'-end sequences
2.3 重组质粒的酶切鉴定
将质粒pMD19-T-1和pMD19-T-3均用Xba I和Sal I双酶切后,以1%琼脂糖凝胶电泳分析,均得到预期大小的两条带。质粒pMD19-T-1的两条带为:2 680 bp(pMD19-T载体)+249 bp(片段)(图2,1),质粒pMD19-T-3的两条带为:2 680 bp(pMD19-T载体)+641 bp(片段)(图2,3),说明得到的2个质粒均为阳性质粒。图2
2.4 3'UTR序列
用3'RACE法扩增的3'端序列大小为565 bp,包括部分非结构蛋白(3D)基因以及紧接着3D基因下游的3'UTR和Poly(A)尾,3'UTR长度为98 nt,Poly(A)长度为19 nt。3'UTR序列从终止密码子UAA到3'端Poly(A)有98 nt(8 101~8 198 nt)。用DNAMAN软件分析3'UTR序列,显示其二级结构含有2个茎环结构,分别位于8 101~8 140和8 146~8 190 nt。
用DNAStar、DNAMAN等软件对O/Akesu/58 CE39A株3'UTR序列与参考毒株的3'UTR序列进行比对。研究表明,O/Akesu/58 CE39A株与参考毒株O/MAY/3/2014(GenBank登录号:KY322672)和O/IND26(54)/2014(GenBank登录号:KJ825807)的核苷酸序列同源性最高,均为89.8%。
注:M: GeneRuler 1Kb Plus DNA Ladder;1:pMD19-T-1酶切产物(2 680 bp+249 bp);3:pMD19-T-3酶切产物(2 680 bp+641 bp)
Note: M: GeneRuler 1Kb Plus DNA Ladder; 1: The products from pMD19-T-1 (2 680 bp+249 bp); 3: The products from pMD19-T-3 (2 680 bp+641 bp)
图2 重组质粒酶切鉴定
Fig.2 Restriction endonuclease digestion identification of recombinant plasmid
2.5 Poly(A)长度
以普通PCR扩增,将得到的多个亚克隆测序,其Poly(A)数量为23、24、25、27、29、43;以3'RACE法扩增,测得亚克隆的Poly(A)数量为19,说明不同扩增方法获得的亚克隆之间Poly(A)长度存在差异。普通PCR和3'RACE法扩增后测得Poly(A)数量差异的可能原因是,3'RACE法扩增时,试剂盒引物中Poly(T)数量可能为19,扩增后Poly(A)数量为19。而普通PCR的6个亚克隆测得Poly(A)数量不同,可能原因为,第一,RNA质量的影响。FMDV RNA为单链结构,容易断裂,所提取的RNA中有断裂的RNA,反转录后cDNA中Poly(A) 长度不同;第二,FMDV RNA长约8 500 nt,基因组长,末端结构复杂,反转录时,反转录引物与模板配对的位置不同,则反转录产物中Poly(A)长度不同。第三,上述2个原因导致反转录产物为全长cDNA和非全长cDNA的混合物,即模板为含不同长度Poly(A)的大量cDNA混合物。进行PCR时,即使模板中Poly(A) 长度相同,但由于下游引物与模板配对是随机的,下游引物与模板结合的位置不同,则扩增的片段中Poly(A)的长度有所不同。况且模板为含不同长度Poly(A)的cDNA混合物,扩增后片段中Poly(A)的长度肯定不同。图3
图3 不同长度的Poly(A)
Fig.3 The different lengths of the Poly(A)
3 讨 论
FMDV 3'UTR和Poly(A)在病毒复制中起着至关重要的作用,其特性和作用并不是很明确。Poly(A)与3'UTR共价相连,可能是病毒有效复制或翻译所必需的结构[13]。Poly (A)与病毒毒力有关,Poly (A)长度与病毒毒力的具体关系尚未定论。为了确定3'UTR和Poly(A) 的作用,需要先对这段序列进行扩增。因此,对3'端序列的扩增显得尤为重要。但FMDV基因组3'端结构复杂,难于扩增。计算机分析发现FMDV China/99的3'端为一个类似三叶草的二级结构[14],研究显示O/Akesu/58 CE39A株的3'UTR含有2个茎环结构,与有的学者[15-16]报道的一致。3'UTR的一部分位于非结构蛋白3D编码区之后,长约100 nt,能形成特殊的高级结构[17],小RNA病毒基因组包括一个Poly (A)区段,该区段可以折叠成特殊结构[18]。上述特殊结构均增加了3'端序列扩增的难度,尤其是Poly(A)的扩增。试验采用普通PCR法和3'RACE法均扩增出了FMDV 3'端序列,为3'UTR和Poly(A)作用的研究奠定了基础。
研究发现FMDV Poly(A) 的长度不同。Chatterjee N K等[19]认为FMDV基因组3'末端Poly(A)尾长达60~80 nt。刘光清等[12]采用3'RACE法克隆得到的Poly(A)长度为56 nt。试验测得Poly(A)数量为19、23、24、25、27、29、43,说明同一毒株的亚克隆,其Poly(A) 长度与基因扩增时所用方法有关。通常Poly(A)要么扩增成功后其长度不同,要么扩增失败,失败的原因可能是3'末端结构复杂。将GenBank中部分FMDV的序列进行比对,发现有的序列具有Poly(A)[如(登录号:AB079061)、(登录号:AF308157)、(登录号:AJ633821)、(登录号:EF552696) 、(登录号:AY593811)和(登录号:HM008917)等],而有的序列没有Poly(A)[如(登录号:AJ539138)、(登录号:AJ539140)、(登录号:AY312589)、(登录号:AH012984) 、(登录号: KJ825804)和(登录号: KJ825802)]。没有Poly(A)的原因可能是研究者与笔者扩增的结果类似,不同亚克隆获得的Poly(A)长度有差异,由于没有确切的长度故而登录时省略;也有可能是试验中未扩增出来,若如此,则要采用不同方法进行多次扩增,以得到Poly(A)。
目前,对Poly(A)尾功能的报道较少,且说法不一,其具体功能仍处于探索阶段。Poly(A)尾在基因组复制方面起着重要作用。Poly (A)的存在能使FMDV转录及时有效地终止[20]。Poly(A)不但具有稳定病毒基因组结构的功能,而且对维持其感染性也有一定的作用[21]。Poly(A)的长度与感染性的关系一直存在争议。认为Poly(A)的长度越长越好,也有人认为O型FMDV Poly (A)的长度与病毒感染性之间的关系很小或没有相关性[21-22];与此相反,C型Poly (A)序列10 nt与40 nt之间的感染性差异在5~16倍,与PV、EMCV、SV等病毒相似[11,21]。苗书魁等[23]认为Poly(A)长度与感染性的关系不是确定的,不是越长越好,或越短越好,而是依毒株而定。因此,通过对FMDV 3'端序列的扩增,为3'UTR和Poly (A)长度的研究,为病毒复制、FMDV结构与功能的研究、Poly(A)长度与感染性关系、感染性克隆构建、致病机理和毒力变异机制等提供参考。
4 结 论
4.1 普通PCR法和3'RACE法均可扩增出FMDV基因组3'末端序列。用3'RACE法扩增的3'端序列大小为565 bp,包括部分非结构蛋白(3D)基因以及紧接着3D基因下游的3'UTR和Poly( A)尾,3'UTR长度为98 nt(8 101~8 198 nt),Poly(A)长度为19 nt。用DNAMAN软件分析3'UTR序列,显示其二级结构含有2个茎环结构,分别位于8 101~8 140和8 146~8 190 nt。
4.2 测得FMDV Poly(A)数量为19、23、24、25、27、29、43,说明同一毒株的亚克隆,其Poly(A) 长度与基因扩增时所用方法有关。据此推测,不同毒株的Poly(A)长度不同,可能是毒株本身属性,也有可能是由扩增方法不同造成。
4.3 Poly(A)要么扩增成功后其长度不同,要么扩增失败,失败的原因可能是3'末端结构复杂,难于扩增。
参考文献(References)
[1] Strohmaier, K. , Franze, R. , & Adam, K. H. . (1982). Location and characterization of the antigenic portion of the fmdv immunizing protein.JournalofGeneralVirology, 59(2): 295-306.
[2] 杨洋,秦晓东,宋一鸣,等. 口蹄疫病毒real-time RT-RPA检测方法的建立与应用. 黑龙江畜牧兽医,2017,(5):172-176.
YANG Yang, QIN Xiao-dong, SONG Yi-ming, et al. (2017). Establishment and application of real-time RT-RPA for rapid detection foot-and-mouth disease virus.HeilongjiangAnimalScineceandVeterinaryMedicine, (5):172-176.(in Chinese)
[3] Munawar Sultana, Mohammad Anwar Siddique, Samina Momtaz, et al. (2014). Complete genome sequence of Foot-and-Mouth Disease Virus serotype O ssolated from Bangladesh.GenomeAnnouncements, 2(1): e01253-13. doi: 10.1128/genomeA.01253-13.
[4] Ryoo, S. , Kim, T. , Nah, J. J. , Sagong, M. G. , Lee, S. , & Lee, K. N. , et al. (2017). Complete genome sequence of a foot-and-mouth disease virus of serotype o isolated from gimje, republic of korea, in 2016.GenomeAnnouncements, 5(10): e01694-16. doi: 10.1128/genomeA.01694-16.
[5] Kim T, Ryoo S, Nah JJ, et al. (2017). Complete genome sequence of a Foot-and-Mouth Disease Virus of serotype O, isolated from Gochang, Republic of Korea, in 2016.GenomeAnnouncements, 5(10): e01671-16. doi: 10.1128/genomeA.01671-16.
[6] Nishi, T. , Ulziibat, G. , Khanui, B. , Myagmarsuren, O. , Morioka, K. , & Yamakawa, M. , et al. (2017). Genome sequence of foot-and-mouth disease virus of serotype o lineage ind-2001d isolated from cattle in in 2015.GenomeAnnouncements, 5(45): e01244-17.
[7] 李爽,张润祥,宋鸽,等.牛Asia 1 型口蹄疫病毒感染性克隆的构建[J].生物工程学报,2009,25(11):1 621-1 626.
LI Shuang, ZHANG Run-xiang, SONG Ge, et al. (2009). Rescue of bovine Asia1 serotype foot-and-mouth disease virus from a full-length cDNA clone [J].ChinJBiotech, 25(11): 1,621-1,626.(in Chinese)
[8] 仝燕许,陈豪泰,潘丽,等. 口蹄疫病毒Asial/JSWX株基因组全长感染性克隆的体外拯救与序列分析.中国兽医科学,2014,44(8):771-780.
TONG Yan-xu, CHEN Hao-tai, PAN Li, et al. (2014). In vitro rescue and sequence analysis of an infectious full-length cDNA clone of foot-and-mouth disease virus Asia 1/JSWX strain.ChineseveterinaryScience, 44(08): 771-780. (in Chinese)
[9] 苗书魁,魏玉荣,魏婕,等.口蹄疫病毒O型OHM/02株全长cDNA感染性克隆的构建[J].动物医学进展,2018,39(1) :1-6.
MIAO Shu-kui, WEI Yu-rong, WEI Jie, et al. (2018). Construction of infectious clone of OHM/02 of foot-and-mouth disease virus serotype O [J].ProgressinVeterinaryMedicine, 39(1):1-6.(in Chinese)
[10] Rajasekhar, R. , Hosamani, M. , Basagoudanavar, S. H. , Sreenivasa, B. P. , Tamil Selvan, R. P. , & Saravanan, P. , et al. (2013). Rescue of infective virus from a genome-length cdna clone of the fmdv serotype o (ind-r2/75) vaccine strain and its characterization.ResearchinVeterinaryScience, 95(1): 291-297.
[11] Arzt, J. , Brito, B. , Pauszek, S. J. , Hartwig, E. J. , Smoliga, G. R. , & Vu, L. T. , et al. (2017). Genome sequence of foot-and-mouth disease virus serotype o lineage ind-2001d collected in in 2015.GenomeAnnouncements, 5(18): e00223-17.
[12] 刘光清,刘在新,谢庆阁.用3'RACE方法扩增并克隆口蹄疫病毒基因组3'末端序列[J].中国兽医学报,2003,23(6):524-526.
LIU Guang-qing, LIU Zai-xin, XIE Qing-ge. (2003). Molecular cloning and analysis of the 3'end sequence of FMDV OH99 strain by 3'RACE [J].ChinJVetSci, 23(6): 524-526.(in Chinese)
[13] Doel, M. T. , & Carey, N. H. . (1976). The translational capacity of deadenylated ovalbumin messenger .Cell, 8(1): 51-58.
[14] Zhang, X. , Liu, Z. , Zhao, Q. , Chang, H. , & Xie, Q. . (2004). Sequencing and analysis for the full-length genome rna of foot-and-mouth disease virus hina/99.ScienceinChina, 47(1): 74-81.
[15] Carrillo, C. , Tulman, E. R. , Delhon, G. , Lu, Z. , Carreno, A. , & Vagnozzi, A. , et al. (2005). Comparative genomics of foot-and-mouth disease virus.JournalofVirology, 79(10): 6,487-6,504.
[16] Biswal, J. K., Subramaniam, S., Ranjan, R., & Pattnaik, B. (2016). Partial deletion of stem-loop 2 in the 3' untranslated region of foot-and-mouth disease virus identifies a region that is dispensable for virus replication.ArchivesofVirology, 161(8): 1-6.
[17] 谢庆阁,口蹄疫[M],北京:中国农业出版社,2004: 23.
XIE Qin-ge. (2004).Foot-and-MouthDisease[M]. Beijing: China Agriculture Press: 23. (in Chinese)
[18] Plotch, S. J. , & Palant, O. . (1995). Poliovirus protein 3AB forms a complex with and stimulates the activity of the viral RNA polymerase, 3dpol.JournalofVirology, 69(11): 7,169-7,179.
[19] Chatterjee, N. K. , Bachrach, H. L. , & Polatnick, J. . (1976). Foot-and-mouth disease virus RNA: presence of 3′-terminal polyriboadenylic acid and absence of amino acid binding ability.Virology, 69(2): 369-377.
[20] 张克山,刘永杰,卢国栋,等.口蹄疫病毒基因组结构及功能最新研究进展[J].中国人兽共患病学报,2011,27(9) :836-838.
ZHANG Ke-shan, LIU Yong-jie, LU Guo-dong, et al. (2011). Recent advances in the structure and function of FMDV genome [J].ChineseJournalofZoonoses, 27(9):836-838.(in Chinese)
[21] Baxt, B. , Grubman, M. J. , & Bachrach, H. L. . (1979). The relation of poly(A) length to specific infectivity of viral RNA: a comparison of different types of foot-and-mouth disease virus.Virology, 98(2): 480-483.
