传统发酵乳制品中微生物多样性研究
2018-01-22,,,,
,, ,,
(内蒙古农业大学,乳品生物技术与工程教育部重点实验室农业部奶制品加工重点实验室,内蒙古呼和浩特 010018)
我国是一个地域辽阔的多民族国家,拥有众多具有独特民族特色的传统食品。传统发酵乳制品作为这类食品的典型代表,其主要由微生物自然发酵各种动物乳而获得,产品的主要发酵为乳酸[1]。我国部分地区居民早在2000多年前就有制作和食用发酵乳制品的习俗,加之不同的气候环境、地理因素以及制作工艺的差异,使得传统发酵乳制品中蕴藏的微生物多样性极为丰富[2-3]。这些微生物对传统发酵乳制品的风味、组织质地、稳定性等方面有很重要的影响[4-5]。
454焦磷酸测序因其具有效率高、通量高、准确率高等特点,被广泛用于微生物多样性的研究。之前也有一些基于该技术对传统自然发酵乳制品如酸牛奶、酸马奶和酸驼奶中细菌多样性的研究报道,但这些研究大多集中于探究传统发酵乳制品中乳酸菌的生物多样性[6-8],而对传统乳制品真菌多样性的研究相对较少。此外,这些研究大多仅停留在微生物多样性的揭示,缺乏对核心菌群及其相关关系的深入探究。全面分析传统发酵乳制品的生物多样性、共有菌群以及共有菌群间相关关系,对于掌握传统发酵乳制品风味物质的产生及变化规律、优化这类产品的生产工艺、实现产品标准化和工业化生产以及科学地筛选开发具有优良品质的工业发酵剂具有重要意义。
本研究基于上述背景,通过454焦磷酸测序技术高通量测序技术对采集自我国新疆地区的酸牛奶、酸马奶和酸驼奶三份样品中微生物多样性、丰度、核心菌群以及核心菌群之间的相关关系进行分析。以期了解传统不同地区和不同类型发酵乳制品中的微生物多样性,为标准化和工业化生产传统发酵乳制品提供数据支持,同时对于开发适合不同发酵乳制品的工业发酵剂提供有意参考。
1 材料与方法
1.1材料与仪器
样品 本研究从新疆自治区特克斯镇一大队局马洪(奇勒乌泽克乡)牧民家中,分别采集了牧民使用传统发酵手段制备的酸牛奶、酸马奶和酸驼奶样品各一份。将样品放置于有冰袋的冷藏箱内迅速运回实验室,并置于-80 ℃冰箱冷藏备用。
5810R型冷冻离心机 德国Eppendorf公司;GDS-8000型凝胶成像仪 美国UVP公司;DYY-12型电泳仪 北京六一公司;PTC-200型梯度PCR扩增仪 美国伯乐公司;2100型生物分析仪 美国安捷伦公司;RocheGSFLX型测序仪 美国Roche454公司;ND-2000C微量紫外分光光度计 美国Nano Drop公司;OMEGA DNA isolation kit(D5625-01)试剂盒 美国OMEGA公司。
1.2实验方法
1.2.1 样品DNA提取及PCR扩增 使用OMEGA DNA isolation kit(D5625-01)试剂盒提取微生物宏基因组,完整度(琼脂糖凝胶电泳结果在500 bp处有单一明亮条带)、纯度(OD260/OD280在1.8~2.0之间)及浓度(在10~20 μg之间)均满足后续实验要求的DNA样品暂存到-20 ℃冰箱备用。
PCR扩增体系:10×PCR缓冲液2.5 μL,10 mmol/L dNTPs mix 2 μL,正向引物1 μL,反向引物1 μL,5 U/μL DNA聚合酶0.3 μL,模板DNA 1 μL,体系用去离子水补充至25 μL。
细菌16S rRNA V1-V3区正向引物为27F(5′-AGAGTTTGATCCTGGCTCAG-3′),其反向引物为533R(5′-TTACCGCGGCTGCTGGCAC-3′)。真菌18S rRNA正向引物3NDF(5′-GGCAAGT CTGGTGCCAG-3′);其反向引物为(5′-ACGGTATCT(AG)ATC(AG)TCTTCG-3′)且在正向引物中加入7个核苷酸标签(barcode)。
扩增条件为:首先94 ℃预变性5 min,然后94 ℃变性1 min,58 ℃退火1 min,72 ℃延伸2 min后重复30个循环,最后72 ℃延伸10 min。扩增产物经1% 琼脂糖凝胶电泳检验合格后,PCR产物置-20 ℃冰箱备用。
1.2.2 454焦磷酸测序 通过产物纯化、安捷伦2100生物分析仪定量混匀,上样测序。焦磷酸测序按照 454 Roche GS-FLX的标准流程进行测试。
1.2.3 序列优化及质控 454测序得到的数据确认引物位置后,认为两端引物之间的片段为可变区,筛选可变区长度大于300 bp的序列。根据样本barcode编号进行筛选,必须为首7位且不允许错配。控制整条序列上质量得分大于20的碱基所占比例高于93%。
1.2.4 生物信息学分析 通过QIIME(v1.7.0)分析平台对进行过质量控制的序列进行物种丰度和多样性分析,主要的操作步骤如下:
通过PyNAST对序列进行校准排齐,在序列100%相似性下进行UCLUST归并,建立无重复的16S rRNA序列集;
在给定相似度97%水平上进行分类操作单元(Operational taxonomic units,OTU)划分;
通过ChimeraSlayer检测属于嵌合体的OTU,随后去除;
将OTU代表性序列通过RDP(Ribosomal Database Project,Release 11.5)和Greengenes(Release 13.8)数据库进行同源性比对,整合两个数据库的比对结果,确定每个OTU最终的分类学地位;
使用FastTree软件绘制基于OTU代表性序列的系统发育进化树,并计算样品的α和β多样性。
1.2.5 多元统计学分析 对传统发酵乳制品中核心细菌和真菌的丰富度指标、多样性指标和基于属分类地位的细菌和真菌相对含量进行相关性分析。通过Origin 8.5软件和R软件(v3.3.2)对QIIME产生的结果进行数据分析和数据可视化处理。
1.2.6 核酸登录号 本研究中所有序列数据已提交至MG-RAST数据库,项目ID号为mgp79277。
2 结果与分析
2.1样品中细菌分析
2.1.1 细菌的丰度和多样性分析 通过454焦磷酸高通量测序,分别获得7367条、15303条和11748条酸牛奶、酸马奶和酸驼奶样品高质量细菌序列。三份传统发酵乳制品样品细菌的稀疏曲线(图1A)和香农指数曲线(图1B)如图1所示。由图1A可知,随着测序深度的增加发现物种的数量也随之增加,但与此同时每个样品的shannon曲线已进入平台期。由此可知,虽然随着测序量的增加有可能会发现新的细菌种系型,但样品细菌的多样性已经不再随测序量的增加而发生变化。因此本研究细菌测序量满足后续生物信息学分析的要求。
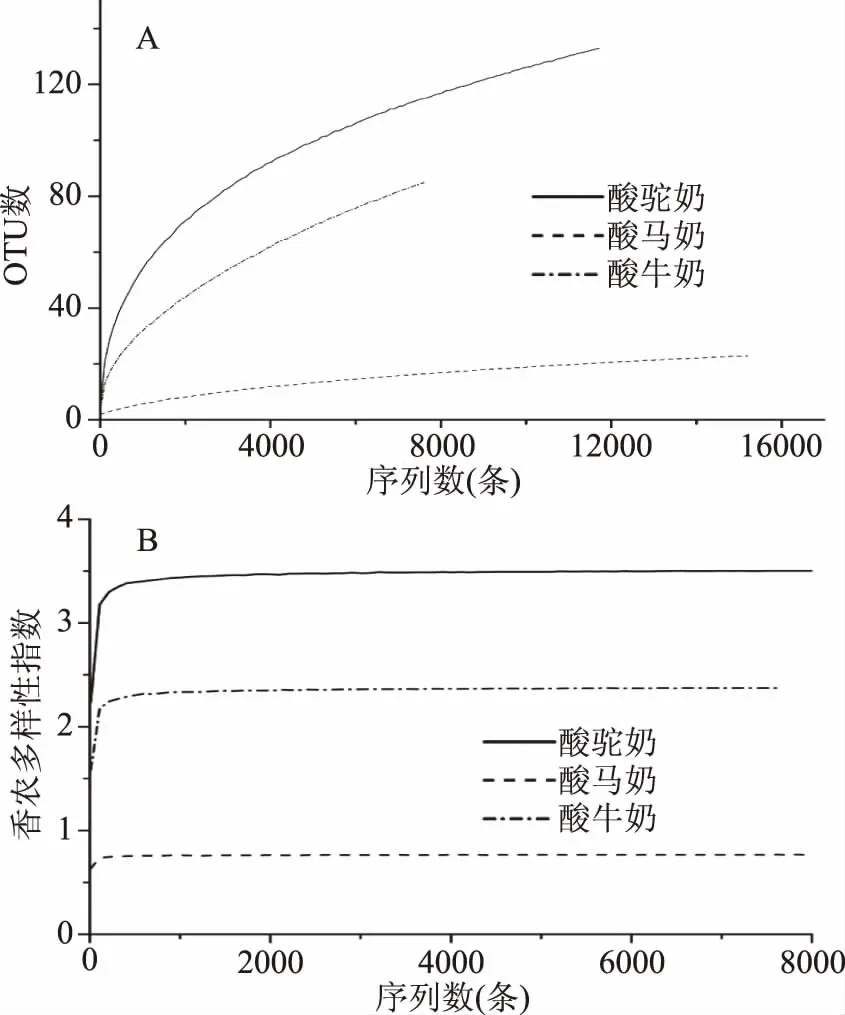
图1 3份传统发酵乳制品样品中细菌的稀疏曲线(A)和香农指数(B)Fig.1 Rarefaction analysis(A)and shannon diversity(B) estimates in bacteria from three traditional fermented dairy products samples
进一步分析发现,酸牛奶、酸马奶和酸驼奶样品分别得到了86个、24个和134个细菌OTUs。与此同时,三份传统发酵乳制品样品的chao1指数分别为136.18、31.94和208.82,simpson指数分别为0.62、0.32和0.82,shannon指数分别为2.37、0.77和3.51。上述结果表明,在微生物丰度和多样性方面酸驼奶高于酸牛奶和酸马奶、酸马奶高于酸牛奶。造成该结果的原因可能是三种传统发酵乳制品原料中蛋白质、VA、Fe、Ca、VB2含量在鲜驼奶、鲜牛奶和鲜马奶中依次降低[9],其原料乳营养成分的不同可能导致了菌群多样性和丰度的不同。
2.1.2 细菌OTU水平上三份传统发酵乳制品样品菌群分析 为了研究三份不同传统发酵乳制品中细菌菌群的共性和差异,本研究在OTU水平上绘制了3份样品细菌OTUs的Venn(维恩)图,如图2所示。三份传统发酵乳制品中,共有的OTUs为6个,包含24777条序列,占全部序列(共计34688条序列)的71.4%。酸牛奶与酸驼奶样品共享26个OTUs,其中包含28110条序列,占全部序列的81.0%。酸马奶样品与酸驼奶样品共享14个OTUs,其中包含25084条序列,占全部序列的72.3%。酸牛奶样品、酸马奶样品与酸驼奶样品,分别拥有59个、9个和99个独有OTUs,分别包含5143、16和1112条独有序列,分别占全部序列(共计34688条序列)的14.8%、0.05%、3.2%。上述结果显示,三份传统发酵乳制品共有的OTU个数虽然少,但是拥有大量的序列数。该结果表明,在三份传统发酵乳制品中存在一部分具有大量序列的共有OTUs。

图2 基于OTU水平对三种传统发酵乳制品菌群分析Fig.2 Based on OTU levels of three types of traditional fermented dairy products bacteria flora analysis
2.1.3 样品中细菌相对含量的比较分析 对样品中的细菌相对含量进行分析,结果发现酸牛奶中细菌分别由Firmicutes(89.79%)、Proteobacteria(9.22%)、Bacteroidetes(0.55%)、Actinobacteria(0.39%)和Deinococcus-Thermus(0.05%)5个菌门组成。酸马奶中细菌分属Firmicutes(99.98%)、Proteobacteria(0.01%)和Bacteroidetes(0.01%)3个菌门。酸驼奶中细菌分别由Firmicutes(82.94%)、Proteobacteria(16.73%)、Bacteroidetes(0.19%)、Actinobacteria(0.12%)、Acidobacteri(0.01%)和Fusobacteria(0.01%)组成。由图3可知,三份传统发酵乳制品均主要以Firmicutes为主。

图3 3份传统乳制品样品中的细菌组成Fig.3 Compositions of bacteria in three traditional dairy products
如图3 所示,在属水平上酸牛奶样品中的细菌主要由Lactococcus、Leuconostoc、Kluyvera、Lactobacillus和Weissella组成,其相对含量分别为66.78%、15.19%、8.34%、4.84%和1.90%。酸马奶样品中的细菌以Lactobacillus为主,其相对含量为99.93%。酸驼奶样品中大于1%的细菌属有Lactobacillus、Lactococcus、Leuconostoc、Acetobacter、Streptococcus、Citrobacter、Pseudomonas和Shigella,其相对含量分别为53.72%、18.72%、6.44%、6.09%、3.94%、3.72%、2.38%和1.51%。由此可见,Lactobacillus是酸马奶和酸驼奶中的主要优势细菌属,发酵酸牛奶中Lactococcus为主要优势细菌属。值得一提的是,本研究还从酸马奶样品中检测到有害菌属——志贺菌属。该菌属的存在可能与自然发酵的开放状态和传统制作工艺的粗放操作有关,预示着传统家庭作坊式的乳制品制作工艺可能存在的潜在安全问题。但由于本研究样本数较少,有害菌属的来源仍需进一步实验进行验证。
Oki 等以焦磷酸测序技术,对蒙古国6个地区的53份传统发酵乳中细菌多样性进行了分析,得到3 个门21 个细菌属,其中属Lactobacillus为优势菌属[10]。Xu等和Liu等采用同样的技术分别对中国新疆和中国西藏地区的传统发酵乳制品中的乳酸菌多样性进行了分析,其结果均显示Firmicutes为绝对优势细菌门Lactobacillus为绝对优势菌属[11-12]。而包秋华等以纯培养和DGGE技术对四川发酵酸牦牛奶制品曲拉进行乳酸菌多样性分析,研究发现乳杆菌属为优势属[13]。呼斯楞等以纯培养和16S rRNA基因序列分析法对内蒙古呼伦贝尔地区传统发酵乳中的乳酸菌进行多样性分析,得到Lactococcuslactissubsp.lactis为内蒙古呼伦贝尔地区传统发酵乳制品的优势菌种占总分离株的25%,其次为Lactobacillusparacasei,占总分离株的20.83%[14]。
以上结果与本实验结果相近,均表明在传统发酵乳制品中存在丰富的细菌多样性。牛奶、马奶和驼奶的营养成分存在差异,不同营养成分导可能致了乳制品中菌群结构存在差异[15]。在三份传统发酵乳制品中,酸驼奶样品的微生物菌群组成相较于其余两种传统发酵乳制品样品的微生物群落更加复杂。而酸马奶的微生物菌群组成在三份传统发酵乳制品中相对简单。
2.2样品中真菌分析
2.2.1 真菌丰度和多样性分析 酸牛奶、酸马奶和酸驼奶样品经过测序后分别得到4375条、7599条和5360条高质量真菌序列,经过OTU划归后分别得到了41个、21个和11个OTUs。三份传统发酵乳制品样品真菌的chao1 指数分别为26.59、11.09和49.94,simpson指数分别为0.48、0.28和0.75,shannon指数分别为1.31、0.95和2.65。样品的真菌的稀疏曲线和香农指数如图4所示。其结果与细菌结果相似,随着测序测序深度的增加本研究将获得真菌的物种数量也随之增加,但与此同时每个样品的shannon曲线已进入平台期。虽然随着测序量的增加新的真菌种系将从样品中被发现,但样品真菌的多样性已经不再随测序量的增加而发生变化。因此本研究的真菌的测序量亦满足后续的生物信息学分析要求。

图4 3份传统发酵乳制品样品中真菌的香农指数图(A)和稀疏曲线(B)Fig.4 Shannon diversity estimates(B)and rarefaction analysis(A) in fungi from three traditional fermented dairy products samples
2.2.2 基于OTU水平对三份传统乳制品样品菌群分析 为了研究三种不同传统发酵乳制品中证明菌群的共性和差异,本研究在OTU水平上绘制了三份样品真菌OTU水平的Venn图,其结果如图5所示。可以看出三份样品包含的244个OTUs,其中包含17334条序列。其中,三份样品共有的OTU为5个,其中包含13936条序列,占全部序列的80.4%。酸牛奶样品与酸马奶样品共享7个OTUs,其中包含16028条序列,占全部序列的92.5%。酸牛奶样品与酸驼奶样品共享9个OTUs,其中包含14210条序列,占全部序列的82.0%。酸马奶样品与酸驼奶样品共享6个OTUs,其中包含14065条序列,占全部序列的81.1%。酸牛奶样品、酸马奶样品与酸驼奶样品,分别拥有30、12和1个独有OTU,分别包含480、405和18条独有序列,分别占样品全部序列的2.8%、2.3%、0.1%。其结果与细菌OTU水平Venn图结果相似,三份传统发酵乳制品中共有的OTUs虽然数目较少,但包含整个序列中大部分的序列。而这部分共有的OTU可能是传统发酵乳制品中核心菌群。

图5 基于OTU水平对三种传统发酵乳制品菌群分析Fig.5 Based on OTU levels of three types of traditional fermented dairy products
2.2.3 样品中真菌含量的比较分析 传统发酵乳制品中真菌的多样性及群落结构明显低于细菌的多样性和群落结构。酸牛奶样品和酸驼奶样品中均包含Ascomycota和Basidiomycota2个真菌门,其相对含量分别为98.83%、0.11%和99.98%、0.02%。酸马奶样品只含有Ascomycota。由此可知Ascomycota为三份传统发酵乳样品的优势真菌菌门。据报道,Ascomycota也是韩国酒精饮料(Korean alcoholic beverages)、可可豆(Coco bean)、发酵白酒(Chinese liquor)的优势菌门,于此同时Ascomycota是真菌界中种类最多的一个门[16-17]。
如图6 所示,从酸牛奶样品中共发现17 个真菌属,其中优势真菌属分别为Galactomyces、Gibberella、Vanderwaltozyma、Wickerhamomyce、Dipodascus,其含量分别为39.36%、37.10%、8.59%、6.33%、5.17%、1.28%和1.23%。酸马奶样品中共发现6个真菌属,其中优势真菌属分别为Vanderwaltozyma和Dipodascus,其相对含量分别为69.43%和30.00%。酸驼奶样品中共发现7个真菌属,其中优势真菌属分别为耐碱酵母属Galactomyces、Vanderwaltozyma、Gibberella,其相对含量分别为84.57%、9.24%和2.72%。Galactomyces是酸牛奶和酸驼奶样品的优势真菌属,酸马奶的优势真菌属为Vanderwaltozyma。
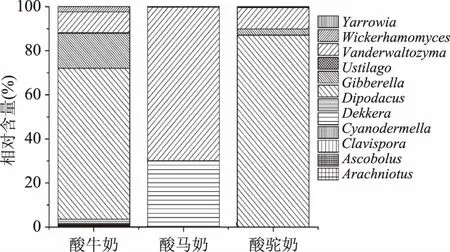
图6 3份传统乳制品样品中的真菌组成Fig.6 Compositions of fungus in threetraditional fermented dairy products
孙志宏等以焦磷酸测序技术,对西藏牦牛奶样品进行生物多样性分析,其研究结果发现Galactomyces为优势菌属[18]。乌仁图雅以同样的方法对内蒙古多部地区的酸牦牛奶的生物多样性进行了分析,其中优势真菌门为Ascomycota、优势真菌属为Galactomyces[19]。而张冬蕾以焦磷酸测序技术,对新疆22份传统发酵乳制品进行生物多样性分析,结果显示其优势菌门为Ascomycota、优势真菌属为Saccharomyces[20]。上述结果与本实验结果一致,其均证明不同发酵类型的传统发酵乳制品菌群构成存在一定的差异。其结果可能是导致传统发酵乳制品口感丰富的原因之一。
2.3共有菌群中细菌与真菌相关关系
图2和图5的结果表明,三份传统发酵样品共享的OTU数目虽少,但包含的序列数却很多,这部分OTU所代表的微生物类群有可能是传统发酵乳样品的“核心微生物群”。此外,每份样品还有一定数量独有的OTU,但这部分OTU包含的序列数相对较少。这进一步说明,传统发酵乳制品中主要优势菌种始终存在,而由于发酵乳原料不同以及加工手段不同会使得一些低丰度的物种存在一定差异。
为探究三份发酵乳中核心发酵菌群中细菌与真菌的相关关系,本研究从三份样品中均含有的菌群出发,根据菌群相对含量利用R软件计算其相关性,结果如图7所示。结果显示,Lactococcus与Galactomyces、Lactococcus与Leuconostoc的相对含量存在显著负相关(p<0.05),而Leuconostoc与Galactomyces、Gibberella与Prevotella的相对含量存在显著正相关(p<0.05)。
据报道在发酵乳制品中,Leuconostoc与真菌或细菌真菌共生体共同参与发酵乳制品的发酵过程[21]。因此在三份乳制品中,Leuconostoc与Galactomyces可能存在互利共生关系,导致其相对含量存在显著正相关。而酵母菌据报道在促进微生物的共生作用、CO2的形成、提高特殊风味和香气起着重要作用[22]。据报道,酵母菌可以分泌脂肪酶,分解脂肪形成游离脂肪酸抑制了乳球菌属的生长;与此同时,乳酸菌产生的苯乳酸、4-羟基-苯乳酸和环肽抑制了酵母菌的生长,其可能是导致Lactococcus与Galactomyces的相对含量呈现显著负相关的原因[23]。但由于本实验的样本数较少,确定发酵乳中细菌和真菌相互作用机理需要进一步实验进行验证。

图7 3份传统乳制品样品中的真菌与细菌的相关关系Fig.7 The correlation between fungi and bacteria in three traditional fermented dairy products
3 结论
本研究采用高通量454焦磷酸测序技术,对从新疆奇勒乌泽克乡同一牧区采集的三份传统发酵酸牛奶、酸马奶和酸驼奶样品中的微生物多样性、核心菌群及核心菌群间的相关关系进行分析。结果发现发现,酸牛奶、酸马奶和酸驼奶样品中的优势细菌属分别为Lactococcus、Lactobacillus和Lactobacillus,优势真菌属依次为Galactomyces、Vanderwaltozyma和Galactomyces。三份传统发酵乳中存在相似的优势菌属,但其菌群结构存在差异。在三份传统发酵样品中存在共有微生物群。且共有菌群中Lactococcus与Galactomyces、Lactococcus与Leuconostoc的相对含量存在显著负相关(p<0.05),而Leuconostoc与Galactomyces、Gibberella与Prevotella的相对含量存在显著正相关(p<0.05)。上述研究成果为工业发酵剂的研发,以及进一步对传统发酵乳制品中的菌群构成、核心菌群及核心菌群间相关关系的研究有一定参考价值。
[1]张和平. 自然发酵乳制品中乳酸菌的生物多样性[J]. 生命科学,2015(7):837-846.
[2]张敏,张艳,兰国伟,等. 高通量测序技术在乳制品微生物多样性中的研究进展[J]. 中国农学通报,2016,32(32):48-52.
[3]Roh S W,Kim K H,Nam Y D,et al. Investigation of archaeal and bacterial diversity in fermented seafood using barcoded pyrosequencing[J]. Isme Journal,2010,4(4):1-16.
[4]李太君,高媛. 发酵乳制品发酵剂的研究进展[J].中国乳业,2014(4):59-61.
[5]杨彦荣. 应用高通量测序研究西藏地区传统发酵牦牛乳中微生物的多样性[D].呼和浩特:内蒙古农业大学,2016.
[6]Aldrete-Tapia A,Escobar-Ramírez M C,Tamplin M L,et al. High-throughput sequencing of microbial communities in Poro cheese,an artisanal Mexican cheese[J]. Food Microbiology,2014,44(6):136.
[7]Sun Z,Liu W,Bao Q,et al. Investigation of bacterial and fungal diversity in tarag using high-throughput sequencing[J]. Journal of Dairy Science,2014,97(10):6085-6096.
[8]Wu X H,Zhang L,Li Y,et al. A survey on composition and microbiota of fresh and fermented yak milk at different Tibetan altitudes[J]. Dairy Science & Technology,2009,89(2):201-209.
[9]王建光,孙玉江,芒来. 马奶与几种奶营养成份的比较分析[J]. 食品研究与开发,2006,27(8):146-149.
[10]Oki K,Dugersuren J,Demberel S,et al. Pyrosequencing analysis of the microbial diversity of airag,khoormog and tarag,traditional fermented dairy products of mongolia[J]. Bioscience of Microbiota Food & Health,2014,33(2):53-64.
[11]Xu H,Liu W,Gesudu Q,et al. Assessment of the bacterial and fungal diversity in home-made yoghurts of Xinjiang,China by pyrosequencing[J]. Journal of the Science of Food & Agriculture,2014,95(10):2007-2015.
[12]Liu W,Xi X,Sudu Q,et al. High-throughput sequencing reveals microbial community diversity of Tibetan naturally fermented yak milk[J]. Annals of Microbiology,2015,65(3):1741-1751.
[13]包秋华,王俊国,刘文俊,等. 传统发酵乳制品曲拉中乳酸菌多样性分析[J]. 中国乳品工业,2016,44(7):12-14.
[14]呼斯楞,刘红新,于洁,等. 内蒙古呼伦贝尔地区传统发酵乳中乳酸菌的多样性分析[J]. 微生物学通报,2016,43(5):984-990.
[15]Jung M J,Nam Y D,Roh S W,et al. Unexpected convergence of fungal and bacterial communities during fermentation of traditional Korean alcoholic beverages inoculated with various natural starters[J]. Food Microbiology,2012,30(1):112.
[16]Illeghems K,Vuyst L D,Papalexandratou Z,et al. Phylogenetic Analysis of a Spontaneous Cocoa Bean Fermentation Metagenome Reveals New Insights into Its Bacterial and Fungal Community Diversity[J]. Plos One,2012,7(5):e38040.
[17]Li X R,Ma E B,Yan L Z,et al. Bacterial and fungal diversity in the traditional Chinese liquor fermentation process[J]. International Journal of Food Microbiology,2011,146(1):31-37.
[18]孙志宏,刘文俊,张和平. 基于宏基因组方法对西藏传统发酵牦牛奶中微生物多样性的研究[J]. 食品科学技术学报,2012,30(4):19-24.
[19]乌仁图雅. 应用454焦磷酸测序技术对传统发酵乳制品微生物多样性的研究[D].呼和浩特:内蒙古农业大学,2014.
[20]张冬蕾.应用焦磷酸测序技术对新疆地区传统发酵乳制品中微生物多样性的研究[D]. 呼和浩特:内蒙古农业大学,2016.
[21]Dunny G M,Cleary P P,McKay L L. Genetics and molecular biology of streptococci,lactococci,and enterococci[M]. American Society for Microbiology,1991.
[22]贺银凤. 传统发酵乳制品中乳酸菌和酵母菌的互作关系[J]. 中国乳品工业,2010,38(10):43-45.
[23]Nielsen M S,Frisvad J C,Nielsen P V. Protection by fungal starters against growth and secondary metabolite production of fungal spoilers of cheese.[J]. International Journal of Food Microbiology,1998,42(1-2):91-99.
