粮棉轮作对土壤中养分及真菌多样性的影响
2018-01-12赵存鹏郭宝生刘素恩王兆晓王凯辉耿军义
赵存鹏,郭宝生,刘素恩,王兆晓,耿 昭,王凯辉,耿军义
(河北省农林科学院 棉花研究所,农业部黄淮海半干旱区棉花生物学与遗传育种重点实验室,国家棉花改良中心河北分中心,河北 石家庄 050051)
棉花是我国主要的经济作物,在农业生产中占有重要的地位。河北省中南部黑龙港流域地区是我国黄河流域棉花生产的优势区域,但由于作物结构相对单一、重茬连作现象非常严重,导致土壤速效养分含量下降,理化性状变差,土壤酶活性降低,微生物群落结构失衡[1-2],从而使棉花体内的各种酶活性改变,生长发育受阻,枯黄萎病和棉铃疫病快速蔓延,产量和纤维品质都受到了影响。
微生物是土壤中重要的有机体系,具有调节植物生长的养分循环,维持土壤中的碳氮循环,分解有机废弃物等作用[3]。根系分泌物对微生物具有选择作用,连作土壤可使微生物的物种多样性和功能多样性水平降低,给植物病原微生物生长繁殖创造了条件,并通过富集,使土传病害加重[4-5]。不同的种植模式可以通过对土壤的理化性质、微生物的环境条件和作物间化感作用来改变土壤微生物的群落结构和功能[6],土壤的群落特征比土壤有机质、养分含量等理化性状能更敏感地对土壤的变化做出响应[7]。利用轮作的种植模式在大豆、马铃薯、黄瓜等作物上均发现土壤中的微生物活性有了明显的改善[5,8-11]。
随着近年来水利条件的逐步改善,冀中南棉区具备了种植粮食作物的条件,华北地区的光热条件充足为棉花-小麦-玉米粮棉轮作两年三熟提供了生态条件。合理倒茬轮作有利于提高该区域土壤生产能力,提高棉花的生物学产量及籽棉产量[12],因此,在河北省中南部传统连作棉区发展粮棉轮作两年三熟种植模式,有利于提高复种指数,在实现稳定棉花生产的同时,大幅度提高河北省粮食的增产能力。
本研究为了提高传统棉区粮棉综合生产能力,开展了在连作棉田、棉花-小麦-玉米两年三熟轮作种植模式下,土壤养分和微生物的变化研究,以期为冀中南连作棉田合理倒茬轮作、提高土壤产出率提供理论指导。
1 材料和方法
1.1 试验概况
试验地在河北省南宫市棉花原种场长期定位试验点进行,该试验点地理位置为37°16′N,115°26′E,属黄河冲积平原,地势平坦,海拔为30 m左右。土壤类型为潮土。属温暖带亚湿润大陆性季风型气候区,年平均气温13.1 ℃,无霜期203 d,年日照时数2 472 h,年平均降雨量476 mm。
试验点自1976年成立以来,试验地及周边一直以种植棉花为主,特别是转Bt基因棉花推广以来,试验地在试验前的近20年间种植的均为抗虫棉。试验设有2个处理,即连作模式的棉田和粮棉轮作的小麦田、玉米田。每个试验小区长70 m,宽10 m,面积为700 m2。每个处理3次重复,随机区组排列。播种前一次施用复合肥为底肥,用量为N 225 kg/hm2,P2O5120 kg/hm2,K2O 180 kg/hm2。棉花蕾铃期(玉米播种时)追施N 150 kg/hm2。
试验自2013年开始,连作棉田于每年4月下旬播种棉花,10月下旬收获;粮棉轮作两年三熟试验则是在收获棉花后,马上整地播种冬小麦,2014年6月收获小麦播种玉米,2015年4月再种植棉花。种植棉花品种为冀2658,小麦品种为邢麦7号,玉米品种为联创808。各种作物栽培管理按照相应的品种要求进行,作物秸秆均做还田处理。
1.2 样品采集与处理
分别于2016年4月(连作棉田整地施用底肥后棉花播种前)、6月(玉米播种后)、8月(棉花花铃期)、10月(作物收获后)采集地下5~25 cm耕层的土样,每个小区采用Z字形取土法取8点,8点土样混匀剔除植物残体,用四分法保留500 g左右,一部分土样自然摊晾,风干后过2 mm 筛用于土壤养分测定,另一部分鲜土样保存于-80 ℃冰箱中,用于微生物多样性分析,为了方便测序和数据分析将连作棉田标为字母MIANH,4,6,8,10月分别标为MIANH1、MIANH2、MIANH3和MIANH4;粮棉轮作模式土壤则标为XIAOM,4,6,8,10月分别标为XIAOM1、XIAOM2、XIAOM3和XIAOM4。
1.3 土壤养分测定
土壤全氮的测定采用凯氏定氮法;土壤速效磷的测定采用NaHCO3浸提-钼锑抗比色法;土壤速效钾的测定采用乙酸铵浸提-原子吸收火焰光度计法[13]。
1.4 土壤总DNA的提取和测定
使用美国MPBIO公司的FastDNA SPIN Kit for Soil试剂盒,依照试剂盒说明提取土壤总DNA。提取后的DNA溶液用超微量紫外分光光度计测定其纯度和浓度,浓度>50 ng/μL的样品在0.8%的琼脂糖凝胶电泳后检查片段是否完整,检查合格后存放在-80 ℃冰箱保存待测序。将冻存的土壤总DNA样品干冰包装送至北京百迈客生物科技有限公司,应用Illumina HiSeq 2500平台进行高通量测序。
1.5 数据处理与分析
测序完成后对原始数据经过QIIME软件进行拼接[14],将拼接得到的序列进行质量过滤[15],并去除嵌合体[16],得到高质量的Tags序列。利用物种分类的 OTU(Operational taxonomic units),在相似性97%的水平上对序列进行聚类[17],以测序所有序列数的0.005%作为阈值过滤OTU[18]。
物种注释采用RDP Classifier[19],置信度阈值为0.8(http://sourceforge.net/projects/rdp-classifier/)。利用的数据库为真菌 ITS:Unite[20](Release 7.0,http://unite.ut.ee/index.php)。
微生物多样性分析采用alpha指数分析和Beta多样性分析。利用Mothur软件(v1.30)进行alpha分析[21];基于R语言平台绘制样本主成分分析、主坐标分析以及环境因子与样本组成相关性分析。组间差异显著性分析采用LefSe (http://huttenhower. sph.harvard.edu/lefse/)分析寻找在所有组间具有显著性差异的物种(LDA值为4.0),再利用Metastats (http://metastats.cbcb.umd.edu/)对组间的物种丰度数据进行t检验得到P值,并对P值进行校正得到q值;最后根据P值(或q值)筛选出导致2组样品组成差异的物种。
2 结果与分析
2.1 不同种植模式下土壤养分变化
通过不同时期对不同种植模式试验区土壤取样分析表明,由于周年生产土地上作物生育期变化和作物类型变化及田间施肥浇水的变化,土壤中氮、磷、钾的含量处于动态变化之中。在棉花和玉米收获时,不同种植模式下,土壤中氮、磷、钾类营养元素的残留,更能反映出轮作后土壤养分变化趋势。截止到10月下旬轮作结束后,轮作模式土壤全氮、速效磷残留含量比连作棉田显著增加,而速效钾则差异不显著(表1)。此外,粮棉轮作模式的土壤中在8月的全氮、速效磷、速效钾含量显著高于棉花连作的地块,可能是由于小麦秸秆还田增加了土壤中养分的含量,速效钾在全年的差异主要为棉花对钾肥的需求量大于小麦、玉米对钾肥的需求。根据分析不同轮作制度下土壤养分的残留量认为,棉花-小麦-玉米两年三熟轮作模式下,种植小麦、玉米后,可减少接茬棉花钾肥施用量。
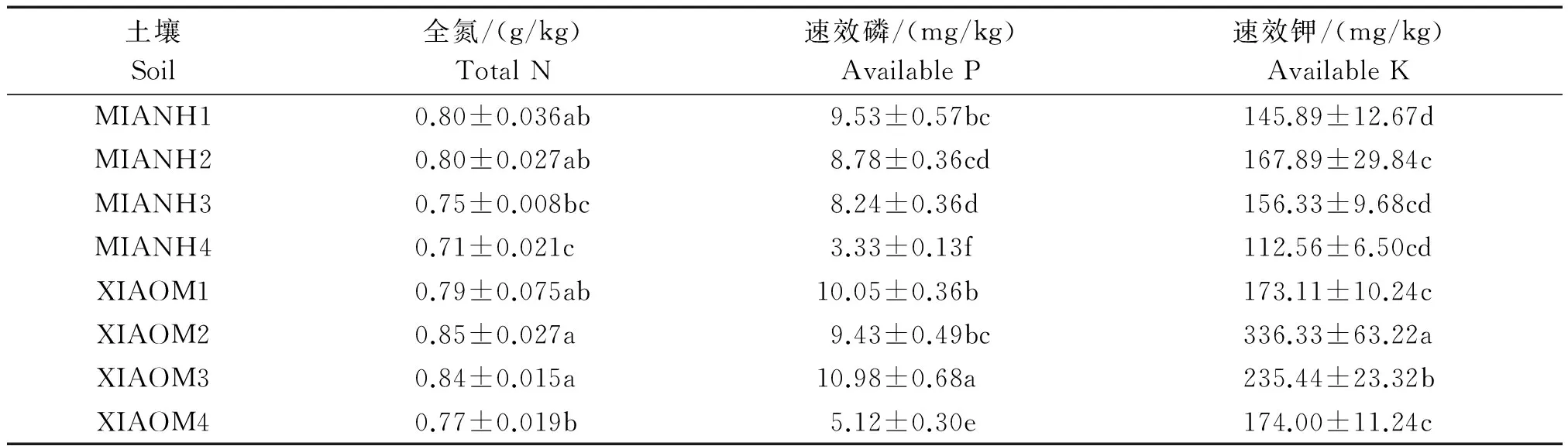
表1 不同种植模式下各样地土壤养分含量Tab.1 The condition of soil nutrient under different cotton planting patterns
注:表中数据为平均值±标准差;同列数值后不同字母表示处理间差异达显著水平(P<0.05)。表2-3,6同。
Note:Data areX± s; Values followed by different letters in a row are significant among treatments at the 0.05 level. The same as Tab.2-3,6.
2.2 不同种植模式下土壤真菌测序结果及OTU分析
本研究24个样品测序共获得1 631 343条有效序列,平均每个样品的测序深度为67 973条有效序列。在97%的相似度水平下,通过聚类得到各样品的OTU个数,OTU越多,则证明菌群的多样性越大。粮棉轮作模式下真菌群落的多样性较棉花连作模式下丰富,OTU个数分别为783,750个。2种模式共有的相似菌群为693个,粮棉轮作模式土壤有90个特有菌群,棉花连作土壤有特有菌群57个。表2为各组各时期的测序结果,从表中可以得到连作棉田进行轮作后能够增加土壤微生物菌群数量,特别是在6月,小麦收获后,粮棉轮作的土壤微生物多样性明显大于棉花连作的土壤,为全年微生物最为活跃的时间点。

表2 不同种植模式下土壤真菌测序结果Tab.2 The results of fungi sequencing of soil under different cotton planting patterns
2.3 不同种植模式对土壤真菌的Alpha多样性的影响
2种种植模式下,土壤真菌的Alpha多样性指数存在明显的差异(表3)。多样性指数Shannon仅在6月棉花连作和粮棉轮作模式中差异达到显著水平,其他时间在2种种植模式中差异均不显著。6月的粮棉轮作模式中的ACE指数和Chao1指数均显著高于棉花连作模式中的指数,同时粮棉轮作模式中Shannon指数又显著低于棉花连作模式中的指数,这主要是由于在小麦收获后,玉米刚播种,试验田真菌的多样性较大,但个体间的分配很不均匀造成的,说明种植作物的改变直接影响着真菌的种类和数量的变化。通过分析土壤养分与真菌群落的个数及Alpha多样性指数的Pearson相关性(表4)可以得知,OTU数与土壤全氮、速效钾的含量具有显著相关性,Alpha多样性指数也与土壤全氮、速效钾的含量具有显著相关性。
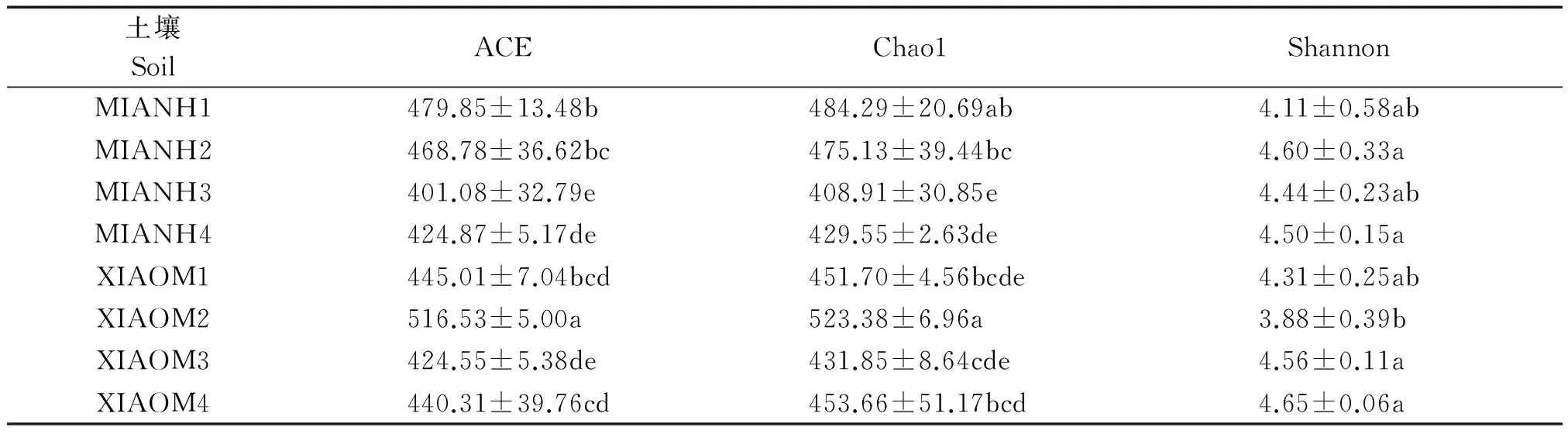
表3 不同种植模式下土壤中微生物 Alpha 多样性指数分析Tab.3 Alpha diversity index of soil fungi under different cotton planting patterns
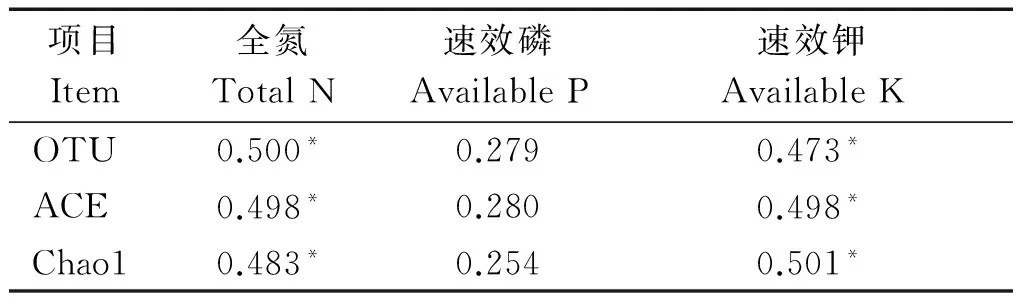
表4 OTU个数、Alpha多样性指数与土壤化学性质 Pearson 相关性分析Tab.4 Pearson correlation coefficients between OTU number,Alpha diversity index and soil chemical
注:*. 0.05 水平具有显著相关性。
Note:*.Correlation is significant at the 0.05 level.
2.4 不同种植模式对土壤真菌群落组成的影响
将OTU的代表序列与微生物参考数据库进行比对可得到每个OTU对应的物种分类信息,进而在各个水平上统计各样品群落的组成。图1为2个种植模式下不同时间点土壤真菌微生物门水平的组成,主要包括子囊菌门(Ascomycota)、担子菌门(Basidiomycota)、接合菌门(Zygomycota)、壶菌门(Chytridiomycota)、球囊菌门(Glomeromycota)等,此外还有一部分真菌目前还未知。连作棉田中子囊菌门所占的比例为32.12%~64.21%,担子菌门所占的比例为12.11%~23.38%,接合菌门所占的比例为5.19%~15.97%,壶菌门所占的比例为0.33%~1.19%,球囊菌门所占的比例为0.16%~2.15%;而轮作模式土壤中子囊菌门所占的比例为34.60%~45.03%,担子菌门所占的比例为8.49%~22.10%,接合菌门所占的比例为3.99%~14.86%,壶菌门所占的比例为0.44%~1.88%,球囊菌门所占的比例为0.21%~1.51%。从图中看,不同的种植模式在不同时间点的真菌微生物的相对丰度差异较为明显。通过微生物组成与土壤化学性质Pearson相关性分析(表5),可知子囊菌门、担子菌门、壶菌门、球囊菌门与土壤的养分含量间没有明显的相关性,而接合菌门与土壤的养分呈极显著的负相关。

图1 不同种植模式下土样中微生物门水平的组成Fig.1 Relative abundance of soil fungi at phylum level under different cotton planting patterns
表6为不同种植模式下土壤微生物纲水平的组成,从表中可以得知,座囊菌纲(Dothideomycetes)在连作模式中的相对丰度为2.20%~17.97%,4月时相对丰度最高,与其他时间点的差异达到显著水平,在轮作模式中的相对丰度为5.16%~9.76%,各时间点差异不显著。球囊菌纲(Glomeromycetes)在连作模式和轮作模式中4月的相对丰度较低分别为0.16%和0.21%,但在6月的相对丰度分别达到1.45%和1.50%,8,10月在连作模式中的相对丰度为2.14%和1.39%,而同期在轮作模式中的相对丰度为0.30%和0.23%,它们之间的差异达到显著的水平,可能是种植玉米会减少球囊菌纲的丰度。

表5 微生物组成与土壤化学性质 Pearson 相关性分析Tab.5 Pearson correlation coefficients between soil chemical characteristics and microbial compositions
注:**.0.01 水平具有显著相关性。
Note:**.Correlation is significant at the 0.01 level.

表6 不同种植模式下土样中微生物纲水平的组成(8 组中至少有 1 组的相对丰度>1%)Tab.6 Relative abundance of soil microorganisms under different cotton planting patterns(At class level, at least one group in eight groups relative abundance>1%) %
2.5 不同种植模式对土壤真菌群落的聚类分析
通过聚类分析,将具有相似Beta多样性的各样本聚类在一起,各处理的结果显示本研究主要聚类为两大分支(图2),即不同的种植模式,各小分支也基本按照取样的时间点进行聚类。采用Beta多样性中主坐标分析法(Principal coordinates analysis,PCoA)分析结果如图3,经分析计算得出PC1为28.92%,PC2为11.91%。不同种植模式下的土壤样品被完全区分,表明连作棉田轮作后,土壤微生物菌群和数量发生较大变化。

图2 不同种植模式不同时间点的Beta多样性分析Fig.2 Beta diversity analysis of soil fungi under different cotton planting patterns of different times

横、纵坐标为导致样品间差异最大的2个特征值,以百分数的形式体现主要影响程度。Horizontal, vertical, to lead to difference between samples of the two largest eigenvalues, influence extent embodies mainly in the form of a percentage.
采用LEfSe(Line discriminant analysis(LDA)effect size)分析绘制进化分支图(图4)。通过分析表明,连作棉田实行与小麦玉米轮作种植后,土壤微生物菌群变化情况如下:在目水平上分别是散囊菌目(Eurotiales)、伞菌目(Agaricales)和粪壳菌目(Sordariales),散囊菌目、伞菌目显著减少,而粪壳菌目显著增加。科水平上伞菌科(Agaricaceae)显著减少,毛球壳科(Lasiosphaeriaceae)显著增加。属水平上分别为柄孢壳菌属(Podospora)的数目显著增加,白环蘑属(Leucoagaricus)的数目显著减少。
3 讨论与结论
土壤中微生物的多样性与植被变化有很大关系,植物凋落物或残留物中碳氮的生化组成、可利用程度、土壤养分含量等均影响着土壤微生物的多样性[22-23]。
国内外学者对作物连作问题进行了长期大量的研究,发现随着连作年限增加,土壤容重相应增加,作物产量则会降低,土壤中真菌数量增加,细菌和放线菌数量减少,连作会使土壤有机质含量减少,有害生物逐渐累积,最终会抑制作物的生长[24-26]。适宜的作物轮作能够减少连作障碍,改善土壤物理性质,促进土壤生物化学过程,提高土壤酶活性,改变土壤微生物区系,引起菌群组成结构发生变化,使土壤中微生物活性增强,有害微生物减少[27]。轮作会增加植物的多样性,种植的作物及其残茬对微生物的群落及其活性产生显著影响[28-29]。

进化分支图由内至外辐射的圆圈代表了由门至种的分类级别;在不同分类级别上的每一个小圆圈代表该水平下的一个分类,小圆圈直径大小与相对丰度大小呈正比;着色原则为将无显著差异的物种统一着色为灰色,其他差异物种按该物种所在丰度最高的分组进行着色。The cladogram radiation from inner to outer circle represents the from the phylum to species on the classification of the level;At different classification level, each small circle represents a classification at this level, and the size of the small circle is proportional to the relative abundance;Principle to be no significant difference of species uniform color is gray, the other differences between species according to the species abundance highest grouping staining.
特别是轮作使有益微生物如固氮菌等增加,有害微生物如腐霉菌和镰孢菌等减少,土壤微生物活性增强。轮作微生物的数量大于连作,细菌和放线菌数量增多,真菌减少。轮作还提高了土壤微生物量碳氮比,改善土壤微生态环境,增加土壤微生物群落多样性,在一定程度上消除连作障碍,避免土传病害的发生,有利于提高作物产量和品质[30]。
随着棉花向优势区域集中发展,我国棉花连作问题日益凸显,棉花连作对土壤微生物及养分影响等问题的研究工作已引起关注。首先棉花连作会造成土壤养分不均衡,一些研究结果认为土壤速效钾含量随棉花连作年限而明显降低,多酚氧化酶随连作年限增加呈现出下降趋势[31]。本研究分析发现,不同作物对土壤中营养元素的吸收和利用存在偏好性。在棉花、小麦、玉米两年三熟轮作模式下,种植小麦、玉米后,可减少接茬棉花氮、磷肥施用量,本研究合理解释了“倒茬如上粪”这一农谚现象。
其次,棉花是受病虫害为害较大作物,特别是枯、黄萎病和棉铃疫病,呈普遍发生趋势,这些病害主要由腐生和寄生真菌引起。Acosta-Martinez 等[32]发现,采用棉花-小麦-玉米轮作比连作更有利于提高土壤细菌多样性,本研究通过土壤DNA高通量测序技术分析2种种植模式的土壤样品,结果显示,通过轮作能够提高土壤真菌的多样性并减少包含许多腐生和寄生有害真菌的散囊菌目菌群种类和数量。随着微生物基因组数据库的丰富,土壤DNA高通量测序技术能够科学准确地反映土壤样品微生物成分和含量,并且不需要进行土壤微生物的培养和分离。本研究还发现,连作棉田含有大量的伞菌科菌群,是由于棉籽壳和棉花秸秆碎渣可以作为伞菌科众多食用菌良好的培养料。当前大部分棉田实行棉花秸秆粉碎还田,多年连作为伞菌科菌群的发展提供了较好的营养条件。粪壳菌属、柄孢壳菌属和粪盘菌属菌群大多腐生于富含有机质的土壤和植物残体上。本研究中这类菌群数量的增加,说明连作棉田轮作小麦、玉米等禾本科作物后,能够增加土壤有机质,为粪壳菌属、柄孢壳菌属和粪盘菌属菌群提供了较好的生长发育条件。综合分析可以看出,通过棉粮轮作,土壤中的微生物优势菌群发生明显变化,减少了土壤中有害真菌数量,合理解释了采用轮作后棉田病害较轻的现象。
通过以上一系列试验分析得出如下结论:通过轮作制度能够实现土壤养分的残效利用并改变土壤养分结构,为茬口衔接节肥技术的实施提供了依据。棉粮轮作后明显改变了土壤中微生物优势菌群,有害菌群明显减少。因此,开展合理的棉粮轮作有助于改良冀中南连作棉田土壤营养状况,有利于增产增收、粮棉双丰。
[1] 张亚楠,王兴祥,李孝刚,等.连作对棉花抗枯萎病生理生化特性的影响[J].生态学报, 2016, 6(14): 4456-4464.
[2] 刘建国,卞新民,李彦斌,等.长期连作和秸秆还田对棉田土壤生物活性的影响[J].应用生态学报, 2008, 19(5): 1027-1032.
[3] 李文娇,杨殿林,赵建宁,等.长期连作和轮作对农田土壤生物学特性的影响研究进展[J].中国农学通报, 2015, 31(3): 173-178.
[4] 李春格,李晓鸣,王敬国.大豆连作对土体和根际微生物群落功能的影响[J].生态学报, 2006, 26(4): 1144-1150.
[5] Yao H Y, Jiao X D, Wu F Z. Effects of continuous cucumber cropping and alternative rotations under protected cultivation on soil microbial community diversity[J]. Plant and Soil, 2006, 284(1/2): 195-203.
[6] Vargas Gil S, Meriles J, Conforto C, et al. Response of soil microbial communities to different management practices in surface soils of a soybean agroecosystem in Argentina[J]. European Journal of Soil Biology, 2011, 47(1): 55-60.
[7] Doran J W, Sarrantonio M, Liebig M. Soil health and sustainability[J]. Advances in Agronomy, 1996, 56(8): 1-54.
[8] Trabelsi D, Ben Ammar H, Mengoni A, et al. Appraisal of the crop-rotation effect of rhizobial inoculation on potato cropping systems in relation to soil bacterial communities[J]. Soil Biology and Biochemistry, 2012, 54(6): 1-6.
[9] 曹 莉,秦舒浩,张俊莲,等.轮作豆科牧草对连作马铃薯田土壤微生物菌群及酶活性的影响[J].草业学报, 2013, 22(3): 139-145.
[10] Mulder C, Boit A,Bonkowski M, et al. A belowground perspective on dutch agroecosystems: how soil organisms interact to support ecosystem services[J]. Advances in Ecological Research, 2011, 44:277-357.
[11] 张立成,邵继海,林毅青, 等.稻-稻-油菜轮作对土壤微生物活性和多样性的影响[J].生态环境学报, 2017, 26(2):204-210.
[12] 祁 虹,王树林,王 燕,等.粮棉轮作与土壤深翻对棉花生育性状及产量的影响[J].天津农业科学, 2016, 22(8): 113-116.
[13] 鲍士旦.土壤农化分析[M]. 3版.北京:中国农业出版社, 2000.
[14] Magoc T, Salzberg S L. FLASH: fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957-2963.
[15] Bolger A M, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data[J]. Bioinformatics, 2014, 30(15): 2114-2120.
[16] Edgar R C, Haas B J, Clemente J C, et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16): 2194-2200.
[17] Edgar R C. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 2010, 26(19): 2460-2461.
[18] Bokulich N A, Subramanian S, Faith J J, et al. Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J]. Nature Methods, 2013, 10(1): 57-59.
[19] Wang Q, Garrity G M, Tiedje J M, et al. Naive bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267.
[20] Koljalg U, Nilsson R H, Abarenkov K, et al. Towards a unified paradigm for sequence-based identification of fungi[J]. Molecular Ecology, 2013, 22(21): 5271-5277.
[21] Schloss P D, Westcott S L, Ryabin T, et al. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Applied and Environmental Microbiology, 2009, 75(23): 7537-7541.
[22] Russo S E, Legge R, Weber K A, et al. Bacterial community structure of contrasting soils underlying Bornean rain forests: Inferences from microarray and next-generation sequencing methods[J]. Soil Biology & Biochemistry, 2012, 55: 48-59.
[23] Campbell B J, Polson S W, Hanson T E, et al. The effect of nutrient deposition on bacterial communities in Arctic tundra soil[J]. Environ Microbiol, 2010, 12: 1842-1854.
[24] 范君华,龚明福,刘 明,等.南疆干旱区连作棉田土壤养分及生物活性的初步研究[J].棉花学报, 2009, 21(2): 127-132.
[25] 刘建国,张 伟,李彦斌,等.新疆绿洲棉花长期连作对土壤理化性状与土壤酶活性的影响[J].中国农业科学, 2009, 42(2): 725-733.
[26] 刘 军,唐志敏,刘建国,等.长期连作及秸秆还田对棉田土壤微生物量及种群结构的影响[J].生态环境学报, 2012, 21(8): 1418-1422.
[27] Asuming-Bremponga S, Gantnerb S, Adikua S K, et al. Changes in the biodiversity of microbial populations in tropical soils under different fallow treatments[J]. Soil Biology & Biochemistry, 2008, 40(11): 2811-2818.
[28] Wright S F, Green V S, Cavigelli M A. Glomalin in aggregate size classes from three different farming systems[J]. Soil & Tillage Research, 2007, 94(2): 546-549.
[29] Tian Y Q, Zhang X E, Liu J, et al. Effects of summer cover crop and residue management on cucumber growth in intensive Chinese production systems: soil nutrients, microbial properties and nematodes[J]. Plant and Soil, 2011, 339(1/2): 299-315.
[30] 吴凤芝,王学征.设施黄瓜连作和轮作中土壤微生物群落多样性的变化及其与产量品质的关系[J].中国农业科学, 2007, 40(10): 2274-2280.
[31] 刘 瑜,梁永超,褚贵新,等.长期棉花连作对北疆棉区土壤生物活性与酶学性状的影响[J].生态环境学报, 2010, 19(7): 1586-1592.
[32] Acosta-Martinez V, Dowd S, Sun Y, et al. Tag-encoded pyrosequencing analysis of bacterial diversity in a single soil type as affected by management and land use[J]. Soil Biology & Biochemistry, 2008, 40(11): 2762-2770.
