大豆sbp基因表达方式分析及其启动子调控活性研究
2018-01-12李珊珊杨晓杰刘丽丽
赵 艳,翟 莹,李珊珊,杨晓杰,刘丽丽
(齐齐哈尔大学 生命科学与农林学院,黑龙江 齐齐哈尔 161006)
蔗糖是大多数高等植物光合作用同化碳的主要运输形式,是植物生长发育中碳水化合物的主要来源。在蔗糖的长距离运输中涉及很多的复合体、转运蛋白等,其中有研究表明,蔗糖结合蛋白也参与蔗糖的转运[1]。杨艳等[2]从大豆中分离了2个蔗糖结合蛋白基因GmSBP2 和GmSBPL。生物信息学分析表明,GmSBP2 编码489 个氨基酸残基组成的蛋白质,GmSBPL编码 504 个氨基酸残基组成的蛋白质,且2个基因编码的蛋白均定位于细胞膜上。颜克亮等[3]利用抑制性消减杂交技术和半定量PCR,发现蔗糖结合蛋白基因在种子胚中高表达。此外,对于蔗糖结合蛋白基因(sbp)的研究较少。
为深入了解大豆sbp基因的功能,对大豆sbp基因的组织表达方式及不同逆境下的表达情况进行研究。根据NCBI中大豆sbp基因序列,通过qRT-PCR方法,检测sbp基因在大豆各组织中(根、茎、叶、花、种子)的相对表达量;在盐、干旱、低温、ABA处理条件下sbp基因的表达。克隆sbp基因5′端上游942 bp的序列,该序列的调控活性继续在转基因烟草中进行研究。
1 材料和方法
1.1 试验材料
大豆(吉豆2号)和烟草(NC89)为齐齐哈尔大学分子生物学实验室保存;大肠杆菌DH5α感受态和EHA105农杆菌感受态购自北京索莱宝公司;TRIzol reagent 购自Invitrogen公司;Oligo(dT)18、M-MLV反转录酶、Recombinant RNase Inhibitor、ExTaq、pMD18-T克隆载体、SYBP Premix Ex TaqⅡ荧光定量试剂盒、限制性内切酶PstⅠ和NcoⅠ均购自TaKaRa公司;X-gluc购自Sigma公司;引物合成、测序由北京六合华大基因公司完成。
1.2 大豆材料处理
逆境及激素条件下材料处理:将大豆水培幼苗分别置于含有NaCl (200 mmol/L) 和PEG8000(20%)的Hoaglands营养液中进行盐和干旱胁迫处理;大豆幼苗置于4 ℃培养箱中进行低温处理;对大豆水培幼苗喷施脱落酸(200 μmol/L,ABA)[4]。在处理0,1,2,5,10,24 h分别取样,迅速放于液氮中,-80 ℃保存备用。
各组织的取材:分别取大豆根、茎、叶、花、成熟种子,迅速放于液氮中,-80 ℃保存备用。
1.3 qRT-PCR
根据RNA提取试剂盒步骤,提取大豆各种备用材料的总RNA;cDNA的合成按照逆转录的步骤分别合成第一条链,作为qRT-PCR反应的模板。选用大豆β-tubulin基因(GMU12286)作为内参基因,上、下游引物分别为5′-GGAAGGCTTTCTTGCATTGGTA-3′和5′-AGTGGCATCCTGGTACTGC-3′[5]。根据NCBI中大豆sbp基因的核苷酸序列(L06038.1),利用Primer Premier 5.0 软件设计检测该基因的引物,上游引物(QF:5′-AAGAGGAGGAAAGCCGTGAA-3′)和下游引物(QR:5′-GAATCAAAGTGGCGAGGAGA
C-3′)。利用BIO-RAD CFX96实时定量PCR仪,反应循环参数为:95 ℃ 10 s,58 ℃ 20 s,72 ℃ 30 s。每次取样点设3次技术重复,试验共设3次生物学重复[6]。
1.4 克隆启动子序列及预测分析
选取大豆幼嫩叶片为材料,采用CTAB法,提取大豆基因组DNA[7]。依据大豆基因组序列(http://www.phytozome.net/soybean)和NCBI中大豆sbp基因序列,设计特异引物扩增大豆sbp基因ATG的5′ 端上游序列942 bp,命名为SP。PCR扩增中的上游、下游引物分别带有PstⅠ与NcoⅠ 酶切位点,上游引物为SF:5′-GGGCTGCAGGAATTTAACAGTGCGTCA
TATG-3′,下游引物为SR:5′-GGGCCATGGGGCTTG
GCAGTGAGGAACA-3′。PCR扩增的反应条件为:预变性94 ℃ 5 min;循环94 ℃ 30 s,52 ℃ 40 s,72 ℃ 40 s,共30个循环;72 ℃后延伸8 min,4 ℃保温。用凝胶回收试剂盒切胶回收PCR扩增片段,回收片段通过连接试剂盒连接到pMD18-T克隆载体上,获得重组质粒pMD18-T-SP,双酶切鉴定后测序。利用DNAMAN软件,对克隆测序片段与大豆基因组中相应片段进行同源性比对。启动子预测分析软件BDGP (http//:www.fruitfly.org/)和PlantCARE(http://www.dna.affrc.go.jp/ PLACE/signalscan.htmL)预测该启动子的转录起始位点和顺式作用元件。
1.5 表达载体的构建和转化烟草
用PstⅠ和NcoⅠ 2个酶,分别双酶切pMD18-T-SP质粒和pCAMBIA1301载体,酶切pMD18-T-SP重组质粒,得到小片段,酶切pCAMBIA1301载体得到大片段,将大、小片段进行连接,得到SP与gus基因融合的表达载体,命名为pCAM-SP。将pCAM-SP载体通过冻融法转入到根瘤农杆菌EHA105中。叶盘法转野生型烟草,获得T0转基因烟草植株。对T0转基因烟草植株进行潮霉素(Hyg)抗性筛选、PCR和RT-PCR分子检测[8-9],PCR检测引物为SF(5′-GGGCTGCAGGAATTTAACAGTGCGTCATATG-3′)和UR(5′-CCCACACTTTGCCGTAATGAG-3′),RT-PCR检测引物为YF(5′-TTCTACACAGCCATCGGTCCA-3′)和YR(5′-TGAAAAAGCCTGAACTCACCG-3′)。同样筛选、鉴定出阳性T1转基因烟草。
1.6 GUS 组织化学染色
GUS 组织化学染色参照Jefferson等[10]的方法进行。阳性T1转基因烟草的幼苗进行干旱处理,条件同处理大豆幼苗的方法;各种处理材料和各组织的收集同大豆幼苗的方法。将处理后的烟草叶片或取自不同组织的烟草叶片,放入GUS染色液中(50 mmol/L 磷酸缓冲液 (pH值7.0),0.5 mmol/L K3(Fe(CN)6),0.5 mmol/L K4(Fe(CN)6),10 mmol/L EDTA,0.1%Triton X-100 和2 mmol/L X-gluc),37 ℃染色12 h,之后材料先后用50%,75%,100%乙醇漂洗,至完全脱色。肉眼观察并拍照。
2 结果与分析
2.1 大豆sbp基因的诱导表达分析
利用qRT-PCR方法,在干旱(PEG)、盐(NaCl)、低温(4 ℃)和激素(ABA)不同条件、不同时间处理下,对大豆sbp基因的相对表达量进行分析。结果显示:大豆sbp基因在干旱胁迫下,随着处理时间的延长,表达量逐渐升高,当处理24 h,相对于没有任何处理的对照,表达量为8.84,说明大豆sbp基因受干旱诱导,且诱导表达量随处理时间增强(图1)。
大豆sbp基因在盐胁迫处理下,相对于没有任何处理的对照,当处理为1.00 h,相对表达量下降80倍左右,以后的2,5,10,24 h处理时间下,相对表达量与处理1 h的表达量基本相同(图2)。大豆sbp基因在低温、ABA条件处理下,表现出与盐胁迫处理下相似的规律(图3,4)。说明大豆sbp基因受盐、低温、ABA诱导,诱导表达下降,下降的表达量不随处理时间而变化。
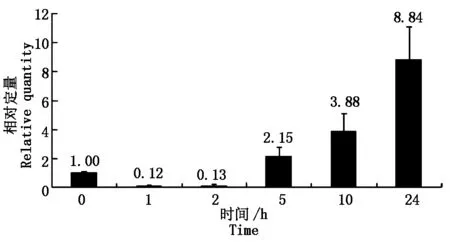
图1 干旱胁迫下大豆植株中sbp基因的表达Fig.1 Relative quantity of soybean sbp gene under drought treatment

图2 盐胁迫下大豆植株中sbp基因的表达Fig.2 Relative quantity of soybean sbp gene under salt treatment
2.2 大豆sbp基因的组织表达分析
利用qRT-PCR方法,大豆β-tubulin基因为内参,在大豆根、茎、叶、花、种子中,检测sbp基因的相对表达量。结果显示(图5):当茎中的相对表达量为1.00时,大豆sbp基因在根、叶、花和种子中表达活性分别为0.13,15.17,4.88,680.84,种子中相对表达是根、茎、叶、花中表达量的45~5 237倍,可以看出,sbp基因在大豆根、茎、叶、花中的相对表达量低,而在大豆种子中的相对表达量较高。

图3 低温胁迫下大豆植株中sbp基因的表达Fig.3 Relative quantity of soybean sbp gene under cold treatment
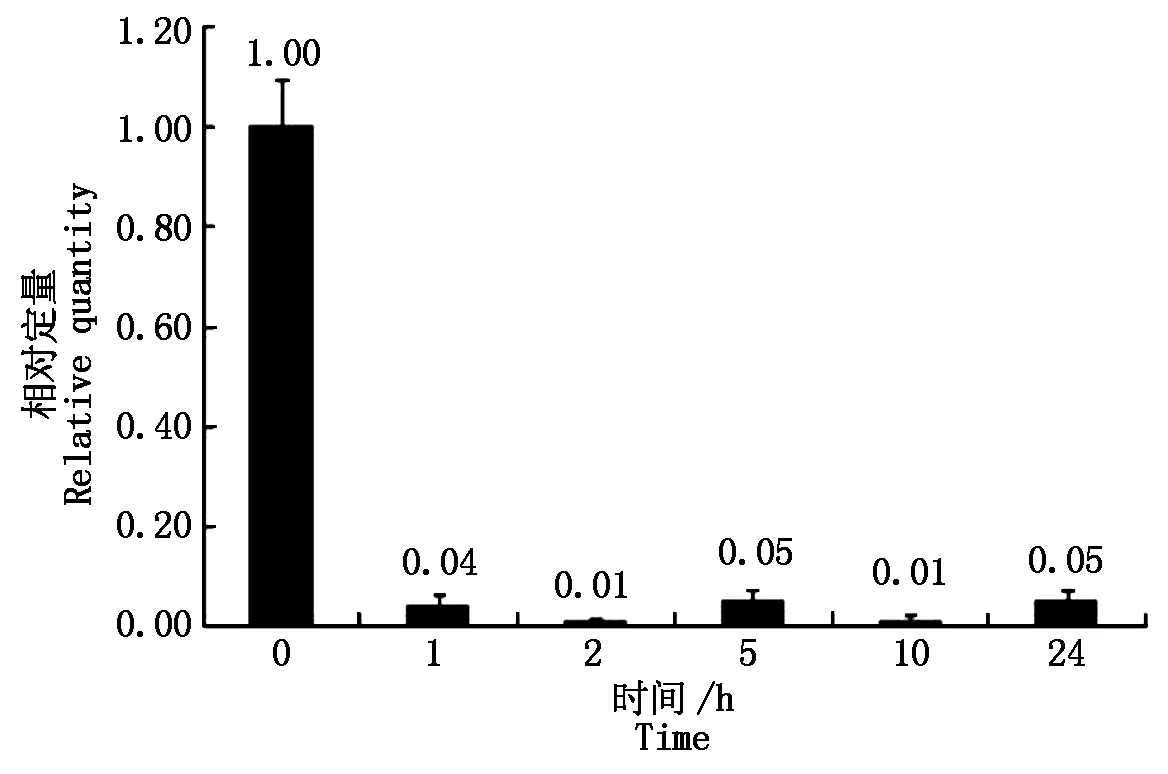
图4 ABA诱导大豆植株中sbp基因的表达Fig.4 Relative quantity of soybean sbp gene under ABA treatment
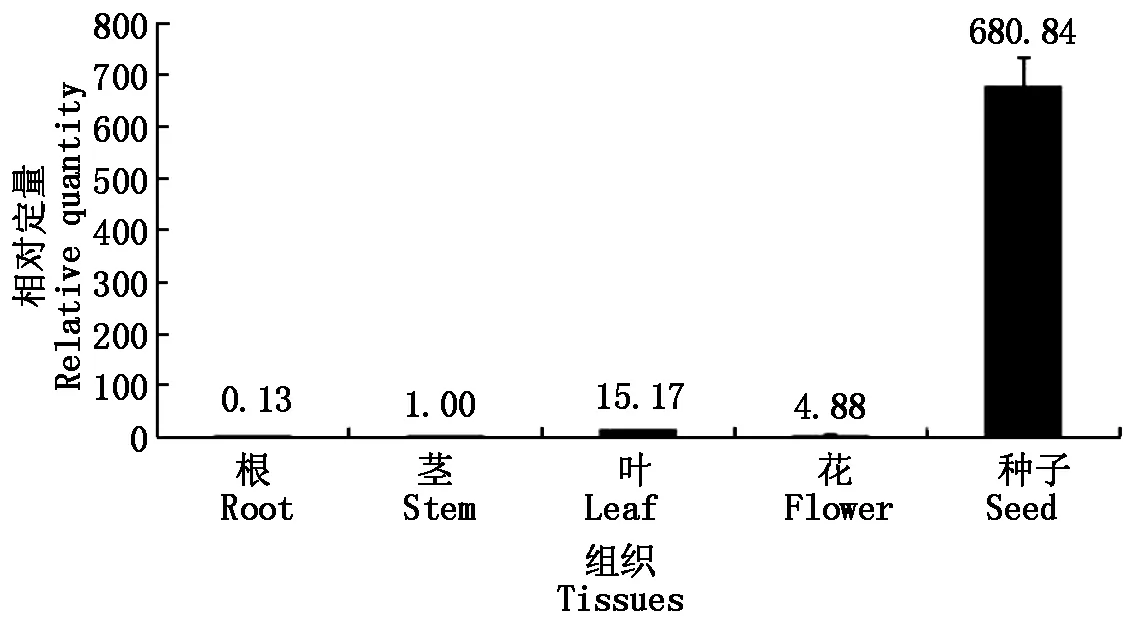
图5 qRT-PCR检测sbp基因在大豆各组织中的表达Fig.5 The expression of soybean sbp gene in tissues of soybean by qRT-PCR
2.3 启动子SP序列的克隆
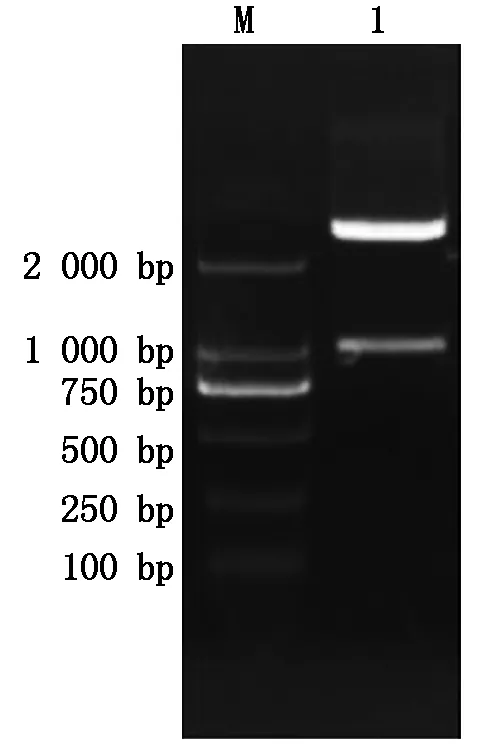
根据NCBI中大豆sbp基因的序列,及大豆基因组序列,扩增sbp基因ATG上游序列,获得长度为942 bp的片段,命名为SP(图6)。将SP片段连接到pMD18-T载体上,获得重组质粒pMD18-T-SP。从大肠杆菌菌液中提取质粒,用PstⅠ和NcoⅠ双酶切鉴定,结果得到与预期相符的片段(图7)。利用DNAMAN软件,对SP序列的测序结果与大豆基因组中相对应的该段序列进行同源性比对,同源性达到100%,说明SP序列在不同大豆品种间无差别。

M.2 000 bp DNA Marker;1. PCR扩增产物SP。M.2 000 bp DNA Marker;1. PCR amplification of SP.
M.2 000 bp DNA Marker;1. 双酶切鉴定。 M.2 000 bp DNA Marker;1. Enzyme digestion indentification.
2.4 大豆SP启动子的预测分析
启动子在线预测软件分析结果表明,根据真核生物启动子序列的特点及启动子预测软件分析,转录起始位点(TSS)为892 bp处的A。此外,在SP启动子中,含有多种常出现在种子特异性启动子中的元件,如对种子特异性表达基因的高水平表达具有重要作用的元件(RY-repeat)[11],胚表达的顺式作用元件(SEF3 motif和SEF4 motif)[12],常出现在参与三酰基甘油合成和植物种子特异表达基因的启动子中的元件(E-box)[13],种子特异表达的顺式作用元件(Skn-1 motif、AATAAA、AACA和ACGT)[14-17]及与激素、逆境诱导相关元件,如ABRE(脱落素响应元件)[18]、W-box(WRKY转录因子的结合位点)[19]、DOF转录因子的结合位点[20](图8)。
2.5 构建表达载体及转基因烟草检测
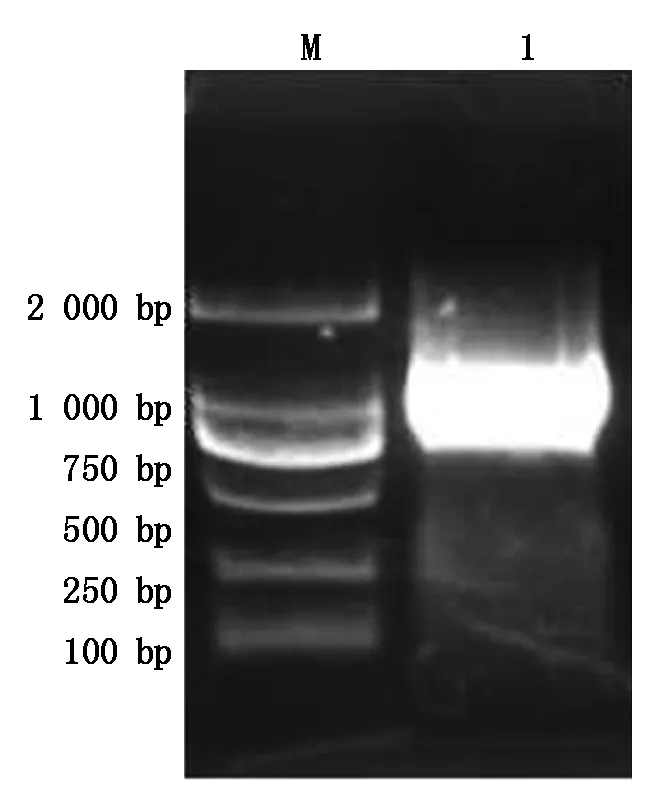
双酶切重组质粒pMD18-T-SP得到的小片段,与双酶切pCAMBIA1301质粒载体得到的大片段进行连接,经转化大肠杆菌和双酶切鉴定,获得植物表达载体pCAM-SP(图9)。通过冻融法,将重组质粒pCAM-SP转化至农杆菌EHA105中(图10)。通过叶盘法,侵染无菌苗烟草叶片,在含有5 mg/L潮霉素的MS培养基上进行培养,待生根后转移土壤中继续生长。利用PCR法和RT-PCR法进行检测,获得阳性T1转烟草植株7株。

图8 SP启动子序列中预测的顺式作用元件Fig.8 Cis-elements of SP promoter

M.2 000 bp DNA Marker;1. pCAM-SP载体的双酶切。M.2 000 bp DNA Marker;1. Enzyme digestion of pCAM-SP vector.
2.6 组织化学染色检测
大豆sbp基因在大豆植株中受干旱诱导,诱导表达量随处理时间增强(图11)。继续对转化pCAM-SP的烟草植株进行干旱处理,如图11所示,不同处理时间下,烟草叶片被染成蓝色的程度不同,随着处理时间增加染色程度增强,当处理24 h时,叶片的染色程度最深,与sbp基因的表达趋势相似。说明SP启动子具有干旱诱导活性,且随诱导时间的增长活性增加。
M.2 000 bp DNA Marker;1. 转化pCAM-SP质粒的农杆菌的PCR检测产物。M.2 000 bp DNA Marker;1. PCR products of transformed Agrobacterium with pCAM-SP vector.
相对于大豆根、茎、叶、花各组织,大豆sbp基因在种子中的相对表达量很高(图5)。对转化pCAM-SP的烟草植株的根、茎、叶、花和种子进行GUS组织化学染色,结果如图12所示,烟草的根、茎、叶基本没有被染色,而种子染色相对较深,说明SP启动子具有种子表达特异性。

图11 干旱胁迫不同时间下转基因烟草叶片的GUS组织化学染色Fig.11 GUS histochemical assays in the transgenic tobacco leaves under drought with different treatment time
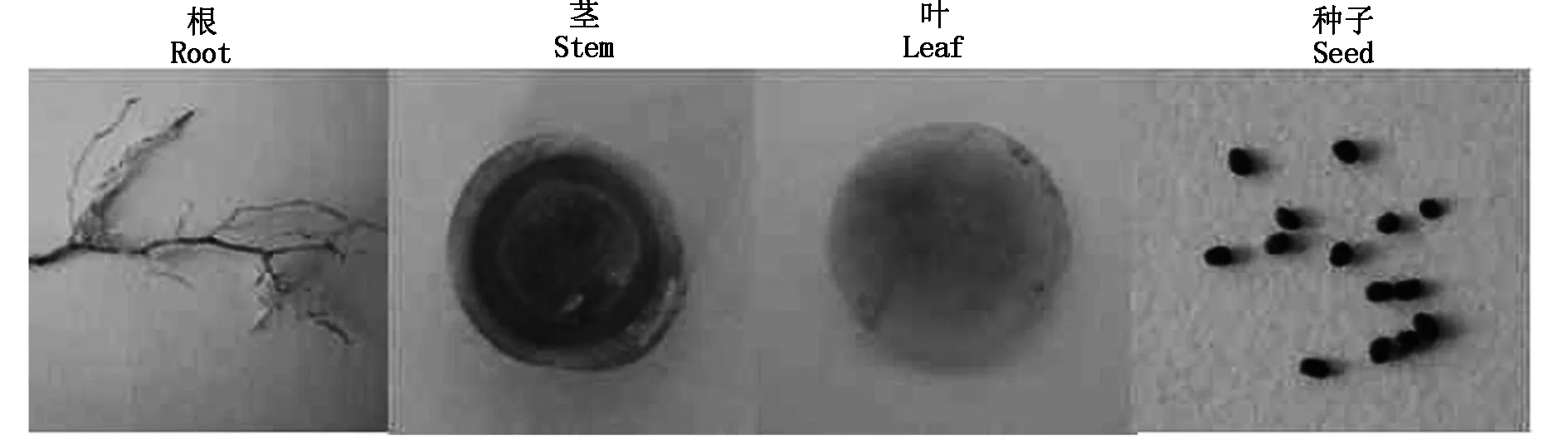
图12 转基因烟草各组织GUS组织化学染色Fig.12 GUS histochemical assays with the transgenic tobacco different tissues
3 结论与讨论
本研究在大豆植株中对大豆sbp基因在不同逆境胁迫和激素处理下的表达情况进行分析。大豆sbp基因在干旱处理下,表达呈上调趋势,表达量在24 h达到最大值,是对照表达量的8.84倍,说明sbp基因受干旱上调诱导明显。在盐、低温和ABA处理条件下,sbp基因的表达量均下降,说明sbp基因受盐、低温和ABA的下调诱导。以上现象可能与大豆sbp基因启动子中存在逆境、激素诱导相关顺式元件,及其位置有关。
本研究在大豆植株中对大豆sbp基因在各组织中的差异表达情况进行分析。sbp基因在大豆种子中相对表达活性很高,是根、茎、叶、花中表达量的45~5 237倍,可能与该时期蔗糖的积累、转运密切相关。从大豆sbp基因在各组织中的表达情况看,该基因为种子特异表达基因。继续克隆sbp基因5′端上游的启动子序列,基因的5′端上游序列中常含有许多的顺式调控元件,往往可能位于距离ATG的5′端上游更远端,所以很多研究中在不明确有效启动子序列时,通常克隆ATG上游2 000 bp的序列,尽可能的包含了较多的调控元件(增强子等)。但是本研究中通过大豆基因组序列发现,sbp基因ATG上游1 000 bp以上为另一基因序列,因此,本研究中克隆了sbp基因ATG上游942 bp作为推测的启动子序列。
本研究继续在转基因烟草中,对SP启动子进行了干旱胁迫诱导和组织表达分析。通过GUS组织化学染色试验,可以看出SP启动子在烟草中具有干旱诱导表达活性,且随着处理时间的增长活性增强;此外,SP启动子在烟草中具有调控下游gus基因在种子中高表达的特性,说明SP启动子兼具胁迫诱导型启动子和种子特异性启动子的特性,在转基因大豆中,SP启动子是否仍具有这两方面的调控活性,仍需试验继续验证。
[1] Kühn C,Quick W P,Schulz A,et al. Companion cell-specific inhibition of the potato sucrose transporter SUT1[J]. Plant Cell and Environment,1996,19(10):1115-1123.
[2] 杨 艳,王 爽,黄丽燕,等.两个大豆GmSBP基因的特征、亚细胞定位及对非生物胁迫的响应[J].生物工程学报,2014,30(11):1709-1719.
[3] 颜克亮,陈 波,姚家玲,等.大豆种子形态建成期与成熟期正反抑制消减文库构建及差异表达基因分析[J].武汉大学学报:理学版,2008,54(2):202-208.
[4] Zhai Ying,Wang Ying,Li Yanjie,et al. Isolation and molecular characterization of GmERF7,a soybean ethylene-response factor that increases salt stress tolerance in tobacco[J]. Gene,2013,513(1):174-183.
[5] Zhao Y,Sha W,Wang Q Y,et al. Molecular cloning and activity analysis of a seed-specificFAD2-1Bgene promoter fromGlycinemax[J]. Cellular and Molecular Biology,2015,61(4):85-89.
[6] Chen Yanhui,Han Yangyang,Zhang Meng,et al. Overexpression of the wheat expansin geneTaEXPA2 improved seed production and drought tolerance in transgenic tobacco plants[J]. PLoS One,2016,11(4):e0153494.
[7] Zhai Ying,Saho Shuli,Sha Wei,et al. Overexpression of soybean GmERF9 enhances the tolerance to drought and cold in the transgenic tobacco[J]. Plant Cell,Tissue and Organ Culture,2017,128(3):607-618.
[8] Li Xiaoshuang,Zhang Daoyuan,Li Haiyan,et al. EsDREB2B,a novel truncated DREB2-type transcription factor in the desert legumeEremospartonsongoricum,enhances tolerance to multiple abiotic stresses in yeast and transgenic tobacco[J]. BMC Plant Biology,2014,14:44.
[9] Wei Zhigang,Qu Zanshuang,Zhang Lijie,et al. Overexpression of poplar xylem sucrose synthase in tobacco leads to a thickened cell wall and increased height[J]. PLoS One,2015,10(3):e0120669.
[10] Jefferson R A,Kavanagh T A,Bevan M W. GUS fusions-beta-glucuronidase as a sensitive and versatile gene fusion marker in higher-plants[J]. EMBO Journal,1987,6(13):3901-3907.
[11] Moreno-risueno M A,Gonzalez,Diazi,et al. FUSCA3 from barley unveils a common transcriptional regulation of seed-specific genes between cereals andArabidopsis[J]. The Plant Journal:for Cell and Molecular Biology,2008,53(6):882-894.
[12] Chung K J,Hwang S K,Hahn B S,et al. Authentic seed-specific activity of thePerillaoleosin 19 gene promoter in transgenicArabidopsis[J]. Plant Cell Reports,2008,27(1):29-37.
[13] Kim M J,Kim J K,Shin J S,et al. The SebHLH transcription factor mediates trans-activation of theSeFAD2 gene promoter through binding to E-and G-box elements[J]. Plant Molecular Biology,2007,64(4):453-466.
[14] Fauteux F,Strömvik M V. Seed storage protein gene promoters contain conserved DNA motifs in Brassicaceae,Fabaceae and Poaceae[J]. BMC Plant Biology,2009,9:126.
[15] Xie Chengjian,Zhang Beibei,Wang De,et al. Molecular cloning and characterization of an achene-seed-specific promoter from motherwort (LeonurusjaponicusHoutt)[J]. Biotechnology Letters,2011,33(1):167-172.
[16] Hu Xinwen,Liu Sixin,Guo Jianchun,et al. Embryo and anther regulation of the mabinlin Ⅱ sweet protein gene inCapparismasaikaiLévl[J]. Functional & Integrative Genomics,2009,9(3):351-361.
[17] Gao Mingjun,Lydiate D J,Li Xiang,et al. Repression of seed maturation genes by a trihelix transcriptional repressor inArabidopsisseedlings[J]. The Plant Cell,2009,21(1):54-71.
[18] Zhao Yan,Saho Shuli,Li Xiaowei,et al. Isolation and activity analysis of a seed-abundant soyAP1 gene promoter from soybean[J]. Plant Molecular Biology Reporter,2012,30(6):1400-1407.
[19] Zhou Qiyun,Tian Aiguo,Zou Hongfeng,et al. Soybean WRKY-type transcription factor genes,GmWRKY13,GmWRKY21,andGmWRKY54,confer differential tolerance to abiotic stresses in transgenicArabidopsis-plants[J]. Plant Biotechnology Journal,2008,6(5):486-503.
[20] Nalbandi K,Kohnehrouz B B,Saeed K A,et al. Isolating barley(HordeumvulgareL.)B1 hordein gene promoter and using sequencing analaysis for the identification of conservedregulatory elements by bioinformatic tools[J]. African Journal of Biotechnology,2012,11(29):7378-7387.
