调控人脑胶质瘤中ezrin表达的miRNAs筛选及鉴定
2017-12-27,,,,,
,,,, ,
(皖南医学院第一附属医院 弋矶山医院 1.循证医学教研室;2.神经外科;3.肿瘤科,安徽 芜湖 241001)
·临床医学·
调控人脑胶质瘤中ezrin表达的miRNAs筛选及鉴定
徐小琳1,江海波2,盛莉莉3,葛瑞祥2,沈军2,毛捷2
(皖南医学院第一附属医院 弋矶山医院 1.循证医学教研室;2.神经外科;3.肿瘤科,安徽 芜湖 241001)
目的:探讨调控人脑胶质瘤中ezrin的miRNAs筛选及鉴定。方法针对ezrin的测序序列,在线使用TargetScan、PITA、MicroRNA.org、miRGator v2.0、miRDB等5种生物信息学软件预测分析可能调控ezrin的miRNA分子,运用荧光素酶报告载体实验检测筛选候选miRNA分子,RT-PCR检测11例胶质瘤组织和10例正常组织中候选miRNA分子mRNA的表达。结果5种生物信息学软件预测出819个可能调控ezrin的miRNAs,根据评分结果同时结合目前国内外研究成果筛选出miR-148a、-204、-205、-34b、-370、-376b、-377、-410、-495、-548a-3p、-630和-96等12个miRNAs。双荧光素酶质粒与ezrin 3′-UTR作用位点突变体实验,证实hsa-miR-204与相应作用位点突变体EZR 204 Mutant无明显作用(P>0.05),RT-PCR结果显示miR-204在脑胶质瘤组织中的表达低于正常脑组织,差异有统计学意义(P<0.01)。结论胶质瘤组织中miR-204可通过靶向调控ezrin表达发挥生物学作用。
miR-204;ezrin;胶质瘤;荧光素酶报告载体;生物信息学
胶质瘤是颅内最常见的恶性肿瘤,占颅内肿瘤的30%~40%,最新的统计结果表明,临床上不论采用何种治疗手段,胶质母细胞瘤患者2年和5年生存率分别为27.2%和9.8%,平均生存期为14.6月[1]。目前,寻找有效的治疗靶点是能否根治胶质瘤的关键。前期研究发现,ezrin在肿瘤中的表达水平与人脑胶质瘤的恶性程度、复发转移及预后密切相关[2]。然而,ezrin在胶质瘤中过表达的机制仍不清楚。miRNA是内源性非编码小分子单链RNA,通过不完全互补的碱基配对方式与靶基因3′-UTR结合,能在转录后水平调控基因表达。本研究拟采用生物信息学预测可能调控ezrin表达的miRNA分子,通过荧光素酶报告载体实验以及临床样本验证,锁定可能调控的miRNA分子,为下一步的研究提供实验基础。
1 材料与方法
1.1 细胞、主要试剂和仪器 PCR仪(TAKARA公司);琼脂糖凝胶电泳仪(Bio-Rad公司);分子凝胶成像系统(Kodak 4000MM P20);凝胶扫描分析系统(英国UVItec公司);人胚肾细胞HEK293T细胞株;miRNA的过表达质粒pLMP(由pLL3.7质粒改造成或miRNA mimics购买于上海吉码公司);双荧光素酶报告基因质粒pLUC、大肠杆菌TOP10菌株、含miRNA表达或靶基因3′-UTR双荧光素酶报告基因的质粒菌株(深圳华安平康生物公司);小量质粒提取试剂盒(OMEGA公司);转染试剂FuGENE®HD(Roche公司);质粒miR-204、376b、630 mimics、miR-204、376b、630 inhibitor(深圳华安平康生物公司);β-actin鼠抗人(武汉三鹰);Lipofectamine 2000转染试剂(美国 Invitrogen公司);DNA(上海Sangon Biotech公司)。人脑胶质瘤组织和因外伤切除的非肿瘤组织均来自我院中心实验室储存的组织。
1.2 实验方法
1.2.1 可调控ezrin的miRNAs预测 我们在线运用生物信息学软件TargetScan、PI TA、microrna.org、miRGator v2.0、miRDB等5种软件进行预测,针对ezrin的测序序列(图1),预测筛选出可能调控ezrin的候选miRNAs。

图1 ezrin测序序列
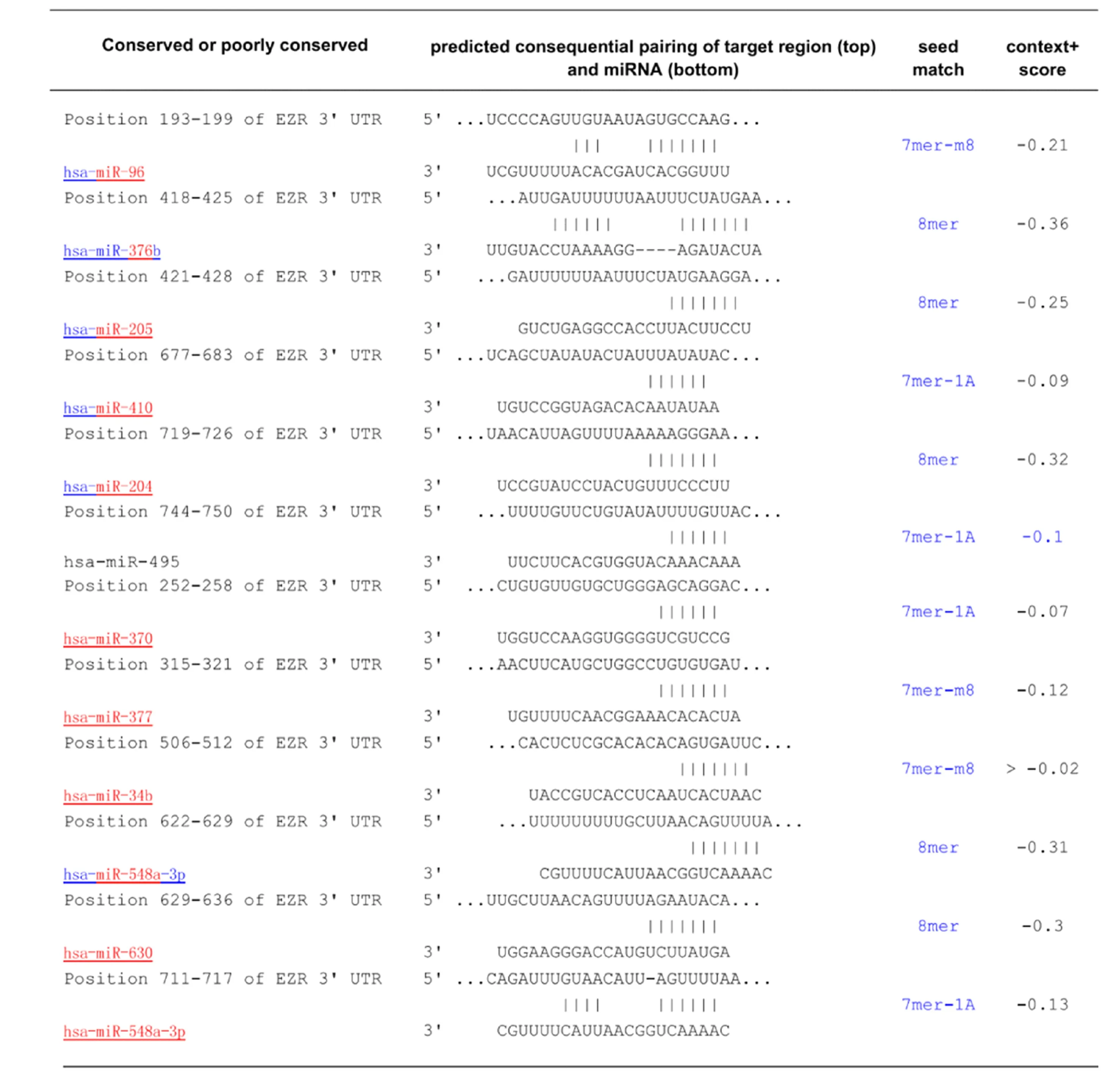
1.2.2 分析候选miRNAs与靶基因ezrin3′-UTR配对结合位点 针对ezrin的测序序列和候选的miRNAs的碱基序列,比照分析候选miRNAs与靶基因ezrin 3′-UTR配对结合位点(表1)。
表1 miRNAs与靶基因ezrin 3′-UTR配对结合位点
1.2.3 双荧光素酶活性检测 将经筛选确定的可能调控ezrin的候选miRNAs,构建ezrin双荧光素酶及miRNA前体表达质粒,共转293T细胞,检测荧光素酶活性(按说明书操作)。
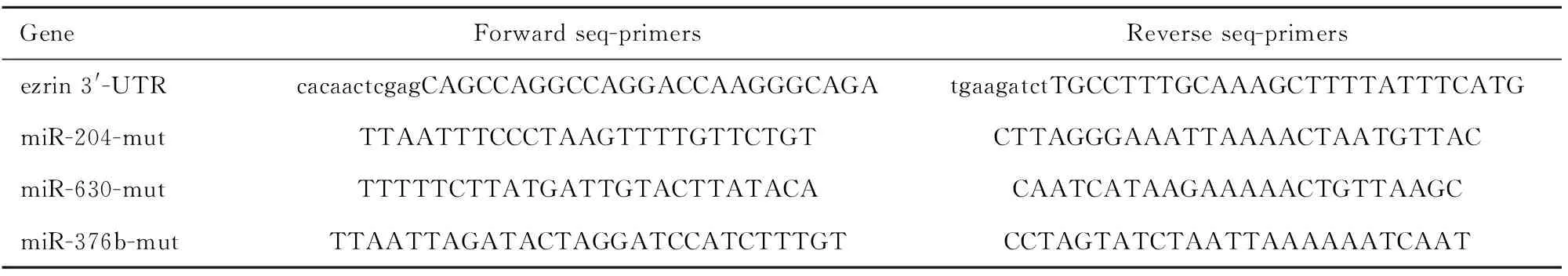
1.2.4 miR-204、-630、-376b及靶基因ezrin的3′-UTR双荧光素酶报告基因质粒构建 利用PCR扩增目的基因序列,并对扩增产物进行琼脂糖凝胶电泳分离及胶回收片段(按说明书操作)。设计miR-204、-376b、-630及靶基因ezrin 3′-UTR引物(表2)。
表2 miR-204,-376b,-630及靶基因ezrin 3′-UTR引物
GeneForwardseq-primersReverseseq-primersezrin3'-UTRcacaactcgagCAGCCAGGCCAGGACCAAGGGCAGAtgaagatctTGCCTTTGCAAAGCTTTTATTTCATGmiR-204-mutTTAATTTCCCTAAGTTTTGTTCTGTCTTAGGGAAATTAAAACTAATGTTACmiR-630-mutTTTTTCTTATGATTGTACTTATACACAATCATAAGAAAAACTGTTAAGCmiR-376b-mutTTAATTAGATACTAGGATCCATCTTTGTCCTAGTATCTAATTAAAAAATCAAT
1.2.5 Real-time PCR检测人脑胶质瘤和正常组织中候选miRNAs的表达 RNA的提取:按照RNA提取步骤进行,用NanoDrop 2000超微量分光光度计测定RNA的纯度和浓度,OD260/OD280比值控制在1.8~2.2;反转录合成 cDNA反应:使用 One Step PrimeScript miRNA cDNA Synthesis Kit 进行反转录反应(按说明书操作);检测miRNAs相对表达量,以RNU-6为内参照,采用2-△△Ct法计算相对表达量。
1.3 统计学分析 采用SPSS 18.0统计软件对数据进行分析,两组间差异比较采用t检验,P<0.05为差异有统计学意义。
2 结果
2.1 可调控ezrin的miRNAs预测结果 在线运用5种生物信息学软件预测出819个可能调控ezrin的miRNAs,其中3种以上软件预测到可调控ezrin的miRNAs有34个(表3)。根据评分结果同时结合目前国内外的研究成果进行筛选,确定miR-148a、-204、-205、-346、-370、-376b、-377、-410、-495、-548a-3p、-630和 -96等12个miRNAs进行候选验证。
2.2 双荧光素酶活性检测结果 将确定的hsa-miR-148a、-204、-205、-346、-370、-376b、-377、-410、-495、-548a-3p、-630和 -96等12个miRNAs,构建ezrin双荧光素酶及miRNA前体表达质粒,共转293T细胞,检测双荧光素酶活性。其中hsa-miR-204(0.082±0.006)、-376b(0.063±0.006)和-630(0.096±0.012)与对照组(0.133±0.002)相比活性下降,差异有统计学意义(t=13.859,P=0.0002;t=19.624,P=0.0001;t=5.122,P=0.007)(图2)。
表3 可调控ezrin的miRNAs预测结果

miRNA预测软件预测软件数量hsa-miR-495TPmi.orgmiRGmiR5hsa-miR-376bTPmi.orgmiRGmiR5hsa-miR-96TPmiRGmi.org4hsa-miR-643TPmiRGmiR4hsa-miR-630TPmiRGmiR4hsa-miR-548mTPmiRGmiR4hsa-miR-410TPmi.orgmiRG4hsa-miR-205TPmi.orgmiRG4hsa-miR-204TPmi.orgmiRG4hsa-miR-183TPmi.orgmiRG4hsa-miR-1323TPmi.orgmiR4hsa-miR-940TPmiRG3hsa-miR-663TPmiRG3hsa-miR-648TPmiR3hsa-miR-623TPmiRG3hsa-miR-595TPmiRG3hsa-miR-566TPmiRG3hsa-miR-548pTPmiR3hsa-miR-548c-3pTPmiR3hsa-miR-548a-3pTPmiR3hsa-miR-377TPmi.org3hsa-miR-370TPmiRG3hsa-miR-346TPmiRG3hsa-miR-338-3pTPmi.org3hsa-miR-300TPmiRG3hsa-miR-211Pmi.orgmiRG3hsa-miR-18bTPmi.org3hsa-miR-18aTPmi.org3hsa-miR-153TPmi.org3hsa-miR-148bTPmi.org3hsa-miR-148aTPmi.org3hsa-miR-1271Pmi.orgmiRG3
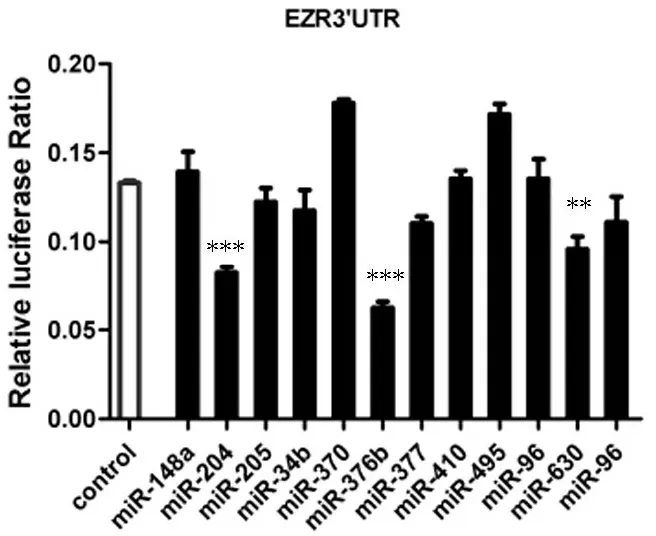
***P<0.001,**P<0.01。
图2 双荧光素酶活性检测结果
2.3 hsa-miR-204、-376b和-630与ezrin 3′-UTR突变体克隆情况及作用位点结果 双荧光素酶活性法与预测ezrin 3′-UTR相应作用位点突变体检测,均无明显作用,说明miR-204、-376b和-630很可能通过预测的作用位点调控ezrin基因(图3)。hsa-miR-204,-630和-376b的表达载体与含ezrin 204、630和376b Mutant基因片段的双荧光素酶报告基因共转染验证试验相对于负对照(不含miRNA的空载体),差异无统计学意义(P=0.836、0.538、0.410)(图4),同时荧光比值分别为负对照的97%、105%和108%。这一结果说明hsa-miR-204和630对ezrin 204 Mutant和630 Mutant的报告基因表达水平无抑制作用,hsa-miR-376b对ezrin 376b Mutant的基因表达水平基本无抑制作用。
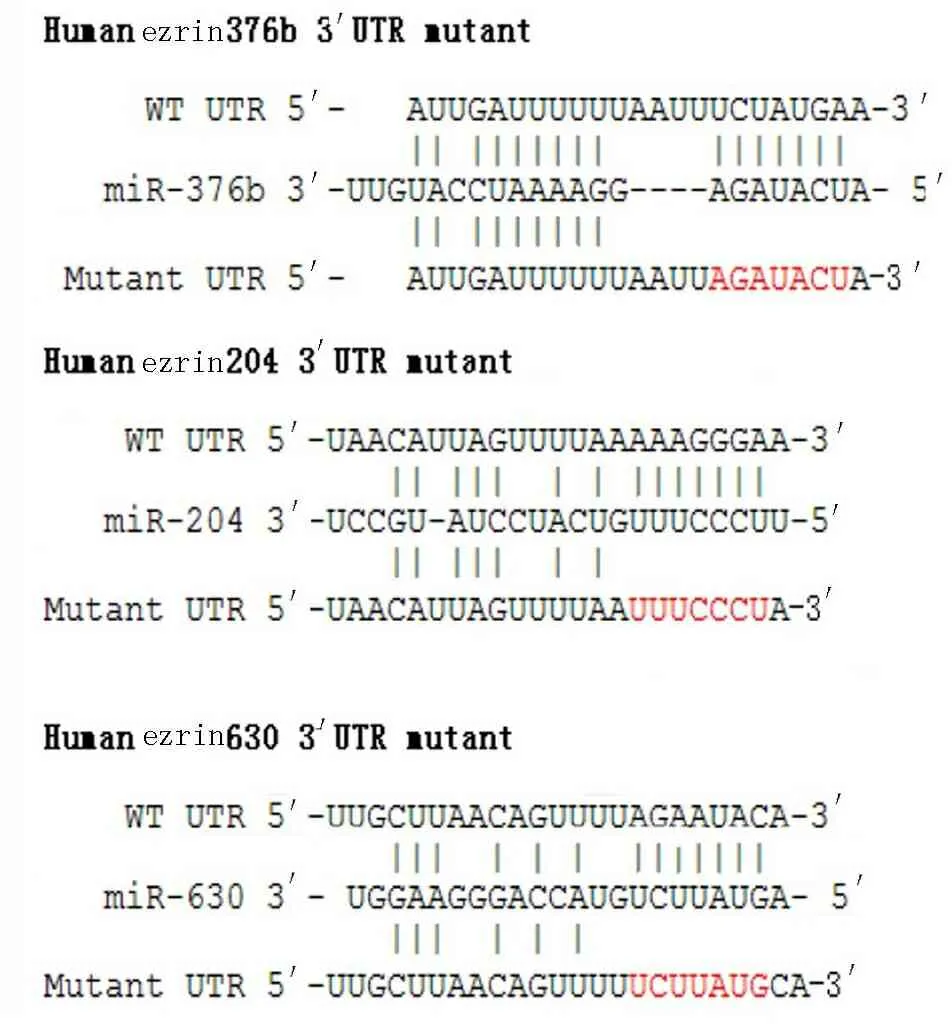
图3 miRNAs与靶基因ezrin 3′-UTR配对结合位点示意图
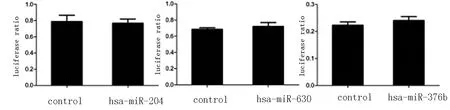
图4 miRNAs双荧光素酶报告载体实验
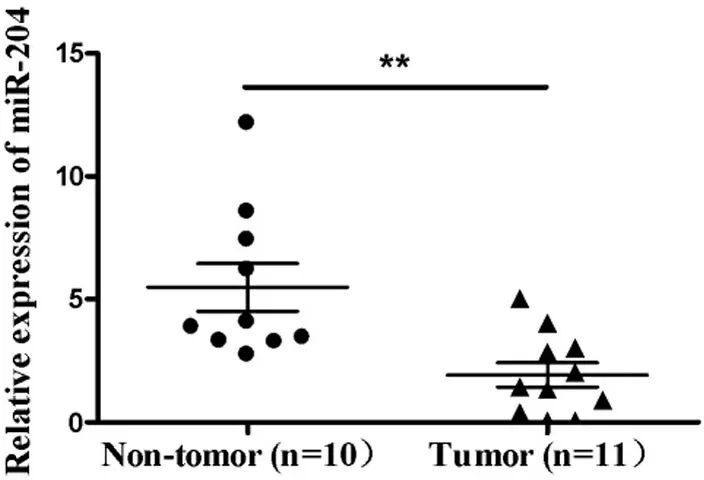
2.4 RT-PCR检测候选miRNAs在组织中表达 对11例胶质瘤组织和10例正常脑组织检测了hsa-miR-204表达,观察到hsa-miR-204在脑胶质瘤组织中的表达低于正常脑组织,差异有统计学意义(1.925±1.645vs. 5.500 ±3.057,t=3.383,P=0.003)(图5),同时发现hsa-miR-376b和-630在正常和胶质瘤组织均能未检测到表达。
**P<0.01。
图5 RT-PCR检测miR-204在组织中表达
3 讨论
miRNA是近年来发现的一种真核生物内源性小分子单链RNA,长约15~25个核苷酸,是一类非编码小RNA。miRNA分子通过不完全互补的方式与靶基因3′非编译区(UTR)结合,通过诱导mRNA的切割降解、翻译抑制或者其他形式的调节机制抑制靶基因的表达,参与生物体的生长、发育、衰老、死亡全部过程的调控。目前为止人类基因组内已发现的miRNA超过1000个,可能调控着基因组内约1/3以上基因表达[3-5]。寻找疾病发生、发展中关键miRNA分子及其靶基因鉴定已成为生命科学领域的研究热点。
我们前期研究发现,ezrin在人脑星形细胞瘤组织中的表达水平与肿瘤的恶性程度、复发转移及预后呈正相关,可以作为抗星形细胞瘤复发转移的治疗靶点[2]。为探索ezrin在胶质瘤中表达失调的机制,我们针对ezrin基因序列,通过生物信息学预测可能调控其表达的miRNA分子,根据评分结果同时结合目前国内外的研究成果进行筛选,预测出hsa-miR-148a、-204、-205、-34b、-370、-376b、-377、-410、-495、-548a-3p、-630和 -96等12个miRNAs可能高度靶向调控ezrin基因的miRNA分子,通过荧光素酶报告载体实验发现其中3个miRNAs即hsa-miR-376b、-204和-630相对于对照组荧光素酶活性下降,差异有统计学意义(P<0.01),提示其对ezrin的表达水平具有抑制作用,进一步采用双荧光素酶质粒与ezrin 3′-UTR作用位点突变体共转293T,证实 hsa-miR-376b、-204和-630可直接结合ezrin 3′-UTR调控ezrin基因表达。我们进一步检测了11例胶质瘤组织和10例正常脑组织hsa-miR-204表达,观察到miR-204在脑胶质瘤组织中的表达低于正常脑组织,差异有统计学意义(P<0.01),同时发现hsa-miR-376b和-630在正常和胶质瘤组织均未能检测到表达。综合以上结果,我们推断miRNA-204可能通过调控ezrin表达在恶性胶质瘤发展过程中发挥重要作用。
目前有文献报道,在肝癌[6-7]、胃癌[8-9]、胰腺癌[10]、肾透明细胞癌[11-12]、乳腺癌[13-14]、子宫内膜癌[15]、脑胶质瘤[16-17]等肿瘤组织中,miRNA-204表达水平均低于同一患者的正常组织中的水平,Lee等[18]在头颈部鳞癌(HNSCC)细胞株中恢复miRNA-204的功能,发现会抑制其相关靶基因的表达,推论出miRNA-204是一种具有肿瘤抑制作用的miRNA。从研究结果来看,miRNA-204在胶质瘤组织中的表达低于非肿瘤组织,然而,作为抑癌基因,其调控ezrin表达的分子机制及其在恶性胶质瘤中的具体作用仍有待下一步研究阐明。
[1] ERIC T WONG,NISE H YAMAGUCHI. Treatment advances for glioblastoma[J]. Expert Review of Neurotherapeutics,2011,11(10):1343-1345.
[2] MAO J,YUAN XR,XU SS,etal. Expression and Functional Significance of Ezrin in Human Brain Astrocytoma[J]. Cell Biochem Biophys,2013,67(3):507-511.
[3] KASINSKI AL,SLACK FJ. Epigenetics and genetics. MicroRNAs en route to the clinic:progress in validating and targeting microRNAs for cancer therapy[J]. Nat Rev Cancer,2011,11(12):849-864.
[4] ESTELLER M. Non-coding RNAs in human disease[J]. Nat Rev Genet,2011,12(12):861-874.
[5] SANDHU S,GARZON R. Potential applications of microRNAs in cancer diagnosis,prognosis,and treatment[J]. Semin Oncol,2011,38(6):781-787.
[6] QIU YH,WEI YP, SHEN NJ,etal. MiR-204 inhibits epithelial to mesenchymal transition by targeting slug in intrahepatic cholangiocarcinoma cells [J]. Cellular Physiology & Biochemistry International Journal of Experimental Cellular Physiology Biochemistry & Pharmacology,2013,32(5):1331-1341.
[7] CUI ZH, SHEN SQ, CHEN ZB,etal. Growth inhibition of hepatocellular carcinoma tumor endothelial cells by miR-204-3p and underlying mechanism[J]. World Journal of Gastroenterology,2014,20(18):5493-5504.
[8] SACCONI A,BIAGIONI F,CANU V,etal. MiR-204 targets Bcl-2 expression and enhances responsiveness of gastric cancer [J]. Cell Death & Disease,2012,3(11):e423.
[9] ZHANG LH,WANG XQ,CHEN PS. MiR-204 down regulates SIRT1 and reverts SIRT1-induced epithelial-mesenchymal transition,anoikis resistance and invasion in gastric cancer cells [J]. Bmc Cancer,2013,13(1):290.
[10] CHEN Z,SANGWAN V,BANERJEE S,etal. MiR-204 mediated loss of Myeloid cell leukemia-1 results in pancreatic cancer cell death [J]. Molecular Cancer,2013,12(1):105.
[11] HALL DP,COST NG,HEGDE S,etal. TRPM3 and miR-204 establish a regulatory circuit that controls oncogenic autophagy in clear cell renal cell carcinoma [J]. Cancer Cell,2014,26(5):738-753.
[12] MIKHAYLOVA O,STRATTON Y,HALL D,etal. VHL-regulated MiR-204 suppresses tumor growth through inhibition of LC3B-mediated autophagy in renal clear cell carcinoma [J]. Cancer Cell,2012,21(4):532-546.
[13] ZENG J, WEI M, SHI R,etal. MiR-204-5p/Six1 feedback loop promotes epithelial-mesenchymal transition in breast cancer [J]. Tumor Biology,2016,37(2):2729-2735.
[14] WEIDONG L,XUEJUN J,QIANBING Z,etal. Decreased expression of miR-204 is associated with poor prognosis in patients with breast cancer [J]. International Journal of Clinical & Experimental Pathology,2014,7(6):3287-3292.
[15] BAO W,WANG HH, TIAN FJ,etal. A T rkB-STAT3-miR-204-5p regulatory circuitry controls proliferation and invasion of endometrial carcinoma cells [J]. Molecular Cancer,2013,12(305):8957-8968.
[16] YING Z, LI Y, WU JH,etal. Loss of miR-204 expression enhances glioma migration and stem cell-like phenotype [J]. Cancer Research,2013,73(2):990-999.
[17] XIA Z,LIU F,ZHANG J,etal. Decreased Expression of MiRNA-204-5p Contributes to Glioma Progression and Promotes Glioma Cell Growth,Migration and Invasion [J]. Plos One,2015,10(7):e0132399.
[18] LEE Y. Network Modeling Identifies Molecular Functions Targeted by miR-204 to Suppress Head and Neck Tumor Metastasis [J]. Plos Computational Biology,2010,6(4):e1000730.
ScreeningandidentificationofmicroRNAstargetingezrininhumanglioma
XUXiaolin,JIANGHaibo,SHENGLili,GERuixiang,SHENJun,MAOJie
Department of Evidence-based Medicine,The First Affiliated Hospital of Wannan Medical College,Wuhu 241001,China
Objective:To screen and identify microRNAs targeting ezrin in human glioma.Methods:On-line software,including TargetScan,PITA,microrna.org,miRGator v2.0 and miRDB,were used to predict potential microRNAs capable of regulating ezrin expression. Dual Luciferase Reporter Assay System was used to determine the luciferase activity of the candidate microRNA genes,and real-time PCR,to detect the mRNA values of the candidate microRNAs in the samples obtained from 11 cases of glioma and brain tissues from 10 healthy subjects.Results:The candidate microRNAs,including miR-148a,-204,-205,-34b,-370,-376b,-377,-410,-495,-548a-3p,-630 and -96 were selected from 819 potential microRNAs that were predicted to regulate the expression of ezrin. DNA constructs expressing the miRNA precursors were co-transfected with pLUC-ezrin 3′UTR,and the results indicated that only transfection with miR-204 expression vector significantly down-regulated the luciferase activity compared to transfection with an empty vector (P>0.05),whereas real-time PCR demonstrated that miR-204 expression was significantly lower than that in samples from non-gliomas samples(P<0.01).Conclusion:MiR-204 in human glioma can direct regulation of ezrin gene,yet correlation and mechanisms of the two proteins need further investigation.
microRNA-204;ezrin;glioma;luciferase reporter vector;bioinformatics
1002-0217(2017)05-0428-05
安徽省自然科学基金项目(1408085MH205;1708085MH231);安徽省教育厅自然科学研究项目(KJ2015A141)
2017-02-15
徐小琳(1973-),女,助理研究员,(电话)15056458756,(电子信箱)xxiaolin66@163.com;盛莉莉,女,副主任医师,(电子信箱)13605535185@163.com,通信作者;毛 捷,男,副主任医师,副教授,(电子信箱)myw921@163.com,通信作者。
R 739.41
A
10.3969/j.issn.1002-0217.2017.05.006
