一株牛源副干酪乳杆菌的分离与鉴定
2017-12-27付强程立坤付石军苗立中王艳沈志强
付强,程立坤,付石军,苗立中,王艳,沈志强
(山东省滨州畜牧兽医研究院,滨州 256600)
一株牛源副干酪乳杆菌的分离与鉴定
付强,程立坤,付石军,苗立中,王艳,沈志强
(山东省滨州畜牧兽医研究院,滨州 256600)
为了分离有研究价值的益生菌,从牛肠道中分离到1株疑似乳酸菌,通过对参考菌株的16S rRNA基因序列的归纳分析,证实存在种间特异性可变区,最终确定为副干酪乳杆菌,分离菌作为一种益生菌在饲料添加剂方面存在重要研究价值。
副干酪乳杆菌;分离鉴定;16S rDNA;序列分析
乳酸菌(Lactobacillus)是一类能发酵碳水化合物产生乳酸的革兰氏阳性菌的通称,乳酸细菌现在主要包括2 3个属,其中比较重要的有乳杆菌属(Lactobacillus)、双歧杆菌属(Bifidobacterium)、链球菌属(Streptococcus)、肠球菌属(Enterococcus)、乳球菌属(Lactococcus)等[1]。作为重要的益生菌,乳酸菌已被广泛地应用于食品、饮料和微生态制剂等行业中,是安全的食品级微生物[2]。因此乳酸菌的分离与鉴定对于后续的研究与应用具有重要的作用。
目前乳酸菌的鉴定方法主要分为表型特征鉴定法和基因型鉴定法。其中16S rDNA鉴定是一种快速获得细菌种属信息的方法,但16S rDNA进化分析不适合物种内或亲缘关系很近的物种[3]。在乳酸菌分类研究领域,干酪乳杆菌群包括L.casei(干酪乳杆菌),L.paracasei(副干酪乳杆菌)和L.rhamnosus(鼠李糖乳杆菌),种群内菌株生理生化特征相似。而16S rRNA基因序列同源性高,其命名和分类地位一直饱受争议[4],目前在副干酪乳杆菌鉴定中应用最广泛的分子生物学技术是16S rDNA序列同源性分析[5],也有研究者采用持家基因hsp60[6]、pheS[7]、recA[8]基因序列分析来揭示群内相近菌株的差异。本试验采用16S rDNA同源性分析以及可变区序列对比,对实验室分离的1株乳酸菌进行鉴定,最终确定为副干酪乳杆菌,证明该方法具有一定的可行性。
1 材料与方法
1.1 试验材料
菌种为从牛肠道中分离纯化的一株乳酸菌,革兰氏阳性,杆状,命名为Ybj。
MRS肉汤培养基:北京路桥有限责任公司;Taq DNA酶、dNTP、10×PCR buffer、MgCl2:宝生物工程(大连)有限公司;溶菌酶:北京科博赛尔生物科技有限公司;DNA纯化回收试剂盒(离心柱型):天根生化科技(北京)有限公司;试验所用引物由生工生物工程(上海)股份有限公司合成;肠球菌菌属生化鉴定管:杭州天和微生物试剂有限公司;化学试剂均为分析纯。
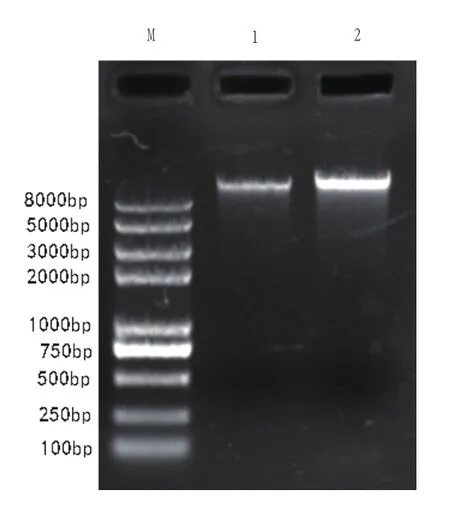
图1 乳酸菌基因组DNA 提取结果
1.2 主要仪器与设备
电泳仪:北京市六一仪器厂DYY 8C型;PCR仪:美国伯乐BIO-RAD PTC-220型;紫外凝胶成像系统:美国A1phaImager HP。
1.3 试验设计
1.3.1 DNA提取
DNA提取参照参考文献[9]并进行适当修改:将筛选出的供试菌株接种到MRS液体培养基中,37℃培养16~17h,取1mL菌液加入到1.5mL离心管中,5 000 r/min离心10min,弃上清。加入50μL TE buffer、100μL(30mg/L)溶菌酶,37℃温育2h,5 000r/min离心10min,弃上清。后续操作使用细菌基因组DNA提取试剂盒,对提取后的DNA溶液取样进行琼脂糖凝聚电泳,剩余DNA置于-20℃保存备用。
1.3.2 乳酸菌16S rDNA的扩增
以提取的基因组D N A为模板进行片段扩增,采用细菌16S rDNA通用引物,正向引物:5'-AGAGTTTGATCCTGGCTCAG-3',反向引物:5'-GGTTACCTTGTTACGACTT-3',PCR扩增采用20μL体系:2×Premix Taq (TaKaRa TaqTMVersion 2.0 plus dye) 10μL,DNA模板 1μL,上游引物(10μmol/L)1μL,下游引物(10μmol/L)1μL,灭菌蒸馏水7μL。PCR反应条件:95℃预变性5min;94℃变性30s,55℃退火45s,72℃延伸1.5min,循环30次;再72℃延伸10min,4℃保存。利用1.5%琼脂糖凝胶电泳检测PCR产物的片段大小是否正确。
1.3.3 转化与质粒提取
采用生产的胶体金回收试剂盒(北京全式金生物技术有限公司)将PCR产物进行回收与纯化。将纯化片段连接pMD-18T,转化至大肠杆菌感受态细胞DH5α,筛选出阳性菌后扩增提取质粒并送大连宝生物工程有限公司测序。
1.3.4 16S rDNA同源性分析和系统发育分析
将所得基因序列与GenBank数据库中已知菌株的16S rDNA序列进行BLAST比对,比对结果用MEGA7.0软件对目的菌株进行系统发育分析。
1.3.5 干酪乳杆菌群16S rDNA 保守序列分析
参考Ward 等[10,11]方法,选取干酪乳杆菌群内6株标准株的16S rDNA序列,用软件MEGA和MegAlign做序列比较,分析可变区核苷酸序列特点,并与待检株Ybj做序列比对。
2 结果与分析
2.1 DNA提取
经检测,提取的基因组DNA为一条亮带,证明DNA提取成功(图1)。
2.2 细菌通用引物的16S rDNA PCR产物检测
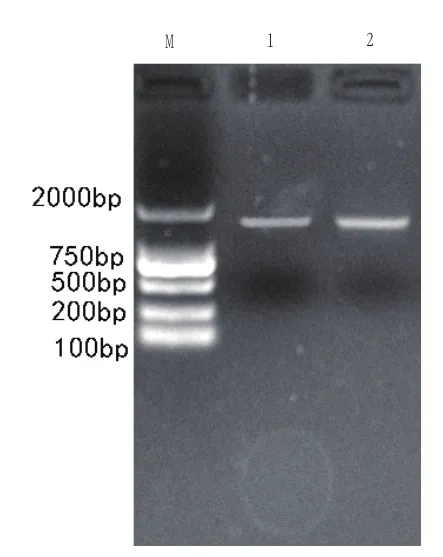
图2 乳酸菌基因组DNA 16S rDNA PCR扩增结果
用细菌通用引物对Ybj株基因组DNA进行扩增验证,扩增产物在约1 500bp处均有条带,与预期结果相符,说明菌株目标片段扩增成功(图2)。
2.3 分离菌与参考株16S rRNA同源性及系统发育树构建及分析
测序结果经chromas软件双向拼接,Ybj株PCR产物全长1 531bp,经NCBI数据库BLAST比对,乳酸菌Ybj与Lactobacillus casei、Lactobacillus paracasei的相似度均达到99.0%以上。说明它们具有很近的亲缘关系,初步认定乳酸菌Ybj为(副)干酪乳杆菌。
参考《乳酸细菌分类鉴定及实验方法》[1],及标准株数据库(http∶//www.straininfo.net/),共选取乳杆菌属18个代表菌株的16S rDNA基因序列,将其16S rDNA序列通过MEGA7.0软件分析,采用Clustral W进行多序列匹配排列,用neighbor-joining获得发育系统树,比较它们之间的进化距离。结果显示,待测菌Ybj与L.casei、L.paracasei的同源性分别为99.8%、99.7%(图3),从系统发育树(图4)可以直观地看出分离菌Ybj与干酪乳杆菌、副干酪乳杆菌处于同一分支,但无法确定属于哪一种。
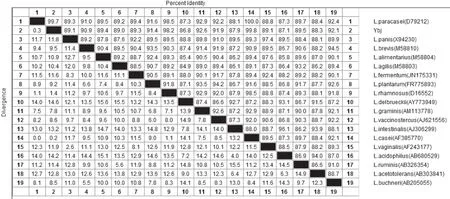
图3 乳杆菌属 16S r DNA 序列比对相似度结果
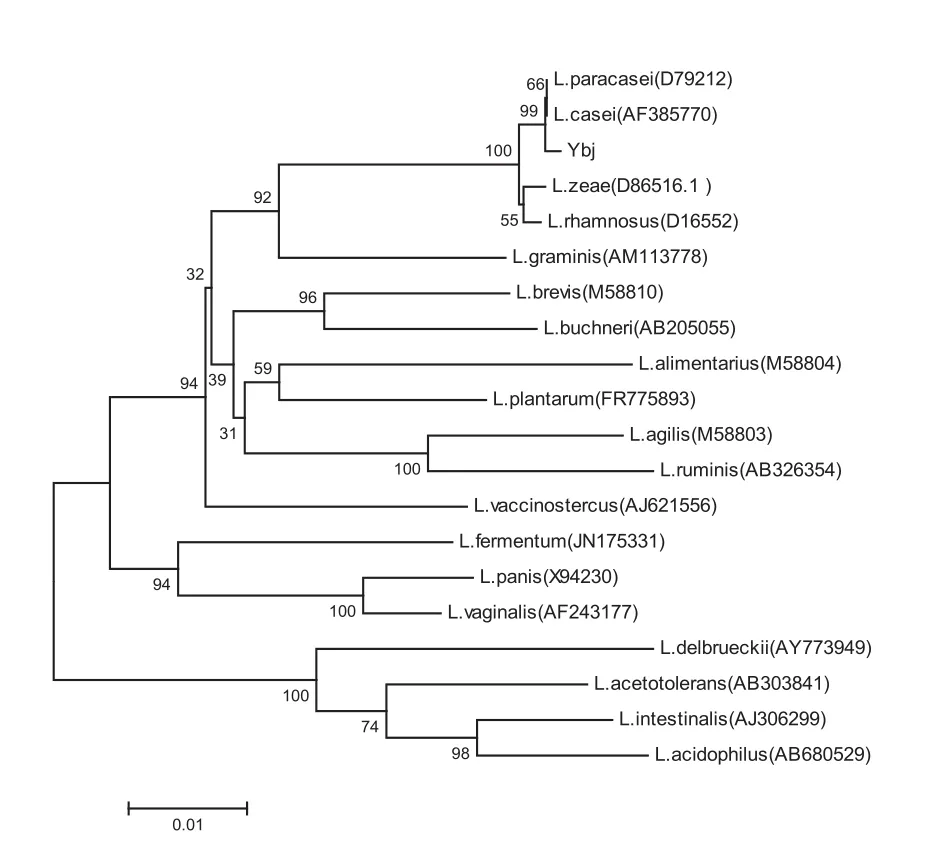
图4 分离菌16S rDNA与18个同属代表序列构建的系统发育树
2.4 干酪乳杆菌群16S rDNA 可变区序列分析
研究发现16S rDNA有10个可变区(V1~V10),其中V1~V3区含有种属特异性序列,可用于种内鉴定[12]。选取与干酪乳杆菌群亲缘关系最近的8株代表株(D79212、D86517、AF469172、KP326371、AB008211、D16552、KT630827、D86516),与分离株Ybj的16S rDNA序列用MegAlign软件进行比对,寻找可变区,结果见图5,以ATCC 334株为参考,其核苷酸序列的第46~84bp存在一明显可变区。
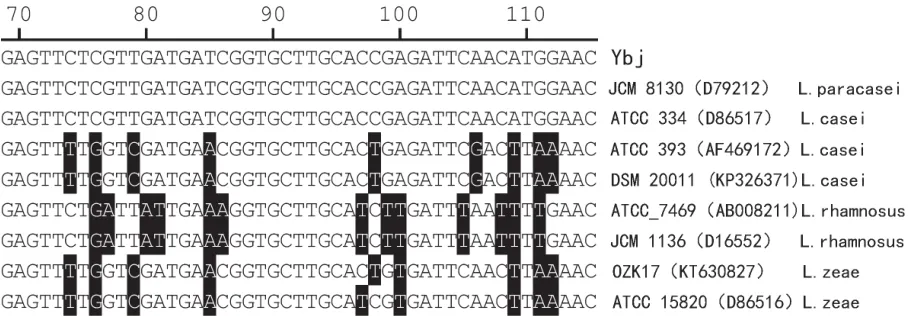
图5 群内代表株16S rDNA可变区核苷酸序列比对分析
如图5所示,分离株Ybj与L.paracaseiJCM 8130株的可变区部分完全符合,与L.caseiATCC 334株的可变区部分也完全符合,但ATCC 334株现已归类于L.paracasei[13],其他菌株均有其种特异性核苷酸序列,与最近的文献报道[11]相符(原文献种特异性反向引物有1碱基有误,应为5'-CCTACTGCTGCCTCCCGTAGGAGT-3'),由此可以确定分离株Ybj为副干酪乳杆菌。
3 讨论
目前不仅在饲料添加剂上还是在表达载体构建上,益生菌的研究方兴未艾,而筛选及分离鉴定是这些工作的基础。由于干酪乳杆菌群存在一种间特异性可变区,本研究仅通过16S rRNA基因序列的归纳分析,即可快捷地鉴定出一株副干酪乳杆菌。当前随着分子技术的发展,出现了随机扩增DNA多态性分析(RAPD)、扩增片段长度多态技术(AFLP)、变性梯度凝胶电泳/温度梯度凝胶电泳(DGGE/TGGE)等多种新技术[5,14],甚至可以达到批量检测,但是实验要求较高,不太适合一般实验室应用,因此16S rDNA序列分析的灵活应用也不失为一种简便的鉴定方法。(副)干酪乳杆菌和鼠李糖乳杆菌被国家食品药品监督管理局批准作为益生菌广泛应用于酸奶、干酪、保健食品和饲料等领域。因此所鉴定出的分离菌具有一定的应用潜力。
[1] 凌代文, 东秀珠. 乳酸细菌分类鉴定及实验方法[M]. 北京:中国轻工业出版社, 1999.
[2] Saad N, Delattre C, Urdaci M, et al. An overview of the last advances in probiotic and prebiotic field[J]. LWT - Food Science and Technology, 2013, 50(1):1-16.
[3] Woo P C, Lau S K, Teng J L, et al. Then and now: use of 16S rDNA gene sequencing for bacterial identification and discovery of novel bacteria in clinical microbiology laboratories[J]. Clinical Microbiology & Infection, 2008, 14(10):908-934.
[4] Felis G E, Dellaglio F, Mizzi L, et al. Comparative sequence analysis of recA gene fragment brings new evidence for a change in the taxonomy of the Lactobacillus casei group[J]. International Journal of Systematic & Evolutionary Microbiology, 2001,51(6):2113-2117.
[5] 周宁, 张建新, 樊明涛,等. 分子分型技术在乳酸菌鉴定及多态性研究中的应用[J]. 食品工业, 2012(5):69-73.
[6] Yu J, Sun Z, Liu W, et al. Phylogenetic study of Lactobacillus acidophilus group, L. casei group and L. plantarum group based on partial hsp60, pheS and tuf gene sequences[J]. European Food Research and Technology, 2012, 234(6):927-934.
[7] 刘光全, 刘勇, 李辉,等. pheS基因序列分析在干酪乳杆菌群种水平鉴定中的应用[J]. 食品与发酵工业, 2011, 37(9):94-97.
[8] 赵思雨, 王辉, 张立冬,等. 利用recA基因对传统发酵乳中干酪乳杆菌群的鉴定和多样性分析[J]. 中国乳品工业, 2016,44(7):8-11.
[9] Naser S, Thompson F L, Hoste B, et al. Phylogeny and identification of Enterococci by atpA gene sequence analysis[J].Journal of Clinical Microbiology, 2005, 43(5):2224-2230.
[10] Ward L J, Timmins M J. Differentiation of Lactobacillus casei,Lactobacillus paracasei and Lactobacillus rhamnosus by polymerase chain reaction.[J]. Letters in Applied Microbiology, 1999,29(2):90-92.
[11] 薛晓晶, 张焱鑫, 陈会君,等. 分子生物学方法鉴定新资源食品中乳杆菌[J]. 检验检疫学刊, 2016, 26(5):1-4.
[12] Dobson C M, Chaban B, Deneer H, et al. Lactobacillus casei,Lactobacillus rhamnosus, and Lactobacillus zeae isolates identified by sequence signature and immunoblot phenotype[J]. Canadian Journal of Microbiology, 2004, 50(7):482.
[13] The type strain of Lactobacillus casei is ATCC 393, ATCC 334 cannot serve as the type because it represents a different taxon,the name Lactobacillus paracasei and its subspecies names are not rejected and the revival of the name 'Lactobacillus zeae' contr[J].International Journal of Systematic & Evolutionary Microbiology,2008, 58(7):1764.
[14] 张家超,孙志宏,刘文俊,等. 适用于乳酸菌分类鉴定的分子生物学技术[J]. 乳业科学与技术, 2009, 32(2):89-93.
Isolation and Identification of a Strain of Lactobacillus casei from Bovine
FU Qiang, CHENG Li-kun, FU Shi-jun, MIAO Li-zhong, WANG Yan
(Shandong Binzhou Animal Science & Veterinary Medicine Academy, Binzhou 256600)
One strain ofLactobacilluswas isolated from bovine intestine in order to separate the valuable probiotics.Followed by 16S rRNA gene sequences analysis, the existence of species specific variable region was con firmed.This strain was eventually identified asLactobacillus paracasei, which may have important research and application in the feed additive industry.
Lactobacillus; Isolation and identification; 16S rDNA; Sequence analysis
S823.4
A
1004-4264(2017)12-0018-04
10.19305/j.cnki.11-3009/s.2017.12.005
2017-03-22
山东省科学技术创新工程-畜禽健康养殖关键技术研究(CXGC2017B02)。
付强(1980-),男,山东菏泽人,助理研究员,硕士,主要从事生物制品研究。
