STGC3基因启动子生物信息学分析及载体构建
2017-12-25,,,,,,,
, ,,, ,,,
(1.南华大学肿瘤研究所,湖南 衡阳 421001;2.南华大学医学院解剖学教研室;3.南华大学医学院2013级临床医学专业;4.湖北省大冶市人民医院病理科)
·基础医学·
STGC3基因启动子生物信息学分析及载体构建
李素云1,2,李佳3,王莉莉4,钟嘉宏3,陈青3,熊文芳3,曾小微3,贺修胜1*
(1.南华大学肿瘤研究所,湖南 衡阳 421001;2.南华大学医学院解剖学教研室;3.南华大学医学院2013级临床医学专业;4.湖北省大冶市人民医院病理科)
目的运用生物信息学预测并分析鼻咽癌抑癌基因STGC3启动子,构建双荧光素酶报告基因表达载体。方法采用生物信息学软件预测STGC3基因5′端上游调控区5 000 bp序列进行启动子预测与分析,克隆最有可能的启动子,构建萤火虫荧光素酶双报告基因重组质粒,重组质粒经MluⅠ和BglⅡ限制性内切酶酶切及测序鉴定。
结果STGC3基因5′端上游-3 046 bp至-46 bp区域内有启动子活性,在-2 992 bp至-69 bp间启动子分值达0.8以上,其中-845 bp至-795 bp间分值最高,达到1.0。在-1 140 bp和-774 bp处检测到GC盒信号;在-441 bp处检测到CAAT盒信号。在-1 805 bp至-1 705 bp和-900 bp至-684 bp间各有一个CpG岛;在-2 348 bp和-948 bp处各有一个转录起始位点。经不同长度的片段缺失比对,取最有可能的283 bp(-1 360 bp至-1 077 bp)、281 bp(-934 bp至-653 bp)和571 bp(-500 bp至+72 bp)启动子片段构建pGL3-en283、pGL3-en281及pGL3-en571三种质粒,质粒经酶切测序,酶切片段大小一致,序列正确。结论根据生物物信息学分析结果,成功构建STGC3基因启动子萤火虫荧光素酶报告基因表达载体,为双荧光素酶报告基因检测系统研究该基因启动子活性提供前期基础。
鼻咽癌; 生物信息学; STGC3基因; 启动子
鼻咽癌(Nasopharyngeal carcinoma,NPC)是头颈部常见的恶性肿瘤之一,起源于人体鼻咽部黏膜上皮,其原发部位隐匿,难以早期发现,恶性度高,且转移早;鼻咽癌发病常见于东南亚地区及我国南方多省,其发病具有种族特异性与地域集聚性,鼻咽癌的发生发展常常涉及到癌基因和/或抑癌基因[1-8]。STGC3基因是课题组前期克隆并分析所得的鼻咽癌抑瘤基因,在GenBank中,基因登录号为AY078383。启动子是位于结构基因5′端上游区域的DNA序列,也是RNA聚合酶结合的部位,是基因转录起始中最重要的结构[9]。本研究利用生物信息学预测、分析并克隆STGC3基因启动子,以期为该基因转录调控元件研究奠定前期基础,为其抑瘤作用机制提供实验依据。
1 材料与方法
1.1质粒与试剂荧火虫荧光素酶报告基因载体pGL3 Enhance购于Promega公司。E.coli.J M 109为中南大学湘雅医学院肿瘤研究所提供。DNA抽提试剂盒购于大连Takara公司,pfu高保真DNA聚合酶购自Fermentas公司,DNA Marker DL2000为广州美津生物公司,限制性内切酶、DNA连接酶购于NEB公司,胶回收试剂盒购于杭州四季青生物工程有限公司,酵母粉及胰蛋白胨购于Oxoid公司,琼脂、琼脂糖购于上海生工公司,甘油购于长沙艾杰生物技术公司。
1.2引物设计与合成在NCBI数据库中,查找STGC3基因gDNA序列,采用生物信息学预测分析得到启动子区,Primer5.0软件行引物设计,DNASTAR软件行酶谱分析,在上游和下游引物5′端分别引入MluⅠ和BglⅡ限制性内切酶酶切位点和保护碱基,交上海生物工程有限公司合成,引物序列见表1。
表1PCR引物序列与产物长度

GenePrimerSequenceProductSize(bp)PGL3⁃en283sense:cgacgcgtTTAGCTGGACATGGTGGCGTantisense:gaagatctATCTCTGTCCCAAAGTGACA283PGL3⁃en281sense:cgacgcgtCCTGAGCGACAGAGTGAGantisense:gaagatctCGAGTAGCTGGGACAACA281PGL3⁃en571sense:cgacgcgtCCTGTAATCCCAGCACTTantisense:gaagatctCTCCTCCATAATGAGACTTT571
1.3分析软件
1.3.1 基因结构分析(NCBI) http://www.ncbi.nlm.nih.gov/genbank
1.3.2 神经网络启动子预测分析(NNPP) http://www.fruitfly.org/seqtools/promoter.html.
1.3.3 转录起始位点及启动子预测分析(Promoter 2.0) http://www.cbs.dtu.dk/services/Promoter
1.3.4 CpG岛预测分析(MethPrimer、Cpgplot) http://www.urogene.org/methprimer;http://www.ebi.ac.uk/Tools/emboss/cpgplot
1.4 STGC3基因启动子质粒的构建提取人外周血中的DNA,以此为模板,采用pfu高保真DNA聚合酶扩增相应的DNA片段。PCR条件为95 ℃预变性3 min后循环,95 ℃变性30 s,57 ℃退火40 s,72 ℃延伸60 s,32个循环后于72 ℃终延伸10 min。扩增产物经1%的琼脂糖凝胶电泳,回收PCR产物。MluⅠ和BglⅡ内切酶双酶切PCR产物及载体pGL3 Enhance,胶回收纯化,将PCR产物与载体大片段进行连接反应,得到的质粒分别命名为pGL3-en283、pGL3-en281及pGL3-en571。重组质粒经MluⅠ和BglⅡ双酶切及测序进行鉴定。
2 结 果
2.1 STGC3基因定位分析在线软件NCBI对STGC3基因结构进行分析,结果显示STGC3基因在GenBank数据库中位于ContigsNT-022517.18上,染色体位于chr3:49,297,518--49,298,749(GRCh37)区域,基因全长为1 232 bp。
2.2 STGC3基因启动子分析在NCBI数据库中,对STGC3基因进行序列比对分析,找出STGC3基因5′端上游5 000 bp序列及基因序列,预测STGC3基因启动子。并对STGC3基因启动子、外显子及内含子序列分析,结果显示STGC3基因cDNA全长1 271 bp,其开放阅读框(ORF)441 bp,为单外显子基因,其中5′端上游3 000 bp序列(chr3:49,295,000-49,298,000)为启动子活跃区。即以STGC3基因转录起始密码子(ATG)的第一个碱基A作为+1,启动子活跃区在-3 046 bp至-46 bp长3 000 bp)区。
提取-3 046 bp至-46 bp间3 000 bp序列,输入启动子软件进行分析,NNPP结果显示,在-2 992(55) bp至-69(2978)bp区启动子分值存在0.8以上序列,其中在-1 360(1 687)bp至-1 048(1 999)bp区含有分值为0.91,即-1 213 bp(1 834 bp)至-1 163 bp(1 884 bp)的启动子片段,在-1 048(1 999)bp至-653(2 394)bp区含有分值为1.0即-845(2 203)bp至-795(2 253)bp启动子片段;在-541(2 506)bp+72bp区含有分值为0.97即-541(2 507)bp至-491(2 557)bp启动子片段。(图1)。
Promoter2.0结果显示,STGC3基因起始密码子上游-3 046 bp至-46 bp区有 两个转录起始位点,分别位于-2 348 bp(700 bp)处和-948 bp(2 100 bp)处(图2)。在-1 140(1 907)bp至-774(2 273)bp处有GC盒,在-441(2 606)bp处有CAAT盒。
NNPP分析结果:

图1 STGC3基因启动子分析结果

图2 STGC3基因转录起始位点分析结果
2.3 STGC3基因5′端CpG岛分析MethPrimer分析比对结果显示,STGC3基因chr3:49,295,000-49,298,000区3 000 bp的序列中,该区域有两个CpG岛:在-1 805(1 242)bp至-1 705(1 342)bp处有一个CpG岛,长101 bp;在-900(2 147) bp至-684(2 363) bp处有另一个CpG岛,长217 bp(图3)。Cpgplot分析比对结果显示,该区域只存在一个CpG岛(图4):在-900(2 147)bp至-684(2 363)bp处,长217 bp。
两个不同的在线软件程序分析结果不同,但二者在-900(2 147) bp至-684(2 363)bp处有一个共同CpG岛区域。且此区包含动子分值1.0的区域,说明该区域可能是STGC3基因的启动子。
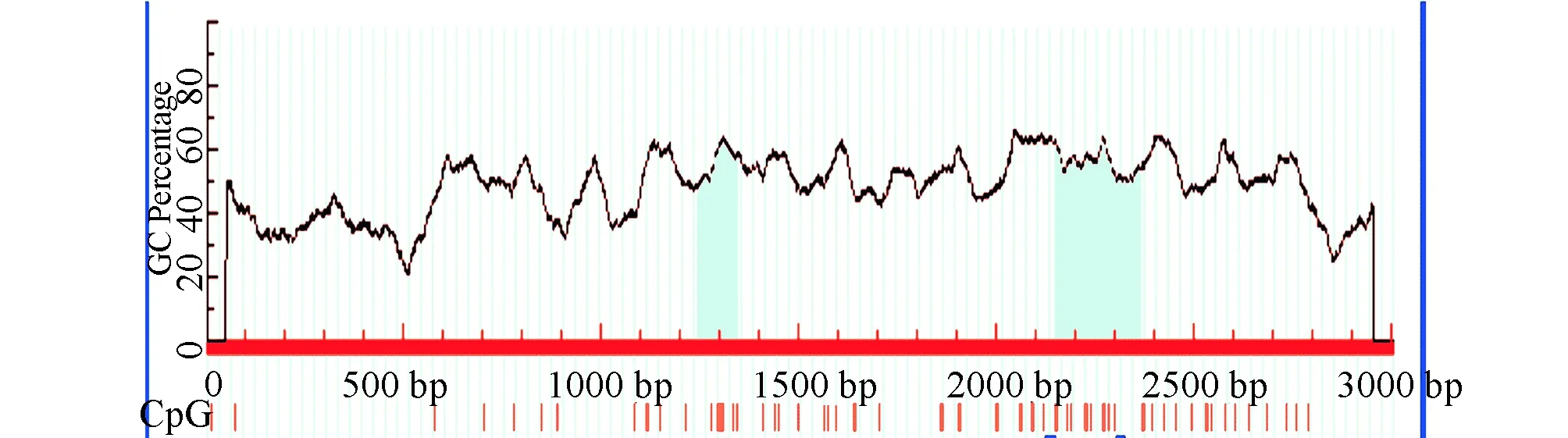
图3 MethPrimer分析CpG岛图
2.4 STGC3基因启动子区生物信息学综合分析
根据生物信息学综合分析,结果显示,STGC3基因预测所得的启动子活性区为-3 046 bp至-46 bp,其中-1 360 bp至-1 048 bp区域包含启动子预测分值为0.91的片段和GC盒;-1 048 bp至-653 bp区域含有启动子预测分值1.0的片段、转录起始位点、CPG岛、GC盒,此区域有可能是STGC3基因启动子核心区;-541 bp至+72 bp区域含有启动子预测分值0.97的片段和CAAT盒,做示意图以明确STGC3基因启动子的基本结构(图5)。对启动子区不同长度缺失分析,最后取最有可能的启动子片段283 bp(-1 360 bp至-1 077 bp)、281bp(-934 bp至-653 bp)、571 bp(-500 bp至+72 bp)片段进行目的片段重组质粒构建(图6)。
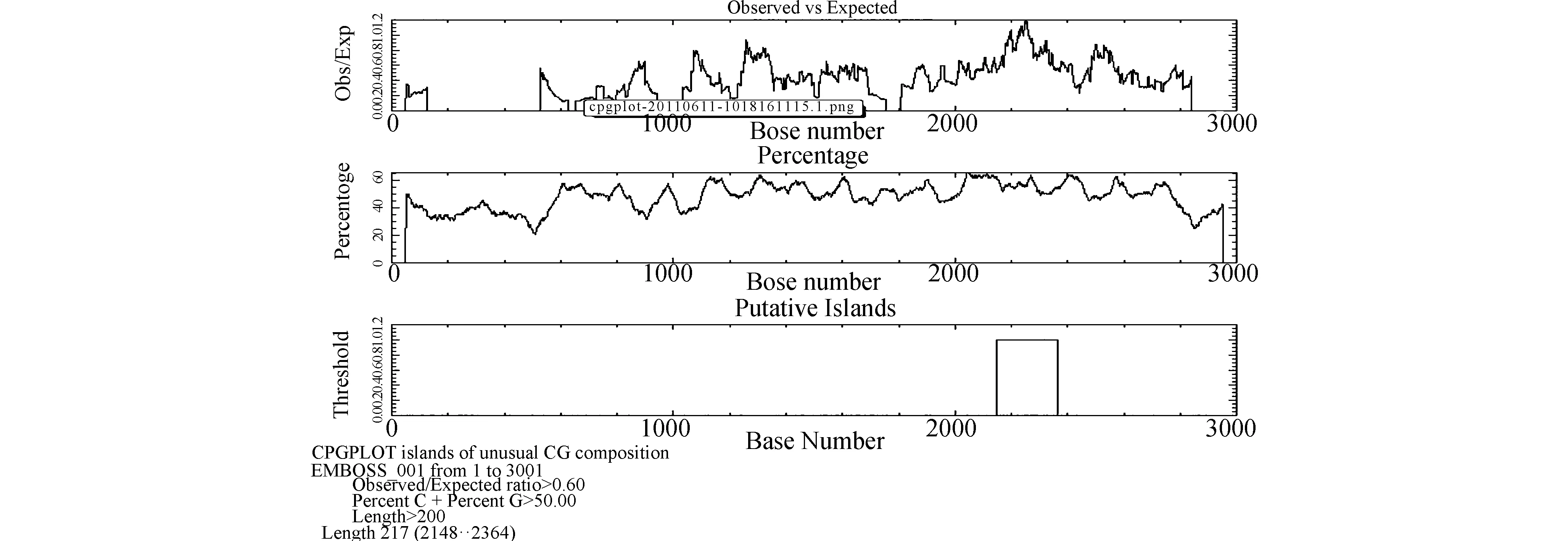
图4 Cpgplot分析CpG岛图

图5 STGC3基因启动子区示意图
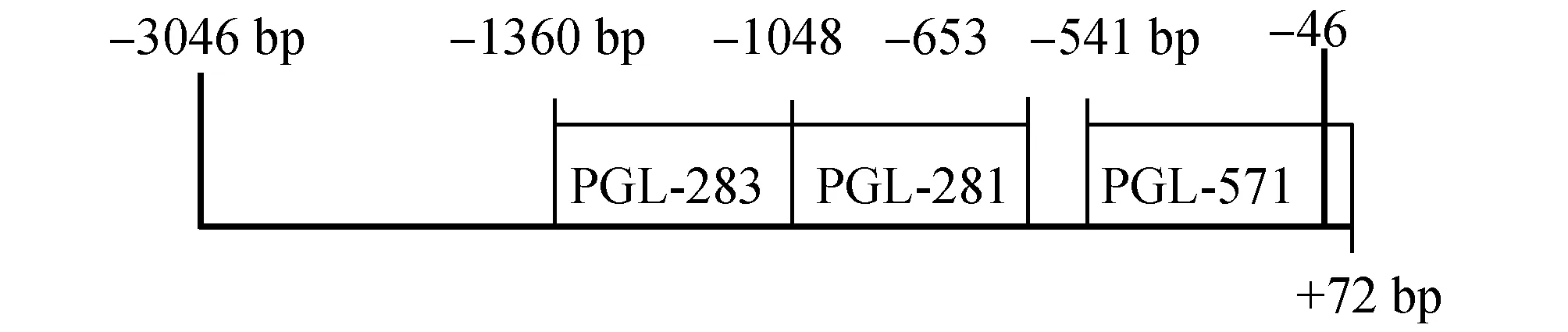
图6 STGC3基因启动子区不同片段载体构建区域示意图
2.5 STGC3基因启动子区目的片段重组载体构建结果
2.5.1 重组质粒双酶切鉴定 利用高保真Pfu酶,以人外周血DNA作为模板,PCR扩增启动子区片段,将扩增片段构建至PGL3-enhance质粒中,得到重组质粒pGL3-en283、pGL3-en281及pGL3-en571。重组质粒经MluⅠ和BglⅡ限制性内切酶酶切,酶切结果显示,重组质粒中插入的片段大小和NCBI软件显示结果一致,片段大小正确(图7)。
2.5.2 重组质粒测序鉴定 重组质粒经双酶切鉴定的阳性菌,扩大培养后,抽提质粒,送Invitrogen公司进行测序,测序结果在blast中进行比对分析,测序图如下(图8~10,红色箭头之间为目的序列),比对分析结果显示,碱基序列正确,表明重组质粒构建成功。
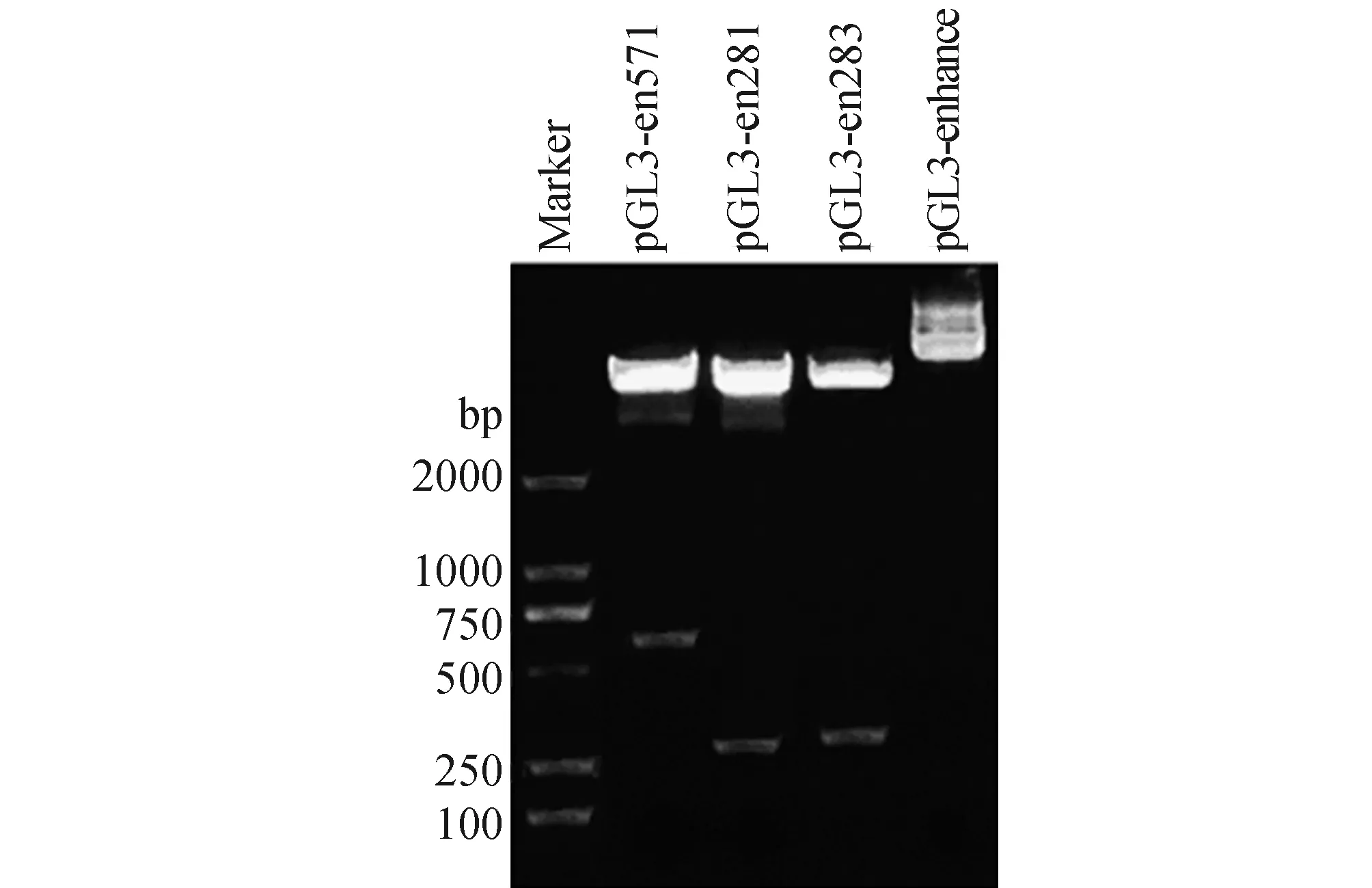
图7 重组质粒酶切电泳
3 讨 论
鼻咽癌是最常见的头颈部恶性肿瘤之一,其发生发展涉及原癌基因和/或抑癌基因,其中抑癌基因的失活在其发生发展中起着更重要的作用。STGC3作为课题组前期克隆的抑瘤基因,其抑瘤功能取得了一定的研究进展,但有关STGC3基因转录调控的机制还不清楚。
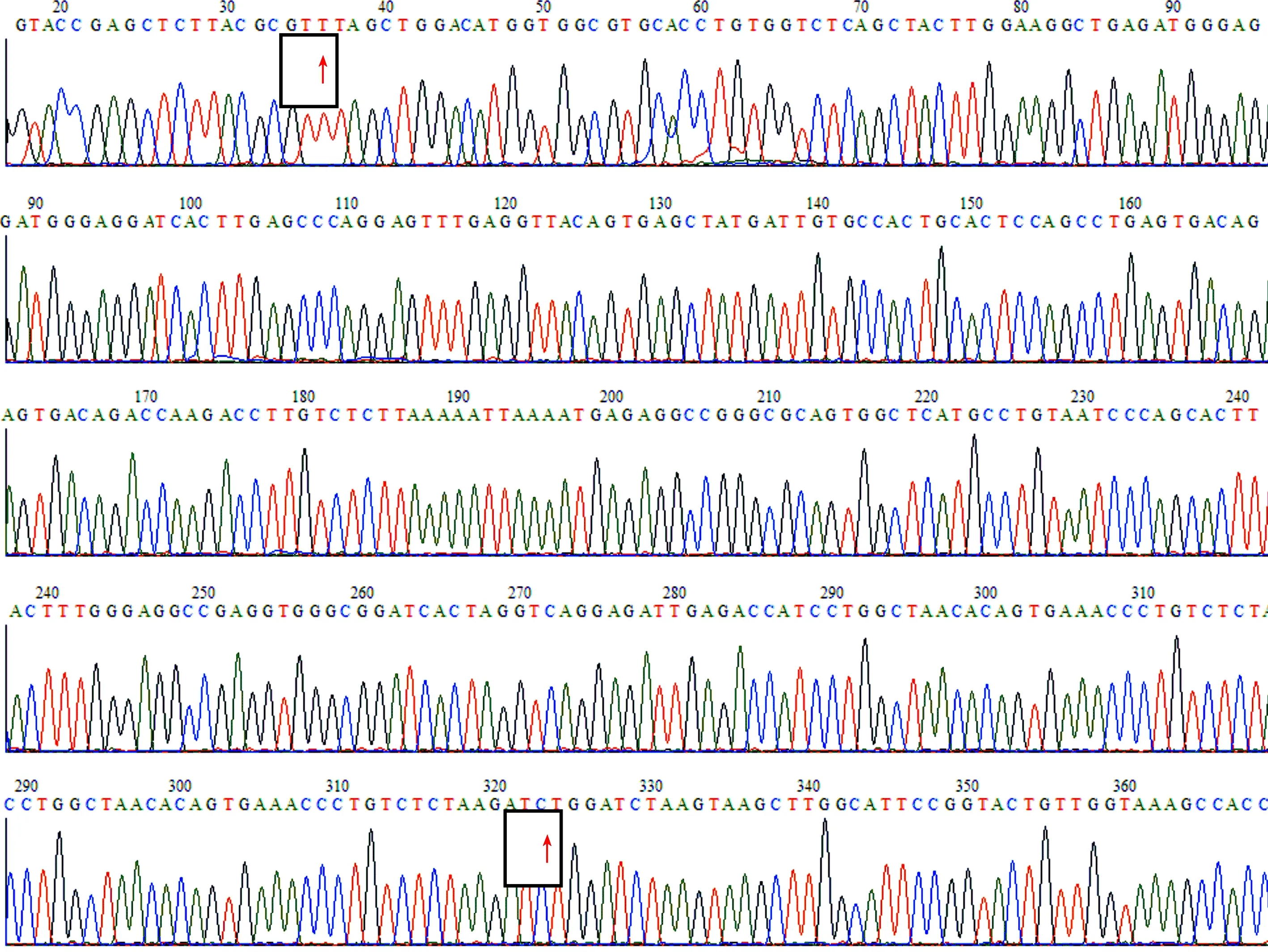
图8 pGL3-en283重组质粒测序结果

图9 pGL3-en281重组质粒测序结果

图10 pGL3-en571重组质粒测序结果
具有转录起始特异性的启动子是一段非编码DNA序列,位于结构基因5′端上游,可使RNA聚合酶活化,并使RNA聚合酶准确结合到DNA模板上,是基因转录调控的核心区域;在转录调控中,扮演着十分重要角色的真核生物启动子,是RNA聚合酶特异性结合位点周围的转录调控元件。
本研究运用生物信息学预测启动子,该方法是基因转录调控机制的重要研究方法,能有效地从基因组中查找已知和末知的的转录调控元件,为研究基因的结构、功能提供可靠依据。在真核生物基因表达调控体系中,基因的转录起始与调控主要通过顺式作用元件、反式作用因子和RNA聚合酶相互协同,共同完成。顺式作用元件对基因转录的起始位点及转录效率起着决定性的作用,基因的转录起始常通过启动子来实现[10-11]。
启动子序列包含核心元件TATA盒、上游核心启动子元件BRE、下游启动子元件DPE、起始子,还包含CAAT盒及GC盒;研究表明约10-20﹪的编码蛋白基因含特定的转录起始位点和TATA盒,大多数基因启动子不含TATA盒,但有多个选择性的启动子区和转录起始位点,约72﹪的基因启动子区含CpG岛,CpG岛是一段约200 bp或更长的DNA序列,其G+C含量较高,且CpG岛占G+C含量达50%以上,许多脊椎动物的启动子区都与CpG岛的位置重合[12-15]。启动子的这些特征是研究工作者利用生物信息学方法研究启动子的重要信息。因此,本研究在NCBI数据库对STGC3基因结构定位分析,对STGC3基因5′端上游5000bp序列进行分析,在软件中,查找该基因的启动子、内含子及外显子,发现该基因启动子区和核心启动子区。利用Promoter 2.0软件预测该基因的转录起始位点,通过MethPrimer与Cpgplot软件预测,发现CpG岛,此外,同时对该3 000 bp序列进行分析,发现STGC3基因无TATA盒,但有GC盒信号,也有CAAT盒信号,经预测打分得到启动子及核心启动子区。
本研究将STGC3基因生物信息学分析得到的结果,根据基因启动子的分析,克隆并构建不同启动子片段报告基因载体,为进一步研究STGC3基因启动活性及转录调控元件,探讨基因表达调控机制提供实验依据。
[1] Chang ET,Adami HO.The enigmatic epidemiology of nasopharyngeal carcinoma[J].Cancer Epidemiol Biomarkers Prev,2006,15(10):1765-1777.
[2] Yu WM,Hussain SS.Incidence of nasopharyngeal carcinoma in Chinese immigrants,compared with Chinese in China and South East Asia:review[J].J Laryngol Otol,2009,123(10):1067-1074.
[3] Caponiqro F,Lonqo F,Lonna F,et al.Treatment approaches to nasopharyngeal carcinoma:a review[J].Anticancer Drugs,2010,21(5):471- 477.
[4] Lo KW,Chung GT,To KF.Deciphering the molecular genetic basis of NPC through molecular,cytogenetic,and epigenetic approaches[J].Semin Cancer Biol,2012,22(2):79-86.
[5] Liao Q,Guo X,Li X,et al.Prohibitin is an important biomarker for nasopharyngeal carcinoma progression and prognosis[J].Eur J Cancer Prev,2013,22(1):68-76.
[6] Adham M,Kurniawan AN,Muhtadi AI,et al.Nasopharyngeal carcinoma in Indonesia:epidemiology,incidence,signs,and symptoms at presentation[J].Chin J Cancer,2012,31(4):185-196.
[7] Ruan HL,Qin HD,Shugart YY,et al.Developing Genetic Epidemiological Models to Predict Risk for Nasopharyngeal Carcinoma in High-Risk Population of China[J].PLoS One,2013,8(2):e56128.
[8] Arnold M,Wildeman MA,Visser O,et al.Lower mortality from nasopharyngeal cancer in the Netherlands since 1970 with differential incidence trends in histopathology[J].Oral Oncol,2013,49(3):237-243.
[9] Stokken J,Manz RM,Flagg A,et al.Synchronous occurrence of nasopharyngeal carcinoma and Hodgkin lymphoma[J].Int J Pediatr Otorhinolaryngol,2014,78(1):154-156.
[10] Juven-Gershon T,Hsu JY,Theisen JW,et al.The RNA polymerase Ⅱcore promoter-the gateway to transcription[J].Curr Opin Cell Biol,2008,20(3):253-259.
[11] Sandelin A,Carninci P,Lenhard B,et al.Mammalian RNA polymerase Ⅱcore promoter:insights from genome-wide studies[J].Nat Rev Genet,2007,8(6):424-436.
[12] 侯德富,关勇军,关瑞,等.人NPCEDRG基因启动子的克隆及CCAAT/NFY结合点初步分析[J].生物化学与生物物理进展,2011,38(8):713-723.
[13] VanniniA,Cramer P.Conservation between the RNA polymerase I,II,and III transcription initiation machineries[J].Mol Cell,2012,45(4):439-446.
[14] Ashraf MA,Shahid AA,Rao AQ,et al.Functional characterization of a bidirectional plant promoter from cotton leaf curl Burewala virus using an Agrobacterium-mediated transient assay[J].Viruses,2014,6(1):223-242.
[15] Duval C,Zaniolo K,Leclerc S,et al.Characterization of the human α9 integrin subunit gene:Promoter analysis and transcriptionalregulation in ocular cells[J].Exp Eye Res,2015,135:146-163.
BioinformaticsanalysisofsTGC3genepromoterandconstructionofdualluciferasereportgeneexpressionVector
LI Suyun,LI Jia,WANG Lili,et al
(CancerResearchInstitute,UniversityofSouthChina,Hengyang421001,Hunan,China)
ObjectiveUsing bioinformatics to predict and analyse the promoter of STGC3 gene,then construct dual luciferase reporter gene expression vector for 5′ Upstream regulated elements of STGC3 gene.MethodsThe bioinformatics was used to predict and analyze the promoter region of STGC3 during gene 5′upstream 5000bp regulatory region,and then the most likely promoters were cloned.The dual-luciferase reporter gene plasmids were constructed,and these recombinant plasmids were identified by the MluⅠand BglⅡ enzyme digestion and DNA sequencing.ResultsThe bioinformatics results show that there are promoter activity from -3 046 bp to-46 bp.The activity value of -2 992 bp to -69 bp is 0.8 and -845 bp to -795 bp is 1.0.There is a GC box in -1 140 bp to -774 bp and a CAAT box in -441 bp.Two CpG islands were detected in -1 805 bp to -1 705 bp and -900 bp to -684 bp.Two transcription start sites(-2 348 bp and -948 bp) were found.The most possible fragments 283bp (-1 360 bp to -1 077 bp),281bP(-934 bp to -653 bp) and 571bP(-500 bp to -72 bp) were chosen to construct plasmids pGL3-en283,pGL3-en281and pGL3-en571.These plasmids were identified by enzyme digestion and sequencing,the size of the fragment and the sequence was correct.ConclusionBased on the results of bioinformatics analysis,the successful construction of STGC3 gene dual luciferase report gene expression vector,provides a preliminary basis for the dual luciferase report gene detection system of the gene promoter activity.
nasopharyngeal carcinoma; bioinformatics; STGC3 gene; promoter
10.15972/j.cnki.43-1509/r.2017.01.011
2016-05-21;
2016-12-19
国家自然科学基金资助项目(2011GZK46)、湖南省科技厅资助项目(2012TT2020)、湖南省大学生研究性学习与创新性实验资助项目(231).
*通讯作者,E-mail:hexiusheng@hotmail.com.
R739.63
A
蒋湘莲)
