Fusarium graminearum sensu stricto和F.asiaticum3种分子鉴定方法的比较
2017-12-20茹艳艳谢淑娜李保叶刘佳中郝俊杰
茹艳艳,谢淑娜,李保叶,孙 静,刘佳中,郝俊杰
(河南省农业科学院 植物保护研究所/农业部华北南部作物有害生物综合治理重点实验室/河南省农作物病虫害防治重点实验室,河南 郑州 450002)
Fusariumgraminearumsensustricto和F.asiaticum3种分子鉴定方法的比较
茹艳艳,谢淑娜,李保叶,孙 静,刘佳中,郝俊杰*
(河南省农业科学院 植物保护研究所/农业部华北南部作物有害生物综合治理重点实验室/河南省农作物病虫害防治重点实验室,河南 郑州 450002)
Fusariumgraminearumsensu stricto和F.asiaticum是中国主要的禾谷镰刀菌复合种(FGSC)成员,为了提高这2个种的鉴定效率,选取48个引起小麦赤霉病和玉米茎腐病的FGSC菌株,比较了系统发育谱系分析、PCR-RFLP(聚合酶链式反应-限制性片段长度多态性)和多重PCR 3种分子鉴定方法。系统发育谱系分析首先扩增菌株的EF-1α和Tri101两个基因的部分序列,测序之后利用MEGA 6.06软件构建系统发育树,结果表明,有11个菌株与F.asiaticum聚在一起,37个菌株与F.graminearumsensu stricto聚为一枝,此法繁琐、复杂、成本高,但鉴定结果准确。PCR-RFLP基于组蛋白H3基因特殊的酶切位点,设计引物H3dStyⅠ/H3R1,通过PCR和酶切反应进行鉴定,电泳检测发现,有11个菌株为F.asiaticum,37个菌株为F.graminearumsensu stricto,该法工作量大且对操作过程要求高。多重PCR基于CYP51A基因设计特异引物FaF/FaR和FgF/FgR进行鉴定,电泳检测结果表明,有11个菌株为F.asiaticum,37个菌株为F.graminearumsensu stricto,此法操作简单、效率高。以上3种分子鉴定方法结果一致,但多重PCR方法具有快速、准确、高效的特点。
禾谷镰刀菌复合种;Fusariumgraminearumsensu stricto;Fusariumasiaticum; 分子鉴定
禾谷镰刀菌复合种(Fusariumgraminearumspecies complex,FGSC)是农业生产中常见的病原菌,可引起多种作物根腐病、茎腐病和穗腐病的发生,不仅使农作物出现减产,造成严重的经济损失,而且产生的多种真菌毒素还影响粮食的安全[1-3]。目前,已经明确禾谷镰刀菌复合种包含至少16个种,不同种表现一定的地理分布特征,其中中国主要为F.asiaticum(进化分枝6)和F.graminearumsensu stricto(进化分枝7)[4-7]。
关于FGSC的概念,最初O’Donnell等[8]在全球收集了数十个代表性菌株,选用Tri101(the trichothecene 3-O-acetyltransferase gene)、RED(a putative reductase gene)、EF-1α(translation elongation factor 1α)、TUB(β-tubulin)、PHO(phosphate permase genes)和URA(UTP-ammonia ligase)6个单拷贝核基因,进行系统发育谱系分析,发现禾谷镰刀菌是一个复合种。之后,Ward等[9]报道了利用多位点基因型分析法(multilocus genotyping assay)进行FGSC的鉴定,该方法基于3个持家基因(RED、MAT和TEF-1)和毒素合成的3个基因(TRI101、TRI12 和TRI3)设计48对引物,通过多重PCR,同时进行种和毒素型的鉴定。Davari等[10]发明了一种灵敏、有效识别FGSC的方法——RCA(rolling circle amplification),该方法通过比较超过40个Fusariumspp.菌株(FGSC、FIESC、FTSC和FOSC)的EF-1α基因序列,设计FGSC特异的探针引物,PCR产物经过1%的琼脂糖凝胶电泳(有条带)或者加入荧光染料(SYBR Green Ⅰ)在紫外(UV)灯下照射(荧光发亮)进行鉴定,此法的特点是灵敏,但不能区分FGSC的不同种。Fernández-Ortuo等[11]研究报道了通过Real-time PCR同时检测FGSC中3个不同种(F.asiaticum、F.ussurianum和F.vorosii),该方法基于镰刀菌的特异基因CYP51C(a sterol 14-alpha demethylase paralogue)设计定量引物进行鉴定,此法具有明显的局限性,只能对已知的这3个种进行鉴定。
由于中国和日本的FGSC主要为F.graminearumsensu stricto和F.asiaticum2个种,因此,研究者基于这2个种,分别建立了系统发育谱系分析、PCR-限制性片段长度多态性(PCR-RFLP)和多重PCR等方法。系统发育谱系分析是基于FGSC菌株的Tri101和EF-1α基因,利用MEGA 6.06软件构建系统发育树,完成种的鉴定[12-13];PCR-RFLP是基于组蛋白H3基因特殊的酶切位点,设计引物H3dStyⅠ/H3R1,通过PCR和酶切反应,完成种的鉴定[14];多重PCR是基于CYP51A基因,设计特异引物FaF/FaR(对F.asiaticum特异)和FgF/FgR(对F.graminearumsensu stricto特异),完成对FGSC种的鉴定[15]。本研究选取引起河南省小麦赤霉病和玉米茎腐病的FGSC菌株,通过比较3种分子鉴定方法,选择出一种快速、准确、高效的F.graminearumsensu stricto和F.asiaticum鉴定方法。
1 材料和方法
1.1 样品收集与菌株分离
2012年,在小麦赤霉病和玉米茎腐病发病的高峰期,采集河南省许昌、漯河、驻马店地区的病样(小麦赤霉病为穗部,玉米茎腐病为病株茎基部第1~2节)。FGSC菌株的分离、单孢纯化和初步鉴定(主要通过观察PDA培养基上的菌落形态和颜色以及液体摇菌培养后分生孢子的形态来鉴定)等参照孙静等[16]和吴全安等[17]的方法,共获得48个FGSC菌株(表1)。

表1 试验所用的48个FGSC菌株
续表1 试验所用的48个FGSC菌株
注:菌株命名的第1个字母“W”和“M”分别代表菌株来自小麦和玉米。
1.2 菌丝收集与DNA提取
对活化的菌株挑取少量新鲜菌丝到PDA平板上,26 ℃黑暗条件下培养4 d后,以灭菌牙签刮取菌丝1 g左右,用组织细胞破碎仪(Bullet Blender,美国)研磨后,采用真菌DNA小量提取试剂盒(上海莱枫生物技术有限公司,产品目录号:DK621-01)提取各菌株的基因组DNA,置于-20 ℃保存备用。
1.3 Tri101和EF-1α基因的PCR扩增、测序及系统发育谱系分析
用引物TRI1015B/TRI1013E[18]和EF1/EF2[19]分别扩增48个菌株的Tri101和EF-1α基因部分序列(表2)。PCR反应体系均为:TaqPCR Master Mix(BIOTEKE公司)12.5 μL、上游引物(5 μmol/L)和下游引物(5 μmol/L)各1 μL、模板DNA(10 ng/μL)1 μL、ddH2O 9.5 μL,总体积为25 μL。反应程序均为:94 ℃ 5 min;94 ℃ 50 s,54 ℃ 50 s,72 ℃ 1 min,35个循环;72 ℃ 10 min。扩增产物使用1.2%的琼脂糖凝胶电泳检测。引物合成及扩增产物序列测定(采用双向测序)均由生工生物工程(上海)股份有限公司完成。

表2 试验所用的引物
注:R=A或G,Y=C或T,S=C或G,I=Inosine(肌苷),N=Nucleoside(核苷)。
通过DNAStar软件的SeqMan模块将Tri101和EF-1α基因序列拼接在一起,人工校对后获得测序菌株的完整序列,然后与GenBank数据库中FGSC 16个种代表性菌株[来自美国农业研究菌种保藏中心(NRRL)]的Tri101和EF-1α基因序列通过ClustalX 1.81(www.clustal.org/clustal2/)进行多重比对,删除多余不匹配的序列,利用MEGA 6.06软件通过最大似然法(maximum-likelihood,ML),根据贝叶斯推断(Bayesian information criterion scores)选择最合适的模型(Kimura-2参数模型,Bootstrap replications的值为1 000)进行系统发育谱系分析[12-13]。
1.4 PCR-RFLP
用引物H3dStyⅠ和H3R1(表2)扩增48个菌株的基因组DNA,获得的PCR产物分别用内切酶StyⅠ(对F.asiaticum特异,酶切片段大小为195 bp和28 bp)或者EcoRⅤ酶切(对F.graminearumsensu stricto特异,酶切片段大小为191 bp和32 bp),其他Fusariumspp.菌株不会出现酶切片段,酶切产物使用2%的琼脂糖凝胶电泳检测,用StyⅠ酶切后电泳条带较小的即为F.asiaticum菌株,用EcoRⅤ酶切后电泳条带较小的即为F.graminearumsensu stricto菌株。PCR反应操作方法参照Suga等[14]的反应体系和程序。
1.5 多重PCR
同时用引物FaF/FaR和FgF/FgR(表2)扩增48个菌株的基因组DNA。多重PCR反应操作方法参照Yun等[15]的反应体系和程序,扩增产物使用1.5%的琼脂糖凝胶电泳检测,F.asiaticum菌株电泳条带大小为292 bp,F.graminearumsensu stricto菌株电泳条带大小为352 bp。
2 结果与分析
2.1 FGSC菌株的系统发育谱系分析
对所研究的48个菌株的Tri101和EF-1α基因部分序列进行了测定。同时从GenBank数据库中下载了已报道的FGSC 16个种(F.asiaticum、F.graminearumsensu stricto、F.vorosii、F.boothii、F.acaciae-mearnsii、F.mesoamericanum、F.austroamericanum、F.brasilicum、F.cortaderiae、F.meridionale、F.aethiopicum、F.louisianense、F.nepalense、Fusariumsp.、F.ussurianum、F.gerlachii)的Tri101和EF-1α基因序列,另外选取F.culmorum和F.pseudograminearum作为参考种,共获得18个种40个菌株(来自NRRL)的Tri101和EF-1α基因序列。将所有序列多重比对后对齐,去掉两端引物和多余部分碱基,用于建树的Tri101和EF-1α基因序列片段为998 bp和598 bp。用MEGA 6.06软件进行Tri101和EF-1α基因部分序列的系统发育谱系分析,结果表明,所研究的48个FGSC菌株中有11个菌株为F.asiaticum(基于Tri101和EF-1α的Bootstrap为96%),37个菌株为F.graminearumsensu stricto(基于Tri101和EF-1α的Bootstrap为97%)(图1)。
2.2 FGSC菌株的PCR-RFLP鉴定
PCR产物酶切之后,用2%的琼脂糖凝胶电泳进行检测,结果表明,用StyⅠ酶切,有11个FGSC菌株的电泳条带与其他菌株相比较小,为F.asiaticum菌株(图2A);用EcoRⅤ酶切,有37个FGSC菌株的电泳条带与其他相比较小,为F.graminearumsensu stricto菌株(图2B)。该鉴定结果与系统发育谱系分析的结果一致。
2.3 FGSC菌株的多重PCR鉴定
多重PCR产物用1.5%的琼脂糖凝胶电泳进行检测,结果表明,有11个FGSC菌株出现292 bp的电泳条带,为F.asiaticum;有37个FGSC菌株出现352 bp的电泳条带,为F.graminearumsensu stricto(图3)。该方法对48个FGSC菌株的鉴定结果与上述2种方法的结果一致。
3 结论与讨论
将FGSC准确鉴定到种是研究其相关病害的基础。目前,常用的是形态鉴定结合分子鉴定的方法。近年来,利用特异性PCR、特征DNA序列等分子技术辅助鉴别FGSC的方法已经被普遍应用[20-22]。Geiser等[23]建立了基于TEF-1α基因部分序列的镰刀菌数据库,根据TEF-1α基因部分序列进行系统发育谱系分析,将FGSC鉴定到种。本研究中,通过比对测序菌株和GenBank数据库中已报道的FGSC 16个种代表性菌株的Tri101和EF-1α基因部分序列,进行系统发育谱系分析,结果表明,有11个FGSC菌株与F.asiaticum聚在一起成为一个分枝,37个FGSC菌株与F.graminearumsensu stricto聚在一起,但这种分子鉴定FGSC的方法繁琐、复杂。有些FGSC具有地理分布特征,单独基于Tri101或者EF-1α基因部分序列进行的系统发育谱系分析,并不能准确鉴定到种,故需要将二者的基因序列拼接之后进行系统发育谱系分析,其结果更准确可靠。系统发育谱系分析方法的特点是准确、利于新发现种的鉴定,特别是对自然界中FGSC内不同种之间通过基因渗入的杂交新种或者基因内重组出现新种的鉴定,对发现新种有重要的意义,但成本较高。
Suga等[14]报道了PCR-RFLP,该法是基于组蛋白H3基因的特殊酶切位点设计特异引物,第一步PCR反应产物片段大小为223 bp,用内切酶StyⅠ酶切后只有F.asiaticum菌株出现195 bp的条带,用EcoRⅤ酶切后只有F.graminearumsensu stricto菌株出现191 bp的条带,将相差28 bp或者32 bp的条带通过电泳显示出来,需要2%或者浓度更高的琼脂糖凝胶。该法对电泳过程有很高的要求,此外PCR反应产物中含有的Buffer、dNTP、镁离子等也可能会影响酶切效率。
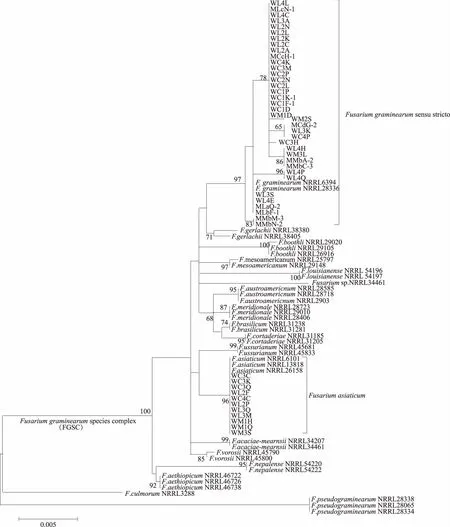
Bootstrap值(%,基于1 000次重复)≥60时显示在系统发育树分支上;比例尺显示了每个位点的核苷酸差异数
Ward等[9]为了对FGSC同时进行种和毒素型的鉴定,设计了利用48对引物的多重PCR,该方法的优点是对已明确的FGSC种进行鉴定,但操作过程繁琐复杂,第一步PCR反应过程需要高保真Taq酶降低碱基错配率,PCR产物经过特殊的仪器设备纯化后作为模板,再利用种和毒素型引物进行等位基因特异性延伸反应,从而完成种的鉴定。Yun等[15]研究报道,基于CYP51A和β-tubulin基因设计了F.asiaticum和F.graminearumsensu stricto的特异引物FaF/FaR、FgF/FgR和FgaF/FgaR(内参引物),用来同时定性和定量检测小麦中的FGSC。本研究利用特异引物FaF/FaR和FgF/FgR进行多重PCR,可将FGSC准确鉴定到种,此方法简单、灵敏、错误率低,电泳检测结果一目了然,但只能用来鉴定F.graminearumsensu stricto和F.asiaticum这2个种,不能对其他14个种或新发现种进行鉴定。
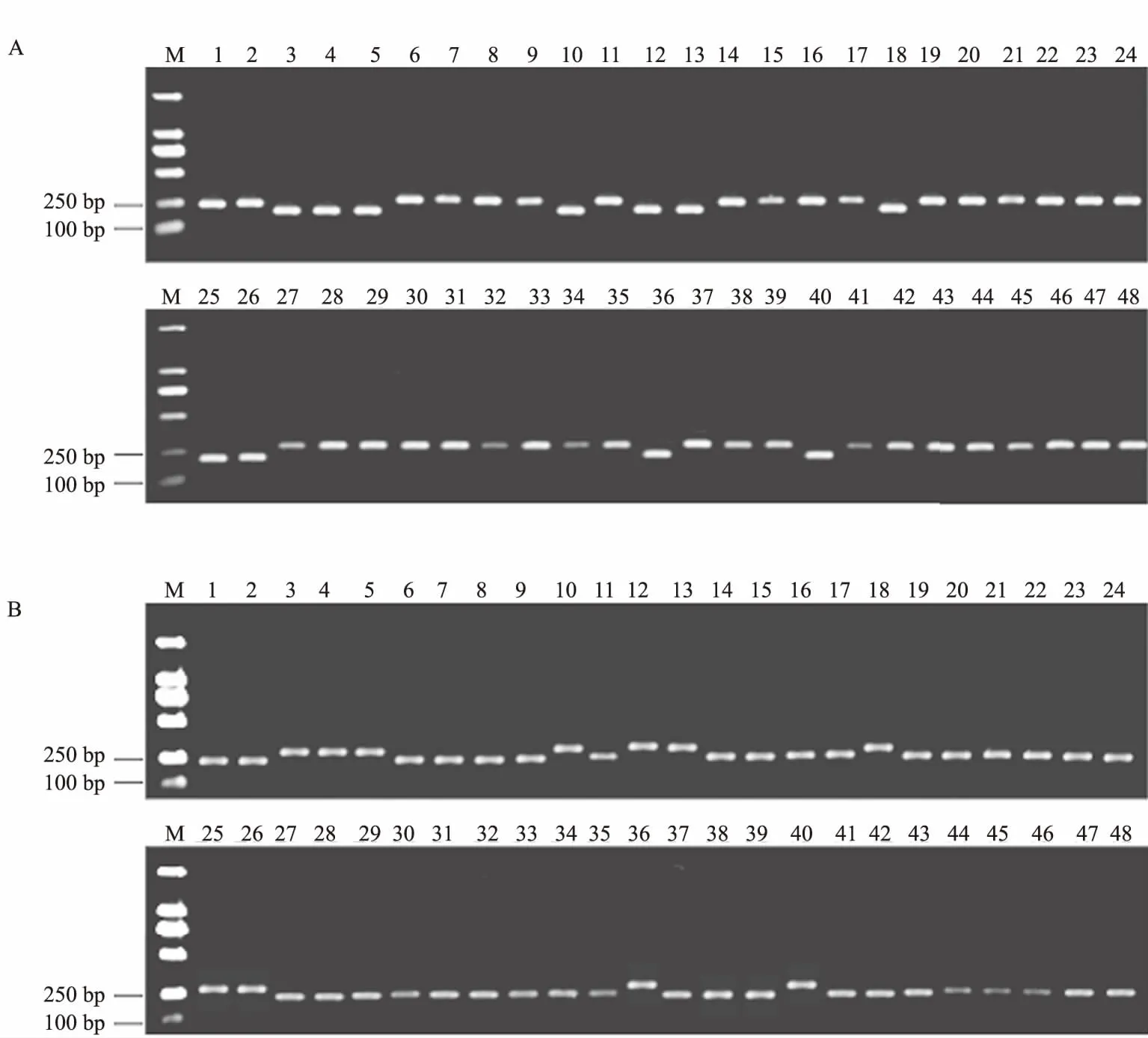
A.StyⅠ酶切; B.EcoRⅤ酶切。M为DL2000 Marker; 3、4、5、10、12、13、18、25、26、36和40为F.asiaticum菌株,其他为F.graminearum sensu stricto菌株,下同

图3 多重PCR鉴定FGSC的电泳检测结果
通过对48个FGSC菌株的鉴定发现,以上3种分子鉴定F.graminearumsensu stricto和F.asiaticum方法的结果一致,其中系统发育谱系分析步骤繁琐、费用高,PCR-RFLP对操作过程要求高且工作量大,而多重PCR快速、准确、高效,但其可能只适合对FGSC引起的中国小麦或玉米相关病害的鉴定,因为F.graminearumsensu stricto和F.asiaticum是中国作物上FGSC的主要成员。
本研究比较了鉴定FGSC的分子方法,发现多重PCR的方法适用于室内大量鉴定F.graminearumsensu stricto和F.asiaticum的工作,其对象包括FGSC菌株、籽粒和植株等,可为中国FGSC相关病害的进一步研究奠定基础。
[1] Miller J D,Greenhalgh R,Wang Y,etal.Trichothecene chemotypes of threeFusariumspecies [J].Mycologia,1991,83(2):121-130.
[2] 孙静,刘佳中,谢淑娜,等.小麦—玉米轮作田镰孢菌的种群结构及其致病性研究[J].河南农业科学,2015,44(5):91-96.
[3] 杨继芝,王继师,龚国淑,等.不同禾谷镰刀菌对小麦产量及其主要性状的影响[J].河南农业科学,2010(9):91-95.
[4] O’Donnell K,Ward T J,Aberra D,etal.Multilocus genotyping and molecular phylogenetics resolve a novel head blight pathogen within theFusariumgraminearumspecies complex from Ethiopia[J].Fungal Genetics and Biology,2008,45(11):1514-1522.
[5] O’Donnell K,Ward T J,Geiser D M,etal.Genealogical concordance between the mating type locus and seven other nuclear genes supports formal recognition of nine phylogenetically distinct species within theFusariumgraminearumclade[J].Fungal Genetics and Biology,2004,41(6):600-623.
[6] Sarver B A,Ward T J,Gale L R,etal.NovelFusariumhead blight pathogens from Nepal and Louisiana revealed by multilocus genealogical concordance[J].Fungal Genetics and Biology,2011,48(12):1096-1107.
[7] Starkey D E,Ward T J,Aoki T,etal.Global molecular surveillance reveals novelFusariumhead blight species and trichothecene toxin diversity[J].Fungal Genetics and Biology,2007,44(11):1191-1204.
[8] O’Donnell K,Kistler H C,Tacke B K,etal.Gene genealogies reveal global phylogeographic structure and reproductive isolation among lineages ofFusariumgraminearum,the fungus causing wheat scab[J].Proc Nat Acad Sci,2000,97(14):7905-7910.
[9] Ward T J,Clear R M,Rooney A P,etal.An adaptive evolutionary shift inFusariumhead blight pathogen populations is driving the rapid spread of more toxigenicFusariumgraminearumin North America[J].Fungal Genetics and Biology,2008,45(4):473-484.
[10] Davari M,van Diepeningen A D,Babai-Ahari A,etal.Rapid identification ofFusariumgraminearumspecies complex using rolling circle amplification (RCA) [J].Journal of Microbiological Methods,2012,89(1):63-70.
[11] Fernández-Ortuo D,Waalwijk C,Van der Lee T,etal.Simultaneous real-time PCR detection ofFusariumasiaticum,F.ussurianumandF.vorosii,representing the Asian clade of theF.graminearumspecies complex[J].International Journal of Food Microbiology,2013,166(1):148-154.
[12] Tamura K,Stecher G,Peterson D,etal.MEGA6:Molecular evolutionary genetics analysis version 6.0[J].Mol Biol Evol,2013,30(12):2725-2729.
[13] Boutigny A L,Ward T J,Van Coller G J,etal.Analysis of theFusariumgraminearumspecies complex from wheat,barley,and maize in South Africa provides evidence of species-specific differences in host preference[J].Fungal Genetics and Biology,2011,48(9):914-920.
[14] Suga H,Karugia G W,Ward T,etal.Molecular characterization of theFusariumgraminearumspecies complex in Japan [J].Phytopathology,2008,98(2):159-166.
[15] Yin Y,Liu X,Ma Z.Simultaneous detection ofFusariumasiaticumandFusariumgraminearumin wheat seeds using a real-time PCR method [J].Letters in Applied Microbiology,2009,48(6):680-686.
[16] 孙静,谢淑娜,刘佳中,等.河南省玉米茎基部镰刀菌的形态和分子鉴定[J].植物病理学报,2014,44(1):8-16.
[17] 吴全安,梁克恭,朱小阳,等.北京和浙江地区青枯病病原菌的分离和鉴定[J].中国农业科学,1989,22(5):71-75.
[18] Proctor R H,McCormick S P,Alexander N J,etal.Evidence that a secondary metabolic biosynthetic gene cluster has grown by gene relocation during evolution of the filamentous fungus Fusarium[J].Mol Microbiol,2009,74(5):1128-1142.
[19] O’Donnell K,Kistler H C,Cigelink E,etal.Multiple evolutionary origins of the fungus causing Panama disease of banana:Concordant evidence from nuclear and mitochondrial gene genealogies[J].Proc Natl Acad Sci USA,1998,95(5):2044-2049.
[20] Reischer G H,Lemmens M,Farnleitner A,etal.Quantification ofFusariumgraminearumin infected wheat by species specific real-time PCR applying a TaqMan probe [J].J Microbiol Methods,2004,59(1):141-146.
[21] Rahjoo V,Zad J,Javan-Nikkhah M,etal.Morphological and molecular identification ofFusariumisolated from maize ears in Iran[J].Journal of Plant Pathology,2008,90(3):463-468.
[22] Hsuan H M,Salleh B,Zakaria L.Molecular identification ofFusariumspecies inGibberellafujikuroispecies complex from rice,sugarcane and maize from Peninsular Malaysia[J].International Journal of Molecular Sciences,2011,12(10):6722-6732.
[23] Geiser D M,Jimenez-Gasco M M,Kang S,etal.FUSARIUM-ID v.1.0:A DNA sequence database for identifyingFusarium[J].European Journal of Plant Pathology,2004,110(5):473-479.
Comparison of Three Molecular Identification Methods for Fusarium graminearum sensu stricto and F.asiaticum
RU Yanyan,XIE Shuna,LI Baoye,SUN Jing,LIU Jiazhong,HAO Junjie*
(Plant Protection Institute,Henan Academy of Agricultural Sciences/Key Laboratory of Integrated Pest Management on Crops in Southern Region of North China/Henan Key Laboratory of Crop Pest Control,Zhengzhou 450002,China)
Fusariumgraminearumsensu stricto andF.asiaticumare major members ofFusariumgraminearumspecies complex(FGSC) in China.In this study,to improve the identification efficiency of these two species,48 members of FGSC collected from plants showingFusariumhead blight (FHB) or maize stalk rot(MSR) were identified using three molecular identification methods.One method was amplification of partial sequences of two genesEF-1αandTri101,and then sequencing and phylogenetic analyses by MEGA 6.06 software.The results showed that 11 isolates were clustered together withF.asiaticumand 37 isolates were clustered together withF.graminearumsensu stricto.This method was cumbersome,complex and costly,but the identification results were accurate.A second method was polymerase chain reaction-restriction fragment length polymorphism(PCR-RFLP).Based on the specific restriction sites of histone H3 gene,primers H3dStyⅠ and H3R1 were designed.11 isolates were identified asF.asiaticumand 37 isolates wereF.graminearumsensu stricto.This method required lots of work and high operation process.The third method was a multiplex PCR.Based on theCYP51Agene,primers FaF/FaR and FgF/FgR were designed.11 isolates were identified asF.asiaticumand 37 isolates wereF.graminearumsensu stricto.This method was simple and efficient.These results were similar,while the multiplex PCR method was more rapid,accurate and efficient.
Fusariumgraminearumspecies complex(FGSC);Fusariumgraminearumsensu stricto;Fusariumasiaticum; molecular identification
S432.4
A
1004-3268(2017)11-0080-07
2017-04-13
河南省农业科学院科研发展专项资金项目(201412008,豫财预[2017]76-11);河南省农业科学院自主创新专项基金项目(2017ZC38)
茹艳艳(1986-),女,河南洛阳人,助理研究员,博士,主要从事玉米病害研究。E-mail:ruyanbingyu@163.com
*通讯作者:郝俊杰(1978-),男,河南登封人,副研究员,博士,主要从事玉米病害研究。E-mail:haojjds@163.com
