基于COⅠ序列绒螯蟹属DNA条形码和遗传多样性研究
2017-12-18王晓梅李晶晶李转转张玲颖
徐 洁,王晓梅,李晶晶,陈 池,李转转,张玲颖
( 1. 天津农学院 水产学院,天津市水产生态及养殖重点实验室,天津 300384;2.天津市水生动物疫病预防控制中心,天津 300402 )
基于COⅠ序列绒螯蟹属DNA条形码和遗传多样性研究
徐 洁1,王晓梅1,李晶晶2,陈 池1,李转转1,张玲颖1
( 1. 天津农学院 水产学院,天津市水产生态及养殖重点实验室,天津 300384;2.天津市水生动物疫病预防控制中心,天津 300402 )
对绒螯蟹属的中华绒螯蟹如东和七里海群体、日本绒螯蟹、合浦绒螯蟹及狭颚绒螯蟹共80条线粒体COⅠ片段进行扩增和测序,并与GenBank中绒螯蟹属的台湾绒螯蟹2条和近方蟹属的绒毛近方蟹19条COⅠ 基因序列进行联配分析。结果显示,101条序列包含44种单倍型,序列组成表现明显的碱基偏倚性。中华绒螯蟹如东、七里海群体与日本绒螯蟹间的遗传距离分别为1.210%和1.078%,明显低于COⅠ基因DNA条形码鉴别种的遗传距离为2%的阈值,表明中华绒螯蟹和日本绒螯蟹为同一物种;而合浦绒螯蟹与中华绒螯蟹如东和七里海群体及与日本绒螯蟹的遗传距离分别为4.823%、5.101%以及5.011%,明显大于2%的鉴别阈值,说明合浦绒螯蟹为独立的种。以绒毛近方蟹为外群,基于群体内及群体间的遗传距离构建的邻接树显示,中华绒螯蟹与日本绒螯蟹聚在一起,合浦绒螯蟹则聚成单系。本文测序的5个群体除狭颚绒螯蟹外,其余均具有遗传多样性,单倍型多样性为0.593±0.144~0.779±0.068,核苷酸多样性为0.00156~0.01336;此外,中华绒螯蟹如东群体与日本绒螯蟹、合浦绒螯蟹和中华绒螯蟹七里海群体分别共享单倍型H1、H2和H3,说明这些蟹类可能有种质资源混杂或是遗传污染的现象。
绒螯蟹;COⅠ基因;DNA条形码;物种鉴定;遗传多样性
绒螯蟹属(Eriocheir)隶属于节肢动物门、甲壳动物亚门、软甲纲、十足目、方蟹科。对于绒螯蟹属内种的分类一直存在争议[1-6],但学者们较为公认的观点是该属包含5个物种,即中华绒螯蟹(E.sinensis)、日本绒螯蟹(E.japonica)、合浦绒螯蟹(E.hepuensis)、狭颚绒螯蟹(E.leptognathus)和台湾绒螯蟹(E.formosa)[7-10]。这些蟹类不仅形态特征有差异,地理分布各不相同,其经济价值也有区别;其中以中华绒螯蟹的经济价值最高,日本绒螯蟹与合浦绒螯蟹次之,这三种蟹俗称均为河蟹[10-11]。自20世纪70年代起,全国的河蟹养殖业蓬勃发展,河蟹的天然苗种不能满足人工养殖的需求,需要从各地引进河蟹苗种,从而导致种质资源混杂和遗传污染。
DNA条形码是利用一段种内较为保守但种间存在明显差异的DNA序列来鉴定物种的技术,现已成为物种鉴定的新兴技术。由于线粒体COⅠ基因进化速度较核DNA快,与其他基因序列如线粒体12S rRNA和16S rRNA 相比,很少发生缺失和插入,因此加拿大圭尔夫大学的Hebert等[12]极力主张将COⅠ基因作为动物物种鉴定的DNA条形码,并于2003年首次提出将一段短的COⅠ基因序列作为动物物种鉴定的通用条形码。COⅠ基因作为DNA条形码在水产动物的物种鉴定和遗传多样性分析上得到广泛应用[13-20],但对绒螯蟹的研究报道甚少[21]。
本文通过分析绒螯蟹样本线粒体COⅠ基因序列,探讨应用DNA条形码技术对绒螯蟹属物种作进一步鉴定,并分析绒螯蟹样本的遗传多样性,以期积累绒螯蟹DNA条形码数据,为了解绒螯蟹种质资源的变化以及为绒螯蟹种质资源的保护和开发与利用提供科学依据。
1 材料与方法
1.1 材料
试验所用的绒螯蟹样本除中华绒螯蟹七里海群体为天津市水生动物疫病预防控制中心于天津市蟹源水产养殖有限公司培育的第6代繁育群体外,其余样本均为本实验室采集的自然群体(表1为绒螯蟹样本的采集信息)。样本采集后,取附肢于95%的乙醇固定、4 ℃保存备用。

表1 绒螯蟹样本采集信息
1.2 DNA的提取、PCR扩增及PCR扩增产物的测序
依据文献[22]的方法略作改进,提取样本附肢肌肉组织的总体DNA。扩增线粒体COⅠ基因片段的引物序列为:COⅠ-L(5′-GGTCAACAAATCATAAAGATATTGG-3′)和COⅠ-H(5′-TAAACTTCAGGGTGACCAAAAAATCA-3′)。引物由生工生物工程(上海)股份有限公司合成。PCR反应体系为25 μL,包括1×PCR buffer,2 mmol/L Mg2+,200 μmol/L dNTP,引物各0.4 μmol/L,Taq酶1.5 U,50~100 ng模板DNA。PCR反应条件为:94 ℃预变性5 min;94 ℃ 1 min,55 ℃ 1 min,72 ℃ 1.5 min,共35个循环;最后72 ℃延伸10 min。
PCR扩增产物经1.5%的琼脂糖凝胶电泳检测,扩增良好的产物经纯化后双向测序。纯化、测序由生工生物工程(上海)股份有限公司完成。本研究测序的COⅠ基因片段序列经GenBank的Blast在线比对(http://blast. ncbi.nlm.nih.gov/Blast.cgi),确认试验所得序列为目的片段。
1.3 数据分析
自GenBank下载COⅠ基因片段长度等于或超过658 bp的方蟹科、近方蟹属的绒毛近方蟹 (Hemigrapsuspenicillatus)19条序列和绒螯蟹属的台湾绒螯蟹的2条序列,与本研究测得的序列进行联配分析。
利用Clustal X 1.81软件对所有序列进行人工校正、序列对齐后,截取658 bp的序列进一步分析。应用DnaSP 5.1软件分析群体的核苷酸多样性指数、单倍型多样性指数,多态位点、单突变位点和简约信息位点;应用MEGA 4.1软件分析序列的碱基组成、碱基的转换与颠换比(R值)、群体内及群体间的遗传距离(Kimura 2-parameter距离),并依据群体内及群体间遗传距离、以绒毛近方蟹为外群,采用邻接法构建分子系统树,系统树分支的置信度采用自展法,重复检测1000次。
2 结果与分析
2.1 线粒体COⅠ基因的序列特征
本研究获得绒螯蟹属5个群体共80个个体的COⅠ基因序列,去掉引物后长度为658 bp,与GenBank下载的COⅠ序列(21条)进行联配分析,101条序列未见插入和缺失现象。其碱基组成及位点变异分析表明所有序列均表现出明显的碱基偏倚性,即CG含量(33.43%~38.22%)明显低于AT含量(61.78%~66.57%);除狭颚绒螯蟹外,所有群体的COⅠ序列均具有变异位点,且碱基转换多于颠换(表2)。
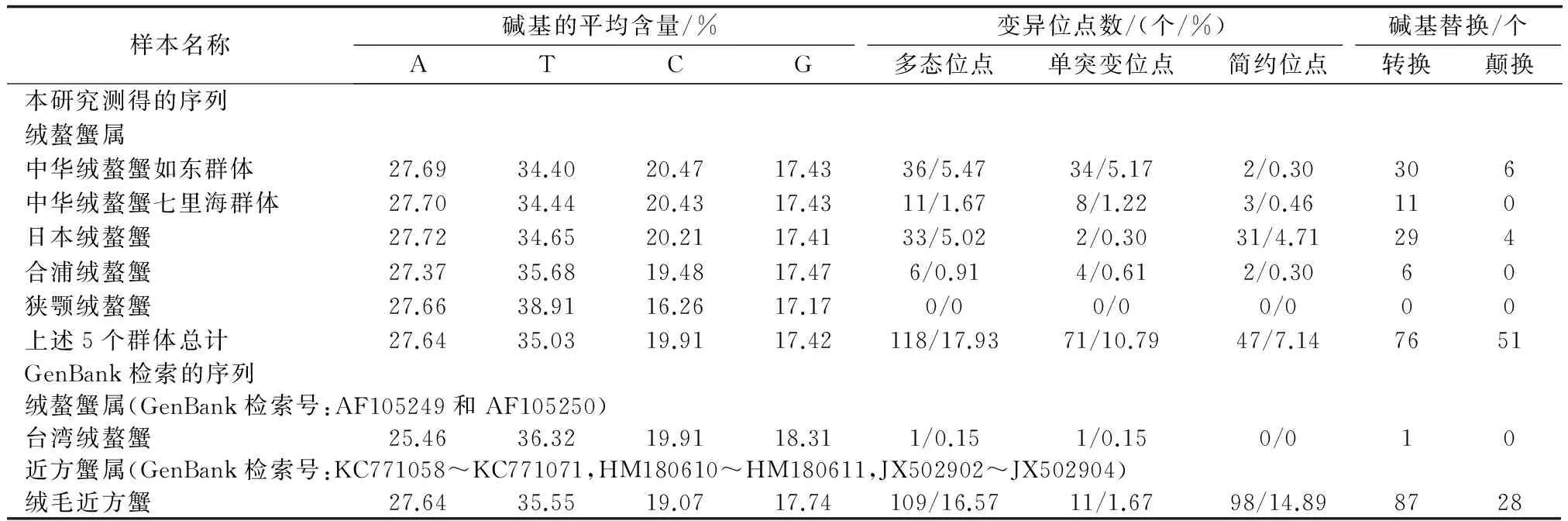
表2 绒螯蟹和绒毛近方蟹COⅠ序列碱基组成及变异信息
2.2 单倍型分布及遗传多样性分析
本研究测序的绒螯蟹属5个群体80条COⅠ序列中检测到23种单倍型,既有群体特有的单倍型,也存在群体共享的单倍型(表3)。其中,中华绒螯蟹如东群体与日本绒螯蟹共享单倍型H1(共5个个体,占2个群体总数的14.3%),与合浦绒螯蟹共享单倍型H2(共10个个体,占2个群体总数的27.8%),与中华绒螯蟹七里海群体共享单倍型H3(共8个个体,占2个群体总数的17.8%)。GenBank下载的21条COⅠ序列中检测到21种单倍型。每个群体单倍型种类、各种单倍型所包含的个体数及在群体中的比例见表3。
所有群体的遗传多样性分析结果显示(表3),狭颚绒螯蟹未检测到遗传多样性,其余6个群体的单倍型多样性指数为0.593±0.144~1.000±0.500,核苷酸多样性指数为0.00152~0.04096。除狭颚绒螯蟹和台湾绒螯蟹外,其他5个群体的Tajimas D中性检验范围为-2.34216~-0.65889,除中华绒螯蟹如东群体外,其余4个群体的中性检验结果均不显著(P>0.10),符合分子水平的突变为中性突变。
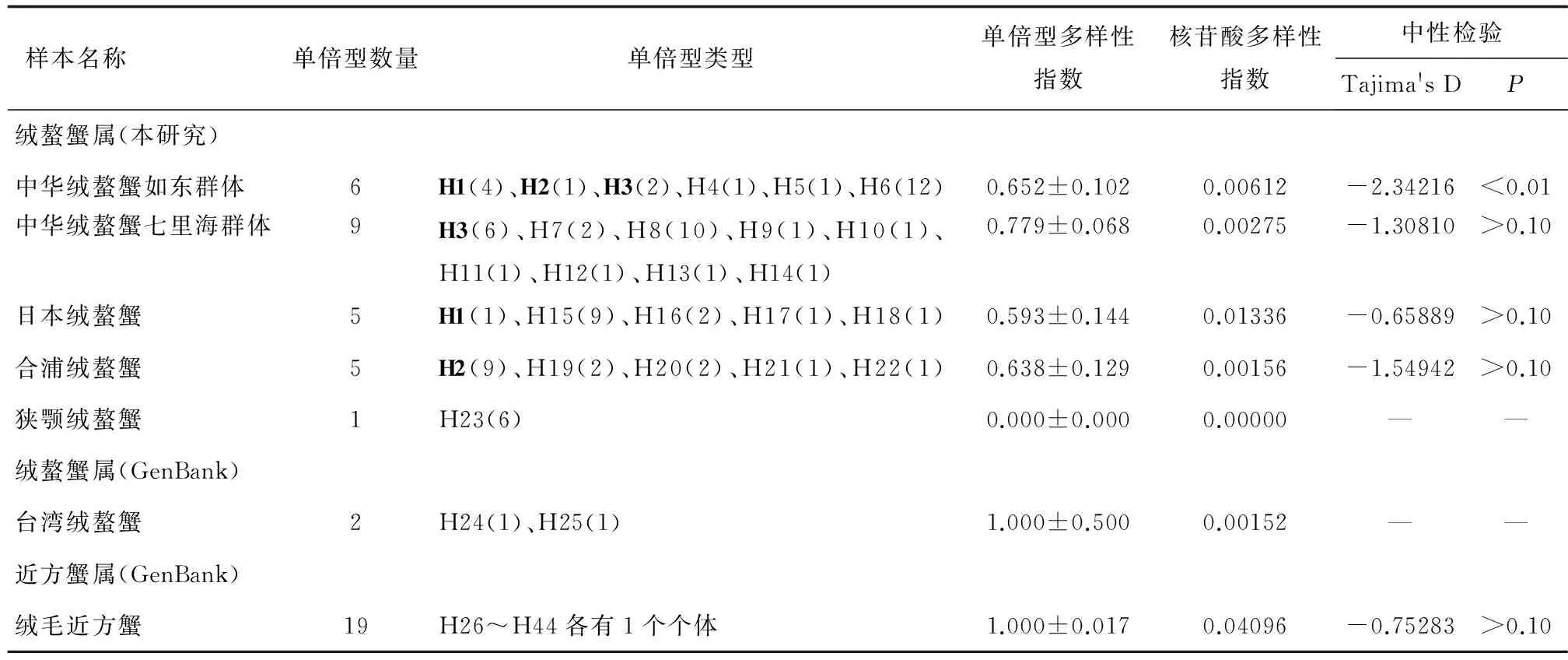
表3 44个单倍型在7个群体中的分布及序列多样性
注:括号内数字表示单倍型的样本个体数,加粗字体表示共享单倍型;GenBank下载序列的检索号见表2.
2.3 遗传距离、R值与分子系统树
应用MEGA软件得出群体内或种内的遗传距离为:中华绒螯蟹如东群体和七里海群体分别为0.000%~5.408%和0.000%~0.766%,日本绒螯蟹为0.000%~4.920%,合浦绒螯蟹为0.000%~0.612%,台湾绒螯蟹为0.152%,绒毛近方蟹为0.152%~16.594%。其平均值以及群体间或种间的遗传距离的平均值和COⅠ序列碱基转换与颠换的比值(R值)见表4。
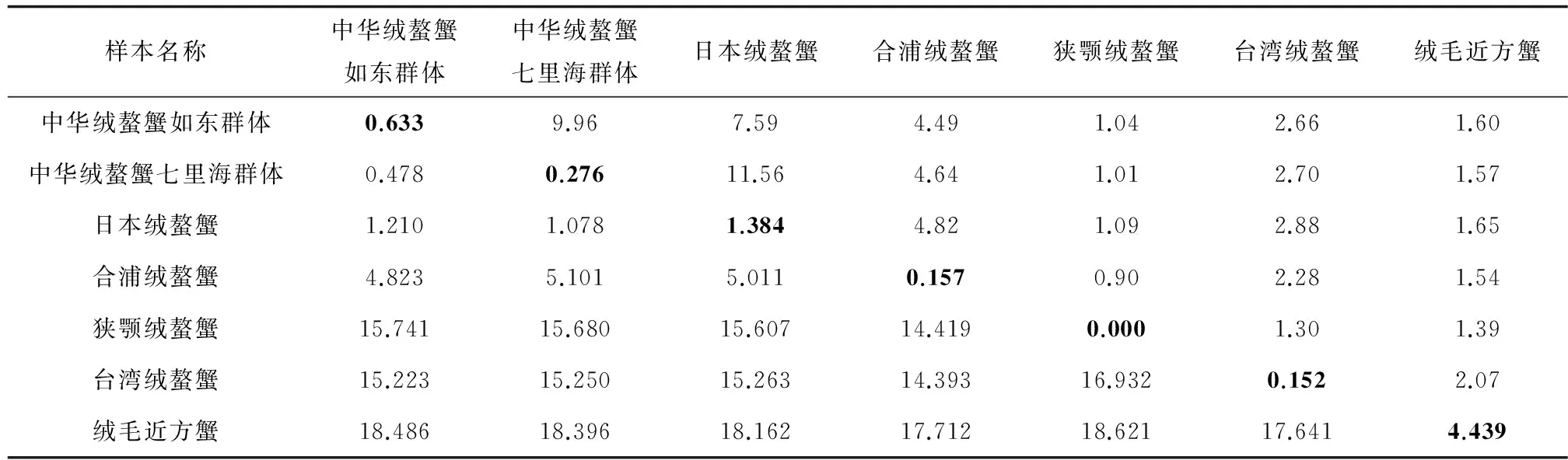
表4 绒螯蟹属和近方蟹属不同分类阶元的平均遗传距离和R值
注:对角线处加粗数字为种群内或种内平均遗传距离(%);对角线下为种群间或种间平均遗传距离(%);对角线上为COⅠ序列的R值.
由表4可知,基于COⅠ序列得出绒螯蟹属群体内或种内的遗传距离为0.000%~1.384%,均小于2%;绒螯蟹属不同群体间或种间的遗传距离明显变大,为0.478%~16.932%;绒螯蟹属6个群体与近方蟹属绒毛近方蟹的遗传距离为17.641%~18.621%。
以绒毛近方蟹为外群,依据群体内及群体间遗传距离,分别构建44种单倍型和群体的邻接系统树(图1和图2)。构建群体邻接树时,本研究测序的5个群体选取所含个体数最多的单倍型序列、而GenBank下载的台湾绒螯蟹和绒毛近方蟹则各任选1条序列。由图1可见,44种单倍型可分为两大支系,一个支系为近方蟹属的绒毛近方蟹,另一支系由绒螯蟹属的6个群体构成;在绒螯蟹属支系中,合浦绒螯蟹、狭颚绒螯蟹和台湾绒螯蟹又各自形成单系,但中华绒螯蟹如东群体、七里海群体和日本绒螯蟹没能各自形成单系。本文分析的7个群体的亲缘关系由近至远的顺序是:中华绒螯蟹如东群体和七里海群体的亲缘关系最近,之后是与日本绒螯蟹、合浦绒螯蟹、狭颚绒螯蟹、台湾绒螯蟹和绒毛近方蟹(图2)。
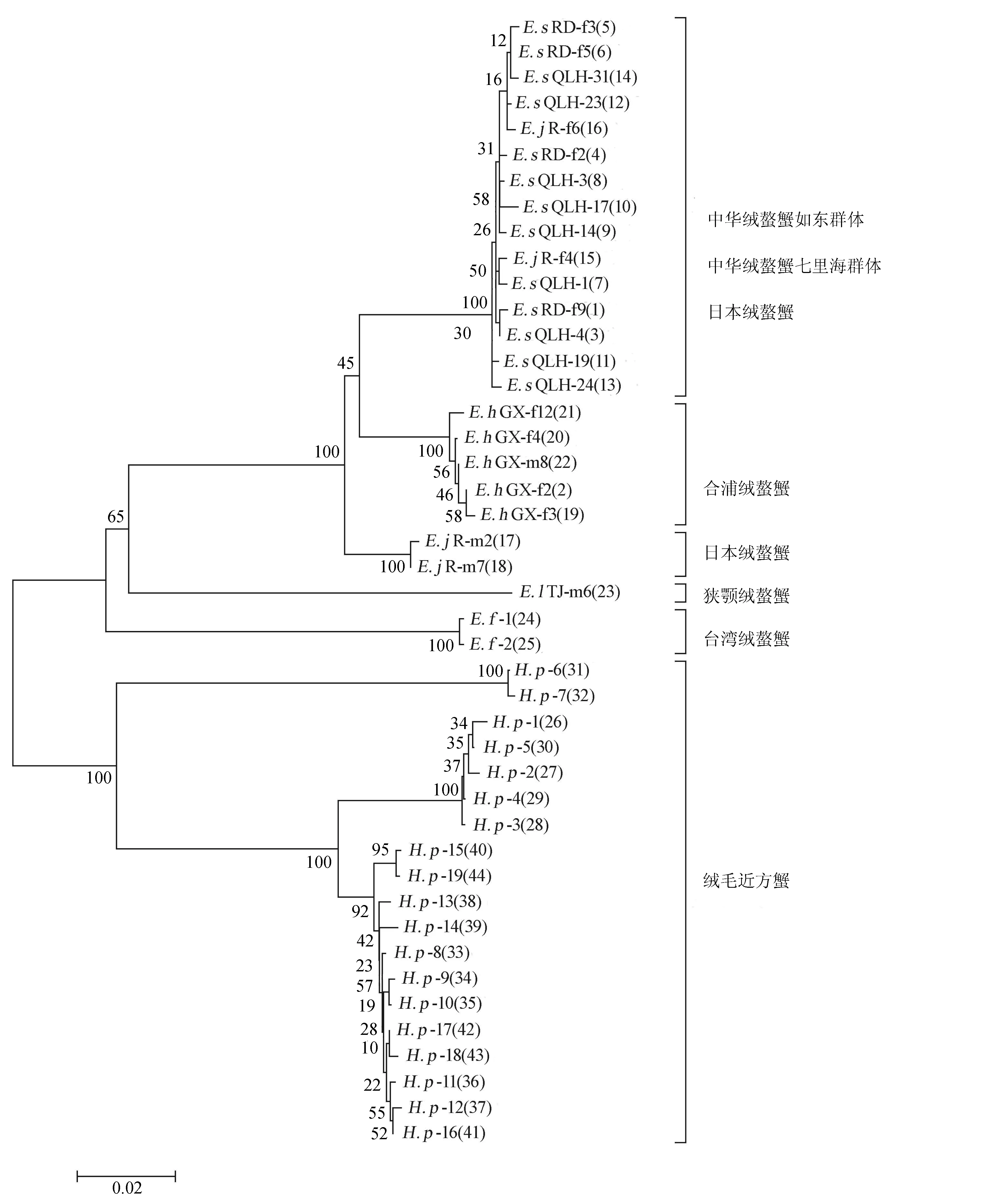
图1 基于群体内及群体间遗传距离构建的 COⅠ基因单倍型的邻接树样本名称中“-”后面的字母f表示雌性、m表示雄性,样本名称中的阿拉伯数字代表样本号,括号内的数字表示单倍型的种类.
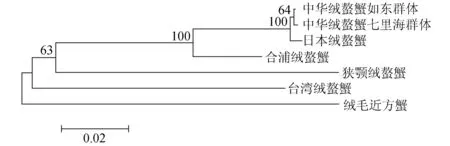
图2 基于群体内及群体间遗传距离建立的7个群体的邻接树
3 讨 论
3.1 序列分析
COⅠ基因是线粒体DNA编码细胞色素c氧化酶亚单位Ⅰ的基因,在蛋白质编码区,很少发生插入和缺失。本研究测序的5个绒螯蟹群体80个个体以及GenBank下载的台湾绒螯蟹2个个体和绒毛近方蟹19个个体的线粒体COⅠ基因序列(658 bp)未发现碱基的插入与缺失。本研究分析的7个群体101条序列,除狭颚绒螯蟹外,均存在变异位点,因台湾绒螯蟹只有2条序列所以仅有单突变位点,其余序列既有单突变位点又有简约位点。在碱基替换上,中华绒螯蟹七里海群体、合浦绒螯蟹和台湾绒螯蟹线粒体COⅠ序列不存在碱基颠换,只存在转换;而中华绒螯蟹如东群体、日本绒螯蟹和绒毛近方蟹线粒体COⅠ序列既有碱基转换又有颠换。在碱基组成上,所有序列均是T含量最高,除狭颚绒螯蟹外,G含量最低;AT含量明显高于GC含量,该结果与COⅠ基因碱基组成中普遍存在的AT含量高于GC含量的现象一致[18,23-24]。
3.2 绒螯蟹属物种的有效性
长期以来,绒螯蟹属内种及亚种的分类存在着较大的分歧。在日本绒螯蟹与中华绒螯蟹的分类上,Li等[25]应用同工酶技术及形态学分析,认为中华绒螯蟹是日本绒螯蟹的不同生态表型群体,应一并称之为日本绒螯蟹;戴爱云[2]通过形态学分析认为日本绒螯蟹可能是中华绒螯蟹的一个亚种;孔晓瑜等[4]的研究结果显示中华绒螯蟹与日本绒螯蟹COⅠ序列间差异较大,倾向于支持存在中华绒螯蟹和日本绒螯蟹两个种或它们为同一种的两个地理亚种的观点;张列士等[26]通过比较日本绒螯蟹与我国各水系中华绒螯蟹的形态特征发现日本绒螯蟹是具有独立形态的种;孙红英等[5]通过对线粒体16S rDNA序列进行分析,认为中华绒螯蟹、合浦绒螯蟹和日本绒螯蟹属于同一个物种E.japonica;王茜等[27]通过形态学分析认为中华绒螯蟹与日本绒螯蟹的差异应在亚种以上水平。在合浦绒螯蟹的分类上,戴爱云[28]将合浦绒螯蟹定名为日本绒螯蟹合浦亚种;Guo等[1]的研究结果表明合浦绒螯蟹是一个独立的物种;堵南山[7]认为日本绒螯蟹合浦亚种很可能是日本绒螯蟹在南方地区的一个种群;谢浩等[3]采用RAPD技术对3种绒螯蟹的亲缘关系进行研究,结果表明合浦绒螯蟹与日本绒螯蟹、中华绒螯蟹之间差异显著,认为合浦绒螯蟹形态分类地位更接近日本绒螯蟹;李孟仙等[9]通过形态学研究认为合浦绒螯蟹是绒螯蟹属一个独立的物种,并将其学名由“日本绒螯蟹合浦亚种”改为“合浦绒螯蟹”。
Costa等[21]对甲壳纲150个物种的DNA条形码(基于线粒体COⅠ序列)的分析结果表明,基于Kimura双参数模型计算的甲壳纲十足目动物种内遗传距离为0.00%~2.57%(平均值为0.46%),86%的物种种内遗传距离低于1%;种间遗传距离为4.92%[雪蟹属(Chionoecete)]~31.39%[端足类钩虾属(Gammarus)],平均为17.16%;属间的遗传距离为11.27%~49.93%,平均为19.75%。
本研究通过DNA条形码分析显示,绒螯蟹属的中华绒螯蟹如东群体和七里海群体、日本绒螯蟹、合浦绒螯蟹、狭颚绒螯蟹和台湾绒螯蟹种内平均遗传距离分别为0.633%、0.276%、1.384%、0.157%、0.000%和0.152%。中华绒螯蟹如东群体与七里海群体间的平均遗传距离为0.478%,与Costa等[21]对十足目动物种内遗传距离的研究结果基本相符。日本绒螯蟹与中华绒螯蟹如东群体、七里海群体间的平均遗传距离分别为1.210%、1.078%,小于日本绒螯蟹群体内的遗传距离(1.384%),均小于Hebert等[29]认为以线粒体COⅠ基因为DNA条形码的最小种间遗传距离为2%的阈值;故本研究结果支持日本绒螯蟹与中华绒螯蟹同属于一个物种的观点。而合浦绒螯蟹与中华绒螯蟹如东群体、七里海群体和日本绒螯蟹间的平均遗传距离分别为4.823%、5.101%、5.011%,明显高于2%的阈值;因此,本研究支持合浦绒螯蟹为绒螯蟹属一个独立物种的观点。此外,本研究中,绒螯蟹属内群体间的遗传距离最小为0.478%(中华绒螯蟹如东群体与七里海群体),最大为16.932%(狭颚绒螯蟹与台湾绒螯蟹);而绒螯蟹属与近方蟹属间的遗传距离最小为17.641%(台湾绒螯蟹与绒毛近方蟹),最大为18.621%(狭颚绒螯蟹与绒毛近方蟹)。该结果与Costa等[21]对十足目动物种内和种间遗传距离的研究结果相符合。
以近方蟹属的绒毛近方蟹为外群,基于群体内及群体间的遗传距离所构建的群体邻接树显示,中华绒螯蟹如东群体与中华绒螯蟹七里海群体的亲缘关系最近,之后依次是与日本绒螯蟹、合浦绒螯蟹、狭颚绒螯蟹、台湾绒螯蟹和绒毛近方蟹,绒螯蟹属中与绒毛近方蟹亲缘关系最近的物种是台湾绒螯蟹。44种单倍型的邻接树显示,绒螯蟹属的6个群体聚成了3大分支,其中狭颚绒螯蟹和台湾绒螯蟹各成一支;而中华绒螯蟹如东群体和七里海群体、日本绒螯蟹以及合浦绒螯蟹形成另一个分支,但在这一分支中,合浦绒鳌蟹又能够单独形成一支。这些结果表明,在分析的绒螯蟹样本中,中华绒螯蟹和日本绒螯蟹亲缘关系近,而与合浦绒螯蟹的亲缘关系较远。
在DNA进化过程中,碱基转换发生的频率要比颠换高得多,利用转换与颠换比值,即R值,可以对序列的饱和度进行分析,一般亲缘关系越近的分类单元之间核苷酸发生颠换的频率越低,表现为R值越大,由R值的大小可初步判断所研究的分类单元之间的亲缘关系[18]。本研究结果显示,中华绒螯蟹七里海群体与日本绒螯蟹间的R值最大,表明两者亲缘关系最近;但中华绒螯蟹如东群体与七里海群体间的R值(9.96)小于中华绒螯蟹七里海群体与日本绒螯蟹间的R值(11.56),可能与中华绒螯蟹如东群体存在物种混杂有关。理论上狭颚绒螯蟹与绒螯蟹其他群体间的R值应比绒毛近方蟹与绒螯蟹其他群体间的R值要大,但本研究结果与之相反,不符合R值越大亲缘关系越近这一规律,可能与狭颚绒螯蟹只具有一种序列单倍型有关,该问题今后将进一步研究。
3.3 绒螯蟹属物种的种质资源
群体的核苷酸多样性指数是衡量群体遗传多样性的重要指标,核苷酸多样性指数越大,群体的遗传多样性越高[30]。葛家春等[31]研究得出长江、辽河、瓯江及其莱茵河4水系中华绒螯蟹样本线粒体COⅠ序列的核苷酸多样性指数分别为0.0028、0.0028、0.0243和0.0017,样本总体的核苷酸多样性指数为0.0169,认为样本总体的遗传多样性指数较高。闫龙等[32]分析得出采自丹东、盘锦、垦利和南京的4个群体中华绒螯蟹样本的线粒体控制区的核苷酸多样性指数分别为0.014、0.016、0.019和0.010,样本总体的核苷酸多样性指数为0.017;也指出分析样本总体的遗传多样性指数较高。本研究测序的5个绒螯蟹群体的分析结果显示,除狭颚绒螯蟹群体无多样性外(核苷酸多样性指数为0.00000),日本绒螯蟹群体的多样性最大(核苷酸多样性指数为0.01336),中华绒螯蟹如东群体次之(核苷酸多样性指数为0.00612),再次是中华绒螯蟹七里海群体(核苷酸多样性指数为0.00275),合浦绒螯蟹的多样性最小(核苷酸多样性指数为0.00156)。说明本研究的两个中华绒螯蟹样本具有一定的遗传多样性,且如东群体的遗传多样性高于七里海群体。群体的核酸序列单倍型多样性指数是衡量群体遗传多样性的另一个指标。本研究中出现了单倍型多样性指数与核苷酸多样性指数不一致的现象,如日本绒螯蟹的单倍型多样性指数最小(0.593±0.144),而核苷酸多样性指数最大(0.01336);中华绒螯蟹七里海群体单倍型多样性指数最大(0.779±0.068),但核苷酸多样性指数较小(0.00275)。日本绒螯蟹的14个个体中多态位点数为33个、单倍型有5种,而七里海群体24个个体的序列多态位点为11个、单倍型有9种。杨帆等[17-18]的研究结果也显示出单倍型多样性和核苷酸多样性不一致的现象。
本研究所测序的5个绒螯蟹群体,单倍型H4~H6为中华绒螯蟹如东群体所特有(占66.67%),其中H6个体最多,占群体的57.14%;H7~H14为中华绒螯蟹七里海群体所特有(占75%),其中H8个体最多,占群体的41.67%;H15~H18为日本绒螯蟹所特有(占92.86%),其中H15个体最多,占群体的64.29%;H19~H22为合浦绒螯蟹所特有(占40%);H23为狭颚绒螯蟹所特有(占100%)。在上述5个群体同时存在单倍型共享现象,中华绒螯蟹如东群体和日本绒螯蟹共享单倍型H1,该单倍型在如东群体占19.05%,而在日本绒螯蟹中占7.14%;中华绒螯蟹如东群体和合浦绒螯蟹共享单倍型H2,该单倍型在如东群体占4.76%,而在合浦绒螯蟹占60%;中华绒螯蟹如东群体与七里海群体共享单倍型H3,该单倍型在如东群体占9.52%,而在七里海群体占25%。由此推测中华绒螯蟹、日本绒螯蟹和合浦绒螯蟹的种质资源存在混杂现象。邱高峰等[33]基于线粒体16S rDNA序列的研究结果表明,各个不同地域的中华绒螯蟹混杂在一起。孙红英等[34]基于线粒体16S rDNA的PCR-RFLP和线粒体Cyt b序列分析显示,长江水系中华绒螯蟹样品中检测出合浦亚种单元型,说明了中华绒螯蟹与合浦绒螯蟹存在种质资源混杂现象。葛家春等[31]基于线粒体COⅠ序列以及闫龙等[32]基于线粒体控制区的序列分析,也证明各水系中华绒螯蟹种质资源混杂确实存在。
由于绒螯蟹养殖的推广,长江蟹苗的管理难度越来越大,再加之不注意保护亲蟹资源,从各地引进蟹苗,导致长江水系中华绒螯蟹养殖遗传污染和种质资源混杂及退化[35-37]。因此应加强对长江水系中华绒螯蟹种质资源的保护,以保护中华绒螯蟹的遗传多样性。
[1] Guo J Y, Ng N K, Dai A Y, et al. The taxonomy of three commercially important species of mitten crab of the genusEriocheirDe Haan, 1835 (Crustacea; Decapoda; Brachyura; Grapsidae)[J]. The Raffles Bulletin of Zoology, 1997, 45(2):445-476.
[2] 戴爱云. 绒螯蟹属支序分类学的初步分析(甲壳总纲:十足目)[J]. 动物分类学报, 1988, 13(1):22-26.
[3] 谢浩, 陆仁后, 项超美, 等. 利用RAPD技术对三种绒螯蟹亲缘关系的研究[J]. 水生生物学报, 1999, 23(2):120-126.
[4] 孔晓瑜, 喻子牛, 刘亚军, 等. 中华绒螯蟹与日本绒螯蟹线粒体COⅠ基因片段的序列比较研究[J]. 青岛海洋大学学报:自然科学版, 2001, 31(6):861-866.
[5] 孙红英, 周开亚, 杨小军. 从线粒体16S rDNA序列探讨绒螯蟹类的系统发生关系[J]. 动物学报, 2003, 49(5):592-599.
[6] 高天翔, 张秀梅, 柳广东, 等. 10个日本绒螯蟹群体与中华绒螯蟹形态的主成分分析[J]. 大连水产学院学报, 2003, 18(4):273-277.
[7] 堵南山. 中华绒螯蟹的同属种类及其英文名称[J]. 水产科技情报, 1998, 25(3):108-109,113.
[8] Chan T Y, Hung M S, Yu H P.Identity ofEriocheirrecta(Stimpson, 1858) (Decapoda: Brachyura), with description of a new mitten crab from Taiwan[J]. Journal of Crustacean Biology, 1995, 15(2):301-308.
[9] 李孟仙, 曾辉. 合浦绒螯蟹Eriocheirhepuensis的形态学研究[J]. 浙江海洋学院学报:自然科学版, 2000, 19(4):327-332.
[10] 张秀梅, 柳广东, 高天翔. 绒螯蟹种质资源研究进展[J]. 青岛海洋大学学报:自然科学版, 2002, 32(4):533-542.
[11] 李思发, 邹曙明. 中国大陆沿海六水系绒螯蟹(中华绒螯蟹和日本绒螯蟹)群体亲缘关系:RAPD指纹标记[J]. 水产学报, 1999, 23(4):325-330.
[12] Hebert P D N,Ratnasingham S, deWard J R. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proceedings of the Royal Society B: Biological Sciences, 2003, 270 (Suppl.):96-99.
[13] Ward R D,Zemlak T S, Innes B H, et al. DNA barcoding Australias fish species [J]. Philosophical transactions of the Royal Society B:Biological Sciences, 2005, 360(1462):1847-1857.
[14] 柳淑芳, 陈亮亮, 戴芳群, 等. 基于线粒体COⅠ基因的DNA 条形码在石首鱼科(Sciaenidae)鱼类系统分类中的应用[J]. 海洋与湖沼, 2010, 41(2):223-232.
[15] 陈军, 李琪, 孔令锋, 等. 基于COⅠ序列的DNA条形码在中国沿海缀锦蛤亚科贝类中的应用分析[J]. 动物学研究, 2010, 31(4):345-352.
[16] Kim D W, Yoo W G, Park H C, et al. DNA barcoding of fish, insects, and shellfish in Korea[J]. Genomics & Informatic, 2012, 10(3):206-211.
[17] 杨帆, 何利军, 雷光春, 等. 中国东南沿海弹涂鱼科常见鱼类的遗传多样性和DNA条形码[J]. 生态学杂志, 2012, 31(3):676-683.
[18] 单云晶, 鲁翠云, 李超, 等. 基于线粒体COⅠ基因序列的5种鲤养殖品种遗传多样性研究[J]. 中国水产科学, 2013, 20(5):931-938.
[19] 张馨月, 刘岩, 张秀梅, 等. 基于COⅠ基因的西南大西洋部分经济鱼类DNA条形码鉴定[J]. 水生生物学报, 2014, 38(6):1161-1167.
[20] 李献儒, 柳淑芳, 李达, 等. DNA条形码在鲱形目鱼类物种鉴定和系统进化分析中的应用[J]. 中国水产科学, 2015, 22(6):1133-1141.
[21] Costa F O,deWaard J R, Boutillier J, et al. Biological identifications through DNA barcodes: the case of the Crustacea[J]. Canadian Journal of Fisheries and Aquatic Sciences, 2007, 64(2):272-295.
[22] Li Q, Park C, Kijima A. Isolation and characterization of microsatellite loci in the Pacific abalone,Haliotisdiscushannai[J]. Journal of Shellfish Research, 2002, 21(2):811-815.
[23] 王敏晓, 程方平, 李超伦, 等. 基于线粒体 cox1 片段序列的胶州湾浮游动物DNA条形码分析[J]. 海洋与湖沼, 2011, 42(5):702-710.
[24] 徐武杰, 邹节新, 白俊, 等. 鄱阳湖流域华溪蟹属DNA条形码[J]. 南昌大学学报:理科版, 2015, 39(4):402-408.
[25] Li G,Shen Q, Xu Z. Morphometric and biochemical genetic variation of the mitten crab,Eriocheir, in southern China[J]. Aquaculture, 1993, 111(1/4):103-115.
[26] 张列士, 姜治忠, 李军. 日本绒螯蟹与不同水系中华绒螯蟹的形态比较[J]. 上海水产大学学报, 2002, 11(2):110-113.
[27] 王茜, 李晓东, 王晓梅, 等. 不同水系绒螯蟹群体外部形态的判别分析[J]. 水产科学, 2006, 25(3):125-128.
[28] 戴爱云. 绒螯蟹属亚种分化的研究(十足目:短尾派)[G]//中国科学院动物研究所系统进化动物学重点实验室,张广学主编. 系统进化动物学重点实验论文集,北京:中国科学技术出版社, 1991:61-71.
[29] Hebert P D N,Cywinska A, Ball S L, et al. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society B:Biological Science, 2003, 270(1512):313-321.
[30] 胡鹏飞, 李晓东, 王晓梅, 等. 不同水系绒螯蟹线粒体DNA的遗传差异和群体遗传多样性研究[J]. 华北农学报, 2008, 23(3):50-55.
[31] 葛家春, 许志强, 李晓晖, 等. 利用线粒体 COⅠ 序列分析 4 水系中华绒螯蟹群体遗传学特征[J]. 中国水产科学, 2011, 18(1):16-22.
[32] 闫龙, 宋娜, 王俊, 等. 基于线粒体控制区的中华绒螯蟹群体遗传多样性分析[J]. 水生生物学报, 2015, 39(3):615-620.
[33] 邱高峰, 徐巧婷, 王丽卿, 等. 四种绒螯蟹分子分类与系统发育[J]. 动物学报, 2001, 47(6):640-647.
[34] 孙红英, 王光跃, 张代臻, 等. 中华绒螯蟹与合浦绒螯蟹两地理亚种的线粒体DNA序列变异[J]. 动物学报, 2005, 51(5):862-866.
[35] 赵乃刚. 长江河蟹种质资源混杂对养蟹业的影响[J]. 内陆水产, 1998, 23(5):2-4.
[36] 谷孝鸿, 赵福顺. 长江中华绒螯蟹的资源与养殖现状及其种质保护[J]. 湖泊科学, 2001, 13(3):267-271.
[37] 林乐峰, 冉炜, 冯宪章. 防止长江水系中华绒螯蟹混杂衰退的综合措施[J]. 渔业致富指南, 2004(6):45-47.
DNABarcodingandGeneticDiversityinGenusEriocheirBasedonCOⅠGeneSequences
XU Jie1, WANG Xiaomei1, LI Jingjing2, CHEN Chi1, LI Zhuanzhuan1, ZHANG Lingying1
( 1.Tianjin Key Laboratory of Aqua-Ecology & Aquaculture, College of Fisheries, Tianjin Agricultural University, Tianjin 300384, China; 2. Tianjin Diseases Prevention and Control Centre of Aquatic Animals, Tianjin 300221, China )
A total of 80 mitochondrial COⅠ gene fragments from fiveEriocheirpopulations includingE.sinensisRudong and Qilihai populations,E.japonica,E.hepuensisandE.leptognathuswere amplified and sequenced, and then the 80 sequences were compared with that of 2 COⅠ gene sequences ofE.formosaand 19 COⅠ sequences ofHemigrapsuspenicillatusrecorded in GenBank. In this study, 44 haplotypes were detected from 101 individuals, and the base composition of sequences showed a significant bias. Based on the analysis of COⅠ gene sequences, the K2P distances betweenE.sinensisRudong population, Qilihai population andE.japonica, being 1.210% and 1.078%, respectively, were significant lower than 2%, which is the threshold of the DNA barcoding species identification, indicating thatE.sinensisandE.japonicawere the same species. However, the K2P distances betweenE.hepuensisand the twoE.sinensispopulations as well asE.japonicawere 4.823%, 5.101% and 5.011%, respectively, which were far beyond 2%, indicating thatE.hepuensiswas an independent species. UsingH.penicillatusas out group, based on K2P distances, neighbor-joining (NJ) method was employed for phylogenetic analysis. NJ trees revealed thatE.sinensisandE.japonicadid not form monophyletic clades, but disturbed, disorderly, whereasE.hepuensiswas clustered into a single branch. Five populations sequenced in our study, with the exception ofE.leptognathus, the rest 4 populations had genetic diversity, the haplotype diversity ranged from 0.593±0.144 to 0.779±0.068 and the nucleotide diversity ranged from 0.00156 to 0.01336. TheE.sinensisRudong population shared the haplotypes H1, H2 and H3 withE.japonica,E.hepuensisandE.sinensisQilihai population, respectively, which suggested that the germplasm resources of the economic crabs were mixed or the phenomenon might be due to the genetic pollution.
Eriocheir; COⅠ gene; DNA barcode; species identification; genetic diversity
10.16378/j.cnki.1003-1111.2017.04.013
Q783
A
1003-1111(2017)04-0480-08
2016-05-31;
2016-08-19.
天津市水产局科技发展计划项目(J2013-21).
徐洁(1993-),女,本科生;研究方向:水产养殖学.E-mail:xujie19937@163.com. 通讯作者:王晓梅(1962-),女,教授;研究方向:水产动物遗传育种及分子生物学.E-mail:xiaomeiw@tjau.edu.cn.
