拟南芥多聚ADP-核糖化修饰蛋白的富集和鉴定
2017-12-14王文静葛晓春
王文静,吴 巧,葛晓春
(1. 复旦大学 遗传工程国家重点实验室,上海 200438;2. 复旦大学 生命科学学院,上海 200438)
拟南芥多聚ADP-核糖化修饰蛋白的富集和鉴定
王文静1,2,吴 巧1,2,葛晓春1,2
(1. 复旦大学 遗传工程国家重点实验室,上海 200438;2. 复旦大学 生命科学学院,上海 200438)
多聚ADP-核糖化修饰[poly(ADP-ribosyl)ation, PARylation]是一种可逆的蛋白质翻译后修饰,该修饰由多聚ADP-核糖聚合酶[poly(ADP-ribose) polymerase, PARP]催化.多聚ADP-核糖化修饰蛋白主要在动物中发现,在植物中少有报道.微生物来源的蛋白质AF1521具有一个能结合多聚ADP-核糖[poly(ADP-ribose), PAR]的macro结构域(macro domain),其突变蛋白AF1521-G42E失去了结合PAR的活性.我们利用AF1521蛋白对PAR的亲和作用来富集拟南芥中的PARylation蛋白质底物并进行质谱分析,同时用AF1521-G42E作为富集过程的负对照.通过这种方法,我们得到多个ADP-核糖化修饰相关蛋白,并对其中一个蛋白GRP7进行了验证.
AF1521; 拟南芥; AF1521-G42E; 多聚ADP-核糖化
多聚ADP-核糖化是一种重要的蛋白质翻译后修饰,它调节细胞内的一系列生理生化过程,如DNA修复[1-2]、转录调控[3]、染色质重塑[4]、细胞死亡[5]、细胞周期进行[6]、RNA剪切[7]、胞内运输[8]、蛋白质降解[9]等.在植物中,多聚ADP-核糖化修饰则与多种生物与非生物胁迫应答过程有关,例如抗病反应、干旱、高温、强光、氧化等非生物胁迫反应[10-11].
PAR修饰由多聚ADP-核糖聚合酶PARP催化[12].PARP与断裂DNA结合后被激活,催化烟酰胺腺嘌呤二核苷酸(Nicotinamide Adenine Dinucleotide, NAD+)中带负电荷的ADP-核糖基团转移到靶蛋白上,PARP的反复催化使得靶蛋白上形成直链或带有支链的多聚ADP-核糖分子,靶蛋白电化学性质发生巨大改变[12-13].靶蛋白上的PAR能够被多聚ADP-核糖糖苷水解酶[poly(ADP-ribose)glycohydrolase, PARG]降解[14].除酵母外,真核生物中都有多个PARP蛋白.拟南芥中有3个: PARP1、PARP2和PARP3[15],其中PARP1和PARP2已被证明具有催化活性,它们被断裂DNA激活后首先进行自我ADP-核糖化修饰,形成PAR链,然后通过PAR链募集与之结合的蛋白并将PAR链转移到靶蛋白上[16].
到目前为止,被多聚ADP-核糖化修饰的蛋白质多在动物中被发现,植物中除了PARP本身及其个别底物外,被报道的修饰蛋白质很少.动物中PAR修饰底物主要包括组蛋白、PARP自身、肿瘤抑制因子p53、泛素连接酶、染色质重塑相关酶、DNA连接酶等[17].除了被PAR修饰的蛋白质外,还有一些蛋白可与PAR结合,已知的PAR结合蛋白大多具有以下结构域: macro域、PAR-结合锌指(PBZ)、WWE域、PAR-结合基序(PBM)、FHA域或BRCT域等[17-19].其中,macro域能够识别PAR的末端ADP-核糖残基,主要存在于组蛋白变体、PARP、PARG、染色质重塑相关酶中[20].大部分PAR结合蛋白也是PAR修饰的底物蛋白,底物蛋白首先结合到PAR上,然后PARP将PAR转移到底物蛋白上.
关于动物中PARylation蛋白的筛选,目前已有多篇报道,他们利用的方法主要有以下几种: 利用亲和蛋白富集PAR相关蛋白[21-22];标记底物分子,检测带有相关信号的靶蛋白[23];根据PAR结合结构域保守序列利用计算机进行预测分析[24].植物中的研究进展较慢,直到2016年,才有了第一篇报道,他们用拟南芥蛋白芯片的方法将10048个拟南芥蛋白进行体外PARylation反应,最后得到54个可能的PARP2的底物[25],这个方法尽管较为直接,但对实验条件的要求较高,本实验室则采用另一种方法,即利用亲和蛋白富集并结合MS分析的方法,来获取PAR相关蛋白.
AF1521蛋白来源于嗜热菌Archaeoglobusfulgidus,是macro域超家族中最简单的分子,可以结合PAR,其大小约为22kDa[20],能够富集PAR底物蛋白以及PAR结合蛋白,其突变体AF1521-G42E为第42位的甘氨酸突变为谷氨酸,失去了结合PAR的能力.在动物中,已成功利用AF1521富集了多种ADP-核糖化相关蛋白[21-22],但植物中尚未有过尝试.本研究中,我们利用AF1521对拟南芥体内的PAR修饰蛋白进行富集实验(pull-down)并进行质谱分析,得到一些PAR相关的蛋白,对其中一个进行了验证,为深入了解多聚ADP核糖化修饰在植物体内的功能奠定了基础.
1 材料与方法
1.1 植物材料、菌株与质粒
拟南芥(Arabidopsisthaliana)突变体parg1为Columbia生态型背景.将种子用70%乙醇消毒后,播撒在1/2 MS培养基上,并置于光照培养箱中,培养条件为: 22℃,光照16h/黑暗8h循环.2周后幼苗用zeocin(Invitrogen公司)处理24h,收集材料液氮速冻待用.质粒扩增大肠杆菌:E.coliDH5α;原核表达大肠杆菌:E.coliOrigami(DE3);中间载体: PCR-bluntⅡ;原核表达载体: pET-32a(+)、pGEX-4T-1;pET-32a(+)-AtPARP1;pGEX-4T-1-AF1521、pGEX-4T-1-G42E(初始AF1521以及AF1521-G42E编码序列来自Ladurner实验室,参见其文章[21]).
pGEX-4T-1-GRP7(用带有BamHⅠ和SalⅠ限制性内切酶位点的引物GRP7-BamHⅠ(5′-GGATCCATG-GCGTCCGGTGATGTTG-3′)和GRP7-SalⅠ(5′-GTCGACTTACCATCCTCCACCACCACC-3′)扩增cDNA,回收大小正确的条带,用T4DNA连接酶(TaKaRa公司)连接到StuⅠ酶切后的PCR-bluntⅡ载体中,转化E.coliDH5α,挑取单菌落摇菌并抽提质粒,测序.得到正确质粒后进行双酶切,与带有BamHⅠ和SalⅠ酶切粘性末端的pGEX-4T-1载体连接,转化E.coliDH5α感受态细胞,挑取单菌落并抽质粒,酶切鉴定.
1.2 方法
1.2.1 蛋白质表达
将蛋白质表达载体转化宿主菌E.coliOrigami(DE3)感受态细胞,过夜培养后将菌落刮至液体LB,37℃、220r/min摇至OD600为0.6~0.8时加入IPTG至终浓度0.2mmol/L.之后16℃继续培养16h.离心收集菌体,重悬后用超声波仪破碎,取上清SDS-PAGE电泳检测目的蛋白的表达.
1.2.2 植物总蛋白抽提
在液氮中研磨zeocin处理的植物材料20g,加入4℃预冷的30mL裂解液(50mmol/L Tris-HCl pH8.0,5mmol/L MgCl2,100mmol/L NaCl,1mmol/L DTT,10% Glycerol,0.1% NP40,cocktail 蛋白酶抑制剂),冰上抽提20min,然后4℃,12000×g离心取上清.
1.2.3 蛋白质亲和富集PAR结合蛋白
将表达AF1521以及AF1521-G42E蛋白的菌体各200mL用PBS缓冲液重悬,超声破碎后离心取上清,菌液上清各与30μL Glutathione Sepharose(Amersham Bioscience公司)室温旋转孵育30min(介质使用前用PBS清洗3次).孵育后用PBS清洗3次.向植物总蛋白抽提物中加入外源NAD+至1mmol/L,超声断裂的拟南芥总DNA 500nmol/L,室温孵育20min以激活PARP活性,提高蛋白质底物的多聚ADP-核糖化修饰程度.在孵育后的蛋白质样品中加入结合了AF1521-G42E的Glutathione Sepharose介质,再次室温旋转孵育1h,之后离心取上清,然后再与结合了AF1521的介质室温孵育1h,用PBS清洗介质3次,流程如图1所示.
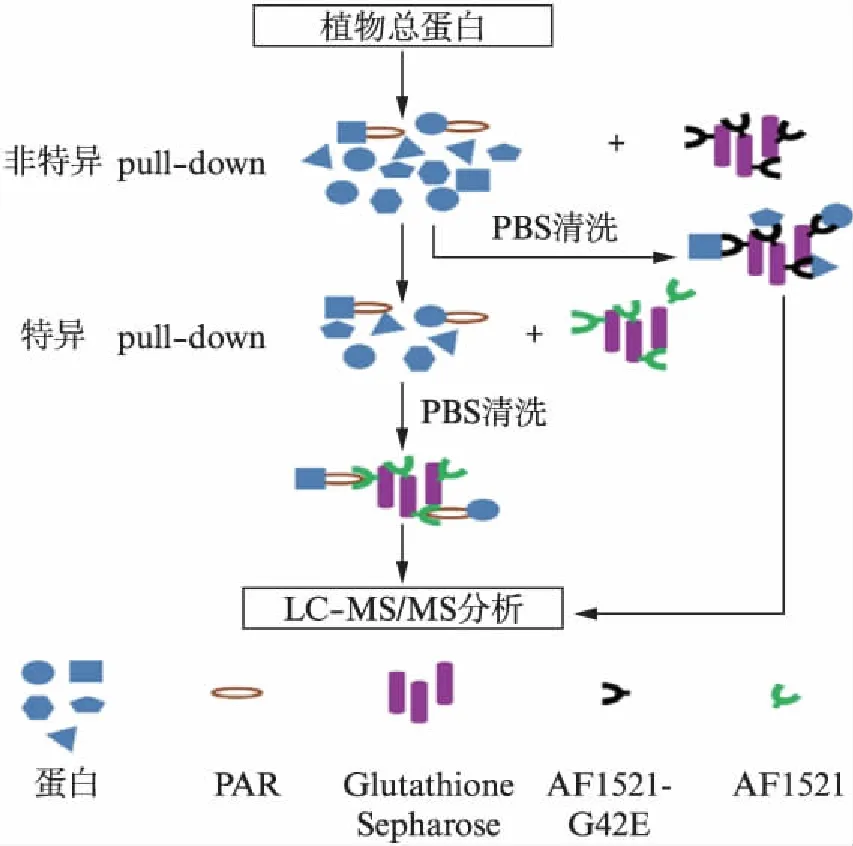
图1 pull-down流程Fig.1 Procedure of pull-down experiments植物材料破碎后,可溶性蛋白首先与结合了GST-AF1521-G42E的介质孵育去除非特异性蛋白,再与结合了GST-AF1521的介质孵育以富集PAR结合蛋白,然后利用质谱分析pull-down的蛋白质.
1.2.4 Western blot分析
向pull-down实验后的介质中加入1×SDS-PAGE样品缓冲液,煮沸10min后离心取上清,取5 μL进行SDS-PAGE电泳,之后转至PVDF膜上.转膜后,用少量TBST缓冲液(20mmol/LTris-HCl pH7.5, 150mmol/L NaCl, 0.05% Tween 20)清洗2次,5%牛奶封闭1h,PAR抗体(Abcam公司)室温孵育1h,TBST缓冲液洗涤4次,各10min,此后二抗室温孵育1h,TBST洗涤4次,各5min,最后利用化学发光成像系统(CLiNX公司,上海)进行荧光信号分析.
1.2.5 LC-MS/MS分析
pull-down所得到的蛋白进行SDS-PAGE电泳,待考马斯亮蓝染料进入分离胶约1cm停止电泳,染色,然后割胶进行质谱分析.质谱分析由复旦大学国家重点实验室的蛋白质分析平台提供技术服务,利用LTQ-Orbitrap Elite质谱仪(Thermo Fisher Scientific,德国)完成.
1.2.6 GRP7与PARP1以及自我修饰的PARP1互作验证
利用PBS缓冲液重悬带有pGEX-4T-1空载体或pGEX-4T-1-GRP7表达载体的宿主菌E.coliOrigami(DE3),超声破碎,12000×g,4℃离心,取上清,分别与Glutathione Sepharose介质室温轻柔旋转孵育30min,使得GST和GST-GRP7结合到介质上,然后PBS洗涤3次,将介质用于pull-down实验.结合了GST tag的介质可作为结合了GST-GRP7介质的对照.超声破碎转化了pET-32a(+)空载体和pET-32a(+)-AtPARP1表达载体的宿主菌E.coliOrigami(DE3),12000×g,4℃离心取上清.表达PARP1的菌液上清分作两部分,一部分用于PARP1的激活,在上清中加入终浓度为1mmol/L的NAD+,以及500nmol/L的断裂DNA,室温温育10min;另一部分加入抑制PARP1活性的3-AB(3-Aminobenzamide)抑制剂.此后,带有GST以及GST-GRP7的介质先与表达His tag的pET-32a(+)空载体菌液上清室温孵育1h,PBS洗涤后一部分介质取样,另一部分则分别与激活以及未激活的PARP1菌液上清室温孵育1h.两种孵育后的介质用PBS洗涤4次,然后所有取样的介质等量在SDS-PAGE上样缓冲液中煮沸,离心后电泳,进行Western blot分析.
2 结果与分析
2.1 GST-AF1521的原核表达
微生物来源的AF1521蛋白具有macro域,能够特异性结合PAR末端的ADP-核糖残基(图2),其突变蛋白AF1521-G42E失去了结合活性.我们将AF1521基因及突变基因AF1521-G42E分别连入pGEX-4T-1表达载体,并在宿主菌E.coliOrigami(DE3)中表达,得到大量带有GST标签的AF1521蛋白以及突变蛋白AF1521-G42E(图3(a),见第406页),并且Glutathione Sepharose纯化后的重组蛋白中杂蛋白较少(图3(b)).
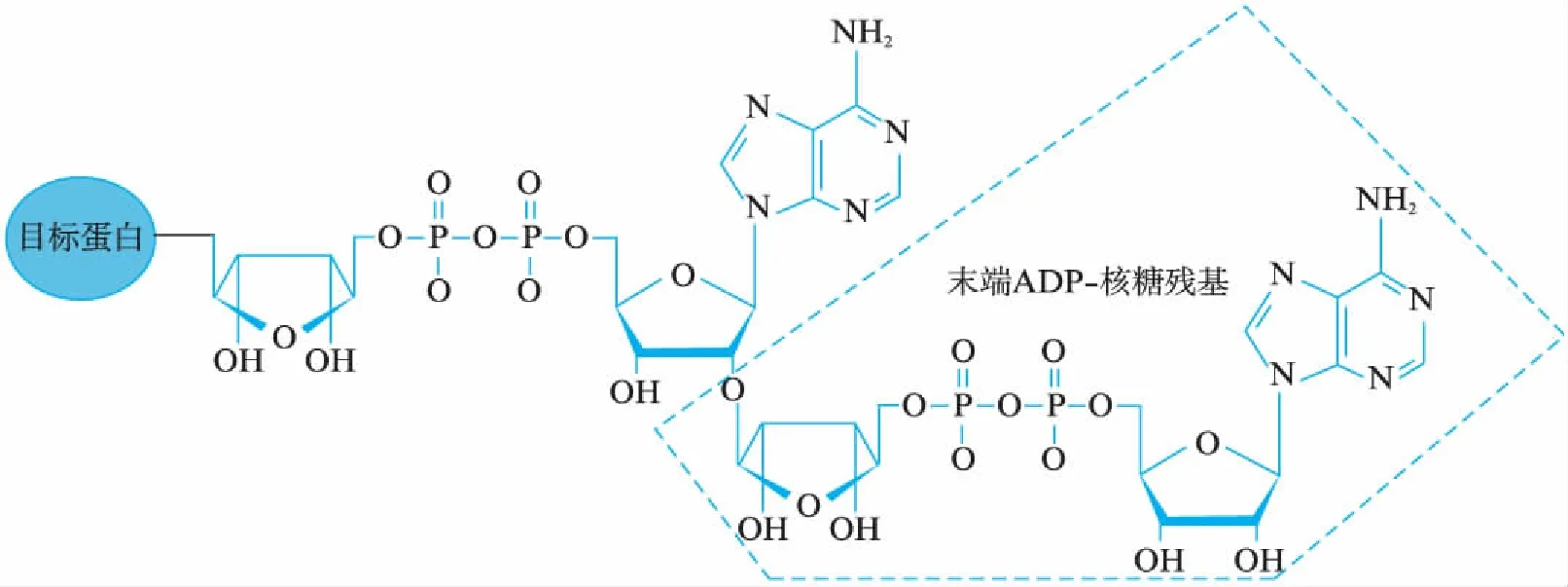
图2 AF1521蛋白能够识别PAR末端ADP-核糖残基(方框表示macro domain识别位点)Fig.2 AF1521 protein can recognize the terminal ADP-ribose residue of PAR(The box indicates the recognition site of macro domain)

图3 GST-AF1521以及GST-AF1521-G42E重组蛋白表达及纯化产物的SDS-PAGE电泳分析Fig.3 SDS-PAGE analysis of the expression and purified products of GST-AF1521 and GST-AF1521-G42E(a) 重组蛋白表达; 1,3为IPTG诱导前蛋白; 2,4为IPTG诱导后蛋白; 1,2为GST-AF1521; 3,4为GST-AF1521-G42E. (b) 纯化后蛋白; 1. GST-AF1521; 2. GST-AF1521-G42E.
2.2 AF1521富集PAR相关蛋白
将蛋白GST-AF1521及GST-AF1521-G42E结合在GST亲和介质上,同时抽提经过zeocin处理的parg1突变体总蛋白.突变体parg1是PARG1基因的T-DNA插入突变体,其中PARG1基因突变,PARG1活性丧失,不能水解PAR,从而可使得体内的PAR修饰蛋白质水平升高[26],有利于PAR修饰蛋白质的富集;zeocin为基因断裂剂,zeocin处理可诱导体内PARP1以及PARP2的表达水平升高并激活其活性,也使得细胞内的PAR修饰蛋白增多.此外,我们在提取植物蛋白后加入PARP活性的限速底物NAD+,以便潜在的PAR修饰蛋白不会因受到NAD+量的限制而失去激活的可能性.利用结合了突变蛋白AF1521-G42E的介质去除一些可能产生非特异性结合的蛋白质,同时也对该介质上结合的蛋白质进行质谱分析,以作为野生型AF1521蛋白所pull-down到的蛋白质的对照.利用PAR抗体进行Western blot实验,结果表明AF1521相对于对照AF1521-G42E确实富集了大量被PAR修饰的蛋白(图4(a)).其中包括了ADP-核糖化修饰的PARP2(图4(b)).此后,将富集的蛋白质进行SDS-PAGE电泳并割胶进行LC-MS/MS分析,得到PAR相关蛋白(表1),一共有14个蛋白质特异性存在于AF1521拉下的蛋白质谱中而没有出现在突变体AF1521-G42E蛋白富集的蛋白质中.

图4 Western blot分析pull-down结果Fig.4 Western blot analysis of the pull-down results(a) 一抗为PAR抗体;(b) 一抗为PARP2抗体;星号表示PARP2条带.
其中多个蛋白属于细胞内高丰度的蛋白或者是与代谢有关的酶,如At1g12900.2(3-磷酸甘油醛脱氢酶,GAPDH)、At5g38410.2(核酮糖-1,5-二磷酸羧化酶/加氧酶)、At3g03780.1(甲硫氨酸合成酶),这些蛋白质可能自身被修饰,也可能因某种原因而被富集.而其他一些蛋白质,不属于组成性存在的蛋白质,它们是At3g09840.1(CDC48A)、At2g21660.2(GRP7)、At5g20010.1(RAN1)、At1g02740.1(MRG1).特别是这些蛋白均存在于细胞核中,与负责PAR修饰的PARP主要位于细胞核中、大部分PAR修饰蛋白均属于细胞核蛋白的预期相符.当然,macro域也能够结合单ADP-核糖残基,但由于植物中尚未发现单-ADP核糖化催化酶,我们也采用了体内孵育以提高蛋白质底物多聚ADP-核糖化修饰程度的实验条件,因此,本实验所富集到的蛋白主要为多聚ADP-核糖相关蛋白,但不排除有单ADP-核糖化相关蛋白的可能.
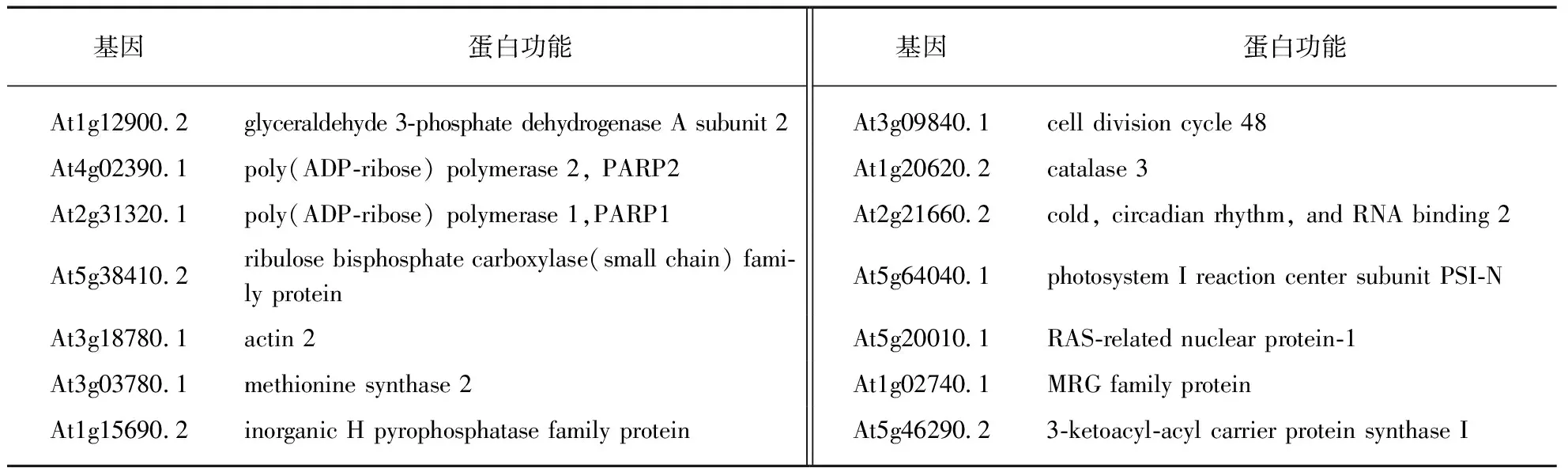
表1 质谱结果Tab.1 The results of LC-MS/MS
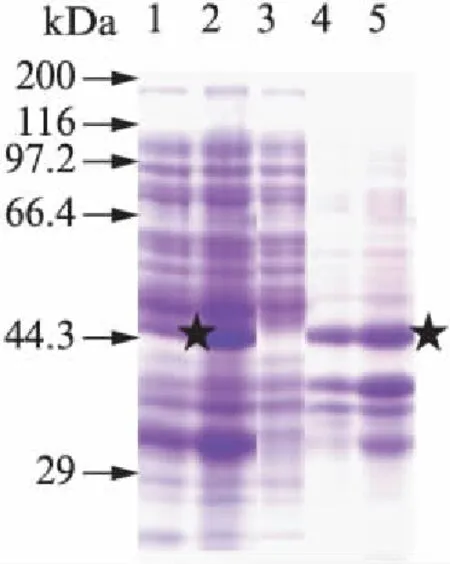
图5 GST-GRP7重组蛋白表达产物的SDS-PAGE电泳分析Fig.5 SDS-PAGE analysis of the expression proteins of E.coli1,4分别为16℃时,诱导后的可溶蛋白与不溶蛋白;2,5分别为37℃时,诱导后的可溶蛋白与不溶蛋白;3.IPTG诱导前蛋白;星号表示GST-GRP7条带.
2.3 蛋白GST-GRP7的诱导表达
质谱鉴定到的蛋白质中,GRP7(表1中At2g21660.2)是已经证实的单ADP-核糖化的底物[27],推测GRP7也有可能被多聚ADP核糖化修饰,因此我们进一步扩增了拟南芥中GRP7的cDNA并构建带有GST tag的载体pGEX-41-1-GRP7,在不同条件下表达目标蛋白,通过SDS-PAGE电泳比较细菌裂解液上清与沉淀中GST-GRP7蛋白含量.结果表明,当诱导温度为37℃,诱导时间为2.5h时,上清中有较多的目的蛋白(图5).
2.4 GRP7与PARP1及被PAR修饰的PARP1体外互作验证
GRP7有可能发生多聚ADP核糖化修饰、或者结合PAR修饰蛋白,我们将GST-GRP7与His-PARP1孵育,以观察GST-GRP7是否可能被PARP1修饰,但未得到预期结果.原因有可能是GRP7没有被PAR修饰而仅仅被单ADP核糖化修饰,或者实验条件不合适以至于不能观察到被PAR修饰的GRP7.GRP7被AF1521富集的另一个原因也有可能是它通过结合PAR或者被PAR修饰的蛋白从而被富集.我们检测了GRP7与PARP1以及被PAR修饰的PARP1的结合活性,Western blot结果显示,GRP7与PARP1以及发生自我修饰的PARP1有相互作用(图6(a)),且GRP7与PARP1的互作明显强于与自我修饰的PARP1的互作(图6(b)).PAR修饰使得PARP1带有大量负电荷,改变了PARP1的带电性从而可能减弱了GRP7与PARP1的互作.

图6 GRP7与PARP1以及自我修饰的PARP1体外互作验证Fig.6 GRP7 associates with PARP1 and activated PARP1 in vitro(a) Anti-His抗体杂交结果,黑色星号表示PARP1条带;(b) Anti-GST抗体杂交结果,黑色和白色星号分别表示GST和GST-GRP7.
3 讨 论
在富集多聚ADP核糖化修饰蛋白时,PARP1和PARP2是植物体内已知的可被多聚ADP-核糖化修饰的蛋白,它们可以进行自我修饰连上多聚ADP-核糖链,因此,如果我们所采用的方法可行的话,应该能在富集到的底物中发现PARP1和PARP2,它们可作为实验结果的内参.
通过对比野生型AF1521蛋白与突变体蛋白AF1521-G42E分别作为饵(bait)时拉下的蛋白质谱,排除那些同时出现的蛋白质,从质谱结果中,我们发现PARP1以及PARP2确实被野生型AF1521蛋白特异性富集,说明该方法富集PAR相关蛋白的方法是可行的.pull-down实验中得到的蛋白可能是PAR修饰或结合蛋白,也可能是与这些蛋白质互作的蛋白.我们进一步选取了GRP7进行体外验证,发现GRP7与PARP1以及自我修饰的PARP1都可互作,但GRP7与PARP1的互作明显强于与被修饰的PARP1的互作,说明PAR修饰可能抑制了GRP7与PARP1的互作,PAR修饰可能调节蛋白质的相互作用.GRP7是hnRNP-like的富含甘氨酸的RNA结合蛋白,能够调控pre-mRNA的剪切,还能够直接结合pri-miRNAs从而阻碍它们的加工[28].PARP1如何参与GRP7的调控,值得进一步研究.
质谱分析得到的蛋白中,RAN1是一种小的GTPase,参与核内外物质运输,可能参与细胞周期调控[29].CDC48A是一种Ⅱ型AAA-ATPase,对于细胞分裂、增殖、分化有着至关重要的作用[30].MRG1是MRG家族的染色质重塑因子[31].已知在动物中,PAR修饰确实参与了细胞周期调控、RNA的剪切以及表观遗传学调控[4,6-7].我们推测植物中PAR修饰确有可能通过与不同蛋白质互作来参与调控这些过程.此外,突变体grp7、mrg1mrg2及多聚ADP-核糖水解酶缺失的突变体parg1都具有早花表型[31-33];而RAN1的过表达则是晚花的[29],这可能意味着PAR修饰可能参与调控开花.
在动物中,Poirier实验室也用该方法富集到了95个蛋白质[22],其中也包括了PARP蛋白,其他还包括RAD50、XRCC1、MSH3蛋白,这些都与DNA损伤响应有关,但在我们富集到的蛋白质中,未发现这些蛋白质的存在,可能是因为本方法pull-down的灵敏度还不够,只富集到了少部分高丰度的PAR相关蛋白,又或者在植物中,PARP调节生理过程的分子机制与动物中有差异,这些蛋白质被修饰状态存在时间可能很短.但是,在动、植物中共同富集到了GAPDH、actin、cell division cycle protein等,说明在动、植物中,这些蛋白质可能具有相同的多聚ADP核糖化调节机制.另外,我们所得到的蛋白质,与2016年Feng等报道的PARP2的底物蛋白质不一样[25],他们的方法中并没有得到植物中已知的多聚ADP核糖化蛋白质底物PARP1和PARP2,而在我们的方法中,这两个蛋白作为内参照,确实被富集,引起差别的原因可能是,我们的方法采用了植物材料进行体内蛋白的pull-down,而他们采用的是利用蛋白质芯片进行体外反应的方法.
目前关于动物的研究报道中,几个不同实验室利用不同方法都筛选到了一些多聚ADP核糖化修饰相关蛋白,这些方法各有优缺点.以[32P]-NAD+为底物的体外标记PAR相关蛋白的方法可以较灵敏地检测到目的蛋白,但是同位素昂贵且操作要求高,该方法未能广泛使用.Gagne等人用小鼠的单克隆抗体成功富集到PAR相关蛋白,但是局限性在于单克隆抗体对短链的PAR(少于20个核糖残基)亲和性很低[22].Gagne实验室还利用失活的PARG-DEAD进行pull-down得到了561个PAR结合蛋白[22].我们采用的方法相对于单克隆抗体和PARG-DEAD富集方法而言,AF1521表达量高、蛋白质小,容易纯化,pull-down流程操作简单,耗资较少,可操作性较高.
植物中,多聚ADP-糖化修饰参与了昼夜节律调控[33]、生物胁迫[34-35]和干旱、氧化胁迫等非生物胁迫调节[36-38].抑制或者沉默PARP可以提高植物对逆境胁迫的抗性[38],除了催化酶本身外,我们对PARP所修饰的蛋白质底物以及它们如何调节其他蛋白质的功能了解并不多,需要更多的手段来寻找PAR修饰相关底物从而研究PAR修饰在体内的调节功能,本研究在这方面提供了有价值的信息.
[1] DANTZER F, SCHREIBER V, NIEDERGANG C,etal. Involvement of poly(ADP-ribose) polymerase in base excision repair [J].Biochimie, 1999,81(1/2): 69-75.
[2] MORTUSEWICZ O, FOUQUEREL E, AME J C,etal. PARG is recruited to DNA damage sites through poly(ADP-ribose)-and PCNA-dependent mechanisms [J].NucleicAcidsRes, 2011,39(12): 5045-5056.
[3] D’AMOURS D, DESNOYERS S, D’SILVA I,etal. Poly(ADP-ribosyl)ation reactions in the regulation of nuclear functions [J].BiochemJ, 1999,342(Pt 2): 249-268.
[4] CAIAFA P, GUASTAFIERRO T, ZAMPIERI M. Epigenetics: poly(ADP-ribosyl)ation of PARP-1 regulates genomic methylation patterns [J].FASEBJ, 2009,23(3): 672-678.
[5] CREGAN S P, DAWSON V L, SLACK R S. Role of AIF in caspase-dependent and caspase-independent cell death [J].Oncogene, 2004,23(16): 2785-2796.
[6] AUGUSTIN A, SPENLEHAUER C, DUMOND H,etal. PARP-3 localizes preferentially to the daughter centriole and interferes with the G1/S cell cycle progression [J].JCellSci, 2003,116(8): 1551-1562.
[7] SHKRETA L, CHABOT B. The RNA splicing response to DNA damage [J].Biomolecules, 2015,5(4): 2935-2977.
[8] SANKPAL U T, ABDELRAHIM M, CONNELLY S F,etal. Small molecule tolfenamic acid inhibits PC-3 cell proliferation and invasioninvitro, and tumor growth in orthotopic mouse model for prostate cancer [J].Prostate, 2012,72(15): 1648-1658.
[9] WANG P, LAYFIELD R, LANDON M,etal. Selective isopeptide bond formation: Coupling ubiquitin(67-76) with histone 2A(114-128) by use of the transfer active ester condensation technique [J].JPeptRes, 1999,53(6): 673-677.
[10] VANDERAUWERA S, DE BLOCK M, VAN DE STEENE N,etal. Silencing of poly(ADP-ribose) polymerase in plants alters abiotic stress signal transduction [J].ProcNatlAcadSciUSA, 2007,104(38): 15150-15155.
[11] ADAMS-PHILLIPS L, BRIGGS A G, BENT A F. Disruption of poly(ADP-ribosyl)ation mechanisms alters responses ofArabidopsisto biotic stress [J].PlantPhysiol, 2010,152(1): 267-280.
[12] AME J C, SPENLEHAUER C, DE MURCIA G. The PARP superfamily [J].Bioessays, 2004,26(8): 882-893.
[13] ALTHAUS F R, RICHTER C. ADP-ribosylation of proteins. Enzymology and biological significance [J].MolBiolBiochemBiophys, 1987,37: 1-237.
[14] SLADE D, DUNSTAN M S, BARKAUSKAYTE E,etal. The structure and catalytic mechanism of a poly(ADP-ribose) glycohydrolase [J].Nature, 2011,477(7366): 616-620.
[15] LAMB R S, CITARELLI M, TEOTIA S. Functions of the poly(ADP-ribose) polymerase superfamily in plants [J].CellMolLifeSci, 2012,69(2): 175-189.
[16] ALTMEYER M, MESSNER S, HASSA P O,etal. Molecular mechanism of poly(ADP-ribosyl)ation by PARP1 and identification of lysine residues as ADP-ribose acceptor sites [J].NucleicAcidsRes, 2009,37(11): 3723-3738.
[17] HOTTIGER M O. SnapShot: ADP-ribosylation signaling [J].MolCell, 2016,62(3): 472.
[18] GIBSON B A, KRAUS W L. New insights into the molecular and cellular functions of poly(ADP-ribose) and PARPs [J].NatRevMolCellBiol, 2012,13(7): 411-424.
[19] LI M, LU L Y, YANG C Y,etal. The FHA and BRCT domains recognize ADP-ribosylation during DNA damage response [J].GenesDev, 2013,27(16): 1752-1768.
[20] KARRAS G I, KUSTATSCHER G, BUHECHA H R,etal. The macro domain is an ADP-ribose binding module [J].EMBOJ, 2005,24(11): 1911-1920.
[21] DANI N, STILLA A, MARCHEGIANI A,etal. Combining affinity purification by ADP-ribose-binding macro domains with mass spectrometry to define the mammalian ADP-ribosyl proteome [J].ProcNatlAcadSciUSA, 2009,106(11): 4243-4248.
[22] GAGNE J P, PIC E, ISABELLE M,etal. Quantitative proteomics profiling of the poly(ADP-ribose)-related response to genotoxic stress [J].NucleicAcidsRes, 2012,40(16): 7788-7805.
[23] FARAONE-MENNELLA M R, DE MAIO A, PETRELLA A,etal. Yeast(ADPribosyl)ation: Revisiting a controversial question [J].JCellBiochem, 2005,94(6): 1258-1266.
[24] GAGNE J P, ISABELLE M, LO K S,etal. Proteome-wide identification of poly(ADP-ribose) binding proteins and poly(ADP-ribose)-associated protein complexes [J].NucleicAcidsRes, 2008,36(22): 6959-6976.
[25] FENG B, MA S, CHEN S,etal. PARylation of the forkhead-associated domain protein DAWDLE regulates plant immunity [J].EMBORep, 2016,17(12): 1799-1813.
[26] ZHANG H, GU Z, WU Q,etal.ArabidopsisPARG1 is the key factor promoting cell survival among the enzymes regulating post-translational poly(ADP-ribosyl)ation [J].SciRep, 2015,5: 15892.
[27] NICAISE V, JOE A, JEONG B R,etal. Pseudomonas HopU1 modulates plant immune receptor levels by blocking the interaction of their mRNAs with GRP7 [J].EMBOJ, 2013,32(5): 701-712.
[28] KOSTER T, MEYER K, WEINHOLDT C,etal. Regulation of pri-miRNA processing by the hnRNP-like protein AtGRP7 inArabidopsis[J].NucleicAcidsRes, 2014,42(15): 9925-9936.
[29] WANG X, XU Y, HAN Y,etal. Overexpression of RAN1 in rice andArabidopsisalters primordial meristem, mitotic progress, and sensitivity to auxin [J].PlantPhysiol, 2006,140(1): 91-101.
[30] PARK S, RANCOUR D M, BEDNAREK S Y. In planta analysis of the cell cycle-dependent localization of AtCDC48A and its critical roles in cell division, expansion, and differentiation [J].PlantPhysiol, 2008,148(1): 246-258.
[31] BU Z, YU Y, LI Z,etal. Regulation ofArabidopsisflowering by the histone mark readers MRG1/2 via interaction with CONSTANS to modulate FT expression [J].PLoSGenet, 2014,10(9): e1004617.
[32] XIAO J, LI C, XU S,etal. JACALIN-LECTIN LIKE1 regulates the nuclear accumulation of GLYCINE-RICH RNA-BINDING PROTEIN7, influencing the RNA processing of FLOWERING LOCUS C antisense transcripts and flowering time inArabidopsis[J].PlantPhysiol, 2015,169(3): 2102-2117.
[33] PANDA S, POIRIER G G, KAY S A. tej defines a role for poly(ADP-ribosyl)ation in establishing period length of theArabidopsiscircadian oscillator [J].DevCell, 2002,3(1): 51-61.
[34] ISHIKAWA K, OGAWA T, HIROSUE E,etal. Modulation of the poly(ADP-ribosyl)ation reaction via theArabidopsisADP-ribose/NADH pyrophosphohydrolase, AtNUDX7, is involved in the response to oxidative stress [J].PlantPhysiol, 2009,151(2): 741-754.
[35] FENG B, LIU C, DE OLIVEIRA M V,etal. Protein poly(ADP-ribosyl)ation regulatesArabidopsisimmune gene expression and defense responses [J].PLoSGenet, 2015,11(1): e1004936.
[36] AMOR Y, BABIYCHUK E, INZE D,etal. The involvement of poly(ADP-ribose) polymerase in the oxidative stress responses in plants [J].FEBSLett, 1998,440(1-2): 1-7.
[37] TIAN R, ZHANG G Y, YAN C H,etal. Involvement of poly(ADP-ribose) polymerase and activation of caspase-3-like protease in heat shock-induced apoptosis in tobacco suspension cells [J].FEBSLett, 2000,474(1): 11-15.
[38] DE BLOCK M, VERDUYN C, DE BROUWER D,etal. Poly(ADP-ribose) polymerase in plants affects energy homeostasis, cell death and stress tolerance [J].PlantJ, 2005,41(1): 95-106.
Identificationofpoly(ADP-ribosyl)atedProteinsinArabidopsis
WANGWenjing1,2,WUQiao1,2,GEXiaochun1,2
(1.SchoolofLifeSciences,FudanUniversity,Shanghai200438,China;2.StateKeyLaboratoryofGeneticEngineering,FudanUniversity,Shanghai200438,China)
poly(ADP-ribosyl)ation is a type of reversible posttranslational modification catalyzed by enzymes known as poly(ADP-ribose) polymerases(PARP). The protein substrates are mostly found in animals and there are rare reports in plants so far. AF1521 protein originated from microorganism possesses a macrodomain, which can recognize and interact selectively with the terminal residue of poly(ADP-ribose)(PAR), whereas the mutant protein AF1521-G42E loses the PAR-binding activity. In our study, we used AF1521 as a selective bait for enriching poly(ADP-ribosyl)ated protein substrates or proteins that interact with them inArabidopsis. In the meantime, AF1521-G42E was used as a control of the experiment. LC-MS/MS was used to analyze the pull-down proteins. In this way,we identified a couple of possible protein substrates of poly(ADP-ribosyl) ation or proteins that related with poly(ADP-ribosyl)ated proteins, and one protein of them, GRP7, was verified byinvitropull-down assay.
AF1521;Arabidopsis; AF1521-G42E; poly(ADP-ribosyl)ation
0427-7104(2017)04-0403-09
2017-02-06
国家自然科学基金(31170169,31070232)
王文静(1990—),女,硕士研究生;葛晓春,女,教授,通信联系人,E-mail: xcge@fudan.edu.cn.
Q943.2
A
