瓦氏黄颡鱼(Pelteobagrus vachellii)脂肪酸去饱和酶FAD2和延伸酶ELOVL5的克隆及表达分析*
2017-12-14覃川杰文正勇袁登越
覃川杰 文正勇 袁登越 邵 婷 龚 全
(1.内江师范学院生命科学学院 长江上游鱼类资源保护与利用四川省重点实验室 内江 641112;2.四川省农业科学院水产研究所 成都 611731)
瓦氏黄颡鱼(Pelteobagrus vachellii)脂肪酸去饱和酶FAD2和延伸酶ELOVL5的克隆及表达分析*
覃川杰1文正勇1袁登越1邵 婷1龚 全2
(1.内江师范学院生命科学学院 长江上游鱼类资源保护与利用四川省重点实验室 内江 641112;2.四川省农业科学院水产研究所 成都 611731)
脂肪酸去饱和酶(FAD, fatty acyl desaturase)及延伸酶(ELOVL, elongases of very long chain fatty acids)在鱼类脂肪代谢过程中发挥了重要作用。利用RT-PCR克隆得到瓦氏黄颡鱼(Pelteobagrus vachellii)肝脏中控制高不饱和脂肪酸合成的(FAD2)和(ELOVL5)基因cDNA 序列。瓦氏黄颡鱼FAD2 cDNA片段长2041bp, 编码447个氨基酸, 含有3个组氨酸簇(HDxGH, HxxHH, QxxHH), 含亚铁血红素结合基序(HPGG)的类似细胞色素b5结构域等。瓦氏黄颡鱼ELOVL5 cDNA片段长1065bp, 编码 294个氨基酸, 含有组氨酸簇(HxxHH)、内质网停留信号(K、R)和 4个 ELO 共有的保守区域(KxxExxDT, QxxFLHxYHH, NxxxHxxMYxYY, TxxQxxQ)等结构域。荧光定量PCR分析表明, FAD2和ELOVL5 mRNA在瓦氏黄颡鱼脑、肝脏的表达量最高, 显著高于肠道、脾脏、肾脏、鳃等组织。结果表明, 瓦氏黄颡鱼具有合成高不饱和脂肪酸的关键酶 FAD2和 ELOVL5, 且肝脏为合成高不饱和脂肪酸的主要场所。
脂肪酸去饱和酶(FAD2); 脂肪酸延伸酶(ELOVL5); 瓦氏黄颡鱼; cDNA
瓦氏黄颡鱼(Pelteobagrus vachellii)是一种偏肉食性的杂食性鱼类, 隶属于鲇形目(Siluriformes)、鲿科(Bagride)、黄颡鱼属(Pelteobagrus), 其肉细嫩、味道鲜美, 深受消费市场青睐(丁瑞华, 1994)。该鱼体型较大、生长快, 已成为我国重要的淡水名优经济养殖鱼类。目前, 吴代武等(2017)发现, 饲料中添加过高的植物蛋白会明显降低黄颡鱼(P.fulvidraco)生长, 饲喂鱼蛋白水解物日粮使黄颡鱼肌肉游离氨基酸含量的升高, 特别是呈味氨基酸含量增加。饲料中添加豆油不会显著影响瓦氏黄颡鱼的增重率、肝体指数和体成分等, 但会影响肌肉中脂肪酸的组成, 增加肌肉中C18:1n-9、C18:2n-6和单不饱和脂肪酸比例(邵婷等,2017)。变换昼夜投喂时间会显著影响肝脏中酰基-CoA去饱和酶(acyl-CoA desaturase), 葡萄糖-6-磷酸酯 酶(glucose-6-phosphatase), γ-谷 氨 酰转肽 酶 1(gamma- glutamyltranspeptidase 1), 胰凝乳蛋白酶 B(chymotrypsin B)等与蛋白、脂肪和糖代谢的相关基因mRNA的表达(Qinet al, 2017)。因此, 深入分析瓦氏黄颡鱼高不饱和脂肪酸的生物合成能力有助于制定合理的饲料配方。
鱼类必需营养素中的高度不饱和脂肪酸(high unsaturation fatty acids, HUFAs), 尤其是二十碳五烯酸(eicosapentaenoic acid, EPA)和二十二碳六烯酸(docosahexaenoic acids, DHA), 它们对维持生物机体的正常机能, 促进生长发育和繁殖, 以及提高成活率等方面起着重要生理作用。水产养殖业中大部分HUFAs的来源主要是鱼油, 但由于其高昂的价格,增加了养殖成本, 导致经济效益降低; 因而, 缺乏高不饱和脂肪酸的豆油等植物油被广泛应用于水产饲料(覃川杰等, 2013)。一般而言, 鱼类可以通过n-3和/或n-6的途径去饱和及碳链的延长作用, 将植物产品中的18:3 n-3及18:2 n-6脂肪酸分别转化为长链高不饱和脂肪酸(Cooket al, 2002)。但不同鱼类合成HUFAs的能力相差比较明显, 如淡水鱼类的高不饱和脂肪酸的合成能力明显优于海水鱼类(Tocheret al,1999, 2006)。因此, 了解和掌握养殖鱼类长链脂肪酸的合成能力, 有助于在鱼类养殖中合理利用不同的脂肪源。研究表明, 脂肪酸去饱和酶Δ6和脂肪酸延伸酶 ELOVL5在动物高效利用植物脂肪过程中发挥了关键作用(Xueet al, 2014), 且脂肪酸去饱和酶和延长酶普遍存在于多数淡水鱼类中(Agabaet al, 2005)。目前, 只从少数几种鱼类克隆了脂肪酸去饱和酶和延伸酶的 cDNA, 且主要集中在海水鱼类, 如军曹鱼Rachycentron canadum, 大西洋鳕Gadus morhua等(许友卿等, 2010; Xueet al, 2014); 赤鲈Perca fluviatilis和草鱼Ctenopharyngodon idellus等淡水鱼类中开展了相关研究(杜嵇华等, 2011; Geayet al,2016)。作者在本文中对瓦氏黄颡鱼的 FAD2和ELOVL5 cDNA序列进行克隆, 并分析该两种基因在瓦氏黄颡鱼中的表达, 以期进一步探讨瓦氏黄颡鱼合成HUFAs的可能性及其机制。
1 材料与方法
1.1 材料
实验材料为鲜活瓦氏黄颡鱼(Pelteobagrus vachellii) (体重约15g/尾), 购于内江某养殖场。将实验鱼置于冰上麻醉, 然后处死, 同时将其肌肉、肝脏、心脏、脾脏、肠道、肾脏、鳃和脑取出, 液氮速冻, 置于–80℃保存备用。
1.2 总RNA提取及cDNA合成
按照 RNAiso Plus (TaKaRa)试剂操作步骤提取瓦氏黄颡鱼肝脏总 RNA; 以肝脏总 RNA为模板,oligo(dT)20为逆转录引物, 按照 PrimeScriptTMRT reagent Kit (TaKaRa)逆转录试剂盒操作步骤合成cDNA。
1.3 瓦氏黄颡鱼FAD2和ELOVL5基因cDNA克隆
根据人、猪以及鱼等物种的 FAD2和 ELOVL5基因保守区域, 设计PCR简并引物(上海生工合成)。以瓦氏黄颡鱼的肝脏cDNA为模板, 采用rTaq DNA聚合酶(TaKaRa), 以简并引物进行 PCR扩增。用Biometra PCR仪扩增, PCR反应参数为: 94℃热启动4min, 然后 94℃变性 30s, 50℃ (FAD2)和 52℃(ELOVL5)分别退火30s, 再72℃延伸1min, 30个循环,72℃ 10min终止反应。
将PCR产物进行1%琼脂糖凝胶电泳, 将含有目的 DNA片段的琼脂糖凝胶切下, 然后使用 Tiangene DNA纯化试剂盒进行纯化。具体操作步骤按天根公司试剂盒的使用说明进行。
1.4 扩增片段的克隆与测序
将PCR产物经2%的琼脂糖凝胶电泳纯化后, 再经HQ & Q.Gel Extraction Kit II (U-gene)回收后, 将其克隆至pMD 18-T载体上(TaKaRa), 转化感受态E.coliJM109, 利用M13正、反向引物, 通过PCR反应,检测得到阳性克隆, 由上海生工公司测序。
1.5 基因片段的序列分析
将测序后所得的瓦氏黄颡鱼 FAD2和 ELOVL5 cDNA片段序列, 在BLAST (http://blast.ncbi.nlm.nih.gov/blast.cgi)进行同源比较。提交NCBI数据库后, 再根据所得到的cDNA序列, 推导其FAD2和ELOVL5氨基酸序列(http://www.ncbi.nlm.nih.gov/gorf/orfig.cgi); 再采用Expert Protein Analysis System (http://us.expasy.org/)以及 ANTHEPROT 2000 V5.2软件推导cDNA片段所得的FAD2和ELOVL5功能位点。
1.6 组织表达分析
按照RNAiso Plus (TaKaRa)试剂操作步骤分别提取9尾瓦氏黄颡鱼肝脏、肌肉、肠道、心脏、脾脏、肾脏、鳃和脑总RNA后, 用Prime ScriptTMRT reagent Kit with gDNA Eraser (Perfect Real Time)试剂合成cDNA。根据已获得瓦氏黄颡鱼 FAD2和 ELOVL5 cDNA设计基因特异性引物分析组织表达, 特异性序列见表 1。以β-actin为内参基因(表 1)(Zhenget al,2010), 采 用 2–ΔΔct法(Livaket al, 2001), 按 照SYBR®Premix Ex TaqTMII试剂操作步骤和 Roche Light Cycler Nano (USA)荧光定量PCR仪分析检测FAD2和ELOVL5 mRNA相对表达量。
1.7 数据统计分析
采用平均值±标准差(Mean±S.D.)表示试验数据,采用SPSS18.0软件处理试验结果。基于单因方差分析基础上, 采用 Duncan多重比较法对组间差异进行检验, 当P<0.05则为存在显著差异。

表1 PCR引物序列Tab.1 The PCR primer sequences
2 结果与讨论
动物体内可通过n-6和n-3途径合成HUFAs。以n-3途径合成高不饱和脂肪酸, 首先第一步反应是由α-亚麻酸(α-linolenic acid, ALA, 18:3n-3)转化为十八碳四烯酸(octadecatetraenoic acid, OTA, 18:4n-3); 而n-6途径则是由前体物质亚油酸(linoleic acid, LA,18:2n-6)转 化 为 γ-亚 麻 酸 (γ-linolenic acid, GLA,18:3n-6); 第一步转化的脱氢反应是 n-3和 n-6 合成途径的限速步骤, Δ6脂肪酸去饱和酶能将 α-亚麻酸和亚油酸分别转化成十八碳四烯酸和亚麻酸, 是由亚油酸和亚麻酸合成高不饱和脂肪酸的限速酶(Tocheret al, 2006)。如果动物体内缺乏Δ6去饱和酶,或该酶活性受到抑制, 就会妨碍机体内亚油酸向γ-亚麻酸(n-6途径)和α-亚麻酸向十八碳四烯酸(n-3途径)的转化, 影响HUFAs的生物合成(Bellet al, 2009)。然而, 不同鱼类Δ6脂肪酸去饱和酶的差异较大; 在大西洋鲑(Salmo salar)等洄游鱼类体内发现两种FAD的存在, 它们分别具有Δ5和Δ6的活性(Hastingset al,2005; Zhenget al, 2005), 而斑马鱼(Danio rerio)FAD则同时具有Δ5和Δ6活性(Hastingset al, 2001)。本实验采用 RT-PCR方法获得瓦氏黄颡鱼 FAD基因的cDNA片段, FAD基因cDNA序列片段1904bp, 包括86bp的5’-UTR、480bp的3’-UTR和1338bp的ORF(图1)。ORF分析获得 FAD完整的 446个氨基酸。经BLASTp比对分析发现, 瓦氏黄颡鱼的FAD2氨基酸序列与其他物种的 FAD2氨基酸序列有着高相似性(图 2)。瓦氏黄颡鱼 FAD2氨基酸与虎头鲨Pangasianodon hypophthalmus(88%, AFN21428.1)、黄颡鱼P.fulvidraco(80%, AJQ20793.1)、墨西哥脂鲤Astyanax mexicanus(76%, XP_007235183.1)、金钱鱼Scatophagus argus(68%, AHA62794.1)等 FAD2均有很高的同源性(表2)。因此可以推断所克隆到的cDNA为 FAD2基因片段, 将所得该基因片段提交至GenBank, 登录号分别为KY853758。瓦氏黄颡鱼FAD氨基酸序列含有多个保守的结构域(图 2), 与草鱼C.idellus、刀鲚Coilia nasus、军曹鱼R.canadum、黄鳝Monopterus albus、黄颡鱼P.fulvidraco等相似(杜嵇华等, 2011; 周秋白等, 2011; 魏广莲等, 2014; Songet al, 2015), 含有八个保守的组氨酸残基, 构成三个保守组氨酸富集区(histidine-richregion), 结构简式表示为HX(3-4)H、HX(2-3)HH、(H/Q)X(2-3)HH (Zhenget al, 2005), 3个保守的组氨酸富集区和1个Fe2+结合形成催化活性中心, 还包括亚铁血红素结合基序(HPGG)的类似细胞色素 b5结构域等(Zhenget al,2009)。Δ6脂肪酸去饱和酶是一种膜结合脂肪酸去饱和酶, 它以细胞色素b5氧化还原酶和NADH作为电子供体, 用来催化甘油酯中的脂肪酸脱氢(Sprecheret al, 1995)。此外, NADH-细胞色素b5还原酶可以通过FAD中的细胞色素b5结构域直接向FAD蛋白催化位点传递电子, 催化甘油酯中的脂肪酸脱氢, 而不需要细胞色素b5蛋白参与(Guillouet al, 2010)。前两个组氨酸簇位于2个跨膜区之间, 第三个组氨酸簇位于第二个跨膜区的后面, 这样三个保守的组氨酸簇与铁离子共同构成了 FAD发挥催化活性的催化中心。2个跨膜区对于它们的细胞定位和形成适合的空间结构十分重要(Loset al, 1998)。
脂肪酸碳链的延伸主要发生于内质网(endoplasmic reticulum, ER)、过氧化物酶体(peroxisome)和线粒体(mitochondrion)中。在内质网上,以脂酰CoA (fatty acyl-CoA)作为起点, 通过缩合、还原、脱水、再还原四个步骤循环完成碳链的延伸, 合成超长链脂肪酸(very long chain fatty acid, VLCFA)(Leonardet al, 2004)。超长链脂肪酸延伸酶(elongase of very long chain fatty acid, ELOVL)家族正是催化超长链脂肪酸合成循环反应中的第一个限速缩合步骤的酶类(Sprecher, 2000)。该家族的蛋白质序列中均含有KxxExxDT、HxxHH、HxxMYxYY、TxxQxxQ等相似的结构域。哺乳动物体内ELOVL家族包含7个成员, 其中ELOVL5参与延长单不饱和脂肪酸(C16、C18)和多不饱和脂肪酸(C18、C20、C22)。它能调控肝脏脂肪及碳水化合物的代谢, 从而影响机体血脂、血糖浓度(Wanget al, 2008)。目前鱼类中已经发现ELOVL5, 并成为HUFA合成代谢研究热点。本实验,采用RT-PCR克隆得到ELOVL5 cDNA片段1164bp,包括 70bp的 5’-UTR、212bp的 3’-UTR 和 882bp的ORF(图3); ORF编码294个氨基酸, BLASTp比对分析表明, 该氨基酸序列与其他鱼类 ELOVL5的氨基酸序列具有较高的相似性(图 4)。因此可以推断所克隆到的cDNA片段为瓦氏黄颡鱼ELOVL5基因, 将所得该基因片段提交至 GenBank, 登录号分别为KY853759。瓦氏黄颡鱼 ELOVL5氨基酸与拉氏巨鲶Pangasius larnaudii(88%, AGR45586.1)、非洲鲶(C.gariepinus, 86%, AAT81405.1)、草鱼(C.idella,81%, ADU04500.1)、墨西哥丽脂鲤(A.mexicanus, 78%,XP_007240116.1)等均有很高的同源性(表2)。瓦氏黄颡鱼ELOVL5基因含有内质网停留信号(K, R)、高度保守的氧化还原中心组氨酸簇(HxxHH, 位于第 2个跨膜区内)以及跨膜区(QxxFLHxYHH, KxxExxDT,TxxQxxQ, NxxxHxxNYxYY)等结构功能域(图4)。同样, 黄颡鱼P.fulvidraco、草鱼C.idella、黄鳝M.albus等鱼类 ELOVL5基因均含有组氨酸簇、碳末端内质网停留信号、多个跨膜区域等, 且在不同鱼类中高度保守(杜嵇华等, 2011; 周秋白等, 2011; Songet al,2015), 以上这些结构域对形成合适构型和其细胞定位有着重要的作用(Loset al, 1998)。

图1 瓦氏黄颡鱼FAD2基因cDNA序列及编码的氨基酸序列Fig.1 Nucleotide sequences of FAD2 cDNA and deduced amino acid sequence of P.vachellii

图2 瓦氏黄颡鱼FAD2基因氨基酸序列与其他物种FADS2基因的氨基酸序列多重比较Fig.2 Alignment of FAD2 of P.vachellii with other homologues using Clustal W program
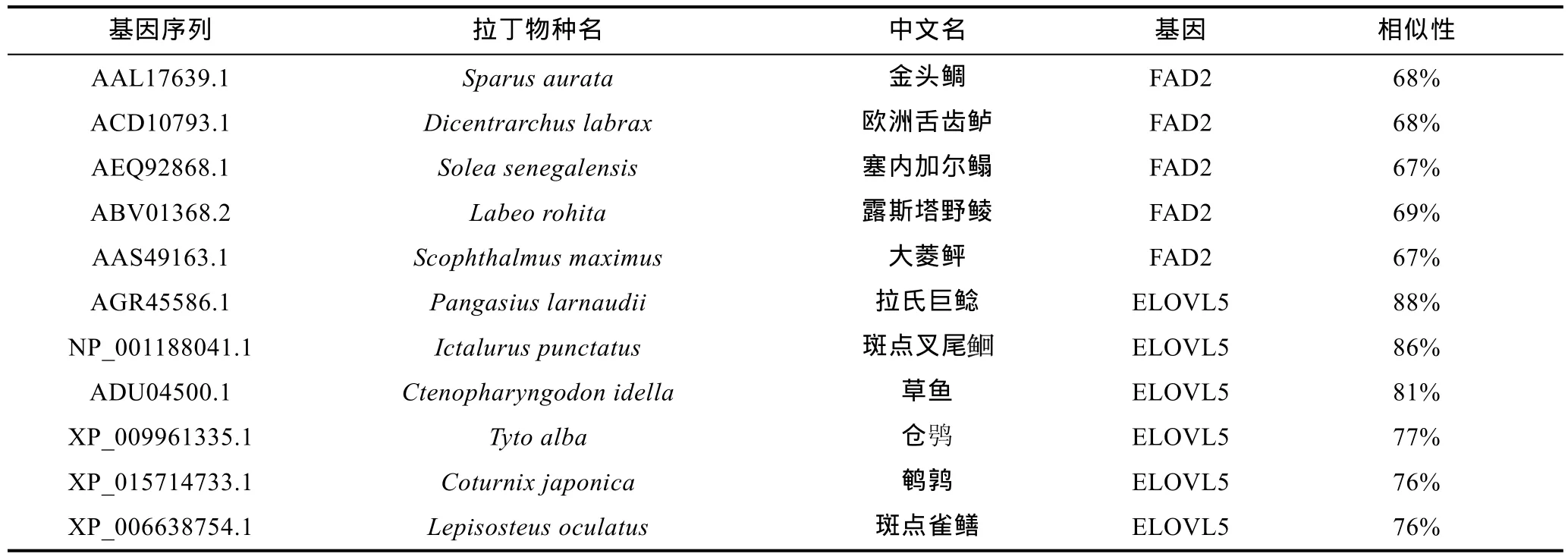
表2 物种名称、序列号和相似性Tab.2 Accession number, scientific name and identity of the members of FAD2 and ELOVL5 amino sequences

图3 瓦氏黄颡鱼ELOVL5基因cDNA序列及编码的氨基酸序列Fig.3 Nucleotide sequences of ELOVL5 cDNA and deduced amino acid sequence of P.vachellii

图4 瓦氏黄颡鱼ELOVL5基因氨基酸序列与其他物种ELOVL5基因的氨基酸序列多重比较Fig.4 Alignment of ELOVL5 of P.vachellii with other homologues using Clustal W program
脂肪酸去饱和酶基因在瓦氏黄颡鱼的 8种组织中均有表达。在肝脏和脑部的表达量最高, 其次为心、肠道、肾、脾、鳃, 而在皮肤和肌肉的表达量相对较低, 见图 5。类似地, 刀鲚 FAD6在脑、肠道、肌肉、肝脏、肾脏、心、鳃等组织中具有表达, 且刀鲚FAD6基因在脑中的表达量最高, 并且极显著地高于其他组织(P<0.01), 其次是肠道(魏广莲等, 2014)。非洲鲶C.gariepinus和赤鲈Perca fluviatilis的FAD2在肝脏和脑中的表达量也显著高于肠道、脾脏、肾脏等组织(Geayet al, 2016; Obohet al, 2016)。黄颡鱼P.fulvidracoFAD2 mRNA在肝脏和脑的表达水平显著高于肌肉、脾脏和鳃组织, 但显著低于肠道(Songet al,2015)。类似地, 大西洋鳕Gadus morhuaΔ6去饱和酶基因在脑中的表达量最高, 其次是肝、肾和肠(Tocheret al, 2006)。虹鳟Oncorhynchus mykiss的Δ6 FAD2基因在肝脏、脑、肠道等组织中有着较高的表达量(Seiliezet al, 2003)。有研究表明, 在幼鱼生长发育的关键阶段, 鱼体的神经组织特别在脑和视网膜中含有高浓度的DHA, 并且DHA和n-3 HUFA的总含量高于其他非神经组织中的含量(Tocheret al, 2006)。FAD2在多种鱼类的脑中均显著高于肠道、肝脏等营养代谢器官, 究其原因, 可能是脑中需要合成或摄取较多的高不饱和脂肪酸, 用以维持其正常的代谢功能的缘故(Naughton, 1981)。

图5 FAD2和ELOVL5 mRNA在瓦氏黄颡鱼组织中的相对表达量Fig.5 Distribution of FAD2 and ELOVL5 mRNA in tissues of P.vachellii
脂肪酸延伸酶基因 ELOVL5在瓦氏黄颡鱼的 8种组织中也均有表达, 其中 ELOVL5 mRNA在瓦氏黄颡鱼脑和肝脏中的表达量最高, 其次是垂体和肠道, 肌肉和血液中的表达水平较低(图 5)。类似地,ELOVL5在非洲鲶C.gariepinus和大西洋鳕G.morhua的组织中均有表达(Xueet al, 2014; Obohet al,2016)。ELOVL5 mRNA在大西洋鳕的脑和鳃组织中的表达水平要高于肌肉、血液、肝脏和心脏; 而ELOVL5a和ELOVL5b mRNA在大西洋鳕中肠道(幽门盲囊), 肝脏和脑的表达水平最高。类似于其他多种淡水鱼类, FAD2和ELOVL2 mRNAs的组织表达水平表明, 肝脏是鱼类长链多不饱和脂肪酸生物合成的主要场所(Moraiset al, 2009)。
3 结论
本实验成功克隆了瓦氏黄颡鱼ELOVL5和FAD2基因cDNA序列, 瓦氏黄颡鱼ELOVL5和FAD2氨基酸序列与其它鱼类ELOVL5和FAD2的氨基酸序列具有较高的相似性, 且含有高度保守的结构域。FAD2和ELOVL5 mRNA在瓦氏黄颡鱼脑、肝脏的表达量显著高于肠道、脾脏、肾脏、鳃等组织。结果表明, 瓦氏黄颡鱼具有合成高不饱和脂肪酸的关键酶FAD2和ELOVL5, 且肝脏可能为合成高不饱和脂肪酸的主要场所。
丁瑞华, 1994.四川鱼类志.成都: 四川科学技术出版社, 451许友卿, 郑一民, 丁兆坤, 2010.军曹鱼Δ6脂肪酸去饱和酶的cDNA序列克隆与基因表达.中国水产科学, 17(6):1183—1191
杜嵇华, 梁旭方, 程佳齐等, 2011.草鱼脂肪酸去饱和酶和延长酶基因 cDNA全序列的克隆与分析.暨南大学学报(自然科学版), 32(5): 513—520
吴代武, 何 杰, 叶元土等, 2017.日粮中鱼蛋白水解物对黄颡鱼生长、体成分和血清生理指标的影响.水产学报,41(3): 415—427
邵 婷, 覃川杰, 袁登越等, 2017.再投喂鱼油对瓦氏黄颡鱼肌肉脂肪酸组成的影响.水生生物学报, 41(1): 139—145
周秋白, 朱长生, 郑 宇等, 2011.黄鳝脂肪酸去饱和酶及延长酶基因cDNA的克隆与表达.江西农业大学学报, 34(1):134—140
覃川杰, 颉 江, 王永明等, 2013.植物油替代鱼油影响鱼类脂肪代谢的研究进展.海洋湖沼通报, (4): 89—100
魏广莲, 徐钢春, 顾若波等, 2014.刀鲚 Δ6脂肪酸去饱和酶cDNA的克隆与组织表达检测.南京农业大学学报, 37(3):138—144
Agaba M K, Tocher D R, Zheng X Zet al, 2005.Cloning and functional characterisation of polyunsaturated fatty acid elongases of marine and freshwater teleost fish.Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology, 142(3): 342—352
Bell M V, Tocher D R, 2009.Biosynthesis of polyunsaturated fatty acids in aquatic ecosystems: general pathways and new directions.In: Kainz M, Brett M T, Arts M T eds.Lipids in Aquatic Ecosystems.New York: Springer, 211—236
Cook H W, McMaster C R, 2002.Fatty acid desaturation and chain elongation in eukaryotes.In: Vance D E, Vance J E.New Comprehensive Biochemistry Volume 36:Biochemistry of Lipids, Lipoproteins and Membranes.4th ed.Amsterdam: Elsevier, 181—204
Geay F, Tinti E, Mellery Jet al, 2016.Cloning and functional characterization of Δ6 fatty acid desaturase (FADS2) in Eurasian perch (Percafluviatilis).Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 191: 112—125
Guillou H, Zadravec D, Martin P G Pet al, 2010.The key roles of elongases and desaturases in mammalian fatty acid metabolism: Insights from transgenic mice.Progress in Lipid Research, 49(2): 186—199
Hastings N, Agaba M, Tocher D Ret al, 2001.A vertebrate fatty acid desaturase with Δ5 and Δ6 activities.Proceedings of the National Academy of Sciences of the United States of America, 98(25): 14304—14309
Hastings N, Agaba M K, Tocher D Ret al, 2005.Molecular cloning and functional characterization of fatty acyl desaturase and elongase cDNAs involved in the production of eicosapentaenoic and docosahexaenoic acids from α-linolenic acid in Atlantic salmon (Salmo salar).Mar Biotechnol, 6: 463—474
Leonard A E, Pereira S L, Sprecher Het al, 2004.Elongation of long-chain fatty acids.Progress in Lipid Research, 43(1):36—54
Livak K J, Schmittgen T D, 2001.Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCtmethod.Methods, 25(4): 402—408
Los D A, Muratan N, 1998.Structure and expression of fatty acid desaturases.Biochimica et Biophysica Acta (BBA)-Lipids and Lipid Metabolism, 1394(1): 3—15
Morais S, Monroig O, Zheng X Zet al, 2009.Highly unsaturated fatty acid synthesis in Atlantic salmon: characterization of ELOVL5- and ELOVL2-like elongases.Marine Biotechnology, 11(5): 627—639
Naughton J M, 1981.Supply of polyenoic fatty acids to the mammalian brain: the ease of conversion of the short-chain essential fatty acids to their longer chain polyunsaturated metabolites in liver, brain, placenta and blood.International Journal of Biochemistry, 13(1): 21—32
Oboh A, Betancor M B, Tocher D Ret al, 2016.Biosynthesis of long-chain polyunsaturated fatty acids in the African catfishClarias gariepinus: Molecular cloning and functional characterisation of fatty acyl desaturase (fads2) and elongase(elovl2) cDNAs7.Aquaculture, 462: 70—79
Qin C J, Gong Q, Wen Z Yet al, 2017.Comparative analysis of the liver transcriptome ofPelteobagrus vachelliiwith an alternative feeding time.Comparative Biochemistry and Physiology Part D: Genomics and Proteomics, 22: 131—138
Seiliez I, Panserat S, Corraze Get al, 2003.Cloning and nutritional regulation of a Δ6-desaturase-like enzyme in the marine teleost gilthead seabream (Sparus aurata).Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology, 135(3): 449—460
Song Y F, Luo Z, Pan Y Xet al, 2015.Three unsaturated fatty acid biosynthesis-related genes in yellow catfishPelteobagrus fulvidraco: Molecular characterization, tissue expression and transcriptional regulation by leptin.Gene,563(1): 1—9
Sprecher H, Luthria D L, Mohammed B Set al, 1995.Reevaluation of the pathways for the biosynthesis of polyunsaturated fatty acids.Journal of Lipid Research,36(12): 2471—2477
Sprecher H, 2000.Metabolism of highly unsaturatedn-3 andn-6 fatty acids.Biochimica et Biophysica Acta (BBA)-Molecular and Cell Biology of Lipids, 1486(2): 219—231
Tocher D R, Ghioni C, 1999.Fatty acid metabolism in marine fish: Low activity of fatty acyl Δ5 desaturation in gilthead sea bream (Sparus aurata) cells.Lipids, 34(5): 433—434
Tocher D R, Zheng X Z, Schlechtriem Cet al, 2006.Highly unsaturated fatty acid synthesis in marine fish: cloning,functional characterization, and nutritional regulation of fatty acyl Δ6 desaturase of Atlantic cod (Gadus morhua L.).Lipids, 41(11): 1003—1016
Wang Y, Torres-Gonzalez M, Tripathy Set al, 2008.Elevated hepatic fatty acid elongase-5 activity affects multiple pathways controlling hepatic lipid and carbohydrate composition.Journal of Lipid Research, 49(7): 1538—1552
Xue X, Feng C Y, Hixson S Met al, 2014.Rise Characterization of the fatty acyl elongase (elovl) gene family, and hepatic elovl and delta-6 fatty acyl desaturase transcript expression and fatty acid responses to diets containing camelina oil in Atlantic cod (Gadus morhua).Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology,175: 9—22
Zheng X Z, Ding Z K, Xu Y Qet al, 2009.Physiological roles of fatty acyl desaturases and elongases in marine fish:Characterisation of cDNAs of fatty acyl Δ6 desaturase andelovl5elongase of cobia (Rachycentron canadum).Aquaculture, 290(1—2): 122—131
Zheng X Z, Tocher D R, Dickson C Aet al, 2005.Highly unsaturated fatty acid synthesis in vertebrates: new insights with the cloning and characterization of a Δ6 desaturase of Atlantic salmon.Lipids, 40(1): 13—24
Zheng K K, Zhu X M, Han Det al, 2010.Effects of dietary lipid levels on growth, survival and lipid metabolism during early ontogeny ofPelteobagrus vachellilarvae.Aquaculture,299(1—4): 121—127
MOLECULAR CLONING AND EXPRESSION OF FATTY ACID DESATURASE AND ELONGASE GENES IN DARKBARBEL CATFISHPELTEOBAGRUS VACHELLII
QIN Chuan-Jie1, WEN Zheng-Yong1, YUAN Deng-Yue1, SHAO Ting1, GONG Quan2
(1.Key Laboratory of Sichuan Province for Fishes Conservation and Utilization in the Upper Reaches of the Yangtze River,Neijiang Normal University,Neijiang641112,China; 2.Fisheries Institute,Sichuan Academy of Agricultural Sciences,Chengdu611731,China)
Understanding the endogenous synthesis of long-chain polyunsaturated fatty acids is of benefit to fish feed formulation.A FADS2 and an ELOVL5 cDNA were cloned containing open reading frames (ORF) of 1338 base pair (bp)and 864 bp specifying proteins of 447 and 294 amino acids, respectively.The deduced amino acid sequence ofPelteobagrus vachelliiFADS2 possessed conserved motif and included the histidine boxes, cytochrome b5 domain,transmembrane regions, and ELOVL 5 containing histidine boxes, endoplasmic reticulum retention signal domain, and transmembrane regions.In addition, FADS2 and ELOVL 5 mRNA distributed mainly in the brain and liver, and expressed significantly higher than that of intestine, spleen, kidney, gill, heart and muscle.These results indicate thatP.vachelliipossessed the desaturase and elongase, and the liver was the main tissues for synthesis of long-chain polyunsaturated fatty acids.
fatty acid desaturase; fatty acid elongase;Pelteobagrus vachellii; cDNA
Q789
10.11693/hyhz20170400088
* 国家自然科学基金项目, 31402305号; 四川省科技厅应用基础项目, 2017JY0161号; 四川省“十三五”育种攻关项目,2016NYZ0024号。覃川杰, 博士, 副教授, E-mail: qinchuanjie@126.com
2017-04-07, 收修改稿日期: 2017-04-25
猜你喜欢
杂志排行
海洋与湖沼的其它文章
- 基于de novo高通量测序的曼氏无针乌贼(Sepiellajaponica)ESTs中微卫星位点筛选与特征分析*
- 泥蚶(Tegillarca granosa)重组铁蛋白富集重金属离子的特性及化学传感器的研究*
- 厚壳贻贝(Mytilus coruscus)金属硫蛋白MT-10:cDNA克隆、结构分析及铜离子胁迫下的表达*
- 舟山海洋沉积物及沿海沉积物流动相石油污染的电阻率特性及其影响因素研究*
- 迟缓型爱德华氏菌(Edwardsiella tarda)诱导牙鲆(Paralichthys olivaceus)TLR1及TLR2基因的表达分析*
- 鲤科经济鱼类马口鱼(Opsariichthys bidens)胚胎发育及仔稚鱼形态与生长观察研究*
