牵牛E3泛素连接酶PNRma1基因的克隆及抗旱相关性分析
2017-12-14代锦绣董轩名倪欢欢董延龙金晓霞
代锦绣,董轩名,倪欢欢,董延龙,金晓霞*
(1.哈尔滨师范大学,哈尔滨 150025;2.黑龙江省农业科学院园艺分院,哈尔滨 150069)
牵牛E3泛素连接酶PNRma1基因的克隆及抗旱相关性分析
代锦绣1,董轩名1,倪欢欢1,董延龙2,金晓霞1*
(1.哈尔滨师范大学,哈尔滨 150025;2.黑龙江省农业科学院园艺分院,哈尔滨 150069)
为研究牵牛E3泛素连接酶对其干旱胁迫的响应情况,利用RT-PCR技术从牵牛中克隆得到1个E3泛素蛋白连接酶基因PNRma1的cDNA全长序列,其序列长度为734 bp,编码244个氨基酸。对牵牛PNRma1蛋白进行二级结构预测,结果表明该蛋白主要由α螺旋(41.88%)和无规则卷曲(37.18%)构成,并且牵牛PNRma1蛋白与野生潘那利番茄的同源性最高(相似度为94%)。在干旱胁迫处理下,PNRma1基因在牵牛根部的表达量总体上与对照相比呈现上升后下降的趋势,其在叶片中表达水平呈现后期上升趋势,试验结果表明,PNRma1基因响应牵牛的干旱胁迫反应,并且在根部(6h)早于叶片(9h)应答干旱信号。
牵牛;PNRma1;生物信息学;表达分析
植物响应逆境胁迫的过程与蛋白质代谢的过程息息相关。在植物蛋白质代谢过程中,某些特定功能蛋白质的降解对于植物响应外界逆境胁迫,维持细胞内稳态和植物体正常的生长发育具有十分重要的作用。
在目前所发现的真核生物体各种蛋白质降解途径中,泛素/26S蛋白酶体系统是其中一个具有高度选择性的降解蛋白质的途径。它是由26S蛋白酶体系统以及泛素启动酶系统共同构成的。其中泛素启动酶系统主要负责对所需要降解的特异性目标蛋白的泛素化[1],它是由三大酶类一起组成的,分别为泛素激活酶E1、泛素结合酶E2和泛素-蛋白连接酶E3。
在泛素/26S蛋白酶体中,泛素-蛋白连接酶E3虽然发现的最晚,但是种类繁多并且功能复杂,已经是当今生物学研究中的热点。它广泛存在与真核生物中,参与植物生长发育过程中的花发育[2]、自交不亲和[3]以及植物衰老[4]等过程,并且在植物的抗病反应、激素反应和逆境胁迫调节中发挥重要的作用[5]。目前发现的泛素-蛋白连接酶E3根据亚基的组成可以分为单亚基型和多亚基型,其中单亚基型主要包括三大类:HETC(homologous to E6-APC terminus)蛋白家族、RING(really interesting new gene)蛋白家族和U-box蛋白家族[6]。Lee等[7]从辣椒中提取出一个E3泛素-蛋白连接酶Rma1H1蛋白,经鉴定它是属于RING-finger类型蛋白,它由Rma1基因编码并能够正调控植物的干旱胁迫反应,从而极大地引起了人们对该类蛋白在应答植物抗逆反应中的兴趣。 Bae等[8]后来也从水稻中分离出辣椒Rma1H1的同源蛋白,该蛋白由osrdcp1基因编码,OsRDCP1同样参与对水稻干旱的正调控;Li等[9]发现了一个RING-finger 类型泛素E3连接酶—OsBBI1蛋白,它的表达受的稻瘟病菌诱导,编码OsBBI1蛋白的基因—osbbi1基因过表达可以提高水稻叶鞘表皮细胞壁厚度从而增强水稻对稻瘟病的抗性;Park[10]等发现了一个RING-finger 类型泛素E3连接酶—OsDSG1蛋白,它是从种子延迟萌发的T-DNA突变群体中鉴定出的,编码该蛋白的基因—osdsg1基因的表达能够延迟种子萌发,因此osdsg1突变体可以增强植物应对干旱和高盐胁迫的耐受性。
本试验通过对牵牛中RING-finger型编码E3泛素连接酶蛋白基因—PNRma1基因的克隆,在此基础上对该基因进行生物信息学分析及预测后,进一步研究其在干旱胁迫下的表达特性,为进一步研究该蛋白在牵牛生长发育及抗性生理过程中的作用提供理论依据。
1 材料与方法
1.1 供试材料
供试材料为牵牛(PharbitisnilL.)。
1.2 试验设计与方法
1.2.1 牵牛RNA的提取。牵牛总RNA的提取使用采用常规Trizol法。RNA质量的检测,用浓度为0.1%的琼脂糖凝胶电泳,检测所提出的牵牛总RNA的质量。
1.2.2 牵牛cDNA第一链的合成。利用反转录试剂盒(TOYOBO)进行反转录反应,反应液的配置如表1所示。

表1 cDNA第一链合成反应液组成
1.2.3 牵牛PNRma1基因cDNA序列克隆
1.2.3.1 牵牛PNRma1基因的引物设计:利用辣椒的Rma1H1的cDNA序列为信息,与已经公布的牵牛基因组序列(http://gkcsuja.solgenomics.net/tools/blast/)进行比对,获得一段基因序列(登陆编号:Solyc07g054080.1),根据这段基因的cDNA序列信息,在Sanger Biotech公司设计特异引物RMA-Q1M和RMA-Q2M(表2)。

表2 PNRma1基因引物名称及序列
1.2.3.2 PCR体系及反应程序:牵牛PNRma1基因的PCR克隆反应体系为:2×ES Tap Master Mix 12.5μL,cDNA 1μL,上下游引物各1μL,ddH2O 9.5μL,总体积25μL。PCR反应的程序是:94℃ 2min,94℃ 30s,58℃ 30s,72℃ 30s,35个循环,最终延长72℃ 2min。PCR扩增产物用浓度为1 %的琼脂糖凝胶进行电泳检测。
1.2.4 牵牛PNRma1基因cDNA序列的回收。利用胶回收试剂盒(原平皓生物公司)说明书对牵牛PNRma1基因cDNA的PCR产物进行回收,回收产物进行1%琼脂糖凝胶电泳检测。
1.2.5 牵牛PNRma1基因的生物信息学分析。根据牵牛PNRma1的氨基酸序列,利用NCBI-blast(http://blast.ncbi.nlm.nih.gov/Blast.cgi)在线分析软件对其序列与其他植物进行比对,为进一步研究PNRma1蛋白氨基酸序列相似性及进化关系,用软件DNAMAN对序列进行多重比对,然后再用软件MEGA 4构建系统进化树;利用ProtParam(http: //web.expasy.org/protparam)分析蛋白质的基本理化性质;利用PSORT Prediction(http: //psort.hge.jp/form.html)预测蛋白质的亚细胞定位;利用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)预测氨基酸的二级结构;利用SWISS-MODEL(http://swissmodel.expasy.org/)预测氨基酸的三级结构。
1.2.6 干旱胁迫下PNRma1基因的表达特性分析。为研究PNRma1基因在牵牛干旱胁迫下的表达特性,对牵牛进行10% PEG6000干旱胁迫处理,在处理后的0,3,6,9,12h提取牵牛植株的根部和叶片的RNA,进行半定量RT-PCR表达分析。
2 结果与分析
2.1 牵牛总RNA的获得与分析
利用Trizol法提取牵牛总RNA,用1%琼脂糖凝胶电泳检验后获得了RNA条带(图1)。电泳后获得的条带较清晰,说明所提取出牵牛总RNA未发生降解,可以用于接下来的反转录。

图1 牵牛RNA的提取
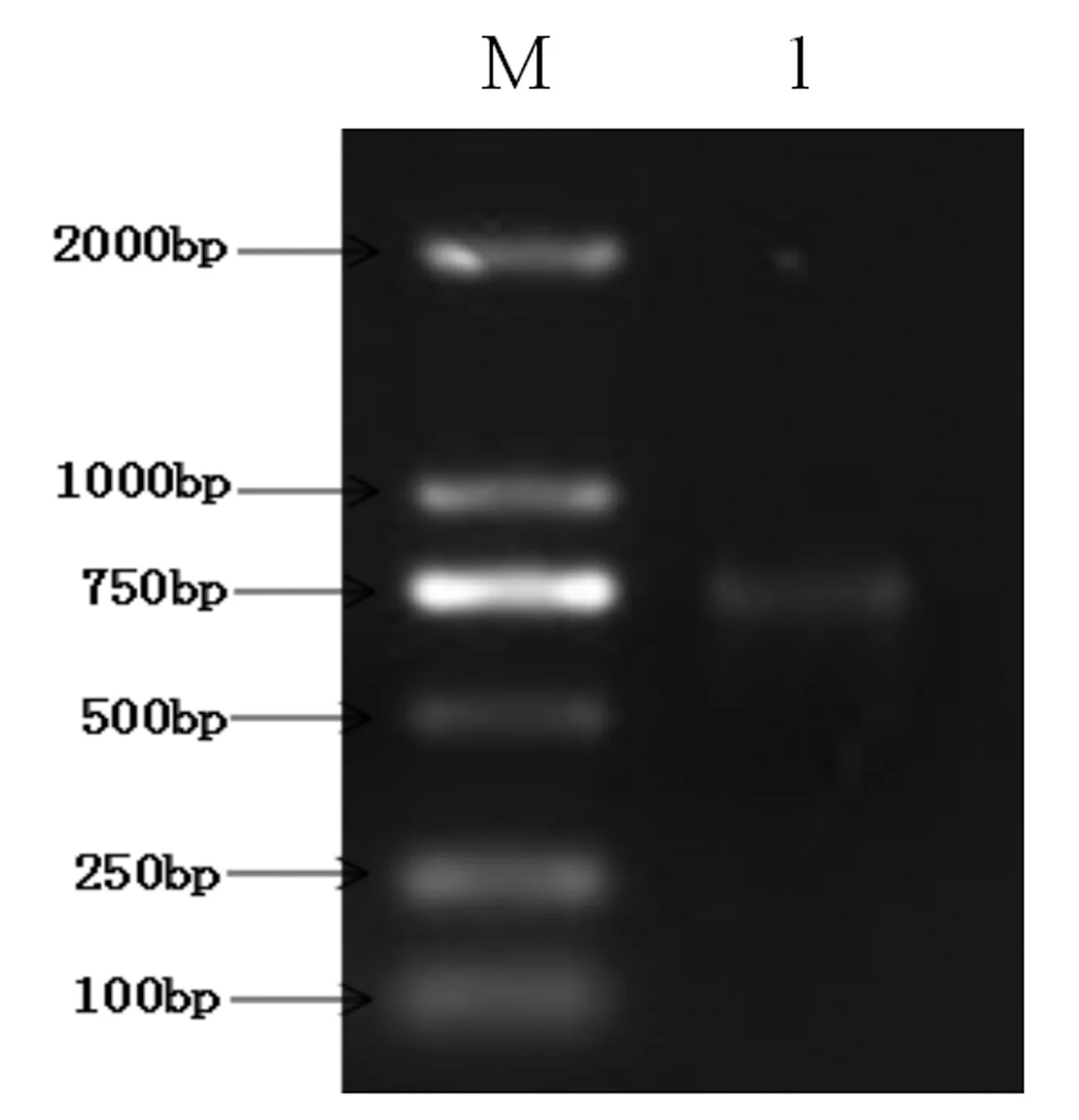
图2 牵牛PNRma1全长cDNA PCR扩增结果M:分子量标准 DL2000 1:PCR产物
2.2 牵牛PNRma1基因cDNA克隆结果
使用牵牛叶片的cDNA为模板,利用设计出的PNRma1引物进行PCR扩增,克隆出牵牛PNRma1基因,回收得到牵牛PNRma1基因,最后电泳检测得到一条750bp左右的目的条带(图2)。
2.3 生物信息学分析
2.3.1 牵牛PNRma1氨基酸同源性分析及系统进化树构建。将克隆得到的cDNA 序列进行生物信息学分析,通过NCBI的ORF Finder程序进行分析,结果显示开放读码区为734 bp,编码244个氨基酸(图3)。
根据已克隆的牵牛PNRma1氨基酸序列在NCBI上与其他植物的同源氨基酸序列进行比对分析,结果表明(图4),牵牛PNRma1氨基酸序列与野生潘那利番茄一致性为94%,与甜椒、番茄和马铃薯的一致性分别为92%、91%、90%,与各种烟草的一致性为80%以上,说明PNRma1基因在这些植物中比较保守。

图3 PNRma1可读框核苷酸序列及氨基酸序列

图4 牵牛PNRma1与其他物种Rma1氨基酸序列保守区域的多重比对
将上述在氨基酸序列与比对较高的植物生成进化树,用于分析牵牛PNRma1与其他物种中相关蛋白的进化关系,结果表明(图5),牵牛PNRma1与番茄同属1个分支;美花烟草和野生烟草的Rma1属于同1个分支;胡萝卜属于并系。
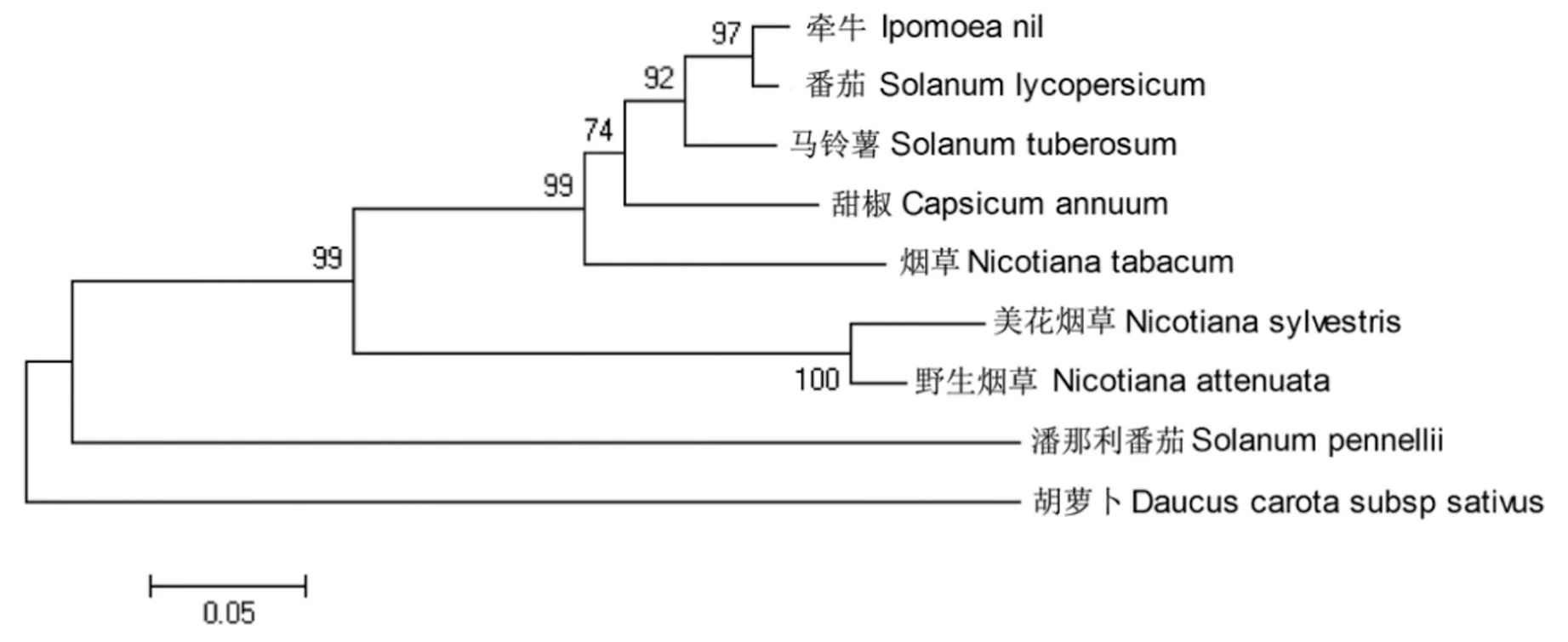
图5 PNRma1氨基酸序列的系统进化树
2.3.2 牵牛PNRma1蛋白的二级结构预测。利用SOPMA对PNRma1蛋白的二级结构进行预测,结果如图6所示,大写行是氨基酸序列,下面的小写行是其对应的二级结构,其中h代表α-螺旋(alpha helix)、e代表延伸链(extended strand)、t代表β-转角(beta turn)、c代表无规则卷曲(random coil)。图中结果表明PNRma1蛋白的二级结构中,h占41.88%,e占14.96%,t占5.98%,c占37.18%。由此推测,PNRma1蛋白含有大量的α-螺旋和无规则卷曲。

图6 PNRma1蛋白二级结构预测
2.3.3 PNRma1蛋白的三级结构预测。利用SWISS-MODEL对PNRma1基因编码的氨基酸三级结构进行预测,如图7所示,牵牛PNRma1蛋白的三级结构区呈现一定的螺旋弯曲结构,其中有4个明显的螺旋结构。

图7 PNRma1蛋白结构域区三级结构预测
2.4 干旱胁迫下,牵牛PNRma1基因的RT-PCR表达特性
为研究PNRma1基因在牵牛受到干旱胁迫下的响应特征,利用半定量RT-PCR技术进行该基因的表达特性分析,结果如图8和图9所示。该基因在牵牛受到干旱胁迫时的根部表达图片看出,其表达在6h时增强显著,显著高于0h初始时期,之后12h时表达量减弱。而在牵牛叶片,PNRma1基因的表达在9h和12h的表达强于初始时期,以上表明PNRma1基因响应牵牛的干旱胁迫反应,并且根部(6h)早于叶片(9h)应答干旱信号,PNRma1基因增强表达显著。

图8 干旱胁迫下牵牛PNRma1基因在根部的表达M:DL2000;1:0h;2:3h;3:6h;4:9h;5:12h

图9 干旱胁迫下牵牛PNRma1基因在叶片的表达M:DL2000;1:0h;2:3h;3:6h;4:9h;5:12h
3 小结与讨论
3.1 小结
本研究通过RT-PCR方法从牵牛中克隆到一个泛素E3连接酶基因PNRma1,cDNA全长为734 bp,可编码244个氨基酸。牵牛PNRma1蛋白与其他植物进行同源性分析,发现其与野生潘那利番茄一致性为94%,与甜椒、番茄和马铃薯的一致性分别为92%、91%、90%,说明该基因在这些植物中保守性较高。对PNRma1蛋白进行生物信息学分析表明,其二级结构中,h占41.88%,c占37.18%,推测PNRma1蛋白含有大量的α-螺旋和无规则卷曲。该蛋白的三级结构区预测含有4个明显的螺旋结构。在以上基础上,进一步研究表明PNRma1基因早期响应牵牛的干旱胁迫反应,基因表达增强显著,并且该基因在根部(6h)早于叶片(9h)应答干旱信号。
3.2 讨论
早在2003年,辣椒Rma1H1作为干旱诱导基因就被最初鉴定出来[11],进而陆续对该基因进行研究[7-8,12]。虽然部分植物Rma1基因已被克隆,但在观赏植物牵牛中尚未见报道。本试验中采用RT-PCR的方法,获得牵牛PNRma1基因cDNA序列(图1、2)。牵牛PNRma1与其他8种植物进行同源性分析,发现其在野生潘那利番茄的同源性最高为94%,其次是甜椒、番茄和马铃薯(图4),这说明PNRma1基因在这些植物中较保守。
对牵牛进行干旱处理,RT-PCR检测结果表明:在干旱胁迫处理下,PNRma1基因在牵牛叶片和根部的表达量在胁迫初期都呈现一次明显的上升(图8、9),总体上表达量与对照相比呈现上升趋势,这与玉米中鉴定出的RING型E3泛素连接酶ZmGW2相似,在玉米苗期不同胁迫处理下,ZmGW2基因在根和叶中均被诱导表达,在干旱胁迫条件下,该基因前期表现出上调表达,而后表达量逐渐下降[13],并且PNRma1基因在根部表达(6h)早于叶片表达(9h)来应答干旱信号。以上说明PNRma1基因是一个受干旱胁迫诱导增强表达的基因且在根部优势表达。
本试验结果表明PNRma1基因可能参与牵牛的干旱应答及信号转导过程,且在不同器官所起的调节作用有一定的差异,其机理需进一步研究。本研究结果为进一步研究牵牛的抗旱分子机理提供了试验基础,为牵牛抗旱遗传改良提供了理论依据和候选基因资源。
[1]吴慧娟,张志刚. 泛素-蛋白酶体途径及意义[J]. 临床与病理杂志,2006,26(1):7-10.
[2]Park B S,Eo H J,Jang I C,et al. Ubiquitination of LHY by SINAT5 regulates flowering time and is inhibited by DET1[J]. Biochemical & Biophysical Research Communications,2010,398(2): 242-6.
[3]于晓敏,蓝兴国,李玉花. 泛素/26S蛋白酶体途径与显花植物自交不亲和反应[J]. 植物学报,2006,23(2): 197-206.
[4]Lin S,Martin R,Mongrand S,et al. RING1 E3 ligase localizes to plasma membrane lipid rafts to trigger FB1-induced programmed cell death in Arabidopsis[J]. Plant Journal for Cell & Molecular Biology,2008,56(4): 550-61.
[5]刘辉志,韩世平. 泛素-蛋白连接酶(E3)—RING-finger蛋白的研究进展[C]. 河南省植物生理学会三十周年庆典暨学术研讨会. 2010.
[6]Hatakeyama S,Yada M,Matsumoto M,et al. U box proteins as a new family of ubiquitin-protein ligases[J]. Biochemical & Biophysical Research Communications,2003,302(4): 635.
[7]Lee H K,Cho S K,Son O,et al. Drought Stress-Induced Rma1H1,a RING Membrane-Anchor E3 Ubiquitin Ligase Homolog,Regulates Aquaporin Levels via Ubiquitination in Transgenic Arabidopsis Plants[J]. Plant Cell,2009,21(2): 622.
[8]Bae H,Kim S K,Cho S K,et al. Overexpression of OsRDCP1,a rice RING domain-containing E3 ubiquitin ligase,increased tolerance to drought stress in rice (Oryza sativa L.)[J]. Plant Science An International Journal of Experimental Plant Biology,2011,180(6): 775-82.
[9]Li W,Zhong S,Li G,et al. Rice RING protein OsBBI1 with E3 ligase activity confers broad-spectrum resistance against Magnaporthe oryzae by modifying the cell wall defence[J]. Cell Research,2011,21(5):835.
[10]Park J J,Yi J,Yoon J,et al. OsPUB15,an E3 ubiquitin ligase,functions to reduce cellular oxidative stress during seedling establishment[J]. Plant Journal,2011,65(2): 194.
[11]Park JA,Cho SK,Kim JE,et al. Isolation of cDNAs differentially expressed in response to drought stress and characterization of the Ca-LEAL1 gene encoding a new family of atypical LEA-like protein homologue in hot pepper (Capsicum annuum L. cv. Pukang)[J]. Plant Sci,2003,165:471-481.
[12]Seo YS,Choi JY,Kim SJ,et al. Constitutive expression of CaRma1H1,a hot pepper ER-localized RING E3 ubiquitin ligase,increases tolerance to drought and salt stresses in transgenic tomato plants[J]. Plant cell reports,2012,31(9),1659-1665.
[13]刘方方,姜涛. 玉米中 RING型 E3泛素连接酶基因ZmGW2的表达分析[J].玉米科学,2013,21(2) :47-51.
CloningandDroughtResistanceAnalysisofE3UbiquitinLigasePNRma1GeneinPharbitisnilL.
Dai Jinxiu1,Dong Xuanming1,Ni Huanhuan1,Dong Yanlong2,Jin Xiaoxia1*
(1.Harbin Normal University,Harbin 150025;2.Heilongjiang Academy of Agricultural Sciences Horticultural branch,Harbin 150069)
The experiment cloned an cDNA sequence of E3 ubiquitin ligase genePNRma1 by RT-PCR method withPharbitisnilL. used as experimental materials. The length ofLeRma1 gene was 734 bp,which encoded 244 amino acids. Two stage structure of PNRma1 protein was predicted and the results showed that the protein was mainly composed of alpha helix (41.88%) and irregular coiled coil (37.18%). Homology analysis showed that the deduced amino acid sequence exhibited high homology withSolanumpennellii(94% similarity). Under drought stress,the expression ofPNRma1 gene compared with the control in the root showed a trend of increase and then decrease,and its expression level in leaves showed upward trend later,the results showed that thePNRma1 gene responded the drought stress response of morning glory and responded to drought signals at the root (6h) earlier than the leaf (9h) response.
PharbitisnilL.;PNRma1; Bioinformatics; Expression analysis
2017-05-04
黑龙江省大学生创新训练计划项目(201510231017)
*通讯作者:金晓霞(1980-),女,博士,副教授,主要从事植物分子生物学研究,E-mail:xiaoxia6195@163.com。
DOI.:10.13268/j.cnki.fbsic.2017.06.002
Q814
A
