玉米干种子基因组DNA提取方法的改进
2017-12-13董永军王陆军郝建平王艳梅李培良王创云邓艳芳李志敏张婷婷
董永军 ,王陆军 ,郝建平 ,王艳梅 ,李培良 ,王创云 ,邓艳芳 ,周 琼 ,李志敏,庞 冰,张婷婷
玉米干种子基因组DNA提取方法的改进
董永军1,王陆军2,3,郝建平1,王艳梅2,3,李培良2,3,王创云2,3,邓艳芳3,周 琼3,李志敏3,庞 冰1,张婷婷1
(1.山西大学生命科学学院,山西太原030006;2.山西省农业科学院作物科学研究所,山西太原030031;3.山西省玉米工程技术研究中心,山西太原030031)
为了实现玉米干种子基因组DNA快速提取,针对玉米干种子淀粉含量高的特点,对传统核酸CTAB提取方法进行了优化与改良,将提取的DNA进行分光光度计及琼脂糖凝胶电泳检测。结果显示,DNA的OD260/OD280值为1.79~1.95,OD260/OD230值为1.90~2.15;琼脂糖凝胶电泳图谱清晰,条带无拖尾现象,说明采用改良方法提取的DNA质量高、无降解。以改良方法提取的DNA样品作为模板进行分子标记检测试验,结果清晰稳定,进一步证明采用改良CTAB法直接从玉米干种子中提取的DNA可以满足SSR和SNP等分子试验的要求。
玉米;干种子;DNA;CTAB
近年来,随着分子育种技术的发展与应用,玉米育种进程显著加快,分子育种越来越受到广大研究者的青睐。提取高质量的核酸是进行玉米分子育种研究的基础,核酸的质量对下游分子试验的影响至关重要。
目前,常用来提取植物基因组DNA的方法有CTAB 法[1-3]、SDS 法[4-5]、酶法[6]和高盐提取法[7]等。其中,CTAB法仍被许多实验室用来提取玉米基因组DNA,提取材料一部分是玉米嫩叶,提取DNA时需要进行发芽、液氮研磨,费时费力;另一部分是玉米干种子,提取周期长,质量欠佳。分子育种研究的样品材料多数为干种子,而其中含有大量的多糖、淀粉、蛋白质等物质[8],如何快速有效地去除这些物质,从玉米干种子中提取到高质量的基因组DNA,是分子育种研究的关键技术。
本研究在传统的CTAB法的基础上,通过对提取试剂、步骤等进行优化,旨在集成一种简单、快速而且可以直接从玉米干种子中提取高质量基因组DNA的新方法,以满足玉米种子纯度快速检测的需要。
1 材料和方法
1.1 材料
1.1.1 供试材料 玉米选自市场上主要的10个杂交品种,其来源列于表1。

表1 玉米品种及来源
1.1.2 试验试剂 2%CTAB缓冲液,氯仿∶异戊醇(24∶1),蛋白酶K,70%乙醇,异丙醇等。
1.1.3 试验仪器 高速冷冻离心机(eppendorf中国有限公司),PCR仪(eppendorf中国有限公司),全自动样品快速研磨仪(上海净信实业发展有限公司),微量紫外分光光度计(北京凯奥科技发展有限公司),垂直电泳仪(北京东方君意电泳设备有限公司)。
1.2 方法
1.2.1 传统CTAB核酸提取方法 其参照SAGHAIMAROOF等[9]的方法进行。
1.2.2 改良CTAB玉米干种子基因组DNA提取方法 破壁,取一粒玉米干种子放入2 mL离心管中,加直径6 mm钢珠1粒,在70 Hz全自动样品快速研磨仪中研磨3 min;裂解,加入800μL预热CTAB缓冲液和 20μL蛋白酶 K,65℃水浴 10 min,13 000 r/min离心1 min,取上清;提纯,加入氯仿∶异戊醇(24∶1)800 μL,振荡 3 min,13 000 r/min 4℃离心5 min,取上清液置于离心管中,重复提纯1次;沉淀,加入0.8倍体积预冷的异丙醇,混匀,-20℃静置 10 min,13 000 r/min 4 ℃离心 5 min,弃上清液;洗涤,用70%的乙醇洗涤2次;保存,将DNA样品风干,加100μL TE Buffer(含RNA酶),37℃保温箱温育2 h,置于-20℃保存。
1.2.3 DNA质量检测 用微量紫外分光光度计测量玉米基因组DNA的OD260/OD280和OD260/OD230值;将DNA样品质量浓度统一稀释到500 ng/μL,取5μL DNA样品进行0.8%的琼脂糖凝胶电泳检测。
1.2.4 分子标记检测 选用SSR分子标记对提取的DNA进行检测,PCR体系为20μL,DNA模板质量浓度为20 ng/μL,检测方法参考郭景伦等[10]的方法进行。SSR引物为 Bnlg1401,序列为 F:5′-CACTCGGTTTTTGCTTAGCC-3′,R:5′-GTGTCGTC GAGTGCATGC-3′。
2 结果与分析
2.1 玉米基因组DNA的纯度和浓度
利用微量紫外分光光度计分别测量改良CTAB法和传统CTAB法提取的玉米干种子DNA的OD值,其测量结果列于表2。

表2 采用改良CTAB法和传统CTAB法所得DNA分光光度计检测结果
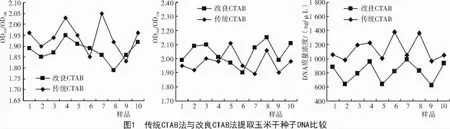
由表2可知,改良CTAB法提取的玉米干种子DNA,其OD260/OD280值介于1.79~1.95,OD260/OD230值介于1.90~2.15,DNA质量浓度介于619.92~986.79 ng/μL;传统CTAB法提取的玉米干种子DNA,其OD260/OD280值介于1.83~2.05,OD260/OD230值介1.89~2.11,DNA质量浓度介于 964.41~1 382.64 ng/μL。当 DNA的 OD260/OD280值为 1.80~1.90,OD260/OD230值在2.00以上时,其质量是最理想的[11]。
从图1可以看出,改良CTAB法提取DNA样品的OD260/OD280平均值为1.88,方差为0.001 9;OD260/OD230平均值为2.04,方差为0.004 9。传统CTAB法提取DNA的OD260/OD280平均值为1.94,方差为0.004 8;OD260/OD230平均值为1.97,方差为0.006 2。传统CTAB法提取的DNA样品OD260/OD280值和OD260/OD230值的偏离度较大,说明利用改良CTAB法提取的DNA样品分子结构完整,纯度高,质量稳定,能够满足玉米分子检测试验对DNA质量的要求。改良CTAB法提取的干种子DNA质量浓度整体比传统CTAB法低约300 ng/μL,但其质量较高,对后期实验影响较小。
2.2 琼脂糖凝胶电泳检测结果
玉米干种子基因组DNA琼脂糖凝胶电泳检测结果如图2所示。由图2可知,1~10号样品,电泳条带无拖尾,DNA无明显降解现象,带型整齐一致;11~20样品,电泳条带弥散,有轻度拖尾现象,存在RNA污染。表明改良CTAB法比传统CTAB法提取的DNA纯度更高,结构更完整,质量更稳定。

2.3 分子标记检测结果
以改良CTAB法提取的玉米基因组DNA作模板进行SSR分子标记检测,结果如图3所示。从图3可以看出,电泳条带稳定、清晰、无杂带,根据条带的迁移位置可以清楚区分不同品种之间的差异。结果表明,利用改良CTAB法提取的玉米干种子DNA完全可以满足SSR分子标记对玉米的真实性、一致性、遗传图谱构建、遗传多样性分析等研究的要求[12-13]。

3 讨论
与传统CTAB法相比,改良CTAB法采用全自动样品研磨仪对玉米材料进行机械粉碎,放弃了液氮研磨,缩短了裂解时间,简化了提取过程,提取周期小于2 h;提取过程不需添加β-巯基乙醇、聚乙烯吡咯烷酮(PVP)等有毒化学试剂,减少了对环境的污染;在提取溶液的配制上,提高Tris-HCl和EDTA的浓度,强化了核酸与其他物质的分离。另外,本研究改良CTAB方法在加入氯仿∶异戊醇之前加入少量蛋白酶K,可以增强细胞膜的破坏、加速核膜的裂解。本试验尝试省略一步氯仿∶异戊醇的提取步骤,虽然可以从样品中提取到DNA,但是所得到的DNA含有大量的杂质,纯度远低于2步氯仿∶异戊醇的提取效果。因此,保留了2步氯仿∶异戊醇的提取步骤,以确保DNA提取产物的纯度和质量。
经微量紫外分光光度计检测,传统CTAB法从玉米干种子中提取的DNA样品,其OD260/OD280值和OD260/OD230值有70%的样品偏离最佳值区间,出现这种现象的原因可能是由于传统CTAB法提取DNA的过程中,RNA去除不彻底,导致OD260/OD280的值偏高;去除杂质不干净,存在糖、盐、无机离子和其他不利因素的污染,导致OD260/OD230值偏低。传统CTAB法提取的DNA浓度比改良CTAB法的高,是由于DNA提取过程中裂解时间长,导致提取的DNA量多。
随着玉米育种进入分子时代,高效且保质保量提取玉米基因组DNA是进行分子育种的首要条件。本试验研究的改良CTAB法可直接提取玉米干种子DNA,适用于SSR,SNP分子标记进行遗传图谱构建、QTL分析等研究[14-18],其提取的DNA质量和浓度完全可以满足实验的要求。
[1]王嘉荟,王琳,王金胜,等.连翘生晒果实和干燥叶片总DNA提取方法比较[J].山西农业科学,2015,43(12):1594-1597.
[2]ALLENGC,FLORESVERGARA MA,KRASYNANSKIS,et al.A modified protocol for rapid DNA isolation from plant tissues using cetyltrimethylammonium bromide[J].Nature Protocols,2006,1(5):2320.
[3]TIWARIK L,JADHAV SK,GUPTA S.Modified CTAB technique for isolation of DNA fromsomemedicinal plants[J].Research Journal of Medicinal Plant,2009,6(1):65-73.
[4]韩玉杰,贾炜珑,王自霞,等.几种提取植物DNA方法的比较[J].山西农业科学,2008,36(7):17-19.
[5]王少斌,程水源,王燕,等.提取板栗DNA的CTAB法和SDS法的比较[J].湖北农业科学,2012,51(14):3101-3103.
[6]胡雅琴,肖娅萍,王孝安,等.几种松科植物基因组总DNA的提取[J].西北植物学报,2004,24(3):523-526.
[7]张富丽,陶李,佟洪金,等.从干种子中快速获取高质量DNA的高盐提取方法[J].中国农学通报,2011,27(24):98-102.
[8]许殊.一种适合MSAP分析谷物干种子的DNA提取方法[J].山东农业大学学报(自然科学版),2014(5):660-664.
[9] SAGHAI-MAROOF M A,SOLIMAN K M,GORGENSEN R A,et al.Ribosomal DNA spacer length polymorphism in barley:mendelian inheritance,chromosomal location and population dynamics[J].Proc Natl Acad Sci USA,1984,81(24):8014-8018.
[10]郭景伦,赵久然,王凤格.适用于SSR分子标记的玉米单粒种子 DNA快速提取新方法[J].玉米科学,2005,13(2):16-17.
[11]田丽波,谷幸幸,商桑,等.苦瓜基因组DNA的提取及ISSR扩增体系的优化[J].中国农学通报,2013,29(4):88-93.
[12]柴美清,原佳敏.SSR分子标记在玉米种质研究中的应用[J].山西农业科学,2011,39(9):1017-1019.
[13]骈跃斌,许晶,武岩军,等.SSR分子标记技术在玉米杂种优势群划分中的应用[J].山西农业科学,2012,40(5):439-441.
[14]宋伟,赵久然,王凤格,等.SSR和SNP标记在玉米分子标记辅助背景选择中的应用比较[J].玉米科学,2016,24(3):57-61.
[15]王文,王金胜.黄芪SSR位点研究[J].山西农业科学,2013,41(4):324-326.
[16]刘兰浩,裴玉贺,穆平,等.玉米SSR连锁图谱构建和抗弯孢菌叶斑病的 QTL分析[J].华北农学报,2009,24(2):27-31.
[17]赵丹.玉米籽粒营养品质性状的QTL分析[D].雅安:四川农业大学,2016.
[18]胡利宗,刘均革,郭晋杰,等.基于玉米BC2F2群体的穗部性状QTL分析[J].华北农学报,2010,25(4):73-77.
欢迎订阅《农业工程》
《农业工程》是由中国农业机械化科学研究院主管、北京卓众出版有限公司主办的农业工程类综合性学术期刊,2011年创刊,月刊,国内统一连续出版物号CN 11-6025/S,国际标准连续出版物号ISSN 2095-1795。办刊宗旨是解读农业工程政策,聚焦农业工程各领域关键技术的创新突破、成套技术系统集成和先进实用技术的推广示范,搭建农业工程交叉学科产学研集成信息平台,推进农业工程技术创新与成果转化,更好地为“三农”服务。
该刊设有装备与机械化、信息与电气化、生物环境与能源、农副产品加工与贮藏、食品科学与工程、水土工程、土地资源管理、设计制造及理论研究、教学研究、农学与生物技术、农业经济管理等栏目,入选国家新闻出版广电总局认定的首批学术期刊和中国科技核心期刊。
《农业工程》每期定价20元,全年240元,欢迎订阅,欢迎投稿。
地址:北京市朝阳区德外北沙滩1号16信箱 邮编:100083
电话:010-64882380/3625 网址:www.d1ae.com
邮箱:nygc_2011@163.com d1ae2011@126.com
Improvement of Genome DNA Extraction Method from Maize Dry Seeds
DONGYongjun1,WANGLujun2,3,HAOJianping1,WANGYanmei2,3,LIPeiliang2,3,WANGChuangyun2,3,DENGYanfang3,ZHOUQiong3,LIZhimin3,PANGBing1,ZHANGTingting1
(1.Collegeof Life Science,Shanxi University,Taiyuan 030006,China;2.Instituteof Crop Sciences,Shanxi Academy of Agricultural Sciences,Taiyuan 030031,China;3.Shanxi Corn Engineering Technology Research Center,Taiyuan 030031,China)
To realize the rapid extraction of maize dry seed genomic DNA,according to the characteristics of high starch content in maize dry seeds,this study optimized and improved the extraction method of traditional nucleic acid CTAB,and analyzed the extracted DNA by spectrophotometer and agarose gel electrophoresis.The results showed that the OD260/OD280value of DNA was between 1.79-1.95,and the OD260/OD230value was between 1.90-2.15.The gel electrophoresis pattern was clear and the band was no tailing,which illustrated that the improved DNA was high degradation.DNA samples extracted by the new method were used as templates to detect the molecular markers,the results were clear and stable.It further proved that DNA extracted from maize dry seeds by modified CTABmethod could meet the requirements of SSRand SNP.
maize;dry seed;DNA;CTAB
S513
A
1002-2481(2017)12-1903-04
10.3969/j.issn.1002-2481.2017.12.01
2017-10-12
国家星火计划项目(2015GA630001);山西省工程技术研究中心项目(201605D141005);山西省农业科学院种业发展专项(2016ZYZX08);山西省重点研发计划项目(201603D221002-7);山西省科技成果转化引导专项(201604D131037);山西省重点研发计划项目(201703D221001-2)
董永军(1990-),男,山西岢岚人,在读硕士,研究方向:玉米分子辅助遗传育种。王陆军、郝建平为通信作者。
