山东省近期育成小麦品种(系)的遗传多样性及其与产量性状的关联分析
2017-12-11程西永吕建华毛瑞喜王春阳李南南李斯深
程西永,吕建华,毛瑞喜,王春阳,李南南,李斯深
(1.山东农业大学农学院/作物生物学国家重点实验室,山东泰安 271018; 2.山东省种子管理总站,山东济南 250100)
山东省近期育成小麦品种(系)的遗传多样性及其与产量性状的关联分析
程西永1,吕建华2,毛瑞喜2,王春阳1,李南南1,李斯深1
(1.山东农业大学农学院/作物生物学国家重点实验室,山东泰安 271018; 2.山东省种子管理总站,山东济南 250100)
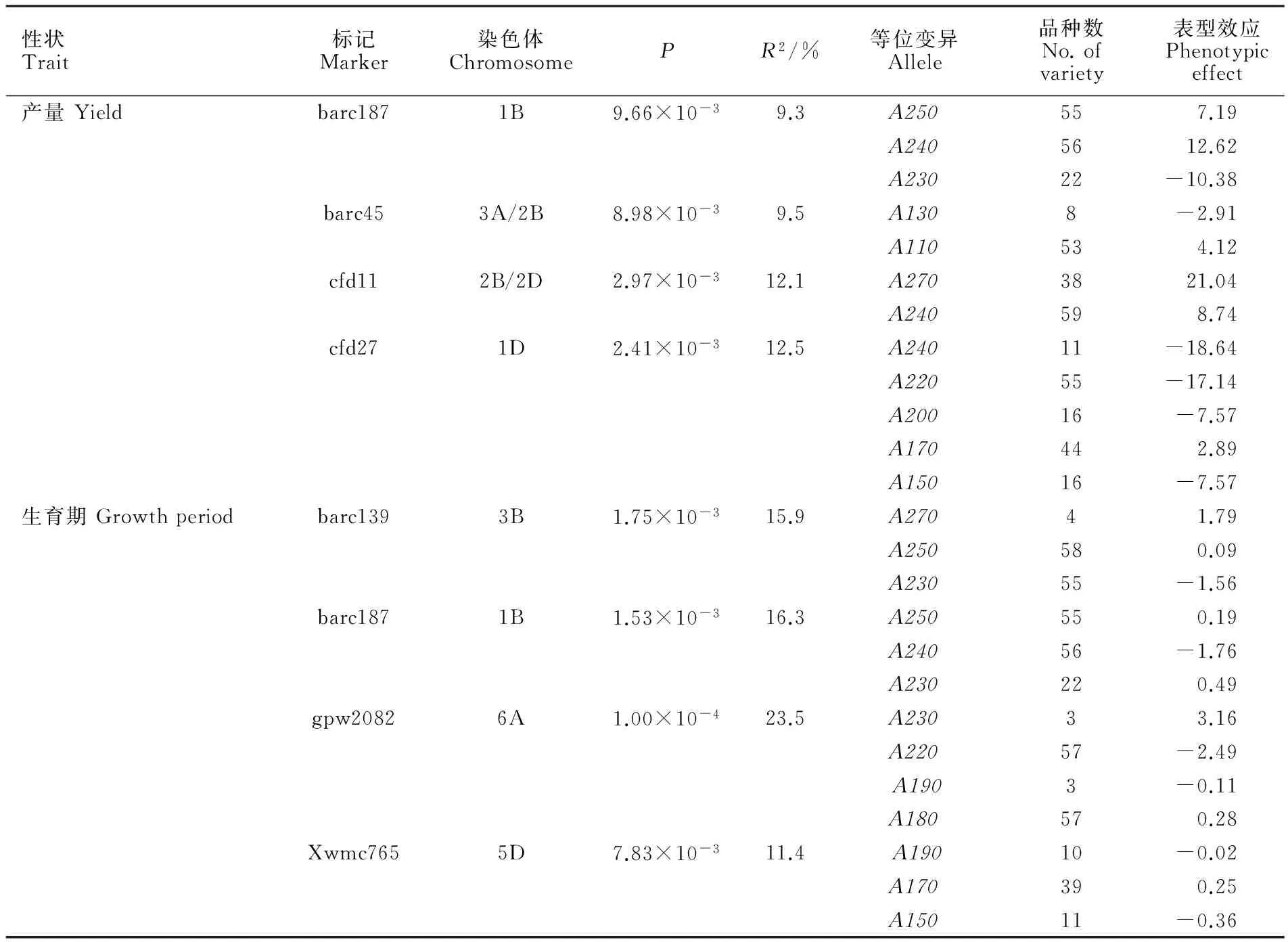
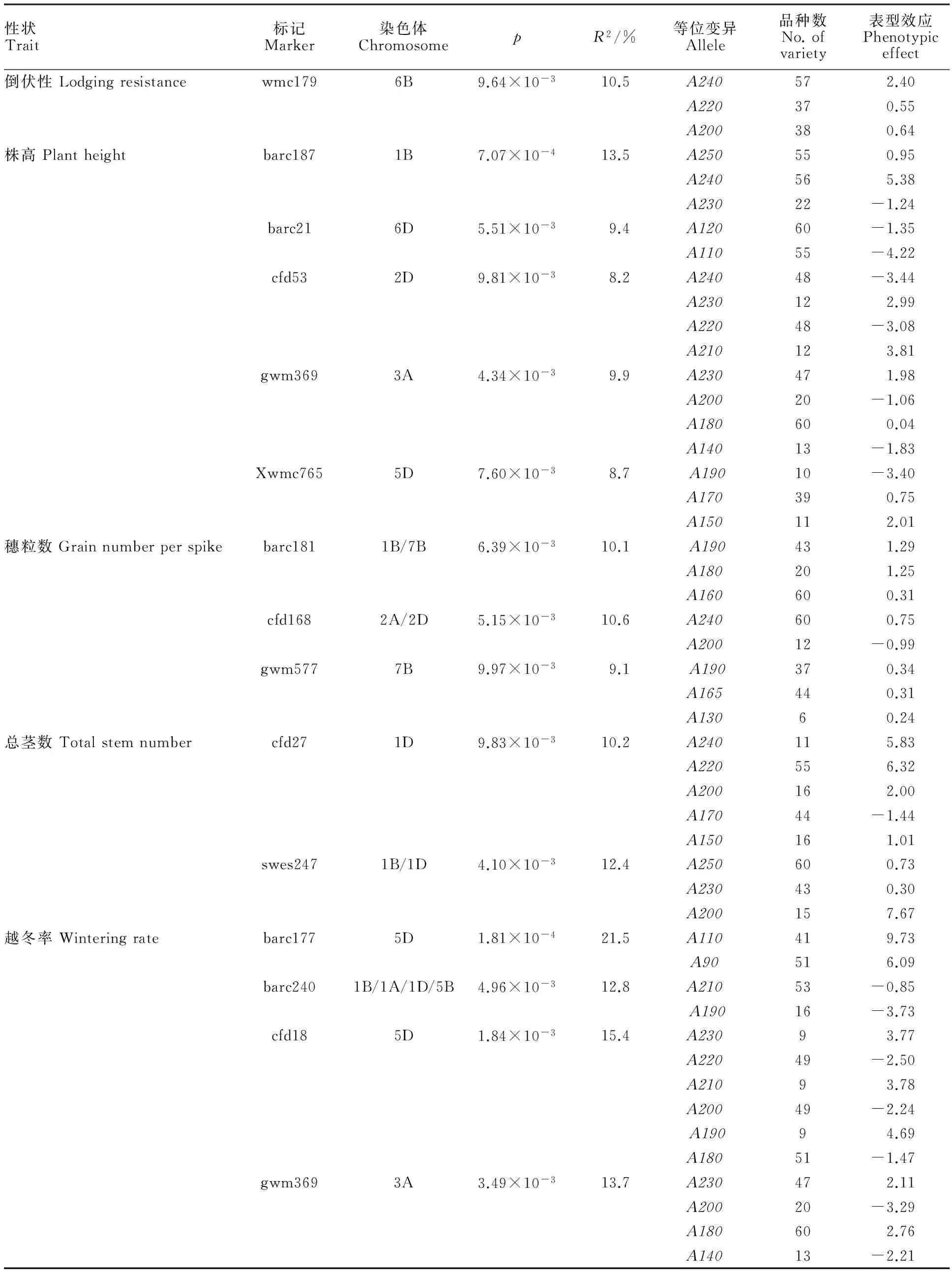
为了解山东省近年来育成品种(系)的遗传多样性,并筛选出与产量性状相关的分子标记及其等位变异,选用58对分布于小麦21条染色体上的SSR标记,对109个山东省近年来育成的品种(系)进行遗传多样性和关联分析。SSR标记多态性分析表明,本研究共检测到176个等位位点,各标记等位位点变化范围为2~6个,平均为3.034个;SSR标记多态性信息量(PIC)变化范围为0.111~0.829,平均为0.552。聚类分析显示,同一育种单位育成的或具有共同亲本的品种往往聚为一类。关联分析表明,与产量性状显著关联(P<0.01)的标记有18对。对相对稳定的等位变异作进一步分析,发掘了一批与产量性状相关的优异等位变异,如增加产量的等位变异 barc187-A240和 cfd11-A270,降低株高的等位变异 barc21-A110、 cfd53-A240和 Xwmc765-A190,增加穗粒数的等位变异 barc181-A190 ,增加总茎数的等位变异 cfd27-A220 和 swes247-A200,提高越冬率的等位变异 barc177-A110。
小麦;SSR标记;遗传多样性;关联分析;等位变异
小麦是我国重要粮食作物之一,有着悠久的种植历史和广阔的种植面积。长期以来,小麦育种家通过杂交育种等方法,育成了大批高产、优质、多抗的小麦品种。然而,随着近年来人工选择的加剧,亲本遗传基础狭窄、遗传多样性水平较低成为影响优质小麦品种选育的主要问题之一[1]。通过对育成品种(系)的遗传多样性分析,可以了解种质资源的遗传信息,指导杂交亲本的选配,提高小麦育种效率[2]。分子标记技术在种质资源的遗传多样性分析中已被广泛应用。其中,SSR分子标记具有稳定性好、多态性高、共显性分离、位点专化性、标记覆盖整个基因组且分布均匀等优点,被应用最为广泛[3-4]。此外,在遗传多样性分析基础上进行关联分析,有利于发掘有效基因,对后期分子标记辅助选择具有重要意义。李小军等[5]利用SSR标记与农艺性状进行关联分析,发现了与穗粒数、产量紧密相关的标记。刘新伦等[6]利用SSR标记与54份小麦品种进行麦长管蚜抗性关联分析,发现了与小麦长管蚜抗性相关联的标记。姜 朋等[7]利用SSR标记与弱筋小麦进行关联分析,发现了与小麦籽粒蛋白质含量关联的标记。本研究采用58对SSR标记对109个山东省近年来育成的小麦品种(系)进行遗传多样性分析,旨在了解山东省近年来育成的小麦品种(系)之间的差异和亲缘关系,拓宽小麦品种的遗传基础;同时通过分子标记与性状的关联分析,进一步发掘与小麦产量等性状相关联的分子标记,为小麦育种工作提供参考。
1 材料与方法
1.1 供试材料和产量性状数据来源
供试材料共109份,包括9个山东省近期育成品种和100个2014-2015年山东省小麦高肥区域试验的品系(表1)。产量性状数据来源于山东省12个区试点调查结果,包括100个新品系和对照品种济麦22。
1.2 基因组DNA的提取和SSR分析
将小麦种子置于铺放双层滤纸的培养皿中,加入适量蒸馏水润湿,待种子露白后,点播在育苗穴盘内,然后置于RXZ智能型人工气候箱内培养。待幼苗长至二叶一心期时,称取幼苗叶片0.2 g,液氮冷冻下快速研磨成粉末,采用CTAB法提取基因组DNA[8]。加入RNaseA(北京天根生化科技有限公司)去除DNA样品中的RNA。用1%琼脂糖凝胶电泳检测所提DNA样品的质量。用超微量蛋白核酸分析仪Bio Drop Duo (Bio Drop, UK)测定DNA浓度。用超纯水将DNA样品稀释到50 ng·μL-1,4 ℃保存备用。
利用20份小麦品种进行SSR标记筛选,凝胶电泳后去除无多态性和扩增条带模糊的标记,最终从陈海梅等[9]、Roder等[10]和Gupta等[11]所开发的83对SSR标记中筛选出分布在小麦21条染色体上的58对SSR标记(表2)用于遗传多样性分析。PCR反应在T1型PCR扩增仪(Biotech, DEU)上进行。反应体系15 μL,包括Reaction Mix 7.5 μL、75 ng DNA、1.5 μL引物、0.2 μL Golden DNA Polymerase、4.3 μL ddH2O。PCR扩增程序:94 ℃预变性5 min;94 ℃变性30 s,50~65 ℃(因引物而异)退火30 s,72 ℃延伸40 s,35个循环;72 ℃延伸10 min,4 ℃保存。PCR扩增产物用8%非变性聚丙烯酰胺凝胶电泳(Native-PAGE),经0.2%硝酸银染色后于白炽灯下观察结果并进行数据统计和拍照。
1.3 数据分析
在相同迁移率位置上有扩增条带赋值“1”,无条带赋值“0”,缺失数据用“9”表示,统计每对标记的带型,构建二元矩阵[12]。多态性信息含量(polymorphism information content, PIC)计算公式如下:
其中,Pij为i位点第j等位变异的频率,标记i的总带型从1到n。
根据Breseghello等[13]的无效等位变异理论,估测位点等位变异的表型效应,计算公式如下:
αi=∑xij/nj-∑Nk/nk
式中,αi表示等位变异的表型效应值,xij表示具有第i个等位变异的第j个材料的表型值,nj表示携带第i个等位变异的材料数,Nk表示具有无效等位变异的第k个材料的表型值,nk表示具有无效等位变异的材料数。如果αi大于0,则认为该等位变异与性状正关联,反之则认为负关联。
采用NTSYS 2.10e软件中similarity程序计算遗传距离,然后导入MEGA 6.0程序中进行Neighbor-Joining聚类分析[14]。以Structure 2.3.1软件分析群体遗传结构,估计最佳群体组群K,其取值范围1~10,将MCMC(Markov Chain Monte Carlo)开始时的不作数迭代(length of burn-in period)设为50 000次,再将不作数迭代后的MCMC设为100 000次,重复20次,计算Q参数。若K值持续增大,参照Evanno等[15]的方法计算ΔK确定K值。应用TASSEL 3.0软件中的GLM模型进行性状和标记之间的关联分析。当标记的P<0.01时认为标记与性状存在显著相关。

表1 小麦品种(系)名称Table 1 Name of wheat varieties(lines)
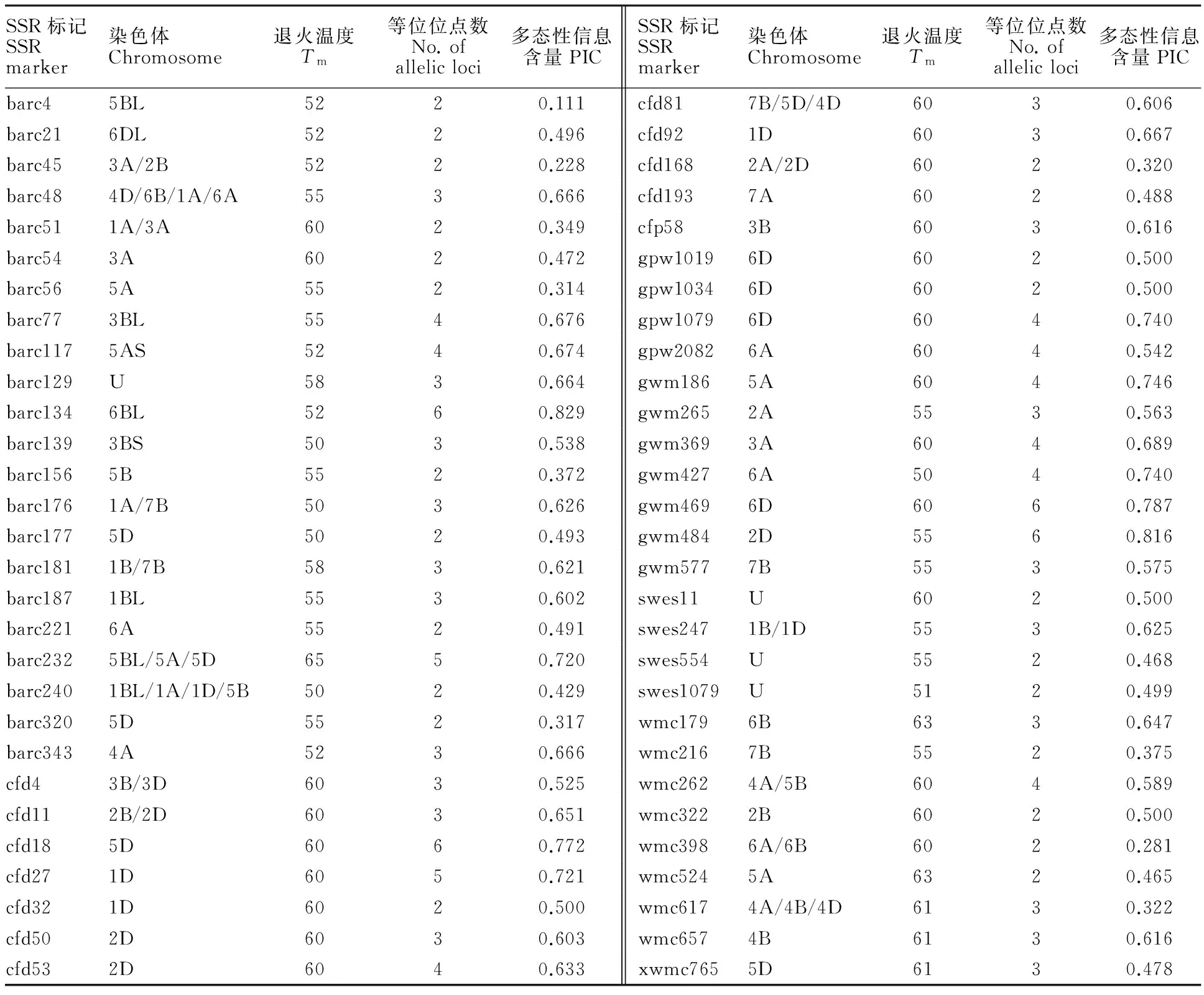
表2 SSR标记及其所在染色体位置、退火温度、等位位点数和位点多态性信息含量Table 2 Information of SSRs and their positions on chromosomes,Tm, number of allelic loci and values of polymorphic information content(PIC)
料及其抗旱指数和加权隶属函数U代表所在染色体未知。
U means the unknown location on chromosome.
2 结果与分析
2.1 SSR标记的多态性分析
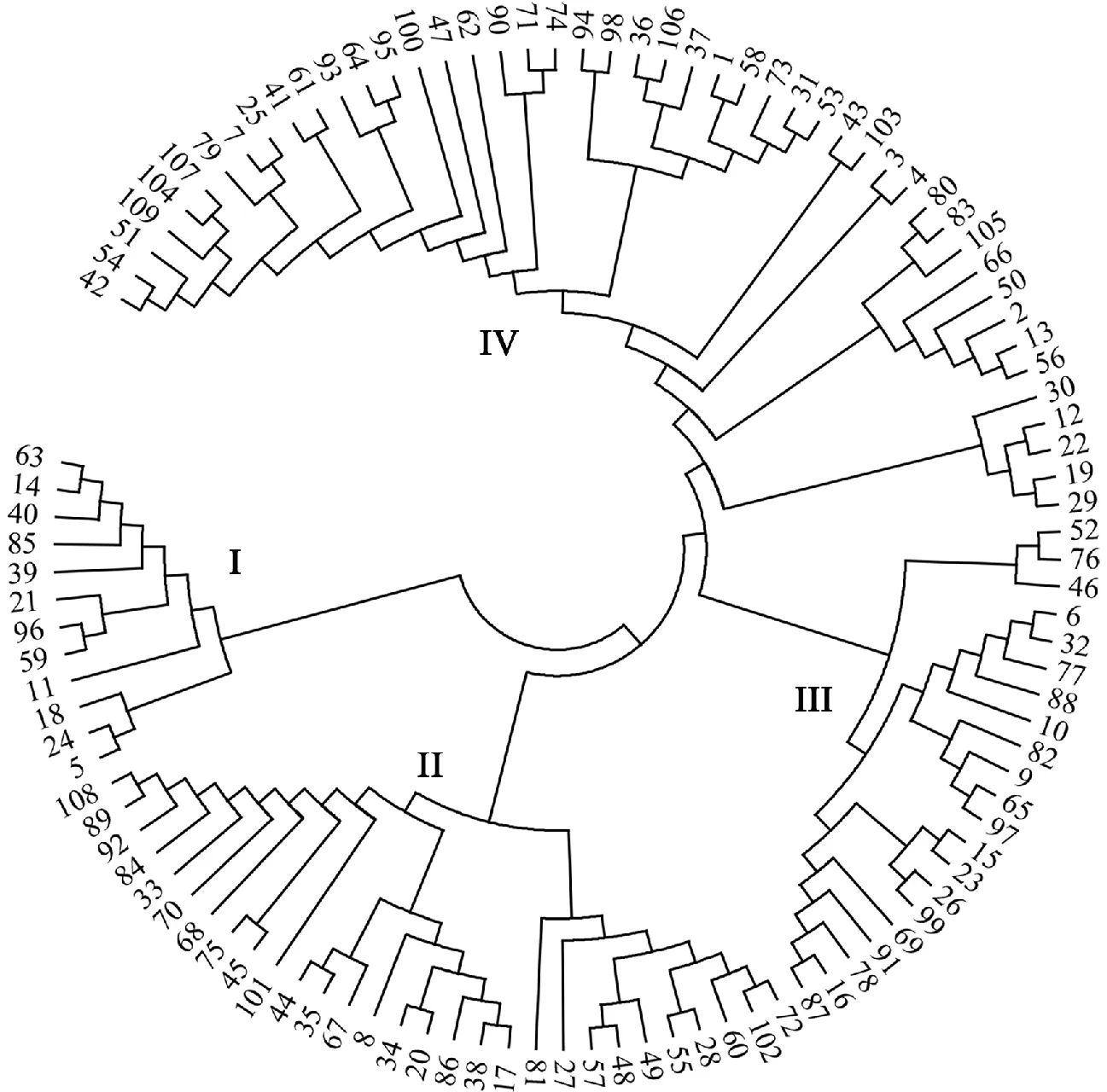
利用58对SSR标记对109个小麦品种(系)的基因组DNA进PCR扩增,共检测出176个等位位点,每对SSR标记的等位位点数目为2~6个,平均3.034个(表2,图1)。根据Botstein等[16]的研究,当PIC≥0.50时,该标记为高度多态性标记;当0.25 根据58对SSR标记的读带数据,利用MEGA 6.0软件进行了聚类分析,109个品种(系)可以聚为4大类(图2)。第I类群包括润农1382、AN02、中麦175、泰田麦118、鲁宁麦1302、东旱1号、山农3050、山农10788、FC0017、淄麦35、东麦15和山农981等12个品种(系)。该类群与其他3个类群关系较远,育成单位也比较复杂,是一个新的类群,在育种中值得注意。 第II类群包括泰农18、裕田麦119、岱麦3166、圣田麦69、乐旱6255、SNW612、岱麦3270、岱麦3640、泰麦1918、山农29、泰农2432、LS4393、淄麦28、HF8327、岱麦2638、山农82573、好麦301、博农麦7号、泰农107、淄麦29、阳光505、鑫秋808、泰山5059、岱麦2173、LS1387、博农麦8号、鲁原231、鲁原502和鲁原304等29个品种(系)。其中,泰农18、泰麦1918为山东省审定品种,山农29和鲁原502为国家审定品种。该类群中许多品种与泰农18有密切亲缘关系,如山农29、LS号、岱麦号、SNW612、淄麦29等均是用泰农18或其姊妹系做亲本之一,可称之为泰农18群。 第III类群包括徐麦1008、济麦45、淄麦27、菏麦0605、济麦31、HF6012、邦麦7号、青麦H055、SNW961、禾元11号、滨麦6号、阳光603、济麦30、鸿翔519、红地167、烟农1212、红地166、红地168、菏麦0666、藁优5766和泰山5328等21个品种(系)。其中,烟农1212正在生产试验中,2016年红地166和菏麦0666通过了山东省品种审定,该类群与第IV类群关系较近。 M:pUC18 marker(北京天根);1~31:菲达1号、临091、QX159、SNW634、山农981、红地166、青麦H012、HF8327、阳光603、鸿翔519、FC0017、ZH4261、Kf2014、AN02、SNW961、济麦45、泰农107、淄麦35、良星638、山农82573、东旱1号、聊麦149、青麦H055、东麦15、山农02-1、邦麦7号、阳光505、博农麦8号、济麦50、青14-7-13、德旱13-10、红地168、乐旱6255、岱麦2638、LS4393、烟5102、泰科麦5366。 M:pUC18 marker(TIANGEN, Beijing); 1-31:Feida 1, Lin 091, QX 159, SNW 634, Shannong 981, Hongdi 166, Qingmai H012, HF8327, Yangguang 603, Hongxiang 519, FC0017, ZH4261, Kf2014, AN02, SNW961, Jimai 45, Tainong 107, Zimai 35, Liangxing 638, Shannong 82573, Donghan 1, Liaomai 149, Qingmai H055, Dongmai 15, Shannong 02-1, Bangmai 7, Yangguang 505, Bonongmai 8, Jimai 50, Qing 14-7-13, Dehan 13-10, Hongdi 168, Lehan 6255, Daimai 2638, LS4393, Yan 5102, Taikemai 5366. 图1标记gwm186(5A)在部分小麦品种(系)中的扩增条带图 Fig.1Amplificationof31wheatvarietieswithprimerpairgwm186(5A) 第IV类群包括济麦50、良星638、聊麦149、ZH4261、青宇14-7-13、良星517、Kf2014、临091、烟农25、泰山6038、临麦4、S13-2、DH51303、SNW634、QX159、烟农24、DH51202、鸿翔1213、德旱13-10、XR-4429、烟农187、菲达1号、泰科麦5366、烟农5158、烟5102、烟农194、泰山6436、泰农3018、济麦34、鑫麦803、良星505、齐都5号、科源026、济麦32、石农086、济麦37、中冠麦3号、济麦22、山农诱02-1、青麦H012、良星667、良星66、良星99、良星77、济麦23、DH51302和FC009等47个品种(系),是最大的一个类群。其中,济麦22和良星66为国家审定品种,良星77、烟农24、临麦4号、登海202(DH51202)、济麦23和峰川9号(FC009)为山东省审定品种,良星99为河北省审定品种,烟5158为山东省和安徽省审定品种。该类群审定品种多、生产上推广面积大。许多品种与济麦22或良星号有密切关系,可称之为济麦22群,总体表现与第II类群差别较大。 对供试的109个小麦品种(系)进行基于一般线性模型的群体结构分析,设定推测亚群数K=1~10,而L(K)虽然随着K值发生变化,但并没有明显的最大值。根据计算出的ΔK值绘制ΔK值随K值变化的折线图,在K=3时,ΔK值出现明显的最大值,随后急剧下降(图3),说明109个小麦品种(系)的最优亚群数为3。这3个亚群分别由37、25、47个品种(系)组成,在群体中所占比例分别为33.94%、22.94%、43.12%。 以100个2014-2015年山东省小麦区试品系和对照品种济麦22为材料,进行58对SSR标记与产量性状关联分析,共获得显著关联标记18对(表3)。与产量显著关联的分子标记有4对(barc187、barc45、cfd11、cfd27),贡献率为9.3%~12.5%;与生育期显著关联的分子标记有3对(barc139、barc187、gpw2082),贡献率为15.9%~23.5%;与株高显著关联的分子标记有5对(barc187、barc21、cfd53、gwm369、Xwmc765),贡献率为8.2%~13.5%;与穗粒数显著关联的分子标记有3对(barc181、cfd168、gwm577),贡献率为9.1%~10.6%;与总茎数显著关联的分子标记有2对(cfd27、swes247),贡献率为10.2%~12.4%;与越冬率显著关联的分子标记有4对(brac177、barc240、cfd18、gwm369),贡献率为12.8%~21.5%;与倒伏性显著关联的分子标记有1对(wmc179),贡献率为10.5%。其中,部分标记与多个性状关联,标记barc187与生育期、株高和产量相关联,贡献率分别为16.3%、13.5%和9.3%;cfd27与产量和总茎数相关联,贡献率分别为12.5%和10.2%;gwm369与株高和越冬率相关联,贡献率分别为9.9%和13.7%。在18对相关联的标记中,有3对标记与性状关联(gpw2082与生育期、barc187与株高、barc177与越冬率)的P值小于0.001,且贡献率较高(分别为23.5%、13.5%、21.5%)。 品种名称详见表1。 Variety names are listed in table 1. 图3 109份小麦材料的群体遗传结构图 在获得与产量性状关联的标记基础上,进行了优异等位变异分析(表3)。产量关联标记barc187、barc45、cfd11和cfd27的等位变异的表型效应范围为-10.38~12.62、-2.91~4.12、8.74~21.04和-18.64~2.89,4对标记等位变异效应最大的分别为 A240、 A110、 A270和 A170;生育期关联标记barc139、barc187、gpw2082和Xwmc765的等位变异的表型效应范围为-1.56~1.79、-1.76~0.49、-2.49~3.16和-0.36~0.25,4对标记等位变异效应最大的分别为 A270、 A230、 A230和 A170;倒伏性关联标记wmc179的等位变异的表型效应范围为0.55~2.40,等位变异 A240的表型效应最大;株高关联标记barc187、barc21、cfd53、gwm369和Xwmc765的等位变异的表型效应范围为-1.24~5.38、-4.22~-1.35、-3.44~3.81、-1.83~1.98和-3.40~2.01,5对标记等位变异效应最大的分别为 A240、 A120、 A210、 A230和 A150;穗粒数关联标记barc181、cfd168和gwm577的等位变异的表型效应范围为0.31~1.29、-0.99~0.75和0.24~0.34,3对标记等位变异效应最大的分别为 A190、 A240和 A190;总茎数关联标记cfd27和swes247的等位变异的表型效应范围为-1.44~6.32和0.30~7.67,2对标记等位变异效应最大的分别为 A220和 A200;越冬率关联标记barc177、barc240、cfd18和gwm369的等位变异的表型效应范围为6.09~9.73、-3.73~-0.85、-2.50~4.69和-3.29~2.76,4对标记等位变异效应最大的分别为 A110、 A210、 A190和 A180。 从表3还可以看出,同一标记不同等位变异的表型效应不同。标记barc187对产量和株高的等位变异效应最大的是 A240,而对生育期的等位变异效应最大的是 A230;标记Xwmc765对生育期的等位变异效应最大的是 A170,而对株高的等位变异效应最大的是 A150;标记gwm369对株高的等位变异效应最大的是 A230,而对越冬率的等位变异效应最大的是 A180。 表3 与表型性状关联的SSR标记及等位变异的表型效应Table 3 SSR markers associated with phenotypic traits and phenotypic effect of alleles (续表3Continuedtable3) 性状Trait标记Marker染色体ChromosomepR2/%等位变异Allele品种数No.ofvariety表型效应Phenotypiceffect倒伏性Lodgingresistancewmc1796B9.64×10-310.5A240572.40A220370.55A200380.64株高Plantheightbarc1871B7.07×10-413.5A250550.95A240565.38A23022-1.24barc216D5.51×10-39.4A12060-1.35A11055-4.22cfd532D9.81×10-38.2A24048-3.44A230122.99A22048-3.08A210123.81gwm3693A4.34×10-39.9A230471.98A20020-1.06A180600.04A14013-1.83Xwmc7655D7.60×10-38.7A19010-3.40A170390.75A150112.01穗粒数Grainnumberperspikebarc1811B/7B6.39×10-310.1A190431.29A180201.25A160600.31cfd1682A/2D5.15×10-310.6A240600.75A20012-0.99gwm5777B9.97×10-39.1A190370.34A165440.31A13060.24总茎数Totalstemnumbercfd271D9.83×10-310.2A240115.83A220556.32A200162.00A17044-1.44A150161.01swes2471B/1D4.10×10-312.4A250600.73A230430.30A200157.67越冬率Winteringratebarc1775D1.81×10-421.5A110419.73A90516.09barc2401B/1A/1D/5B4.96×10-312.8A21053-0.85A19016-3.73cfd185D1.84×10-315.4A23093.77A22049-2.50A21093.78A20049-2.24A19094.69A18051-1.47gwm3693A3.49×10-313.7A230472.11A20020-3.29A180602.76A14013-2.21 根据标记的凝胶电泳图,用“A+不同条带大小”表示该标记不同的等位变异。 ‘A+different fragment sizes amplified with a marker’ indicates different alleles of the marker. 遗传多样性分析是后期进行关联分析进而发掘有利基因的基础和前提[17-18]。本研究利用SSR标记进行遗传多样性分析,确定供试材料在不同标记上所含有的等位变异及基因型,在一定程度上反映了材料间的亲缘关系。聚类分析发现,山东省近期育成的品种(系)中,具有共同亲本或同一育种单位育成的品种往往聚为一类,如来源于同一育种单位的良星66、良星77和良星99聚在IV类;亲缘关系较近的泰农18、山农29和淄麦28聚在II类。该结果在程 斌等[19]、蒲艳艳等[12]的研究中均有所体现。本试验发现,济麦22、良星66、良星77等主要推广品种相似性较高,这与彭 芹等[20]试验结果一致。聚类分析是研究品种(系)遗传差异、杂交亲本选育的重要依据。倪中福等[21]利用65对SSR标记分析了23个小麦品种(系)D染色体组的遗传多样性,供试材料间的平均遗传距离为0.428。耿惠敏等[22]利用43对SSR标记分析了40份河南审定小麦品种的遗传多样性,供试材料间平均遗传距离为0.404。本试验利用58对SSR标记对山东省小麦品种(系)进行分析,供试品种(系)间遗传距离为0.04~0.55,平均遗传距离为0.269,遗传距离较近,相似性较高,说明山东省近期育成的品种(系)遗传多样性水平较低,遗传基础狭窄。如第IV类群品种(系)数目高达47个,且与第III类群关系较近,在育种亲本选配时应该注意增加遗传多样性。造成这种现象的主要原因可能是育种中大量使用相同的骨干亲本或以骨干亲本衍生材料做亲本。因此,本试验结果可以为小麦育种过程中亲本的选择提供一定的理论依据,避免相似亲本的重复利用,有利于拓宽遗传基础,提高小麦品种(系)的遗传多样性。 近年来,关联分析在作物中得到广泛应用。张国华等[23]利用64对SSR、2对EST-SSR、47对功能标记和黄淮麦区小麦4个环境变量下的产量性状进行关联分析,发现49对标记与4个环境的产量性状及其均值显著关联。司二静等[24]利用86对SSR标记和156份大麦材料进行关联分析,得到了18个与株高、穗长、芒长、穗粒数和千粒重等性状相关联的标记。Agrama等[25]利用123对SSR标记分析了92个水稻品种,得到与产量、千粒重、粒长和粒宽显著关联的分子标记。本研究分析了58对标记与产量性状的相关性,共检测到18对与小麦产量性状相关联的SSR标记。结果显示,barc45(3A/2B)与产量显著相关,barc177(5D)与小麦的越冬率显著关联,barc181(1B/7B)与小麦穗粒数具有显著相关性,这与武玉国等[26]、靳 婷[27]、宋彦霞[28]的研究结果一致,说明试验结果较为可靠,可以用作分子标记辅助选择。本试验还发现,与穗粒数相关的标记还有cfd168(2A/2D)和gwm577(7B),前人在2A、2D和7B上均发现过与穗粒数相关联的标记或者QTL[29-30]。本研究还发现了两个与总茎数关联的标记,分别为cfd27(1D)与swes247(1B/1D),尚未见相关报道。本研究也发现了一些优异等位变异,在育种中应注意利用。 [1] TANKSLEY S D,MCCOUCH S R.Seed banks and molecular maps:Unlocking genetic potential from the wild [J].Science,1997,277(5329):1063. [2] 赖 勇,贾建磊,王晋民,等.外引大麦SSR标记遗传多样性及其与农艺性状的关联分析[J].麦类作物学报,2017,37(2):198. LAI Y,JIA J L,WANG J M,etal.Analysis of genetic diversity and association with agronomic traits in barley(HordeumvulgareL.) introduced from abroad using SSR markers [J].JournalofTriticeaeCrops,2017,37(2):198. [3] LUO H Y,WANG X J,ZHAN G M,etal.Genome-wide analysis of simple sequence repeats and efficient development of polymorphic SSR markers based on whole genome re-sequencing of multiple isolates of the wheat stripe rust fungus [J].PloSOne,2015,10(6):e0130362. [4] FISCHER D,BACHMANN K.Onion microsatellites for germplasm analysis and their use in assessing intra- and inter-specific relatedness within the subgenusRhizirideum[J].TheoreticalandAppliedGenetics,2000,101(1-2):153. [5] 李小军,徐 鑫,刘伟华,等.利用SSR标记探讨骨干亲本欧柔在衍生品种的遗传[J].2009,42(10):3397. LI X J,XU X,LIU W H,etal.Genetic diversity of the founder parent Orofen and its progenies revealed by SSR markers [J].ScientiaAgriculturaSinica,2009,42(10):3397. [6] 刘新伦,李志超,王亚娟,等.抗麦长管蚜小麦的遗传多样性及SSR标记与麦长管蚜抗性的关联分析[J].农业生物技术学报,2015,23(3):297. LIU X L,LI Z C,WANG Y J,etal.Genetic diversity analysis of aphid- resistant wheat(Triticumaestivum) and correlation analysis between SSR markers and resistance to the English grain aphid(Sitobionavenae) [J].JournalofAgriculturalBiotechnology,2015,23(3):297. [7] 姜 朋,张平平,张 旭,等.弱筋小麦宁麦9号及其衍生系的蛋白质含量遗传多样性及关联分析[J].作物学报,2015,41(12):1831. JIANG P,ZHANG P P,ZHANG X,etal.Genetic diversity and association analysis of protein content in weak gluten wheat Ningmai 9 and its derived lines [J].ActaAgriculturaeBoreali-occidentalisSinica,2015,41(12):1831. [8] 李荣华,夏岩石,刘顺枝,等.改进的CTAB提取植物DNA方法[J].实验室研究与探索,2009,28(9):15. LI R H,XIA Y S,LIU S Z,etal.CTAB-improved method of DNA extraction in plant [J].ResearchandExplorationinLaboratory,2009,28(9):15. [9] 陈海梅,李林志,卫宪云,等.小麦EST-SSR标记的开发、染色体定位和遗传作图[J].科学通报,2005,50(20):2211. CHEN H M,LI L Z,WEI X Y,etal.Development of EST-SSR markers,chromosome location and genetic mapping in wheat [J].ChineseScienceBulletin,2005,50(20):2211. [10] RDER M S,KORZUN V,WENDEHAKE K,etal.A microsatellite map of wheat [J].Genetics,1998,149(4):2009. [11] GUPTA P,BALYAN H,EDWARDS K,etal.Genetic mapping of 66 new microsatellite(SSR) loci in bread wheat [J].TheoreticalandAppliedGenetics,2002,105(2):416. [12] 蒲艳艳,程 凯,李斯深,等.山东省近期育成小麦品种遗传多样性的SSR分析[J].分子植物育种,2011,9(4):443. PU Y Y,CHENG K,LI S S,etal.Genetic diversity of recent wheat cultivars in Shandong province using SSR markers [J].MolecularPlantBreeding,2011,9(4):443. [13] BRESEGHELLO F,SORRELLS M E.Association mapping of kernel size and milling quality in wheat(TriticumaestivumL.) cultivars [J].Genetics,2006,172(2):1165. [14] 罗 凯,卢会翔,吴正丹,等.中国西南地区甘薯主要育种亲本的遗传多样性及群体结构分析[J].中国农业科学,2016,49(3):597. LUO K,LU H X,WU Z D,etal.Genetic diversity and population structure analysis of main sweet potato breeding parents in southwest China [J].ScientiaAgriculturaSinica,2016,49(3):597. [15] EVANNO G,REGNAUT S,GOUDET J.Detecting the number of clusters of individuals using the software STRUCTURE:A simulation study [J].MolecularEcology,2005,14(8):2613. [16] BOTSTEIN D,WHITE R L,SKOLNICK M,etal.Construction of a genetic linkage map in man using restriction fragment length polymorphisms [J].AmericanJournalofHumanGenetics,1980,32(3):321. [17] HARRIS B P,STOKESBRY K D E.The spatial structure of local surficial sediment characteristics on Georges Bank,USA [J].ContinentalShelfResearch,2010,30(17):1840. [18] WANG M L,ZHU C S,BARKLEY N A,etal.Genetic diversity and population structure analysis of accessions in the US historic sweet sorghum collection [J].TheoreticalandAppliedGenetics,2009,120(1):13. [19] 程 斌,张淑英,张明霞,等.山东省近年育成小麦品种(系)的遗传多样性分析[J].山东农业科学,2016,48(9):19. CHENG B,ZHANG S Y,ZHANG M X,etal.Genetic diversity analysis of recent bred wheat varieties(lines) in Shandong province [J].ShandongAgriculturalSciences,2016,48(9):19. [20] 彭 芹,戴 双,郭赛欢,等.1950年以来山东省主推小麦品种的遗传多样性演变[J].分子植物育种,2012,10(2):235. PENG Q,DAI S,GUO S H,etal.The evolution of genetic diversity of wheat varieties released in Shandong province since 1950 [J].MolecularPlantBreeding,2012,10(2):235. [21] 倪中福,张义荣,梁荣奇,等.普通小麦D染色体组微卫星分子标记遗传差异研究[J].作物学报,2003,29(1):149. NI Z F,ZHANG Y R,LIANG R Q,etal.Genetic diversity of D-genome revealed by SSR markers in wheat(TriticumaestivumL.) [J].ActaAgronomicaSinica,2003,29(1):149. [22] 耿惠敏,刘红彦,宋玉立,等.40个河南省审定小麦品种遗传多样性的SSR标记分析[J].西北农业学报,2005,14(2):30. GENG H M,LIU H Y,SONG Y L,etal.SSR analysis of genetic diversity among forty released wheat cultivars in Henan province [J].ActaAgriculturaeBoreali-occidentalisSinica,2005,14(2):30. [23] 张国华,高明刚,张桂芝,等.黄淮麦区小麦品种(系)产量性状与分子标记的关联分析[J].作物学报,2013,39(7):1192. ZHANG G H,GAO M G,ZHANG G Z,etal.Association analysis of yield traits with molecular markers in Huang-Huai river valley winter wheat region,China [J].ActaAgronomicaSinica,2013,39(7):1192. [24] 司二静,张 宇,汪军成,等.大麦农艺性状与SSR标记的关联分析[J].作物学报,2015,41(7):1068. SI E J,ZHANG Y,WANG J C,etal.Association analysis between SSR markers and agronomic traits in barley [J].ActaAgronomicaSinica,2015,41(7):1068. [25] AGRAMA H A,EIZENGA G C,YAN W.Association mapping of yield and its components in rice cultivars [J].MolecularBreeding,2007,19(4):348. [26] 武玉国,吴承来,秦保平,等.黄淮冬麦区175个小麦品种的遗传多样性及SSR标记与株高和产量相关性状的关联分析[J].作物学报,2012,38(6):1018. WU Y G ,WU C L,QIN B P,etal.Diversity of 175 wheat varieties from Yellow and Huai river valleys facultative wheat zone and association of SSR markers with plant height and yield related traits [J].ActaAgronomicaSinica,2012,38(6):1018. [27] 靳 婷.不同基因型小麦的抗寒性及SSR分子标记分析[D].合肥:安徽农业大学,2014:34. JIN T.The cold resistance of different wheat genotype and SSR analysis [D].Hefei:Anhui Agricultural University,2014:34. [28] 宋彦霞.小麦抽穗期及其它农艺性状的QTL分析[D].雅安:四川农业大学,2005:67. SONG Y X.QTL analysis of wheat spike and other agronomic traits [D].Ya’an:Sichuan Agricultural University,2005:67. [29] BRNER A,SCHUMANN E,FÜRSTE A,etal.Mapping of quantitative trait loci determining agronomic important characters in hexaploid wheat(TriticumaestivumL.) [J].TheoreticalandAppliedGenetics,2002,105(6-7):933. [30] MARZA F,BAI G H,CARVER B F,etal.Quantitative trait loci for yield and related traits in the wheat population Ning 7840 × Clark [J].TheoreticalandAppliedGenetics,2006,112(4):691. GeneticDiversityofRecentWheatVarietiesinShandongProvinceandTheirAssociationAnalysiswithYieldTraits CHENGXiyong1,LÜJianhua2,MAORuixi2,WANGChunyang1,LINannan1,LISishen1 (1.College of Agronomy, Shandong Agricultural University/State Key Laboratory of Crop Biology, Tai'an, Shandong 271018, China; 2.Shandong Seed Management Station, Jinan, Shandong 250100, China) The objective of this study is to reveal the genetic diversity of wheat varieties in recent years in Shandong province and to identify the association between SSR markers and important agronomic traits. A number of 109 wheat varieties of Shandong province were analyzed using 58 SSR markers on 21 chromosomes. Totally, 176 alleles were detected among 109 varieties. For a single SSR locus, 2-6 alleles could be detected, with an average of 3.034. The range of polymorphism information content(PIC) was 0.111-0.829, with an average of 0.552. Cluster analysis showed that most of the varieties with the common parent or from the same breeding institution were trend to be clustered into one group. Eighteen markers were significantly associated with plant yield related traits(P<0.01). Some favorable alleles associated with yield traits in multiple environments were discovered, such as barc187-A240 and cfd11-A270 for increasing yield, barc21-A110, cfd53-A240 and Xwmc765-A190 for reducing plant height, barc181-A190 for increasing kernel number per spike, cfd27-A220 and swes247-A200 for increasing the number of all stems and barc177-A110 for increasing the wintering rate. Wheat; SSR; Genetic diversity; Association analysis; Allele 时间:2017-11-14 网络出版地址:http://kns.cnki.net/kcms/detail/61.1359.S.20171114.1027.002.html 2017-03-11 2017-09-25 山东省自然科学基金项目(ZR2010CZ003);山东省农业良种工程项目 E-mail:chengxiyong2010@163.com 李斯深(E-mail:ssli@sdau.edu.cn) S512.1;S330 A 1009-1041(2017)11-1399-102.2 聚类分析

2.3 群体结构分析
2.4 关联分析

2.5 关联位点优异等位变异分析
3 讨 论
