磺胺类抗性基因的产生及演变研究进展*
2017-12-07杨再福陈勇航孙冉冉张姚姚王亚楠
汪 涛 杨再福 陈勇航 孙冉冉 张姚姚 王亚楠
(东华大学环境科学与工程学院,上海 201620)
磺胺类抗性基因的产生及演变研究进展*
汪 涛 杨再福#陈勇航 孙冉冉 张姚姚 王亚楠
(东华大学环境科学与工程学院,上海 201620)
磺胺类抗生素是人工合成的高效广谱抗菌药,广泛用于畜牧养殖、水产养殖和人类疾病治疗中。但其长期排放和累积残留会诱导产生抗生素抗性基因(ARGs)。ARGs作为一种新型的环境污染物,在环境中的持久性残留、复制、传播和扩散比抗生素本身的危害更大。通过对磺胺类ARGs及耐药微生物产生和传播进行综述,提出“渐变→累积→突变”的ARGs演变模型。最后针对中国提出磺胺类ARGs未来的研究重点。
磺胺类抗生素 抗生素抗性基因 演变模型
磺胺类抗生素是重要的人兽共用药,具有广谱抗革兰氏阳性和革兰氏阴性菌活性,由于其价格低廉,被广泛用于临床、畜牧养殖和水产养殖中[1]3。由于磺胺类抗生素大范围无节制的使用,使其频繁进入环境,成为“假持久性”物质。磺胺类抗生素可诱导产生抗生素抗性基因(ARGs)[2]1512。ARGs作为一种新型污染物,其潜在的生态风险和对公众健康的威胁已受到国内外广泛关注[3-6]。冀秀玲等[7]在上海市某地养殖场土壤中检出多种磺胺类ARGs。在水环境中,越南北部的城市运河、养鱼池、养虾池也发现了磺胺类ARGs[8]。此外,PEI等[9]在美国科罗拉多州北部河流沉积物中也发现磺胺类ARGs。世界卫生组织(WHO)已将ARGs作为21世纪威胁人类健康的最大挑战之一,并宣布将在全球范围内对控制ARGs进行战略部署[10]。因此,研究磺胺类ARGs及其耐药微生物传播与分布以及ARGs风险评价对控制磺胺类ARGs污染具有重要意义。
本研究综述了磺胺类抗生素,ARGs和耐药微生物的产生、传播与分布最新研究进展,并提出“渐变→累积→突变”的ARGs演变模型,以期为磺胺类抗生素、ARGs以及ARGs风险评价等研究提供参考。
1 磺胺类抗生素、ARGs及耐药微生物
1.1 磺胺类抗生素的生产及环境存在现状
目前,常用的磺胺类抗生素有磺胺甲恶唑(SMZ)、磺胺二甲氧嘧啶(SMZ)、磺胺嘧啶(SD)等。在欧洲,磺胺类抗生素是使用量第二大的兽药抗生素[11],我国从20世纪40年代开始生产磺胺类药物,生产品种多达30余种,年产量在1 000 t左右,2003年产量突破2万t[12]。KIM等[13]研究表明,磺胺类抗生素的排泄率为67%(质量分数,下同)~90%,以原药或代谢物的形式扩散到环境中。与其他国家相比,我国环境中磺胺类抗生素总体浓度与检出率均较高,如珠江河流中磺胺类抗生素的浓度比美国、日本等发达国家要高得多[14]。
1.2 环境中磺胺类ARGs
磺胺类抗生素的抗菌机制主要是其化学结构类似于对氨基苯甲酸,可与对氨基苯甲酸竞争二氢叶酸合成酶(DHPS),阻断细菌中二氢叶酸的生物合成,进而抑制以二氢叶酸为底物的四氢叶酸的生物合成过程,从而影响核酸前体物嘌呤和嘧啶的合成,从而抑制细菌的正常生长和繁殖[15]。
目前发现的磺胺类ARGs有sul1、sul2、sul3、sulA[16]。sul1基因被认为是Ⅰ类整合子3’保守片段中的一部分,sul2基因通常在各种类型的质粒中都有发现[17]1325。sul3是在2003年分离的猪致病性大肠杆菌分离株的接合性质粒中发现的[18]。细菌对磺胺类抗生素的抗性与DHPS基因(folP)的突变或DHPS的替代基因有关[1]16。
近年来,磺胺类ARGs在不同环境介质中均有检出。STOLL等[19]检测了德国和澳大利亚地表水中24种ARGs的分布情况,结果显示sul1、sul2检出率最高,达77%~100%。XIONG等[20]研究了流溪河流域中ARGs分布特点,均检测出sul1、sul2、sul3,其中sul1丰度为4.19×10-3~1.72×10-2拷贝数/mL。CHEN等[21]在江苏畜禽养殖废水中检测到高丰度sul1和sul2,且sul1和sul2与16S rRNA拷贝数的比值分别为38.4、16.2。污水处理厂已成为磺胺类ARGs的潜在储存库,MAO等[22]在污水处理厂中检出丰度较高的sul1和sul2,在污水和脱水污泥中丰度分别为(6.7±7.2)×105拷贝数/mL和(2.2±2.8)×1011拷贝数/g。BYRNE BAILEY等[23]从英国林肯郡农田土壤菌株中检测到磺胺类ARGssul1、sul2、sul3,其中sul1的检出率最高。在食用动物体内检测到大量耐药菌和相应的ARGs。金明兰等[24]对养殖场空气中360株大肠杆菌(Escherichiacoli)进行磺胺类ARGs检测,结果显示sul1、sul2检出率较高,sul3检出率较低,并得出养殖场的抗生素使用种类、建场时间、饲养规模与空气中大肠杆菌的磺胺类抗生素、ARGs呈正相关。
1.3 磺胺类耐药微生物种类与分布
自1937年磺胺类药物上市后不久,就报道有多种耐药菌株存在[1]14。近年在畜禽体内及畜禽排泄物中检测到的磺胺类耐药菌株多为肠球菌属(Enterococcus)和大肠杆菌,土壤及水体中多为杆菌。磺胺类耐药菌在环境中的种类及分布见表1。
1.4 磺胺类ARGs的传播
人兽药物是环境中磺胺类抗生素的两个主要来源。养殖业把磺胺类抗生素加入饲料,可诱导生物体产生耐药菌株。ENNE等[17]1327从英国猪肉中分离出的大肠杆菌大多具有耐药性并普遍检测到sul2的存在,耐药菌株再通过动物性食用产品进入人体。同时,耐药菌株也可随着禽畜的排泄物经雨水冲刷和地表径流等多种途径进入土壤、河流、湖泊或渗入地下水中[27]。
水平基因转移(HGT)是磺胺类ARGs传播的主要途径,HGT可通过细菌的可移动遗传元件如接合性质粒、转座子、整合子及基因组岛等在同种甚至不同种菌株间发生,这加速了磺胺类ARGs的传播扩散[28]。Ⅰ类整合子作为可移动的DNA片段,能够捕获外源ARGs并将其水平传播,董洪燕等[29]分离的鸡白痢沙门菌磺胺耐药菌株中94.4%的菌株携带sul1,且88.9%的sull基因与Ⅰ类整合子同时存在。LUO等[30]7224发现,沉积物中整合子基因与磺胺类ARGs的相对拷贝数具有良好的线性关系(R2=0.889)。TOLEMAN等[31]发现ISCR9和ISCR10两种遗传元件目前只在嗜麦芽寡养单胞菌属(Stenotrophomonasmaltophilia)中存在,并常与sul2基因相联系。环境中磺胺类ARGs的HGT还受到其他因素影响,如JI等[32]监测了上海市多个养殖场粪肥、土壤中磺胺类ARGs和重金属,发现sulA、sul3与Cu、Zn、Hg含量显著相关。

表1 环境中磺胺耐药菌种类及分布
2 ARGs风险评价
2.1 ARGs风险评价的研究进展
目前,有关环境中的抗生素耐药菌株及ARGs对人类及动物健康的风险评价方法还较少,主要的研究方法还是采用流行病学方法[33]。徐冰洁等[34]研究了ARGs在环境中的来源、传播扩散及生态风险,定性描述了ARGs对生态环境和人类健康带来的潜在危害。牛卫杰等[35]研究了典型城镇化地区河流ARGs分布特征与风险评价,结果显示污水处理厂中四环素抗性基因tetC污染较严重。多指标决策分析(MCDA)也被用于ARGs风险评估,MCDA可用于评估不同来源的共选择剂(如清洁剂、杀虫剂、金属、纳米材料)对环境中总的耐药微生物风险所产生的相对影响,然而MCDA法过于依赖专家观点,有很大的主观判断偏移的可能[36]。ASHBOLT等[37]在美国环境保护署(USEPA)建立的微生物风险评价(MRA)的基础上提出了人群健康风险评价体系(HHRA),主要包括:(1)对环境中选择压力下抗生素抗性发展过程的解释;(2)分析确定环境热点区域内水平转移过程及速率;(3)对传统“剂量—效应”评价方法进行修订,确定环境中各类耐药微生物风险浓度及传播途径。此外,病原菌对农药的抗性风险评价可以作为ARGs风险评价的参考,王文桥等[38]研究了植物病原菌对杀菌剂抗性风险评估的研究,主要有初步探测、回归分析、早期评估以及分类评估4种方法。徐颖等[39]根据抗药性突变体的突变率较高判断rpsL基因突变引起的水稻白叶枯病菌对链霉素的抗性风险较高。
2.2 ARGs的演替
1960年,磺胺类ARGs的sul1、sul2由于质粒转移被识别确认,sul1存在于Tn型整合子上,sul2基因位于incQ组的微小质粒上;sul3在2003年分离的猪致病性大肠杆菌分离株的接合性质粒中被发现,之后TAVERNA等[40]从人体大肠杆菌中提取出sul3。MASKELL等[41]对肺炎双球菌(Pneumococcus)磺胺耐药菌株的研究表明,sulA上有3~6个碱基的重复。LUO等[30]7222研究海河流域河水和沉积物中ARGs发现,sul1、sul2检出率为100%,在沉积物中的丰度数量级为1011拷贝数/g,是水体中的120~2 000倍,且其丰度与抗生素含量呈显著正相关,说明磺胺类抗生素选择性富集了磺胺类ARGs。王娜等[42]研究磺胺类耐药菌中ARGs的DHPS替代基因表达规律时发现,随着磺胺嘧啶暴露浓度的增加,DHPS替代基因的表达随之显现出上升趋势,且大多数DHPS替代基因呈现出明显的剂量—效应关系,同时发现在同一菌株内磺胺类ARGs的表达规律为sul1>sul2>sul3。邹世春等[43]在北江河水中7个样品中均检出磺胺类ARGssul1和sul2,并发现sul1和sul2的含量水平与该区域水中磺胺类抗生素含量分布具有一定的相关性,表明抗生素是诱导产生ARGs的重要原因。
随着抗生素的过度使用,细菌对抗生素的耐药性已由单一耐药逐渐发展为多重耐药性,目前的研究也发现大多数耐药菌株的表型为多重耐药性。细菌多重耐药性的产生与环境中的抗生素多重选择压力密切相关[44]。SCHMIDT等[45]从虹鳟鱼养殖场分离出的气单胞菌属(Aeromonas)除了抗四环素外,大部分也抗磺胺嘧啶或甲氧苄氨嘧啶。
滥用抗生素使得近年来出现超级细菌,每年全世界有50%的抗生素种类被滥用,而我国甚至接近80%[2]1512。近年来已有20多个国家发现超级细菌感染,包括欧洲、北美、非洲及亚洲地区,而在我国宁夏回族自治区、福建省、杭州市以及台湾省等地也均有超级细菌感染的病例报道。超级细菌指临床上出现的多重耐药菌,如耐甲氧西林的金黄葡萄球菌(MRSA)、鲍曼不动杆菌(Ab),抗万古霉素的肠球菌(VRE)、耐多药肺炎链球菌(MDRSP)、多重耐药性结核杆菌(MDR-TB)以及碳青霉烯肺炎克雷伯菌(KPC)等[46]。2010年英国媒体爆出南亚发现新型超级细菌NDM-1,耐药性极强可全球蔓延。2011年德国爆发了“毒黄瓜”事件,疫情短期内在欧洲至少9个国家蔓延,33人确认死亡,超过3 000人受感染,包括至少470人出现肾功能衰竭并发症。引起本次疫情的O104:H4血清型肠出血性大肠杆菌是一种新型高传染性有毒菌株,该菌株携带氨基糖苷类、大环内酯类、磺胺类等抗生素的耐药基因,导致抗生素治疗无效[47]。2013年以英国为发源地的超级细菌已经开始在多个国家被发现,据美国媒体报道,这种超级细菌被称为LA-MASA超级细菌,主要存在于禽类体内,感染率极高,但是对人体危害很小。2016年5月26日,美国卫生官员报告,美国发现首例对所有已知抗生素有抵抗力的细菌感染病例,如果这种超级细菌传播,可能造成严重危险[48]。
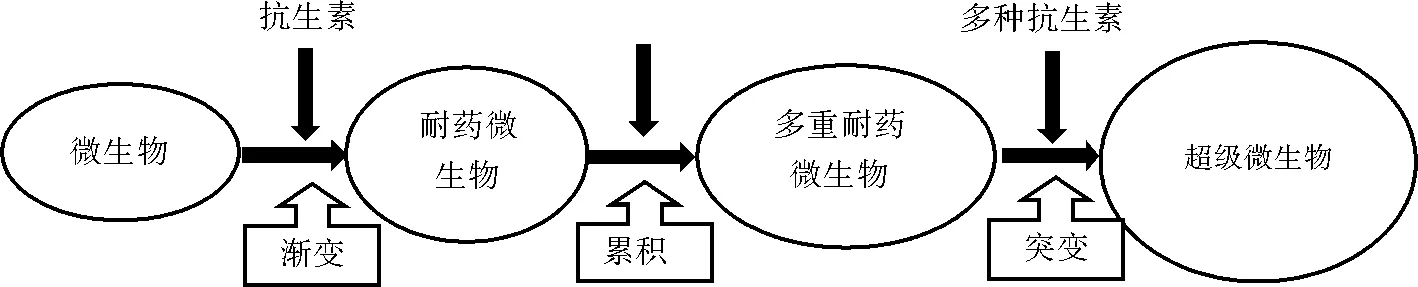
图1 “渐变→累积→突变”演变模型Fig.1 The “germination,cumulation,mutation” evolution model
综上,本研究提出“渐变→累积→突变”的ARGs演变模型(见图1),由于抗生素大量使用,对环境中微生物产生选择性压力使环境微生物出现耐药性,继而使耐药微生物体内ARGs不断积累,发展成多重耐药微生物,进一步突变成为超级细菌。
3 结论与展望
我国磺胺类抗生素使用量逐年增长,导致环境中大量抗生素残留和sul1、sul2、sul3、sulA等ARGs的检出,世界多地出现携带磺胺类ARGs的耐药微生物,对生态环境和人体健康产生潜在不利影响。根据ARGs的发展规律,提出ARGs的“渐变→累积→突变”演变模型。
未来迫切需要完善我国抗生素及ARGs的风险评估体系,包括生态环境风险评估和健康环境风险评估,加强生态毒理作用机制研究。确定抗生素抗性试验以期填补风险评价的数据不足,改进剂量效应关系,确定适合抗生素耐药微生物的剂量效应关系。此外,应建立环境中磺胺类ARGs检测分析方法、开展磺胺类ARGs在环境中迁移、转化相关研究,确定我国磺胺类ARGs的污染区域、种类和水平,建立我国磺胺类ARGs污染的数据库。
[1] 王娜.环境中磺胺类抗生素及其抗性基因的污染特征及风险研究[D].南京:南京大学,2014.
[2] 汪涛,杨再福,陈勇航,等.地表水中磺胺类抗生素的生态风险评价[J].生态环境学报,2016,25(9).
[3] PRDDEN A,PEI R,HEATHER STORTEBOOM A,et al.Antibiotic resistance genes as emerging contaminants:studies in Northern Colorado[J].Environmental Science & Technology,2006,40(23):7445-7450.
[5] ALLEN H K,DONATO J,WANG H H,et al.Call of the wild: antibiotic resistance genes in natural environments[J].Nature Reviews Microbiology,2010,8(4):251-259.
[6] 罗义,周启星.抗生素抗性基因(ARGs)——一种新型环境污染物[J].环境科学学报,2008,28(8):1499-1505.
[7] 冀秀玲,刘芳,沈群辉,等.养殖场废水中磺胺类和四环素抗生素及其抗性基因的定量检测[J].生态环境学报,2011,20(5):927-933.
[8] PHUONG HOA P T,NONAKA L,HUNG V P,et al.Detection of thesul1,sul2,andsul3 genes in sulfonamide-resistant bacteria from wastewater and shrimp ponds of north Vietnam[J].Science of the Total Environment,2008,405(1/2/3):377-384.
[9] PEI R,KIM S C,CARLSON K H,et al.Effect of river landscape on the sediment concentrations of antibiotics and corresponding antibiotic resistance genes (ARG)[J].Water Research,2006,40(12):2427-2435.
[10] 陈琳琳,李宝泉.新型抗生素抗性基因传播元件ISCR及其生态风险[J].应用生态学报,2015,26(10):3215-3225.
[11] SARMAH A K,MEYER M T,BOXALL A B.A global perspective on the use,sales,exposure pathways,occurrence,fate and effects of veterinary antibiotics (VAs) in the environment[J].Chemosphere,2006,65(5):725-759.
[12] 李云开,刘世荣,张克强,等.磺胺类药物在农田生态系统中迁移转化过程的研究进展[J].中国畜牧兽医,2007,34(12):141-144.
[13] KIM Y,JUNG J,KIM M,et al.Prioritizing veterinary pharmaceuticals for aquatic environment in Korea[J].Environmental Toxicology & Pharmacology,2008,26(2):167-176.
[14] 王丹,隋倩,赵文涛,等.中国地表水环境中药物和个人护理品的研究进展[J].科学通报,2014,59(9):743-751.
[15] SKÖLD O.Sulfonamide resistance:mechanisms and trends[J].Drug Resistance Updates,2000,3(3):155-160.
[16] SU H C,YING G G,TAO R,et al.Occurrence of antibiotic resistance and characterization of resistance genes and integrons in Enterobacteriaceae isolated from integrated fish farms in south China[J].Journal of Environmental Monitoring,2011,13(11):3229-3236.
[17] ENNE V I,LIVERMORE D M,STEPHENS P,et al.Persistence of sulphonamide resistance inEscherichiacoli,in the UK despite national prescribing restriction[J].The Lancet,2001,357(9265).
[18] PERRETEN V,BOERLIN P.A new sulfonamide resistance gene (sul3) inEscherichiacoliis widespread in the pig population of Switzerland[J].Antimicrobial Agents & Chemotherapy,2003,47(3):1169-1172.
[19] STOLL C,SIDHU J P,TIEHM A,et al.Prevalence of clinically relevant antibiotic resistance genes in surface water samples collected from Germany and Australia[J].Environmental Science & Technology,2012,46(17):9716-9726.
[20] XIONG W,SUN Y,DING X,et al.Antibiotic resistance genes occurrence and bacterial community composition in the Liuxi River[J].Frontiers in Environmental Science,2014,2:1-6.
[21] CHEN B,HAO L J,GUO X Y,et al.Prevalence of antibiotic resistance genes of wastewater and surface water in livestock farms of Jiangsu Province,China[J].Environmental Science & Pollution Research,2015,22(18):13950-13959.
[22] MAO D,YU S,RYSZ M,et al.Prevalence and proliferation of antibiotic resistance genes in two municipal wastewater treatment plants[J].Water Research,2015,85(6):458-466.
[23] BYRNE BAILEY K G,GAZE W P.Prevalence of sulfonamide resistance genes in bacterial isolates from manured agricultural soils and pig slurry in the United Kingdom[J].Antimicrobial Agents & Chemotherapy,2009,53(2):692-702.
[24] 金明兰,孟庆玲,赵玉鑫,等.养殖场空气中E.coli磺胺类抗生素的抗性[J].环境化学,2017,36(3):472-479.
[25] GLENN L M,LINDSEY R L,FOLSTER J P,et al.Antimicrobial resistance genes in multi-drug resistantSalmonellaentericaserovars isolated most frequently from animals,retail meat,and humans in the U.S. and Canada[J].Microbial Drug Resistance,2013,19(3):175-184.
[26] WANG N,YANG X,JIAO S,et al.Sulfonamide-resistant bacteria and their resistance genes in soils fertilized with manures from Jiangsu Province,Southeastern China[J].Plos One,2014,9(11):e112626.
[27] 童蕾,姚林林,刘慧,等.抗生素在地下水系统中的环境行为及生态效应研究进展[J].生态毒理学报,2016,11(2):27-36.
[28] 朱永官,欧阳纬莹,吴楠,等.抗生素耐药性的来源与控制对策[J].中国科学院院刊,2015,30(4):509-516.
[29] 董洪燕,缪晓斌,李鑫,等.鸡白痢沙门菌分离株的耐药性及磺胺类耐药机制研究[J].中国家禽,2010,32(9):29-33.
[30] LUO Y,MAO D,RYSZ M,et al.Trends in antibiotic resistance genes occurrence in the Haihe River,China[J].Environmental Science & Technology,2010,44(19).
[31] TOLEMAN M A,BENNETT P M,BENNETT D M,et al.Global emergence of trimethoprim/sulfamethoxazole resistance in stenotrophomonas maltophilia mediated by acquisition ofsulgenes[J].Emerging Infectious Diseases,2007,13(4):559-565.
[32] JI X L,SHEN Q H,LIU F,et al.Antibiotic resistance gene abundances associated with antibiotics and heavy metals in animal manures and agricultural soils adjacent to feedlots in Shanghai;China[J].Journal of Hazardous Materials,2012,235/236:178-185.
[33] 史密伟,朱晓磊,唐文忠.抗生素抗性基因环境效应的研究进展[J].环境保护科学,2015,41(6):123-128.
[34] 徐冰洁,罗义,周启星,等.抗生素抗性基因在环境中的来源、传播扩散及生态风险[J].环境化学,2010,29(2):169-178.
[35] 牛卫杰,罗雅,贾俊娜,等.典型城镇化地区河流抗生素抗性基因分布特征与风险评价[J].水利水电技术,2012,43(8):53-56.
[36] SEMENZIN E,CRITTO A,RUTGERS M,et al.Integration of bioavailability,ecology and ecotoxicology by three lines of evidence into ecological risk indexes for contaminated soil assessment[J].Science of the Total Environment,2008,389(1):71-86.
[37] ASHBOLT N J,AMÉZQUITA A,BACKHAUS T,et al.Human health risk assessment (HHRA) for environmental development and transfer of antibiotic resistance[J].Environmental Health Perspectives,2013,121(9):993-1001.
[38] 王文桥,马志强,张小风,等.植物病原菌对杀菌剂抗性风险评估[J].农药学学报,2001,3(1):6-11.
[39] 徐颖,祝晓芬,周明国.水稻白叶枯病菌室内链霉素抗性菌株的诱导及其抗性分子机制和风险评价[J].农药学学报,2013,15(1):127-128.
[40] TAVERNA M,BAILLET A,BAYLOCQ FERRIER D.Sulphonamide resistance genesul3 found inEscherichiacoliisolates from human sources[J].Journal of Antimicrobial Chemotherapy,2003,52(6):1022-1024.
[41] MASKELL J P,SEFTON A M,HALL L M.Mechanism of sulfonamide resistance in clinical isolates ofStreptococcuspneumoniae[J].Antimicrobial Agents & Chemotherapy,1997,41(10):2121-2126.
[42] 王娜,杨晓洪,郭欣妍,等.磺胺类耐药菌中抗性基因sul的表达规律[J].生态毒理学报,2015,10(5):75-81.
[43] 邹世春,朱春敬,贺竹梅,等.北江河水中抗生素抗性基因污染初步研究[J].生态毒理学报,2009,4(5):655-660.
[44] 张伟,李天保,杨秀生,等.抗生素的使用和耐药性浅析[J].齐鲁渔业,2008,25(1):37-38.
[45] SCHMIDT A S,BRUUN M S,LARSEN J L,et al.Characterization of class 1 integrons associated with R-plasmids in clinicalAeromonassalmonicidaisolates from various geographical areas[J].Journal of Antimicrobial Chemotherapy,2001,47(6):735-743.
[46] 宋兰兰.ICU多重耐药菌感染的预防与控制[J].工企医刊,2014,27(2):694.
[47] 苏建强,黄福义,朱永官.环境抗生素抗性基因研究进展[J].生物多样性,2013,21(4):481-487.
[48] 中国新闻网.美现“超级细菌”可抵抗所有已知抗生素[EB/OL].(2016-05-27)[2017-01-05].http://discovery.163.com/ 16/0527/17/BO3DR3AI000125LI.html?baike.
Reviewontheproductionandevolutionofsulfonamideresistancegenes
WANGTao,YANGZaifu,CHENYonghang,SUNRanran,ZHANGYaoyao,WANGYanan.
(CollegeofEnvironmentalScienceandEngineering,DonghuaUniversity,Shanghai201620)
Sulfonamides were synthetic broad spectrum antibiotics which were extensively used in animal husbandry,aquaculture and human disease treatment. The antibiotics resistance genes (ARGs) would be induced because of long term residue of sulfonamide. As a new type of environmental pollutants,ARGs was more harmful than antibiotics because of its persistence,replication,transmission and spread in the environment. Accordingly,the production and propagation of ARGs were reviewed. The “germination,accumulation,mutation” evolution model of ARGs was proposed. The significance of relative research was proposed for China.
sulfonamides; antibiotics resistance genes; evolution model
汪 涛,男,1993年生,硕士研究生,研究方向为环境风险与污染场地修复。#
。
*上海市科技攻关项目(No.033919457)。
10.15985/j.cnki.1001-3865.2017.11.019
2017-05-10)
