基质辅助激光解吸电离飞行时间质谱在临床微生物检验中的应用与发展
2017-12-02张嵘
张 嵘
基质辅助激光解吸电离飞行时间质谱在临床微生物检验中的应用与发展
张 嵘
基质辅助激光解吸电离飞行时间质谱; 微生物鉴定; 细菌耐药性; 分子流行病学
Application and development of MALDI-TOF MS in clinical microbiology laboratory
基质辅助激光解吸电离飞行时间质谱(MALDI-TOF MS)自20世纪90年代开始成功用于细菌的快速鉴定,其原理是根据不同细菌的特征性蛋白指纹图谱进行细菌鉴定,打破了传统生化反应方法烦琐的鉴定流程,大大缩短了检测时间,降低了检测成本,且具有准确、高通量等优势,为临床微生物检验带来了革命性的发展。MALDI-TOF MS在临床微生物实验室的应用主要涵盖以下方面:①病原微生物的鉴定;②细菌耐药性的检测;③某些特定菌株的分子流行病学分析。
1 MALDI-TOF MS在病原菌鉴定中的应用
1.1 培养后病原微生物鉴定
MALDI-TOF MS对培养后临床病原菌的快速检测已十分成熟,有文献评估临床常用的2台质谱仪——布鲁克公司Biotyper MS和物生梅里埃公司的VITEK MS的鉴定性能,其对临床常见病原菌的鉴定准确率均达95%以上[1]。此外,MALDI-TOF MS在厌氧菌、真菌、分枝杆菌及其他少见或难培养细菌的鉴定方面与传统方法相比较具有明显优势。
采用传统方法鉴定厌氧菌周期长,鉴定困难,有些菌种之间很难区分,不能满足临床实验室快速准确的要求。随着MALDI-TOF MS数据库不断完善,MALDI-TOF MS对临床分离的厌氧菌鉴定准确率已经达到90%以上[2]。与“金标准”16S rRNA相比,197株临床分离厌氧菌,MALDI-TOF MS鉴定准确率为94.9%[3]。真菌亦是临床鉴定的难题,质谱对临床常见白念珠菌、近平滑念珠菌、热带念珠菌、新生隐球菌等鉴定总准确率能达96%以上,但是对数据库中未包含的菌种,例如弗比恩毕赤酵母无法给出鉴定结果,需要自建库进行补充[4]。由于菌丝和细胞壁破碎困难,丝状真菌是目前质谱鉴定的瓶颈,有文献报道经复杂的前处理后,390株临床分离的丝状真菌的鉴定准确率仅为85.6%[5]。
质谱的另一个优势是对细菌复合群的鉴定,常规的生化反应均难将复合群区分到种。Biotyper MS的数据库对醋酸钙-鲍曼不动杆菌复合群的区分能力可达到100%[6],VITEK MS对于链球菌属,尤其对肺炎链球菌和其他链球菌之间的区分准确率可达99.1%(115/116)[7]。结核分枝杆菌复合群菌种同源性高,鉴定困难,Biotyper MS可鉴定至种的水平,结核分枝杆菌和牛分枝杆菌的鉴定准确率在96%左右[8],对非结核分枝杆菌复合群细菌鉴定准确率为98.4%(65/66)[9]。目前国产质谱仪对临床革兰阴性杆菌的鉴定准确率可达到98.05%,但是对某些复合群的鉴别仍然有待提高[10]。
质谱用于支原体的鉴定目前还有一定的困难,主要由于数据库有限,样本混合其他细菌,或合并多种支原体感染,干扰质谱仪鉴定的准确性。目前质谱可通过自建库或直接分析某一种如肺炎支原体特征峰进行鉴定[11]。
1.2 临床样本直接检测细菌
质谱针对培养后病原菌的鉴定可缩短鉴定时间,但仍需培养18~24 h,而将质谱运用于临床样本的直接检测,可大大缩短检测周转时间(turnaround time,TAT),对于临床细菌及真菌感染患者的早期诊断和治疗有重要意义。
鉴于血流感染的重要性,MALDI-TOF MS用于阳性血培养标本的快速检测一直是研究的热点,标本前处理能除去血液中血细胞以及其他非细菌成份的干扰,对直接检测尤为重要。差速离心的方法在菌量大于107cfu/mL时准确率在87%左右[12];分离胶方法检出率约59%;Sepsityper商品化试剂盒可检出88%细菌,阴性菌鉴定准确率更高[13];合适的裂解剂(如SDS和saponin)可提高直接检测的检出率。从目前文献综合评估,Sepsityper具有标准化、准确率高的优点,但操作稍复杂,且成本较裂解剂及分离胶方法高。
目前MALDI-TOF MS直接检测血培养还存在一些问题。临床常用血培养瓶如BacT/ALERT活性炭需氧瓶内活性炭吸附细菌导致细菌浓度降低,前处理后检测效果不佳,需要鉴定短时间培养后生成菌膜,需要耗时4~8 h[14];革兰阳性菌的鉴定准确率低于革兰阴性菌;无法可靠地区分肺炎链球菌与草绿色链球菌[15];混合感染样本只能检测一种细菌或无法给出鉴定结果[16];对厌氧菌的鉴定准确率在75%左右等。国内外对MALDI-TOF MS运用于临床标本的直接鉴定都持乐观态度,中国临床微生物质谱共识专家组以及上海医学会检验医学专科委员会临床微生物学组均发表了关于血培养直接质谱检测共识,将MALDI-TOF MS用于血培养直接检测作为未来的手段。
除了血培养阳性标本外,MALDI-TOF MS还可用于尿液标本的直接检测。尿液样本经离心后洗涤,对菌量>105cfu/mL的尿液样本检出率为92.7%,尤其是革兰阴性杆菌显示很好的符合性[17]。采用SDS前处理,可大大提高方法准确度。文献对比发现过滤(78.9%)和固体培养(84.2%)比差速离心(68.4%)有更高的准确率,MALDI-TOF MS在尿液样本的直接检测具有巨大潜力[18]。其他无菌体液标本的快速检测报道不多,主要为脑脊液,已有报道检出脑膜炎患者脑脊液中肺炎链球菌、布鲁菌和肺炎克雷伯菌[19-20]。
2 MALDI-TOF MS在细菌耐药性检测中的研究与应用
MALDI-TOF MS应用于细菌耐药性的检测是质谱在微生物检验中的另一个拓展。根据目前已有的报道,大致可分为:①直接寻找耐药菌中特异峰,采用特定的软件分析获得耐药和敏感细菌的差异性(直接分析法);②根据细菌产生特异性酶水解抗生素,检测抗生素峰图的变化,确认是否耐药(酶水解法);③曲线下面积计算法;④其他方法。
2.1 直接分析法
在2000年,Edwards-Jones等[21]首次提出了采用MALDI-TOF MS快速区分MRSA和MSSA,随后更多的研究验证了MALDI-TOF MS对MRSA和MSSA具有区分能力。Biotyper MS采用分析软件区分MRSA组的准确率为83.3%,MSSA组为90.9%[22],见图1;VITEK MS通过建立SuperSpectra,选择3个特征峰区分MRSA和MSSA,准确率达到84%和91%[23],见图2。同理,MALDI-TOF MS对耐万古霉素肠球菌也可进行快速区分,采用GA算法其特异度和灵敏度分别为92.40%和99.18%[24]。2014年,Lau等[25]发现质荷比为11109 Da是blaKPC表达pKpQIL肽的特征峰,通过该特征峰可快速区分产KPC型碳青霉烯酶的肺炎克雷伯菌。
2.2 酶水解法
肠杆菌科细菌、铜绿假单胞菌和鲍曼不动杆菌对β内酰胺类和碳青霉烯类抗生素的耐药机制大多数是由于产生水解抗生素的酶,如CTX-M、KPC、NDM和OXA酶等。2011年首次报道MALDI-TOF MS检测碳青霉烯酶,整个过程1~2.5 h即可完成[26]。随后大量文献报道,针对革兰阴性杆菌β内酰胺酶以及碳青霉烯酶耐药菌株的检测,通过孵育水解抗生素区分耐药株和敏感株[27-28]。胡燕燕等[29]比较产KPC型碳青霉烯酶肠杆菌科细菌在不同药物浓度下水解厄他培南的能力,推荐0.1 g/L的厄他培南作为水解底物来检测产KPC型肠杆菌科细菌。为了加快水解时间,有研究报道,添加NH4HCO3等促进水解酶活性物质从而提高肠杆菌科细菌KPC酶水解美罗培南的能力,将灵敏度从76%提高至98%[30]。
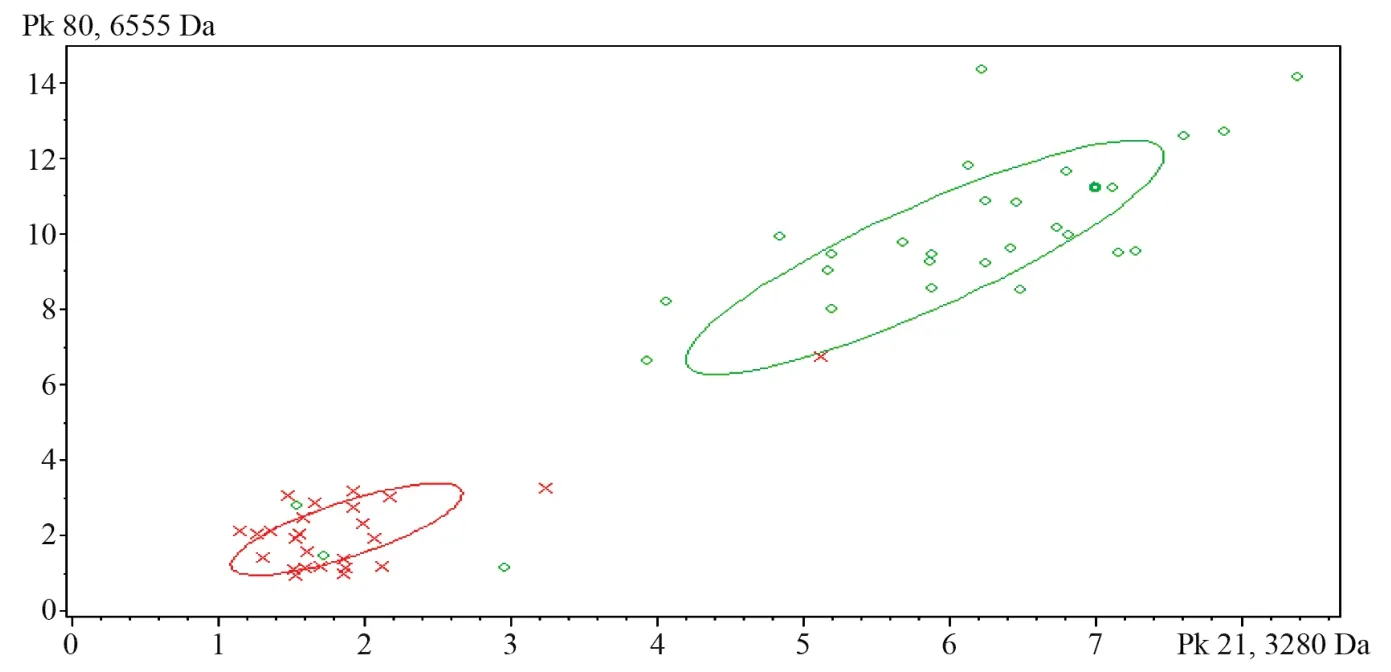
图1 MRSA(红色×)和MSSA(绿色○)的二维分析图Figure 1 Two-dimensional profiling of MRSA (red×) and MSSA (green ○) isolates
图2 MRSA(红色峰)和MSSA(蓝色峰)的区分Figure 2 The SuperSpectra of MRSA (red peaks) and MSSA (blue peaks)
2.3 曲线下面积法
2009年,Marinach等[31]发现白念珠菌在不同浓度氟康唑的压力下,其表达的蛋白组分会发生改变,用MALDI-TOF MS能定性地表现出来。基于此原理,近2年兴起一种确定细菌耐药的新方法——曲线下面积法:细菌在含有抗生素的溶液中培养,敏感菌株会被抗生素抑制,细菌浓度大大降低,在质谱峰图上有直观的体现。根据孵育前后质谱峰曲线下面积的比值(cutoff值)来判断细菌对该药物是否耐药,可定量得到细菌对该抗生素的MIC值,见图3。2014年,Lange等[32]检测108株肺炎克雷伯菌对碳青霉烯类药物的敏感性,结果与E试验相比,该方法灵敏度和特异度分别为97.3%和93.5%。2016年,Sparbier等[33]对大量药物进行半定量的分析,包括青霉素类、头孢菌素类、碳青霉烯类、氟喹诺酮类以及氨基糖苷类,该方法能够快速测定抗生素对细菌的MIC值,鉴定与药敏结果在短时间内完成。
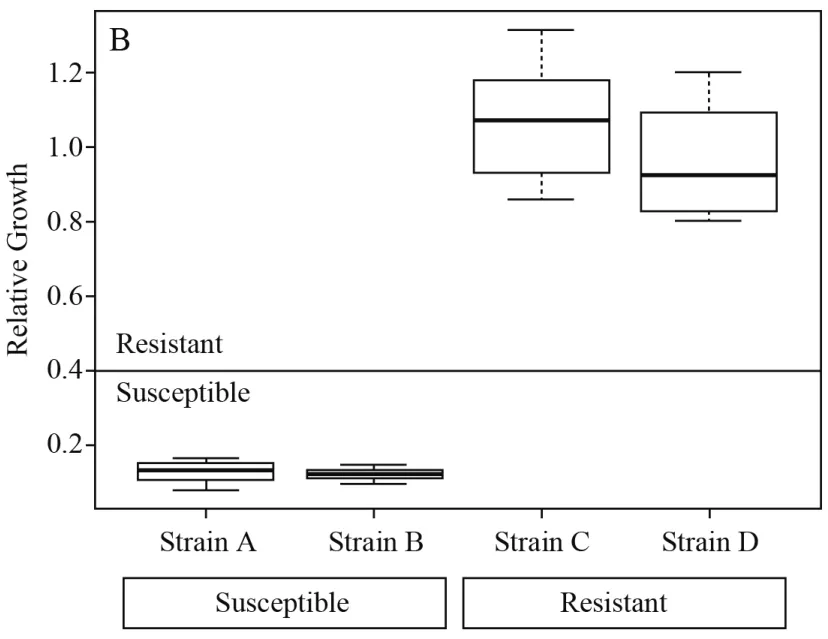
图3 敏感株(A,B)与耐药株(C,D)曲线下面积比值与cutoff值的比较Figure 3 Comparison of area ratio and cutoff value between the sensitive strains (A, B) and resistant strains (C, D)
2.4 其他方法
外膜蛋白缺失是导致细菌耐药的另一重要原因,Cai等[34]首次采用质谱快速检测外膜蛋白缺失引起碳青霉烯类耐药的肺炎克雷伯菌,与SDSPAGE比较,MALDI-TOF MS具有快速、准确、直观等优势。Hu等[35]对外膜蛋白缺失的大肠埃希菌快速检测,进一步验证了质谱检测外膜蛋白的可靠性。
近年来发展的耐药快速检测方法还包括采用核素标记生长培养基中特定氨基酸。细菌生长程度不同,培养基中核素标记的氨基酸消耗程度不同,通过检测核素反映细菌的耐药情况。目前研究采用这种方法检测MRSA,孵育3 h甚至更少时间即可得到结果[36]。
质谱技术在耐药性检测中的应用已经显示出巨大潜力,如能与培养后纯菌落鉴定相结合,可将TAT时间缩短24~48 h,如与直接样本检测相结合,则可将报告临床时间提早48~54 h。
3 MALDI-TOF MS在流行病学的应用研究
质谱除了对细菌耐药的快速检测外,还可用于聚类分析和主成份分析,为医院感染和传染性疾病的监测、防控提供方法。传统的细菌同源性主要是采用多位点序列分型(MLST)、脉冲场凝胶电泳(PFGE)等分子生物学分析手段对细菌管家基因以及特异的限制性内切酶剪切后的DNA片段进行研究,质谱同源性分析主要是基于检测细菌的蛋白质,因此在分型方法上与MLST和PFGE存在差异。2011年,首次采用质谱技术对金黄色葡萄球菌进行分型,通过13个特征峰,找到不同的CC群组与组之间的差异[37],见图4。后续有研究者利用质谱对不同ST型的金黄色葡萄球菌ST17和ST1进行系统发育树分析,结果显示同一ST型之间菌株分布集中,不同ST型之间距离明显[38]。黄永禄等[39]采用Biotyper MS对产ESBL大肠埃希菌最常见ST型ST131和ST405型进行聚类分析,两组之间存在明显分簇情况,灵敏度和特异度分别为97.9%和93.3%,VITEK MS通过建立SuperSpectra,对ST131和非ST131型大肠埃希菌采用自建数据库并进行分析和验证,得到两者区分的灵敏度和特异度分别为86.6%和95.1%[40]。在小样本量的比较中,MALDI-TOF MS分型结果与“金标准”PFGE分型情况完全相同,且具有更快速、成本更低的优势。虽然质谱用于分型尚处于研究阶段,但有希望成为一种新的快速分型方法。
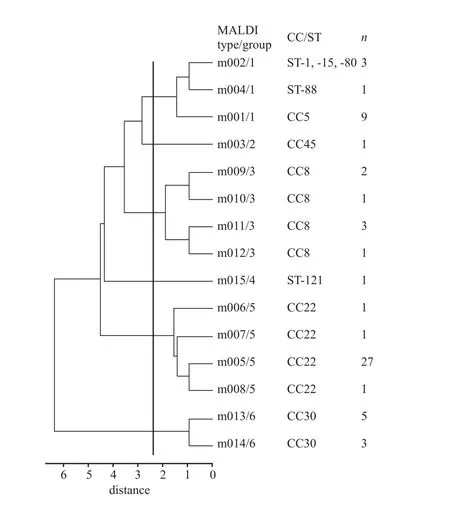
图4 MALDI分型与CC分型的聚类分析树状图Figure 4 Dendrogram representation of hierarchical cluster analysis of the MALDI types and CC types
4 总结和展望
MADLI-TOF MS经过数十年的发展,为临床微生物实验室带来了革命性的发展。其具有的快速、准确、灵敏、高通量等特点,加上操作简单和低耗材的优势在临床微生物实验室被推广。现阶段MADLI-TOF MS在微生物检测中还存在很大的发展和提升空间,随着技术的不断发展,相信在不久的将来,一定会成为微生物常规工作的一大支柱。
[1]MARTINY D, BUSSON L, WYBO I, et al. Comparison of the Microflex LT and VITEK MS systems for routine identification of bacteria by matrix-assisted laser desorption ionization-time of flight mass spectrometry[J]. J Clin Microbiol, 2012, 50(4):1313-1325.
[2]LI Y, GU B, LIU G, et al. MALDI-TOF MS versus VITEK 2 ANC card for identification of anaerobic bacteria[J]. J Thorac Dis, 2014, 6(5): 517-523.
[3]HANDAL N, BAKKEN JORGENSEN S, SMITH TUNSJO H,et al. Anaerobic blood culture isolates in a Norwegian university hospital: identification by MALDI-TOF MS vs 16S rRNA sequencing and antimicrobial susceptibility profiles[J]. APMIS,2015, 123(9): 749-758.
[4]BADER O, WEIG M, TAVERNE-GHADWAL L, et al.Improved clinical laboratory identification of human pathogenic yeasts by matrix-assisted laser desorption ionization time-offlight mass spectrometry[J]. Clin Microbiol Infect, 2011, 17(9):1359-1365.
[5]BECKER PT, DE BEL A, MARTINY D, et al. Identification of filamentous fungi isolates by MALDI-TOF mass spectrometry:clinical evaluation of an extended reference spectra library[J].Med Mycol, 2014, 52(8): 826-834.
[6]ESPINAL P, SEIFERT H, DIJKSHOORN L, et al. Rapid and accurate identification of genomic species from theAcinetobacter baumannii(Ab) group by MALDI-TOF MS[J]. Clin Microbiol Infect, 2012, 18(11): 1097-1103.
[7]BRANDA JA, MARKHAM RP, GARNER CD, et al.Performance of the Vitek MS v2. 0 system in distinguishingStreptococcus pneumoniaefrom nonpneumococcal species of theStreptococcus mitisgroup[J]. J Clin Microbiol, 2013, 51(9):3079-3082.
[8]QUINLAN P, PHELAN E, DOYLE M. Matrix-assisted laser desorption/ionisation time-of-flight (MALDI-TOF) mass spectrometry (MS) for the identification of mycobacteria from MBBacT ALERT 3D liquid cultures and Lowenstein-Jensen (LJ)solid cultures[J]. J Clin Pathol, 2015, 68(3): 229-235.
[9]MEDIAVILLA-GRADOLPH MC, DE TORO-PEINADO I,BERMUDEZ-RUIZ MP, et al. Use of MALDI-TOF MS for identification of nontuberculousMycobacteriumspecies isolated from clinical specimens[J]. Biomed Res Int, 2015 : 854078.
[10]范欣,肖盟,徐志鹏,等. 国产基质辅助激光解吸电离飞行时间质谱系统Clin-TOF-Ⅱ MS与Bruker Biotyper质谱系统在革兰阴性菌的鉴定效能评估[J]. 中华检验医学杂志, 2017,40(1): 41-45.
[11]XIAO D, ZHAO F, ZHANG H, et al. Novel strategy for typingMycoplasma pneumoniaeisolates by use of matrix-assisted laser desorption ionization-time of flight mass spectrometry coupled with ClinProTools[J]. J Clin Microbiol, 2014, 52(8): 3038-3043.
[12]CHRISTNER M, ROHDE H, WOLTERS M, et al. Rapid identification of bacteria from positive blood culture bottles by use of matrix-assisted laser desorption-ionization time of flight mass spectrometry fingerprinting[J]. J Clin Microbiol, 2010, 48(5): 1584-1591.
[13]KLEIN S, ZIMMERMANN S, KOHLER C, et al. Integration of matrix-assisted laser desorption/ionization time-of-flight mass spectrometry in blood culture diagnostics: a fast and effective approach[J]. J Med Microbiol, 2012, 61(Pt 3): 323-331.
[14]KOHLMANN R, HOFFMANN A, GEIS G, et al. MALDITOF mass spectrometry following short incubation on a solid medium is a valuable tool for rapid pathogen identification from positive blood cultures[J]. Int J Med Microbiol, 2015, 305(4-5): 469-479.
[15]PROD'HOM G, BIZZINI A, DURUSSEL C, et al. Matrixassisted laser desorption ionization-time of flight mass spectrometry for direct bacterial identification from positive blood culture pellets[J]. J Clin Microbiol, 2010, 48(4): 1481-1483.
[16]SCHUBERT S, WEINERT K, WAGNER C, et al. Novel,improved sample preparation for rapid, direct identification from positive blood cultures using matrix-assisted laser desorption/ionization time-of-flight (MALDI-TOF) mass spectrometry[J]. J Mol Diagn, 2011, 13(6): 701-706.
[17]FERREIRA L, SANCHEZ-JUANES F, GONZALEZ-AVILA M, et al. Direct identification of urinary tract pathogens from urine samples by matrix-assisted laser desorption ionization-time of flight mass spectrometry[J]. J Clin Microbiol, 2010, 48(6):2110-2115.
[18]VERON L, MAILLER S, GIRARD V, et al. Rapid urine preparation prior to identification of uropathogens by MALDITOF MS[J]. Eur J Clin Microbiol Infect Dis,2015,34(9):1787-1795.
[19]NYVANG HARTMEYER G, KVISTHOLM JENSEN A, BOCHER S, et al. Mass spectrometry : pneumococcal meningitis verified andBrucellaspecies identified in less than half an hour[J]. Scand J Infect Dis, 2010, 42(9): 716-718.
[20]SEGAWA S, SAWAI S, MURATA S, et al. Direct application of MALDI-TOF mass spectrometry to cerebrospinal fluid for rapid pathogen identification in a patient with bacterial meningitis[J].Clin Chim Acta, 2014, 435 : 59-61.
[21]EDWARDS-JONES V, CLAYDON MA, EVASON DJ, et al. Rapid discrimination between methicillin-sensitive and methicillin-resistantStaphylococcus aureusby intact cell mass spectrometry[J]. J Med Microbiol, 2000, 49(3): 295-300.
[22]胡燕燕,蔡加昌,周宏伟,等. 基质辅助激光解吸/电离飞行时间质谱仪快速鉴别甲氧西林耐药和甲氧西林敏感金黄色葡萄球菌的研究[J]. 中华微生物学和免疫学杂志, 2015, 35(1): 42-45.
[23]SHAN W, LI J, FANG Y, et al. Rapid identification of methicillin-resistantStaphylococcus aureus(MRSA) by the Vitek MS Saramis system[J]. Curr Microbiol, 2016 , 72(1):29-32.
[24]NAKANO S, MATSUMURA Y, KATO K, et al. Differentiation ofvanA-positiveEnterococcus faeciumfromvanA-negativeE.faeciumby matrix-assisted laser desorption/ionisation time-offlight mass spectrometry[J]. Int J Antimicrob Agents, 2014, 44(3): 256-259.
[25]LAU AF, WANG H, WEINGARTEN RA, et al. A rapid matrix-assisted laser desorption ionization-time of flight mass spectrometry-based method for single-plasmid tracking in an outbreak of carbapenem-resistantEnterobacteriaceae[J]. J Clin Microbiol, 2014, 52(8): 2804-2812.
[26]BURCKHARDT I, ZIMMERMANN S. Using matrix-assisted laser desorption ionization-time of flight mass spectrometry to detect carbapenem resistance within 1 to 2. 5 hours[J]. J Clin Microbiol, 2011, 49(9): 3321-3324.
[27]JUNG JS, POPP C, SPARBIER K, et al. Evaluation of matrix-assisted laser desorption ionization-time of flight mass spectrometry for rapid detection of beta-lactam resistance inEnterobacteriaceaederived from blood cultures[J]. J Clin Microbiol, 2014, 52(3): 924-930.
[28]HOYOS-MALLECOT Y, RIAZZO C, MIRANDA-CASAS C, et al. Rapid detection and identification of strains carrying carbapenemases directly from positive blood cultures using MALDI-TOF MS[J]. J MicrobiolMethods, 2014, 105 : 98-101.
[29]胡燕燕, 孙谦, 蔡加昌, 等. 基质辅助激光解析/电离飞行时间质谱仪检测KPC型碳青霉烯酶的研究[J]. 中华微生物学和免疫学杂志, 2012, 32(6): 561-565.
[30]PAPAGIANNITSIS CC, STUDENTOVA V, IZDEBSKI R, et al. Matrix-assisted laser desorption ionization-time of flight mass spectrometry meropenem hydrolysis assay with NH4HCO3, a reliable tool for direct detection of carbapenemase activity[J]. J Clin Microbiol, 2015, 53(5): 1731-1735.
[31]MARINACH C, ALANIO A, PALOUS M, et al. MALDI-TOF MS-based drug susceptibility testing of pathogens: the example ofCandida albicansand fluconazole[J]. Proteomics, 2009, 9(20): 4627-4631.
[32]LANGE C, SCHUBERT S, JUNG J, et al. Quantitative matrix-assisted laser desorption ionization-time of flight mass spectrometry for rapid resistance detection[J]. J Clin Microbiol,2014, 52(12): 4155-4162.
[33]SPARBIER K, SCHUBERT S, KOSTRZEWA M. MBTASTRA: A suitable tool for fast antibiotic susceptibility testing?[J].Methods, 2016, 104 : 48-54.
[34]CAI JC, HU YY, ZHANG R, et al. Detection of OmpK36 porin loss inKlebsiellaspp. by matrix-assisted laser desorption ionization-time of flight mass spectrometry[J]. J Clin Microbiol,2012, 50(6): 2179-2182.
[35]HU YY, CAI JC, ZHOU HW, et al. Rapid detection of porins by matrix-assisted laser desorption/ionization-time of flight mass spectrometry[J]. Front Microbiol, 2015, 6 : 784.
[36]SPARBIER K, LANGE C, JUNG J, et al. MALDI biotyperbased rapid resistance detection by stable-isotope labeling[J]. J Clin Microbiol, 2013, 51(11): 3741-3748.
[37]WOLTERS M, ROHDE H, MAIER T, et al. MALDI-TOF MS fingerprinting allows for discrimination of major methicillinresistantStaphylococcus aureuslineages[J]. Int J Med Microbiol,2011, 301(1): 64-68.
[38]LARTIGUE MF, KOSTRZEWA M, SALLOUM M, et al.Rapid detection of "highly virulent" Group BStreptococcusST-17 and emerging ST-1 clones by MALDI-TOF mass spectrometry[J]. J MicrobiolMethods, 2011, 86(2): 262-265.
[39]黄永禄, 李佳萍, 顾丹霞, 等. 采用基质辅助激光解析电离飞行时间质谱快速检测产KPC大肠埃希菌ST131和ST405分型的研究[J]. 中国感染与化疗杂志, 2016, 16(4): 460-464.
[40]MATSUMURA Y, YAMAMOTO M, NAGAO M, et al.Detection ofEscherichia colisequence type 131 clonal group among extended-spectrum beta-lactamase-producingE. coliusing VITEK MS Plus matrix-assisted laser desorption ionizationtime of flight mass spectrometry[J]. J MicrobiolMethods, 2015,119: 7-9.
R378
A
1009-7708 ( 2017 ) 06-0703-06
10.16718/j.1009-7708.2017.06.018
浙江大学附属第二医院检验科, 杭州 310009。
张嵘(1972—),女,博士,主任技师,主要从事微生物方面研究。
张嵘,E-mail:brigitte_zx@163.com。
2017-02-27 修回日期:2017-06-20
